Introduction
Phosphatidylethanolamin methyltransferase (PEMT) Enzyme katalysieren die kovalente Bindung von einem oder mehreren Methylgruppen mit S -adenosylmethionine (SAM) als Methylgruppendonator auf PE, PE oder Monomethyl-dimethyl-PE zu Monomethyl-PE, Dimethyl-PE und / oder geben Phosphatidylcholin (PC). Diese Enzyme sind fast allgegenwärtig in Tierzellen und Pilze. Sie können auch in einigen Anlagen 1 und etwa 10% der Bakterien, insbesondere solche, die mit Eukaryonten 2 interagieren gefunden werden.
PEMTs sind nicht nur, indem sie zur Herstellung von PC, die Hauptlipidklasse in tierischen Zellen, aber auch von der Erfüllung anderer wichtigen zellulären Funktionen, die für die Biologie der Zelle. Bei Säugetieren sind PEMTs hauptsächlich in der Leber, wo sie für normale Sekretion von Very Low Density Lipoprotein erforderlich sind, ausgedrückt, und sie auch ernährungsbedingte Fettleibigkeit 3, Atherosklerose 4 beitragen und Insulin Resistance 5. Zusätzlich werden Säugetier PEMT auch in Adipozyten exprimiert, wenn auch in den unteren Ebenen, und beteiligen sich an Fettablagerung 6, 7. PEMT Rolle bei der Krebsentwicklung 8. Apoptose 9, und das Zellwachstum 10 wurden ebenfalls demonstriert. In Bakterien haben PEMT Enzyme gezeigt, wichtig für die normale Zellwachstum 2, Virulenz 2 und Symbiose mit der Wirtspflanze 11 zu sein.
Das Ziel und Grundprinzip der vorliegenden Protokolls ist es, PEMU-Aktivität aus Gesamtzellextrakten zu messen, ohne die Notwendigkeit, um das Enzym zu reinigen. Zwei unterschiedliche Protokolle wurden entwickelt, um PEMT Aktivität zu messen. Die erste und häufigste misst die Übertragung von tritiiertem Methylgruppe von radioaktivem SAM auf PE, die das Thema dieses Artikels. Dieses Protokoll wurde ursprünglich entwickelt, um PEMU-Aktivität zu messen aus Hefe 12 und Säugerzellen (Leber) 13, eine underst gewinnenVerknüpfen der PC-Biosynthese in diesen Zellen als auch die Spezifität dieser Enzyme zu bestimmen. Später wurde diese Technik auf andere Zelltypen, wie beispielsweise Bakterien 2 (unter Verwendung eines basischen pH-Wertes für den Assay, obwohl 15) und Protozoenparasiten 14 angewendet. Diese Technik kann mit Gesamtzellextrakten sowie gereinigtes Enzym verwendet werden, und können möglicherweise zu jedem Zellextrakt System angewendet werden. Ein nichtradioaktiver Assay wurde ebenfalls entworfen, das auf die enzymatische Quantifizierung von S -adenosylhomocysteine, der Trans Produkt der SAM 16 stützt. Letztere Assay kann einfacher sein, da es sich nicht um die Radioaktivität, aber es ist nur gereinigte Enzyme geeignet.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Zellextrakt Vorbereitung
- Wachsen die Leishmania-Zellen in einem sterilen Plastikflasche mit luftdichten Deckel verschlossen bei 26 ºC in einem Medium mit M199 1x machte mit 20 mM HEPES pH 7,4, 100 U / ml Penicillin, 100 ug / ml Streptomycin, 5 ug / ml Häm ergänzt 0,35 g / L NaCO 2 H, 0,1 mM Adenin, und 2 & mgr; g / ml biopterin ohne Schütteln. Ernte der Zellen durch Zentrifugation bei 1.500 g für 5 min bei 4 ºC, wenn sie eine Zelldichte von 1-2 x 10 7 / ml zu erreichen.
- Verwerfen Sie den Überstand mit einer Pipette und waschen Sie die Zellen durch Resuspendieren des Zellpellets mit einer serologischen Pipette in die Hälfte der Kultur Volumen von kaltem phosphatgepufferter Kochsalzlösung (137 mM NaCl, 2,7 mM KCl, 10 mM Na 2 HPO 4, 1,8 mM KH 2 PO 4, pH 7,4). Entsorgen Zellüberständen nach BL2 Sicherheitsrichtlinien.
- Zentrifuge Zellen erneut bei 1500 g für 5 min bei 4 ºC. Überstand verwerfen mit einer Pipette. Gehen to mit dem nächsten Schritt oder Schnapp einfrieren das Zellpellet in flüssigem Stickstoff zur Langzeitlagerung bei -80 ° C (bis zu drei Monaten).
- Bereiten 2x Lysepuffer (0,5 M Sucrose, 0,1 M Tris-HCl, pH 7,5, 2 mM EDTA, und 2x-Protease-Inhibitor-Cocktail) und halten Sie sie bei 4 ° C auf Eis.
- Zellpellet (frisch oder gefroren) in gleichen Volumen 2x Lysepuffer. Hinzufügen 1x Volumen von Glasperlen. Vortex kräftig bei 4 ° C für 10 min.
- In 2 Volumen 1x Lysepuffer und mischen. Centrifuge Zellextrakte bei 1.500 × g bei 4 ° C für 10 min pelletiert ungebrochen Zellen und Kerne.
- Den Überstand mit einer Pipette in ein frisches kühles Zentrifugenröhrchen und bewahren Zellextrakten auf Eis bis zum Abschluss des Experiments.
2. Bestimmen Sie die Proteinkonzentration der Zellextrakt unter Verwendung von Protein Estimation Kit Wie Bicinchoninsäure-Assay
- Bereiten Sie die Bicinchoninsäure (BCA) (1 ml / Röhrchen) durch Mischen des BCA und Kupfer (II) sulfate in einem Verhältnis von 49: 1 (v / v).
- Vorbereitung der Proteinstandards von 0, 10, 20, 30, 40, 50 und 60 ug / ml durch Verdünnen einer 10 mg / ml Rinderserumalbumin (BSA) Stammlösung in 1 ml-Aliquots der BCA-Lösung.
- Add 2 ul Zellen Extrakte in 1 ml BCA-Lösung in Duplikaten. Standards und Proteinproben inkubieren für 10 Minuten in einem vorgewärmten 60 ° C Wasserbad.
- Transfer Proben für 3 min Eis. Die Extinktion der Standards und der Proteinproben mit einem Spektrophotometer bei einer Wellenlänge von 562 nm.
- Berechnung der Proteinkonzentration der Zellextrakte unter Verwendung der BSA-Standard als Referenz, wie in dem Protokoll des Herstellers beschrieben. Verdünnte Zellextrakten zu einer Proteinkonzentration von 10 mg / ml mit 1x Lysepuffer ml.
3. enzymatischen Assay in 200 ul pro Rohr
HINWEIS: Führen Sie die folgenden Schritte in einer chemischen Abzugshaube.
- Testen Sie jede Probe in zweifacher Ausfertigung in einem 15 mlkonischen Rohr. Es werden 20 & mgr; l 1 M Tris-HCl pH 7,5 pro Röhrchen und bewahren Sie es auf Eis. Vorbereitung wurden 2 ml Chloroform / Methanol (1: 1 (v / v); Stopp-Lösung) bei Raumtemperatur für jedes Röhrchen.
- Pipettieren Sie 20 ul 1 M Tris-HCl pH 7,5, in jeder 15 ml konischen Röhrchen auf Eis.
- Folgen Strahlenschutzrichtlinien von hier an. Füge das Äquivalent von 0,06 um (0,2 uCi) S - [Methyl- 3 H] Adenosyl-L-Methionin und 50 & mgr; M kaltes SAM pro Röhrchen für insgesamt 50,06 uM SAM. Add x ul kaltem Wasser mit x = 200- (20 (für Puffer) + 20 (für die Zellextrakte) + Volumen von kaltem und radioaktivem SAM) pro Röhrchen.
- Übertragen Sie jede konische Röhrchen in eine vorgewärmte 30 ºC Wasserbad. Hinzufügen zu jedem Röhrchen 20 ul Zellextrakte (Äquivalent von 200 ug Protein), um die Reaktion zu starten. Inkubation für die gewünschte Zeit (0-45 min).
- Stoppen der Reaktion durch Zugabe von 2 ml Chloroform / Methanol (1: 1; v / v; Stopplösung) in jedes Röhrchen. Übertragen Sie die konische Röhrchen auf Raumtemperatur (20-256; C).
4. Lipidextraktion
HINWEIS: Führen Sie die folgenden Schritte in einer chemischen Abzugshaube.
- Fügen Sie 700 ul Wasser zu jedem Röhrchen die enzymatische Reaktion enthaltenden Probe. Vortex kräftig für 30 Sekunden. Zentrifuge bei 1500 × g für 5 min bei RT, die organische von der Wasserphase trennen.
- Übertragen die untere organische Phase in ein neues 15 ml konischen Röhrchen mit einer Pipette. 1 ml Wasser in jedes "Unterphase" Rohr und Wirbel enthält kräftig für 30 Sekunden. Zentrifuge wieder bei 1.500 xg für 5 min, um die organische von der Wasserphase zu trennen.
- Übertragen die untere organische Phase wird in einem Scintillationsröhrchen mit einer Pipette. Trockenproben unter einem Strom von N 2. Entsorgen Sie die Wasserphasen, die das nicht-radioaktive eingebaut SAM und die radioaktiven konische Röhrchen nach Strahlungs Richtlinien.
- 2 ml / Röhrchen Szintillationsflüssigkeit. Messen Sie die eingebaute Radioaktivität mit einem Scintillations counter gemäß dem Protokoll des Herstellers und die Verwendung Instruments.
- Berechnung der enzymatischen Aktivität in nmol / mg Protein unter Verwendung der folgenden allgemeinen Gleichung:
cpm Wert x 10 3 x [Gesamt (radioaktive und kalt) Konzentration von SAM (mM)] x 5 spezifische Aktivität des radioaktiven SAM (Ci / mmol) x [Konzentration von radioaktivem SAM (mM)]
HINWEIS: der obige Protokoll kann für eine SAM-Konzentration abhängige oder einem Protein abhängig PEMT Test geändert werden. SAM konzentrationsabhängigen enzymatischen Assay wird die Zeitkonstante (15 min, die in dem linearen Bereich ist) und verschiedenen Mengen von kalter SAM gehalten, während für Protein abhängigen PEMT Assay SAM-Konzentration werden dem Assay hinzugefügt (wählten wir 0,05 mM) und Zeit konstant (15 min) gehalten wird. Auch der pH-Wert des Puffers kann wie erforderlich, wenn einige PEMT Enzyme haben eine unterschiedliche optimale pH-Wert verändert werden.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Figur 1 zeigt eine zeitabhängige PEMT Assay, der mit Leishmania Gesamtzellextrakt als Enzymquelle mit endogenen PE als Substrat durchgeführt wurde. Die Menge der Radioaktivität in der organischen Phase wurde durch Szintillationszählung quantifiziert. Die erhaltenen Zahlen wurden genutzt, um die Menge an tritiiertem Methylgruppen auf PE übertragen berechnen. Die PEMT Aktivität war linear für ca. 20 min. Es erreichte dann ein Plateau bei rund 30 min, wonach es blieb für weitere 15 Minuten konstant. Wie erwartet, wurde PEMT Aktivität nachgewiesen, wenn keine Zellextrakte wurden auf den Test (Figur 2) aufgenommen. Ferner wurde diese Aktivität in Gegenwart von 100 uM Octadecyltrimethylammoniumbromid, die ein Inhibitor L. aufgehoben Haupt PE Methyltransferasen LMJ PEM1 und LMJ PEM2 14. PEMU-Aktivität war auch Proteinkonzentration dependent, und diese Aktivität war linear proportional zu der Menge an Protein für den Enzymtest (Figur 3) angelegt wird. Schließlich wurde eine SAM-Konzentration abhängig PEMT Assay durchgeführt, bei denen steigende Konzentrationen von SAM wurden (Abbildung 4) getestet. PEMU-Aktivität erreichte ein Plateau bei SAM-Konzentration von etwa 0,5 mm. Alle zusammen, diese vier Assays demonstrieren, dass PEMT Aktivität ist spezifisch und kann von Gesamtzellextrakten, ohne die Notwendigkeit, das Enzym (e) zu reinigen, gemessen werden.
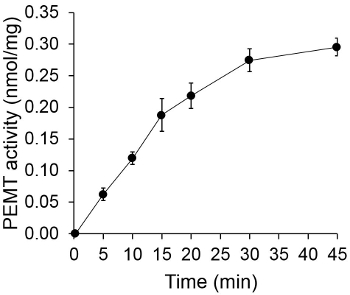
Abbildung 1 Zeitabhängige PEMT Assay. Der Enzymtest wurde zweimal in zweifacher Ausführung mit 0,2 mg ganzen Leishmania Zellextrakten als eine Funktion der Zeit durchgeführt. PEMT Aktivität wird als nmol Methylgruppen auf PE übertragen pro mg Protein und p repräsentierter hr. Bevor sie dem Assay-Lösung, die SAM hinzugefügt: für Zeit "O", Zellextrakte wurden zunächst mit 2 ml Stopplösung aus Chloroform / Methanol (1, Vol 1) hergestellt gemischt. Standardabweichungen werden gezeigt.
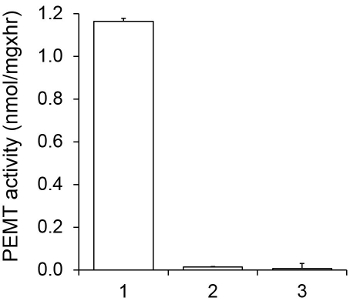
Abbildung 2. Spezifität der PEMT Aktivität. PEMT Assay wurde doppelt in zweifacher Ausführung für 15 min in Gegenwart von 0,05 mM SAM ausgeführt. 1, 0,2 mg Proteinextrakt; 2, keine Zellextrakt; 3, 0,2 mg Protein und 0,1 mM Octadecyltrimethylammoniumbromid. Standardabweichungen werden gezeigt.
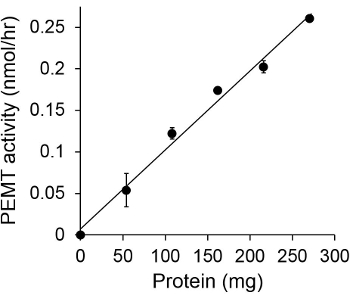
Abbildung 3. Protein abhängigen PEMU-Test. Der Enzymtest wurde zweimal durchgeführt, in derDuplikat in Abwesenheit (Punkt "O") oder in Gegenwart von verschiedenen Mengen von Leishmania-Proteine mit 0,05 mM SAM 15 min. Standardabweichungen werden gezeigt.
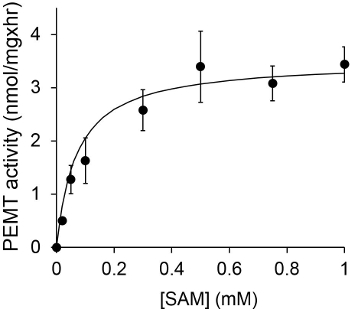
Figur 4. Die PEMT Assay wurde doppelt in zweifacher Ausführung mit 0,2 mg ganzen Leishmania-Zellextrakten in der Gegenwart verschiedener Konzentrationen von SAM für 15 min durchgeführt. Die Standardabweichungen werden gezeigt.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Diese einfache, schnelle PEMT Assay ermöglicht die Quantifizierung von methylierten Formen von PE, die aus der Übertragung von radioaktiver Methylgruppen von SAM auf PE mit Gesamtzellextrakt als Proteinquelle führt. Es ist schnell, empfindlich, reproduzierbar, und auch für die gereinigten Enzyme 17 geeignet. Monomethyl- oder Dimethyl-PE zu dem Assay zugegeben werden, wenn die Methyltransferase von Interesse spezifisch auf diesen Substraten anstatt PE 12,13,18,19. Wenn gereinigt PEMT Enzym verwendet wird, können PE zu dem Assay zugesetzt werden. Eine Einschränkung dieses Protokolls ist, daß der Test nicht die Reaktionsprodukte (Monomethyl-PE, Dimethyl-PE oder PC) zu identifizieren. Allerdings ist die Identität der Reaktionsprodukte (Monomethyl-PE, Dimethyl-PE, PC) kann durch eindimensionale Dünnschichtchromatographie wie in 20,21 beschrieben, analysiert werden. Ferner können einige der SAM Abbauprodukte wie S -adenosylhomocysteine und 5'-Methylthioadenosin PEMT Aktivität hemmen by Feedback-Hemmung. Jedoch Leishmania besitzt einen S -adenosylhomocysteine Hydrolase 22, spaltet S -adenosylhomocysteine in Adenin und S -ribosylhomocysteine und eine methylthioadenosin Phosphorylase, die Adenin und methylthioribose-1-phosphat 23 erzeugt. Es ist jedoch nicht bekannt, ob S -adenosylhomocysteine Hydrolase und Methylthioadenosin Phosphorylase Aktivitäten hoch genug, um wirksam zu metabolisieren S -adenosylhomocysteine und Methylthioadenosin jeweils so daß keine Hemmung der Aktivität auftritt PEMT sind. In dem Fall S -adenosylhomocysteine Hydrolase und / oder 5'-methylthioadenosin metabolischen Enzyms sind in der Zelle von Interesse fehlt, Zugabe der jeweiligen gereinigten rekombinanten Enzyme zu dem Assay zugegeben, um die Rückkopplungshemmung durch SAM Abbauprodukte 24,25 entlasten, 26.
Es gibt vier wichtige Schritte in diesem Protokoll: i)die Protease-Inhibitor-Cocktail-Pulver zu dem Lysepuffer kurz vor der Verwendung (Schritt 1.1) aufgenommen; ii) die Gesamtzellextrakten sind in den folgenden Stunden nach der Herstellung (nach Schritt 1.4 verwendet werden); iii) in den Lipid-Extraktionsschritte (Schritte 4.2 und 4.4), ist Vorsicht angewendet werden, um nicht zu übertragen eine der Interphase oder Wasserphase, die das überschüssige tritiierten SAM enthält, und iv) die radioaktiven S aliquotieren - [Methyl- 3 H] Adenosyl-L-methionin und Kalt Reagenz nach Empfang als wiederholte Zyklen von Einfrieren und Auftauen verschlechtern sie in 5'-methylthioadenosin und Homoserinlacton, gefolgt von Hydrolyse zu Adenin und S -pentosylmethionine 26,27,28, die mangels entfallen kann messbaren PEMT Aktivität. Schlechte Gesamtzellextrakten kann auch ohne die enzymatische Aktivität verantwortlich sein. In diesem Fall kann die Qualität des Zellextraktes durch Messen andere bekannte enzymatische Aktivität beurteilt werden. Last, mangelnde PEMU-Aktivität kann auf der PEMU en vertrauenEnzym spezifisch für Monomethyl-PE oder Dimethyl-PE anstatt PE. Zugabe von Monomethyl-PE und / oder Dimethyl-PE Substrate zu dem Assay, wiederhergestellt PEMT Aktivität.
Eine alternative, nicht-radioaktive SAM abhängigen Methyltransferase-Assay wurde entwickelt, das auf die enzymatische Quantifizierung von Homocystein, die Transmethylierungsprodukte der SAM 29 stützt. Dieses Protokoll wurde erfolgreich in PE spezifische Methyltransferase-Aktivität zu messen 16 angewendet. Unglücklicherweise ist dieser Test nur für gereinigtes Enzym und nicht für Gesamtzellextrakt, und nicht die Identität der Reaktionsprodukte zeigen nicht.
Die vorliegende Protokoll kann möglicherweise zu jedem Zelltyp angewandt werden. Zusätzlich kann dieser Assay verwendet werden, um potenzielle spezifisch für eine PEMT von Interesse im Zusammenhang mit der Erprobung neuer antimikrobieller Verbindungen gegen bakterielle Infektionen oder neuartiger Therapeutika gegen Diät-induzierten Atherosklerose bekämpfen Medikamente testen, AdipositasUnd Insulinresistenz.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Kein Interessenkonflikt erklärt
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



