Introduction
Fosfatidiletanolamina metiltransferasi (PEMT) enzimi catalizzano l'attacco covalente di uno o più gruppi metilici utilizzando S -adenosylmethionine (SAM) come il gruppo metilico donatore sul PE, PE o monometil-dimetil-PE dare monometil-PE, dimetil-PE e / o fosfatidilcolina (PC). Questi enzimi sono quasi onnipresenti nelle cellule animali e funghi. Possono anche essere trovati in alcune piante 1 e circa il 10% dei batteri, in particolare quelli che interagiscono con eucarioti 2.
PEMTs sono rilevanti alla biologia della cellula non solo contribuendo alla produzione di PC, che è la classe principale lipidi nelle cellule animali, ma anche compiendo altre importanti funzioni cellulari. Nei mammiferi, PEMTs sono espressi principalmente nel fegato dove sono richiesti per il normale secrezione di lipoproteine a bassissima densità e contribuiscono anche all'obesità indotta dalla dieta 3, aterosclerosi 4, e l'insulina resistonoANCE 5. Inoltre, PEMT mammiferi sono espressi anche negli adipociti, anche se a livelli più bassi, e partecipare a deposizione di grasso 6, 7. Ruolo PEMT nello sviluppo del cancro 8, 9 apoptosi, e la crescita delle cellule 10 sono stati anche dimostrato. Nei batteri, enzimi PEMT hanno dimostrato di essere importante per la normale crescita delle cellule 2, la virulenza 2, e simbiosi con la pianta ospite 11.
L'obiettivo e le motivazioni del presente protocollo è quello di misurare l'attività PEMT da estratti cellulari interi, senza la necessità di purificare l'enzima. Due protocolli distinti sono stati sviluppati per misurare l'attività PEMT. Il primo e più comune misura il trasferimento del gruppo metilico triziata da SAM radioattivo su PE, che è l'argomento di questo articolo. Questo protocollo è stato originariamente sviluppato per misurare l'attività PEMT dal lievito 12 e cellule di mammifero (fegato) 13 per ottenere un understANDing di biosintesi PC in queste cellule, nonché per determinare la specificità di questi enzimi. Successivamente, questa tecnica è stata applicata ad altri tipi di cellule, come i batteri 2 (utilizzando un valore pH basico per il test se 15) e protozoi parassiti 14. Questa tecnica può essere utilizzata con estratti cellulari interi nonché enzima purificato, e può potenzialmente essere applicato a qualsiasi sistema di estratto cellulare. Un test non radioattivo è stato progettato che si basa sulla quantificazione enzimatica di S -adenosylhomocysteine, il prodotto di transmetilazione SAM 16. Quest'ultimo test può essere più conveniente in quanto non comporta radioattività ma è adatto solo per gli enzimi purificati.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. cella estratto Preparazione
- Crescere le cellule Leishmania in una bottiglia di plastica sterile, sigillato con tappo a tenuta d'aria a 26 ° C in un mezzo fatto di 1x M199 supplementato con 20 mM HEPES pH7.4, 100 U / ml di penicillina, 100 mg / ml di streptomicina, 5 mg / ml eme , 0,35 g / L Naco 2 H, adenina mM 0.1, e 2 mg / ml biopterina senza agitare. Raccogliere le cellule per centrifugazione a 1.500 g per 5 minuti a 4 ° C quando raggiungono una densità cellulare di 1-2 x 10 7 / ml.
- Eliminare il surnatante con una pipetta e lavare le cellule risospendendo il pellet cellulare con una pipetta sierologica a metà del volume del cultura del fosfato salino freddo tamponata (137 mM NaCl, 2.7 mM KCl, Na 10 mm 2 HPO 4, 1.8 KH mM 2 PO 4, pH7.4). Smaltire surnatanti cellulari secondo le linee guida di sicurezza BL2.
- Centrifugare le cellule di nuovo a 1.500 g per 5 minuti a 4 ° C. Eliminare il surnatante con una pipetta. Procedere to il prossimo passo elastico, congelare il pellet di cellule in azoto liquido per la conservazione a lungo termine a -80 ° C (fino a tre mesi).
- Preparare 2x tampone di lisi (0,5 M di saccarosio, 0,1 M TrisHCl, pH7.5, 2 mM EDTA, e inibitore della proteasi 2x cocktail) e tenerlo a 4 ° C su ghiaccio.
- Risospendere il pellet cellulare (fresco o congelato) in volume uguale di tampone di lisi 2x. Aggiungere il volume 1x di perline di vetro. Vortex vigorosamente a 4 ° C per 10 min.
- Aggiungere 2 volumi di tampone di lisi 1x e mescolare. Estratti cellulari centrifugare a 1500 xg a 4 ° C per 10 minuti a pellet cellule intatte e nuclei.
- Trasferire il surnatante con una pipetta in una provetta da centrifuga fresca aperta e mantenerlo estratti cellulari in ghiaccio fino al completamento dell'esperimento.
2. Determinare la concentrazione della proteina del estratto cellulare utilizzando proteine Stima Kit Come bicinconinico Acid Assay
- Preparare la soluzione di acido bicinconinico (BCA) (1 ml / tubo) mescolando il BCA e rame (II) sulfate in un rapporto di 49: 1 (v / v).
- Preparare gli standard proteici di 0, 10, 20, 30, 40, 50, e 60 ug / ml diluendo un / ml albumina di siero bovino (BSA) soluzione madre 10 mg in 1 ml aliquote della soluzione BCA.
- Aggiungere 2 ml di cellule estratti in 1 ml di soluzione di BCA in duplicato. Incubare standard e campioni di proteine per 10 minuti in un bagno d'acqua a 60 ° C pre-riscaldato.
- Campioni di trasferimento di ghiaccio per 3 min. Misurare l'assorbanza degli standard e dei campioni di proteine con uno spettrofotometro ad una lunghezza d'onda di 562 nm.
- Calcolare la concentrazione proteica degli estratti cellulari utilizzando lo standard BSA come riferimento, come descritto nel protocollo del produttore. Diluire estratti cellulari ad una concentrazione proteica di 10 mg / ml con tampone di lisi 1x.
3. enzimatica Assay in 200 pl per Tubo
NOTA: Eseguire le seguenti operazioni in una cappa chimica.
- Testare ciascun campione in duplicato in 15 mltubo conico. Preparare 20 ml 1 M TrisHCl pH 7,5 per tubo e tenerlo in ghiaccio. Preparare 2 ml di cloroformio / metanolo (1: 1 (v / v); soluzione di stop) a temperatura ambiente per ogni provetta.
- Pipettare 20 ml di 1 M pH 7,5 TrisHCl in ciascun tubo da 15 ml su ghiaccio.
- Seguire le linee guida di sicurezza di radiazioni da qui in poi. Aggiungere l'equivalente di 0,06 mM (0,2 pCi) S - [Methyl- 3 H] adenosil-L-metionina e 50 mM freddo SAM per provetta per un totale di 50,06 mM di SAM. Aggiungi x ml di acqua fredda dove x = 200- (20 (per il buffer) + 20 (per estratti cellulari) + volume di freddo e radioattivi SAM) per provetta.
- Trasferire ogni tubo conico a 30 ° C bagno di acqua preriscaldata. Aggiungere a ciascuna provetta 20 l di estratti cellulari (pari a 200 mg di proteine) per attivare la reazione. Incubare per il tempo desiderato (da 0 a 45 min).
- Arrestare la reazione aggiungendo 2 ml di cloroformio / metanolo (1: 1; v / v; soluzione di arresto) ad ogni provetta. Trasferire il tubo conico di RT (20-256; C).
4. lipidi Estrazione
NOTA: Eseguire le seguenti operazioni in una cappa chimica.
- Aggiungere 700 ml di acqua a ciascuna provetta contenente il campione reazione enzimatica. Vortex energicamente per 30 sec. Centrifugare a 1500 xg per 5 minuti a RT per separare il organica dalla fase acquosa.
- Trasferire la fase organica inferiore in un nuovo tubo da 15 ml con una pipetta. Aggiungere 1 ml di acqua per ogni "fase inferiore" contenente provetta e agitare vigorosamente per 30 secondi. Centrifugare nuovamente a 1.500 xg per 5 min per separare il organica dalla fase acquosa.
- Trasferire la fase organica inferiore in un tubo scintillazione con una pipetta. Campioni secco sotto flusso di N 2. Smaltire le fasi d'acqua contenenti la SAM radioattivo non incorporato e tubi coniche radioattive come da linee guida di radiazione.
- Aggiungere 2 ml / tubo di scintillazione liquida. Misurare la radioattività incorporata con un paio di scintillazionenter secondo il protocollo dei costruttori e l'uso dello strumento.
- Calcolare l'attività enzimatica in nmol / mg proteina usando la seguente equazione generale:
valore cpm x 10 x 3 [totale (radioattivi e freddo) concentrazione di SAM (mM)] x 5 attività specifica di SAM radioattivi (Ci / mmol) x [concentrazione di SAM radioattivi (mM)]
NOTA: il protocollo di cui sopra può essere modificato per una concentrazione SAM dipendente o una proteina dipendente test PEMT. Per concentrazione SAM saggio enzimatico dipendente, il tempo viene mantenuta costante (15 min, che è nel range lineare) e varie quantità di freddo SAM vengono aggiunti al dosaggio mentre per proteina dipendente dosaggio PEMT, concentrazione SAM (abbiamo scelto 0,05 mM) e tempo sono mantenuto costante (15 min). Anche il pH del tampone può essere cambiata come necessario se alcuni enzimi PEMT hanno un diverso valore ottimale del pH.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
La figura 1 mostra un tempo dipendente PEMT dosaggio, che è stata effettuata con Leishmania estratto cellula intera come fonte dell'enzima utilizzando PE endogena come substrato. La quantità di radioattività nella fase organica è stata quantificata mediante conteggio a scintillazione. I numeri ottenuti sono stati utilizzati per calcolare la quantità di gruppi metilici triziata trasferiti verso PE. L'attività PEMT era lineare per circa 20 min. È quindi raggiunto un plateau a circa 30 minuti, dopo di che è rimasto costante per altri 15 min. Come previsto, l'attività PEMT non è stato rilevato quando non estratti cellulari sono stati aggiunti al test (Figura 2). Inoltre, tale attività è stata abolita in presenza di 100 mM octadecyltrimethylammonium bromuro, che è un inibitore di L. principali metiltransferasi PE LMJ PEM1 e LMJ PEM2 14. Attività PEMT era anche proteine concentrazione dependent, e questa attività è linearmente proporzionale alla quantità di proteine applicata per il saggio enzimatico (Figura 3). Infine, una concentrazione SAM dosaggio PEMT dipendente è stata effettuata, in cui concentrazioni crescenti di SAM sono stati testati (Figura 4). Attività PEMT raggiunto un plateau a concentrazioni SAM di circa 0,5 mm. Tutti insieme, questi quattro saggi dimostrano che l'attività PEMT è specifico e può essere misurata da estratti cellulari totali senza la necessità di purificare l'enzima (s).
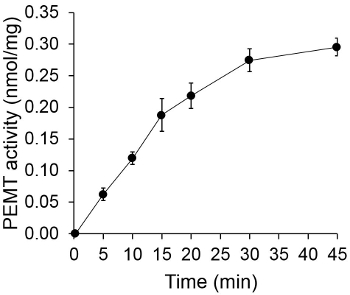
Figura 1. tempo dipendente dosaggio PEMT. Il saggio enzimatico è stato eseguito in duplicato due volte con 0,2 mg di estratti cellulari interi Leishmania in funzione del tempo. Attività PEMT è rappresentato come gruppi metilici nmol trasferiti verso PE per mg di proteine e per ora. Per il tempo "O", estratti cellulari sono stati miscelati con 2 ml di soluzione di arresto in cloroformio / metanolo (1: 1 in volume) prima di essere aggiunto alla soluzione test contenente SAM. Sono mostrati deviazioni standard.
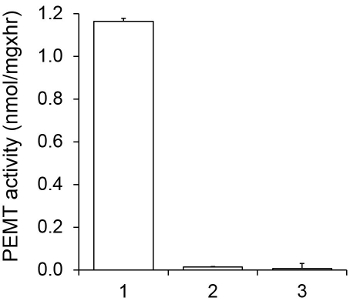
Figura 2. Specificità PEMT attività. PEMT saggio è stata effettuata due volte in duplicato per 15 min in presenza di 0,05 mM SAM. Estratto proteico 1, 0,2 mg; 2, nessun estratto cellulare; 3, 0,2 mg di proteine e 0,1 mm bromuro octadecyltrimethylammonium. Sono mostrati deviazioni standard.
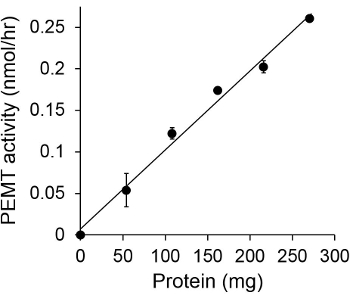
Figura 3. Proteine test PEMT dipendente. Il dosaggio enzimatico è stato eseguito due volte induplicare in assenza (punto 'O') o la presenza di diverse quantità di proteine Leishmania con 0,05 SAM mM per 15 min. Sono mostrati deviazioni standard.
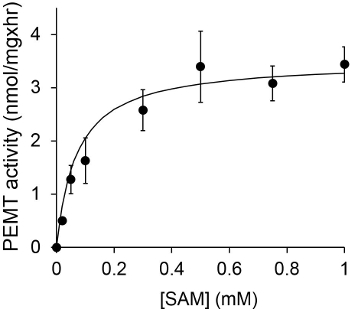
Figura 4. Il saggio PEMT stata effettuata due volte in doppio con 0,2 mg di estratti di cellule intere Leishmania in presenza di varie concentrazioni di SAM per 15 minuti. Le deviazioni standard sono mostrati.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Questa semplice, rapido test PEMT consente la quantificazione di forme metilate di PE che deriva dal trasferimento di gruppi metilici radioattive da SAM su PE utilizzando estratto cellulare tutto come fonte proteica. È veloce, sensibile, riproducibile e adatto anche per enzimi purificati 17. Monomethyl- o dimetil-PE possono essere aggiunti al test se il metiltransferasi di interesse specifico per questi substrati anziché PE 12,13,18,19. Se viene utilizzato purificato enzima PEMT, PE può essere aggiunto al dosaggio. Una limitazione di questo protocollo è che il test non identifica i prodotti reazioni (monometil-PE, dimetil-PE, o PC). Tuttavia, l'identità dei prodotti di reazione (monometil-PE, dimetil-PE, PC) può essere ulteriormente analizzato mediante una cromatografia su strato sottile dimensionale come descritto in 20,21. Inoltre, alcuni dei prodotti di degradazione SAM come S -adenosylhomocysteine e 5'-metiltioadenosina potrebbe inibire l'attività PEMT bl'inibizione di feedback a. Tuttavia, Leishmania possiede una idrolasi S -adenosylhomocysteine 22, che scinde S -adenosylhomocysteine in adenina e S -ribosylhomocysteine, e un fosforilasi metiltioadenosina, che produce adenina e methylthioribose-1-fosfato 23. Tuttavia, non è noto se S -adenosylhomocysteine idrolasi e attività fosforilasi metiltioadenosina sono abbastanza alto per metabolizzare efficientemente S -adenosylhomocysteine e metiltioadenosina, rispettivamente, in modo che non si verifichino inibizione dell'attività PEMT. Nel caso S idrolasi -adenosylhomocysteine e / o 5'-metiltioadenosina enzima metabolico sono assenti nella cella di interesse, l'integrazione dei rispettivi enzimi ricombinanti purificati, può essere aggiunto al test per alleviare l'inibizione di feedback da SAM prodotti di degradazione 24,25, 26.
Ci sono quattro passaggi critici di questo protocollo: i)il cocktail inibitore di proteasi in polvere deve essere aggiunto al tampone di lisi appena prima dell'uso (passo 1.1); ii) l'intera cella estratti debbono essere utilizzati nei seguenti ore dopo la preparazione (dopo il punto 1.4); iii) durante le fasi di estrazione dei lipidi (passi 4.2 e 4.4), cautela deve essere applicato a non trasferire qualsiasi dell'interfase o acqua di fase, che contiene l'eccesso triziata SAM, e iv) Aliquotare S radioattivi - [Methyl- 3 H] adenosil-L-metionina e reattivo freddo al momento della ricezione da ripetuti cicli di congelamento e disgelo degradare in 5'-metiltioadenosina e lattone omoserina seguita da idrolisi di adenina e S -pentosylmethionine 26,27,28, che può spiegare la mancanza di attività PEMT misurabile. Estratti di cellule intere Bad può anche essere responsabile di alcuna attività enzimatica. In questo caso, la qualità dell'estratto cellulare può essere valutata misurando un'altra nota attività enzimatica. Infine, la mancanza di attività PEMT può contare sulla PEMT enzyme specificamente concepita per monometil-PE o dimetil-PE anziché PE. L'aggiunta di monometil-PE e / o substrati di dimetil-PE per il campione potrebbe ripristinare l'attività PEMT.
Un'alternativa, SAM non radioattivo dosaggio metiltransferasi dipendente è stato sviluppato, che si basa sulla quantificazione enzimatica dell'omocisteina, i prodotti di transmetilazione SAM 29. Questo protocollo è stato applicato con successo per misurare PE attività metiltransferasi specifica 16. Purtroppo, questo saggio è adatto solo per enzima purificato e non per intero estratto cellulare, e non rivela l'identità dei prodotti di reazione o.
Il presente protocollo può eventualmente essere applicato a qualsiasi tipo di cellula. Inoltre, questo test può essere usato per testare potenziali farmaci specifici per un PEMT di interesse nel contesto della sperimentazione di nuovi composti anti-microbici per combattere le infezioni batteriche o nuove terapie contro l'aterosclerosi indotta dalla dieta, obesità, E insulino-resistenza.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Nessun conflitto di interessi dichiarati
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



