Introduction
포스파티딜 에탄올 아민의 메틸화 된은 (PEMT) 효소 모노-PE, 디메틸 PE 및 / 또는 제공하기 위해 PE, 모노 - PE 또는 디메틸 PE 상 메틸기 공여체로서 S의 -adenosylmethionine (SAM)을 사용하여 하나 이상의 메틸 그룹의 공유 결합을 촉진 포스파티딜콜린 (PC). 이 효소는 동물 세포와 곰팡이에 거의 어디에나 있습니다. 이들은 또한 몇몇 식물 1 세균의 약 10 %, 진핵 2와 상호 작용하는, 특히에서 찾을 수있다.
PEMTs이뿐만 아니라, 다른 중요한 세포 기능을 다함으로써 동물 세포에서 주요 지질 클래스 PC의 생산에 기여함으로써 세포 생물학에 적합하다. 포유류에서 PEMTs 주로 그들은 매우 저밀도 지질 단백질의 정상적인 분비에 필요한 간에서 발현되며, 또한식이 유도 비만 (3), 동맥 경화증 (4)에 기여하고, 인슐린 저항ANCE 5. 또한, 포유류 PEMT은 비록 낮은 수준으로, 지방 세포에서 발현, 지방 침착 (6, 7)에 참여하고 있습니다. 암 발생 8, 9 사멸 및 세포 증식 10 PEMT 역할도 입증되었다. 박테리아, PEMT 효소는 숙주 식물 (11)와 정상 세포 성장 2, 2 병독성과 공생 중요한 것으로 밝혀졌다.
본 프로토콜의 목적 및 근거는 효소를 정제 할 필요없이 전체 세포 추출물로부터 PEMT 활성을 측정하는 방법이다. 두 가지 프로토콜 PEMT 활성을 측정하기 위해 개발되었다. 첫 번째 가장 일반적인 하나는이 문서의 주제 PE, 상 방사성 SAM에서 삼중 메틸 그룹의 전송을 측정한다. 이 프로토콜은 원래 underst을 얻기 위해 효모 및 포유 동물 세포 (12) (간) (13)로부터 PEMT 활성을 측정하기 위해 개발되어왔다이들 세포에서 PC 생합성 AND 작업뿐만 아니라 이들 효소의 특이성을 결정한다. 나중에,이 기술 및 원생 동물 기생충 14 (15 비록 분석 용 염기성 pH 값을 이용하여) (2) 박테리아와 같은 다른 세포 유형에 적용되고있다. 이 기술은 전체 세포 추출물뿐만 아니라, 정제 된 효소로 사용할 수 있고, 잠재적으로 모든 세포 추출물 시스템에 적용 할 수있다. 비 방사성 분석도의 S -adenosylhomocysteine, SAM (16)의 transmethylation 생성물의 정량 효소에 의존하는 설계되었다. 이 방사능을 포함하지 않는 한 후자의 분석은 더 편리 할 수 있지만 정제 효소에만 적합합니다.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. 세포 추출물 준비
- 20 mM의 HEPES pH 7.4로, 100 U는 / ㎖ 페니실린, / ㎖ 스트렙토 마이신, 5 μg의 / ㎖ 헴 (heme) 100 μg의 보충 1X M199 만든 배지에서 26 ºC에서 기밀 캡으로 밀봉 멸균 플라스틱 병에 슈만 편모충 세포를 성장 흔들림없이, 0.35 G / L 나코 2 H, 0.1 mM의 아데닌, 2 μg의 / ㎖ biopterin. 그들은 1-2 X 107 / ml의 세포 밀도에 도달했을 때 4 ºC에서 5 분간 1,500g에서 원심 분리하여 세포를 수확.
- 피펫으로 뜨는을 취소하고 차가운 인산염 완충 식염수의 문화의 볼륨 (137 mM의 염화나트륨, 2.7 밀리미터의 KCl의 절반에 혈청 학적 피펫으로 세포 펠렛을 재현 탁하여 세포를 씻어, 10 mM의 나 2 HPO 4, 1.8 mM의 KH 2 PO 4, pH 7.4로). BL2 안전 지침에 따라 세포의 상층 액을 폐기하십시오.
- 원심 분리기 세포를 다시 4 ºC에서 5 분 1,500g에서. 피펫으로 상층 액을 버린다. T를 진행또는 다음 공정 o를하면 -80 ºC (최대 세 개월)에 장기 저장을 위해 액체 질소에서 세포 펠렛을 동결 스냅.
- 배 용해 버퍼 (0.5 M 자당, 0.1 M TrisHCl, pH7.5, 2 mM의 EDTA 및 배 프로테아제 억제제 칵테일)를 준비하고 얼음에 4 ºC에서 보관하십시오.
- 배속 용해 완충액의 동일 부피 (신선하거나 냉동) 세포 펠릿을 재현 탁. 유리 구슬의 1X 볼륨을 추가합니다. 소용돌이 적극적으로 10 분 동안 4 ºC에서.
- 1X 용해 버퍼의 2 볼륨을 추가하고 혼합한다. 10 분 동안 4 ºC에서 1,500 XG에 원심 분리기 세포 추출물은 끊어지지 세포와 핵 펠렛합니다.
- 신선한 시원한 원심 분리기 튜브에 피펫으로 뜨는을 전송하고 실험이 완료 될 때까지 얼음에 세포 추출물을 유지합니다.
2. 이러한 Bicinchoninic 산 분석으로 단백질 평가 키트를 사용하여 세포 추출물의 단백질 농도를 결정
- bicinchoninic 산을 준비 (BCA) 솔루션 (BCA)과 구리 (II)를 혼합하여 (1 ㎖ / 튜브)의(49)의 비율 ulfate : 1 (V / V).
- 단백질 표준을 제조 0, 10, 20, 30, 40, 50, 및 BCA 용액 1 ml의 분취 량으로 10 ㎎ / ㎖ 소 혈청 알부민 (BSA) 원액을 희석하여 60 ㎍ / mL로.
- 중복에 BCA 용액 1 ml의 세포 추출물의 2 μl를 추가합니다. 미리 예열 60 ℃ 수조에서 10 분 동안 표준 및 단백질 시료를 배양한다.
- 전송 샘플은 3 분 동안 얼음. 562 nm의 파장에서 분광 광도계 표준 및 단백질 샘플의 흡광도를 측정한다.
- 제조사의 프로토콜에 기술 된 바와 같이 참조로서 BSA 표준을 사용하여 세포 추출물의 단백질 농도를 계산한다. 1X 용해 완충 용액과 10 밀리그램 / 단백질의 농도로 세포 추출물을 희석.
튜브에 200 μL 3. 효소 분석
참고 : 화학 후드에서 다음 단계를 수행하십시오.
- 15 ml의 중복 각 샘플을 테스트원뿔 튜브. 튜브 당 20 μL (1) M TrisHCl pH가 7.5을 준비하고 얼음에 보관하십시오. 각각의 튜브에 대한 실온에서; (솔루션을 정지 1 (V / V) 1) 클로로포름 / 메탄올 2 ㎖를 준비합니다.
- 피펫 얼음에 각 15 ML 원뿔 튜브에 1 M TrisHCl의 pH 7.5의 20 μL.
- 여기에에서 방사선 안전 지침을 따르십시오. SAM의 50.06 μm의 총 튜브 당 [메틸 3 H] 아데노-L 메티오닌 50 μm의 차가운 SAM - 0.06 μM (0.2 μCi를) S에 해당하는 추가. 찬물 μL 추가 x 여기서 x = 200 - ((20) (+ 세포 추출물 20 () + 추위와 방사성 SAM의 볼륨) 버퍼) 튜브 당.
- 미리 예열 30 ºC 물을 욕조에 각 원뿔 튜브를 전송합니다. 반응을 시작하기 위해 각 튜브에 세포 추출물 (단백질 200 μg의 동일)의 20 μl를 추가합니다. 원하는 시간 (0-45 분)에 대한 품어.
- 클로로포름 / 메탄올 2 ㎖ 첨가하여 반응을 정지 (1 : 1, V / V, 정지 용액) 각각의 튜브를. (실온 20 ~ 25을 원뿔 튜브로 이동6; C).
4. 지질 추출
참고 : 화학 후드에서 다음 단계를 수행하십시오.
- 효소 반응 샘플을 포함하는 각 튜브에 물을 700 μl를 추가합니다. 적극적으로 30 초 동안 소용돌이. RT에서 5 분 동안 1,500 XG 원심 분리기는 물, 유기 상으로부터 분리한다.
- 피펫 새로운 15 ML 원뿔 튜브에 낮은 유기 상을 전송합니다. 30 초 동안 적극적으로 튜브와 소용돌이를 포함하는 각각의 "낮은 단계"에 물 1 ML을 추가합니다. 다시 원심 분리기에서 5 분 동안 1,500 XG에서 수상으로부터 유기물을 분리.
- 피펫 섬광 관에 낮은 유기 상을 전송합니다. N 2의 스트림에서 건조 샘플. 비 방사성 혼입 SAM 방사선 지침에 따라 방사성 원추형 튜브를 함유하는 물 상을 폐기.
- 2 ㎖ / 섬광 액체의 튜브를 추가합니다. 섬광 COU와 통합 된 방사능을 측정nter 제조 업체의 프로토콜과 기기의 사용에 따라.
- 하기 일반 식을 이용을 nmol / mg의 단백질의 효소 활성을 계산한다 :
CPM 값 × 10 3 × [SAM (MM)의 총 (방사성 차가운) 농도] 방사성 SAM (Cl / mmol)의 X의 X 5 특정 활동 [방사성 SAM의 농도 (MM)]
참고 : 위의 프로토콜에 의존 SAM 농도 또는 단백질에 의존 PEMT 분석을 위해 변경 될 수 있습니다. SAM 농도 의존 효소 분석을 위해, 시간 동안 단백질에 의존 PEMT 분석, SAM의 농도 분석에 추가됩니다 상수 (선형 범위에서 15 분) 차가운 SAM의 다양한 양을 유지 (우리는 mM의 0.05를 선택) 및 시간 상수 (15 분) 유지됩니다. 또한 완충액의 pH는 약 PEMT 효소는 다른 최적의 pH 값을 가질 경우, 필요에 따라 변경 될 수있다.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
도 1은 기판으로서 PE를 사용하여 내인성 효소 원으로서 Leishmania의 전체 세포 추출물로 행하여, 시간 종속 PEMT 분석을 보여준다. 유기 상 중의 방사능의 양을 섬광 계수에 의해 정량화 하였다. 얻어진 번호는 PE 삼중 전사 메틸기의 양을 계산하는데 이용되었다. PEMT 활성은 약 20 분 동안 선형이었다. 그런 다음, 또 다른 15 분 동안 일정한 머문 후 약 30 분에서 정체기에 도달했다. 예상대로 더 셀 추출물 분석 (그림 2)에 추가되지 때, PEMT 활동은 검출되지 않았다. 또한,이 활성의 억제제 L. 100 μM의 옥타 데실 브로마이드의 존재하에 폐지 주요 체육 methyltransferases LMJ PEM1 및 LMJ PEM2 (14). PEMT 활성은 또한 단백질 농도 dependen이었다t,이 활성은 효소 분석 (도 3)에 대한인가 된 단백질의 양에 정비례 하였다. 마지막으로, SAM 농도 의존적 PEMT 분석이되는 SAM의 농도 증가 (도 4)을 시험 하였다, 행했다. PEMT 활성은 0.5mm 정도의 SAM 농도에 도달 고원. 모두 함께, 이러한 분석은 네 PEMT 활동이 특정 효소 (들)를 정제 할 필요없이 전체 세포 추출물로부터 측정 될 수 있음을 보여준다.
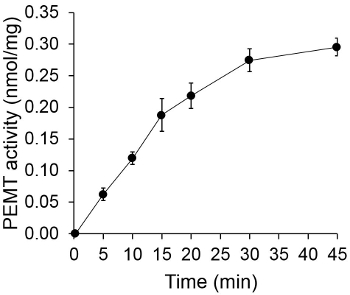
도 1 시간 의존적 PEMT 분석. 효소 분석은 시간의 함수로서 전체 Leishmania의 세포 추출물을 0.2 mg의 중복으로 두번 수행 하였다. PEMT 활성 단백질 (P)의 당 mg PE 전사 nmol의 메틸 그룹으로 표현되는어 시간. SAM을 포함하는 분석 용액에 첨가되기 전에, (부피 기준으로 1, 1) 타임 "O"를 들어, 세포 추출물을 먼저 클로로포름 / 메탄올로 이루어지는 정지 용액 2 ㎖와 혼합 하였다. 표준 편차가 표시됩니다.
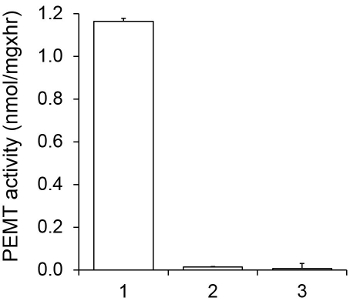
PEMT 분석 0.05 mM의 SAM의 존재에 15 분 동안 중복에 두 번 실시 하였다 특이 PEMT 활동. 그림 2. 1, 0.2 mg의 단백질 추출물; 2, 아니 세포 추출물; 3, 0.2 mg의 단백질 및 0.1 mM의 옥타 데실 브로마이드. 표준 편차가 표시됩니다.
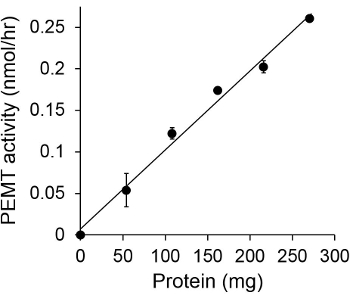
도 3 종속 PEMT 단백질 분석은. 효소 분석은 두 번 수행 된15 분 동안 0.05 mM의 SAM으로 부재 요점 ( 'O') 또는 리 슈만 편모충 단백질의 다양한 양의 존재를 복제. 표준 편차가 표시됩니다.
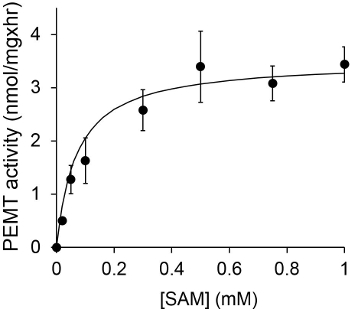
도 4는 분석 PEMT 15 분 동안 SAM의 다양한 농도의 존재 하에서 리 슈만 편모충 전체 세포 추출물을 0.2 mg의 중복으로 두번 수행 하였다. 표준 편차를 나타낸다.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
이 단순한 빠른 PEMT 분석은 단백질 원으로 전체 세포 추출액을 이용하여 PE 상 SAM에서 방사성 메틸기의 전달로 인한 PE의 메틸화 된 형태의 정량을 허용한다. 그것은 빠르고, 민감한, 재현성, 정제 효소 17도 적합하다. 관심 메틸화 된 오히려 PE 12,13,18,19보다 이들 기판에 특정한 경우 Monomethyl- 또는 디메틸 PE는 상기 분석에 첨가 될 수있다. 정제 PEMT 효소가 사용되는 경우, PE는 분석에 첨가 될 수있다. 이 프로토콜의 한계는 분석이 반응 제품 (모노-PE, 디메틸 PE 또는 PC)를 식별하지 않습니다. 20, 21에서 설명하지만, 반응 생성물 (모노-PE, 디메틸 PE, PC)의 아이덴티티는 또한 하나의 차원 박층 크로마토 그래피에 의해 분석 될 수있다. 또한, 이러한 S의 -adenosylhomocysteine 및 5'- methylthioadenosine로서 SAM 분해 산물의 일부는 PEMT 활동 B를 억제 할 수있다Y 피드백 억제. 그러나, 리 슈만 편모충는 S의 -adenosylhomocysteine 가수 분해 효소 (22), 보유하고있는 절단 S -adenosylhomocysteine 아데닌과 S의 -ribosylhomocysteine 및 아데닌과 methylthioribose -1- 인산 (23)을 생성 methylthioadenosine 포스로. 그러나, S의 -adenosylhomocysteine 가수 분해 효소 및 methylthioadenosine 포스 활동 PEMT 활동의 더 억제가 발생하지 않도록 효율적으로 각각 S의 -adenosylhomocysteine 및 methylthioadenosine을 대사 할 정도로 높은 여부를 알 수 없습니다. 인스턴스에서 각 정제 된 재조합 효소를 첨가 SAM 분해물 24,25 의한 피드백 억제를 완화하기 위해 상기 분석에 첨가 될 수있는 관심 셀에 존재 -adenosylhomocysteine 가수 분해 및 / 또는 5'-methylthioadenosine 대사 효소이며, S, (26).
이 프로토콜의 네 가지 중요한 단계가 있습니다 : I)프로테아제 억제제 칵테일 분말은 사용 직전에 용해 버퍼 (단계 1.1)을 첨가 할 수있다; ⅱ) 추출물) 단계 이후에 1.4 제제 (이후 다음 시간 이내에 사용하여야한다 전체 셀; ⅲ) 지질 추출 단계 (4.2 및 4.4 단계),주의 과량 삼중 SAM을 포함하는 계면 또는 물 위상 중 하나를 전송하지 적용되어야하고, ⅳ) 방사성 S 나누어지는 데시 - [메틸 - (3) H] 아데노-L 메티오닌과 동결의 반복주기 등의 수령시 차가운 시약 및 부족으로 설명 할 수있다 아데닌과 S -pentosylmethionine 26,27,28에 가수 분해 5'-methylthioadenosine과 호모 세린 락톤,로 저하 해동 측정 PEMT 활동. 나쁜 전체 세포 추출물도없는 효소 활동을 담당 할 수있다. 이 경우, 세포 추출액의 품질은 다른 공지의 효소 활성을 측정함으로써 평가 될 수있다. PEMT 활성의 마지막 부족 PEMT EN에 의존 할 수있다효소는 모노-PE 또는 디메틸 PE보다는 PE에 해당되는. 모노-PE 및 / 또는 분석에 디메틸 PE 기판 첨가 PEMT 활성을 복원 할 수있다.
대안으로, 비 - 방사성 SAM 종속 메틸화 분석은 호모시스테인의 효소 부량, SAM (29)의 transmethylation 제품에 의존하는, 개발되고있다. 이 프로토콜은 성공적 PE 특정 메틸화 16 활성을 측정하기 위해 적용되어왔다. 불행히도,이 분석은 전체 세포 추출물만을 정제 된 효소에 적합하지 않고, 어느 반응 생성물의 신원을 공개하지 않는다.
본 프로토콜은 가능한 모든 세포 유형에 적용될 수있다. 또한,이 분석이 다이어트에 의한 동맥 경화에 대한 세균 감염 또는 새로운 치료제 싸울 새로운 항균 화합물 테스트의 맥락에서 관심 PEMT 특정 잠재적 인 약물을 테스트하는 데 사용할 수 있습니다, 비만및 인슐린 저항성.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
관심 없음 충돌 선언하지 않습니다
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



