Introduction
Phosphatidyléthanolamine méthyltransférase (OGERP) enzymes catalysent la fixation covalente d'un ou plusieurs des groupes méthyle à l'aide de S -adenosylmethionine (SAM) comme le groupe méthyle donneur sur PE, monométhyl-PE ou diméthyl-PE pour donner monométhyl-PE, le diméthyl-PE et / ou phosphatidylcholine (PC). Ces enzymes sont presque omniprésents dans les cellules animales et les champignons. Ils peuvent également être trouvés dans des plantes 1 et environ 10% des bactéries, en particulier celles qui interagissent avec les eucaryotes 2.
PEMTs correspondant à la biologie de la cellule, non seulement en contribuant à la production de PC, qui est la principale classe de lipides dans les cellules animales, mais aussi en remplissant d'autres fonctions cellulaires importantes. Chez les mammifères, PEMTs sont principalement exprimés dans le foie où ils sont nécessaires pour la sécrétion normale de la lipoprotéine de très basse densité et ils contribuent également à l'obésité induite par l'alimentation 3, 4 athérosclérose, et l'insuline résistentance 5. En outre, OGERP mammifères sont également exprimée dans les adipocytes, mais à des niveaux inférieurs, et de participer à des dépôts de graisse 6, 7. OGERP rôle dans le développement du cancer 8, 9 apoptose, et 10 de la croissance cellulaire a également été démontrée. Chez les bactéries, les enzymes OGERP se sont révélés être importants pour la croissance normale de la cellule 2, 2 virulence, et symbiose avec la plante hôte 11.
Le but et la justification du présent Protocole est de mesurer l'activité OGERP à partir d'extraits de cellules entières, sans la nécessité de purifier l'enzyme. Deux protocoles distincts ont été développés pour mesurer l'activité OGERP. La première et la plus commune mesure le transfert d'un groupe méthyle tritiée de SAM radioactifs sur PE, qui est le sujet de cet article. Ce protocole a été développé à l'origine pour mesurer l'activité de la levure OGERP 12 et des cellules de mammifères (foie) 13 pour obtenir un understanding biosynthèse de PC dans ces cellules ainsi que pour déterminer la spécificité de ces enzymes. Par la suite, cette technique a été appliquée à d'autres types tels que des bactéries de cellules (2 à l'aide d'une valeur de pH basique pour le dosage si 15) et des protozoaires parasites 14. Cette technique peut être utilisée avec des extraits de cellules entières, ainsi que l'enzyme purifiée, et peut potentiellement être appliqué à tout système d'extrait cellulaire. Un essai non radioactif a également été conçu qui repose sur la quantification enzymatique de -adenosylhomocysteine S, le produit de transméthylation de 16 SAM. Ce dernier dosage peut être plus pratique, car elle ne comporte pas de radioactivité, mais elle ne convient que pour des enzymes purifiées.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Extrait 1. Préparation des cellules
- Cultiver les cellules de Leishmania dans une bouteille en plastique stérile scellé avec chapeau d'air étanche à 26 ºC dans un milieu en 1x M199 supplémenté avec 20 mM HEPES pH 7,4, 100 U / ml de pénicilline, 100 ug / ml de streptomycine, 5 ug / ml hème , 0,35 g / L NaCO 2 H, mM adénine 0,1 et 2 ng / ml bioptérine sans agitation. Récolter les cellules par centrifugation à 1500 g pendant 5 min à 4 ° C quand ils atteignent une densité cellulaire de 1-2 x 10 7 / ml.
- Jeter le surnageant avec une pipette et laver les cellules par remise en suspension du culot de cellules avec une pipette sérologique à la moitié du volume de la culture d'une solution saline de phosphate froide tamponnée (NaCl 137 mM, KCl 2,7 mM, Na 10 2 HPO 4, 1,8 mM de KH 2 PO 4, pH 7,4). Éliminer des surnageants de cellules selon les directives de sécurité BL2.
- Centrifuger les cellules à nouveau à 1500 g pendant 5 min à 4 ºC. Jeter le surnageant avec une pipette. Procéder to l'étape suivante ou enclencher geler le culot de cellules dans de l'azote liquide pour stockage à long terme à -80 ºC (jusqu'à trois mois).
- Préparer 2x tampon de lyse (0,5 M de saccharose, 0,1 M TrisHCl, pH 7,5, EDTA 2 mM, et l'inhibiteur de protéase de 2x cocktail) et le conserver à 4 ° C sur de la glace.
- Remettre en suspension le culot cellulaire (frais ou congelé) dans un volume égal de tampon de lyse 2X. Ajouter 1x volume de billes de verre. Vortex vigoureusement à 4 ºC pendant 10 min.
- Ajouter 2 volumes de tampon de lyse 1x et mélanger. Des extraits cellulaires de centrifugeuses à 1500 g à 4 ° C pendant 10 min pour sédimenter les cellules et des noyaux intacts.
- Transférer le surnageant avec une pipette dans un tube de centrifugeuse fraîche et la maintenir extraits cellulaires sur la glace jusqu'à la fin de l'expérience.
2. Déterminer la concentration en protéines de l'extrait cellulaire utilisant le kit Protein Estimation Comme Bicinchoninic Acid Test
- Préparer l'acide bicinchoninique (BCA) solution (1 ml / tube) en mélangeant le BCA et de cuivre (II) sulfate dans un rapport de 49: 1 (v / v).
- Préparer les étalons de protéine de 0, 10, 20, 30, 40, 50, et 60 ug / ml par dilution d'un ml d'albumine de sérum bovin (BSA) solution à 10 mg / des stocks en aliquotes de 1 ml de la solution de BCA.
- Ajouter 2 ul d'extraits de cellules dans 1 ml de solution de BCA en double. Incuber des normes et des échantillons de protéines pendant 10 min dans un bain d'eau à 60 ° C préchauffé.
- Échantillons de transfert de la glace pendant 3 min. Mesurer l'absorbance des étalons et des échantillons de protéines avec un spectrophotomètre à une longueur d'onde de 562 nm.
- Calculer la concentration en protéine des extraits cellulaires en utilisant la BSA comme étalon de référence, comme décrit dans le protocole du fabricant. Diluer extraits cellulaires à une concentration en protéine de 10 mg / ml avec du tampon de lyse 1x.
3. enzymatique de dosage dans 200 pi par tube
REMARQUE: Effectuer les étapes suivantes dans une hotte chimique.
- Tester chaque échantillon en double en 15 mltube conique. Préparer 20 pi 1 M TrisHCl pH 7,5 par tube et le garder sur la glace. Préparation 2 ml d'un mélange chloroforme / méthanol (1: 1 (v / v); solution d'arrêt) à température ambiante pendant chaque tube.
- Pipet 20 pl de 1 M TrisHCl pH 7,5 dans chaque tube de 15 ml conique sur la glace.
- Suivez les directives de sécurité de rayonnement à partir d'ici. Ajouter l'équivalent de 0,06 uM (0,2 uCi) S - [méthyl-3H] adénosyl-L-méthionine et 50 pm SAM froid par tube pour un total de 50,06 M de SAM. Ajouter x ul d'eau froide où x = 200 (20 (pour le tampon) + 20 (pour les extraits cellulaires) + volume de froid et radioactifs SAM) par tube.
- Transférer chaque tube conique à un bain d'eau à 30 ºC préchauffé. Ajouter à chaque tube 20 ul d'extraits cellulaires (équivalent de 200 pg de protéine) pour démarrer la réaction. Incuber pendant le temps désiré (0 à 45 min).
- Arrêter la réaction en ajoutant 2 ml d'un mélange chloroforme / méthanol (1: 1, v / v; solution d'arrêt) à chaque tube. Transférer le tube conique à RT (20-256; C).
4. Extraction des lipides
REMARQUE: Effectuer les étapes suivantes dans une hotte chimique.
- Ajouter 700 ul d'eau à chaque tube contenant l'échantillon de réaction enzymatique. Vortex vigoureusement pendant 30 sec. Centrifuger à 1500 xg pendant 5 min à température ambiante pour séparer le organique de la phase aqueuse.
- Transférer la phase organique inférieure dans un nouveau tube conique de 15 ml avec une pipette. Ajouter 1 ml d'eau à chaque "phase inférieure" et contenant le tube vortex vigoureusement pendant 30 sec. Centrifuger de nouveau à 1500 x g pendant 5 min pour séparer la phase organique de l'eau.
- Transférer la phase organique inférieure dans un tube à scintillation avec une pipette. Les échantillons secs sous un courant de N 2. Éliminer les phases de l'eau contenant de la SAM radioactif non incorporé et les tubes coniques radioactives conformément aux lignes directrices de rayonnement.
- Ajouter 2 ml / tube à scintillation liquide. Mesurer la radioactivité incorporée avec un cou de scintillationnter selon le protocole du fabricant et l'utilisation de l'instrument.
- Calculer l'activité enzymatique en nmol / mg de protéine en utilisant l'équation générale suivante:
valeur cpm x 10 x 3 [total (radioactifs et froid) concentration de SAM (mM)] x 5 activité spécifique de SAM radioactif (Ci / mmol de) Les x [concentration de SAM radioactif (mM)]
REMARQUE: le protocole ci-dessus peut être modifiée pour une concentration SAM dépendante ou un test de OGERP dépendante de la protéine. Pour le dosage enzymatique dépendant de la concentration de SAM, le temps est maintenue constante (15 min, ce qui est dans la plage linéaire) et diverses quantités de SAM froid sont ajoutés à l'essai tandis que pour dépendante dosage de OGERP protéique, la concentration de SAM (nous avons choisi 0,05 mM) et le temps sont maintenus constants (15 min). De plus, le pH du tampon peut être modifiée selon les besoins, si certaines enzymes OGERP ont une valeur de pH optimal différent.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
La figure 1 montre un dosage de OGERP dépendant du temps, qui a été réalisée avec Leishmania extrait de cellules entières comme source d'enzyme endogène PE en utilisant comme substrat. La quantité de radioactivité dans la phase organique a été quantifiée par comptage par scintillation. Les chiffres obtenus ont été utilisés pour calculer le montant de groupes méthyle tritiatés transférés sur PE. L'activité de OGERP était linéaire pendant environ 20 min. Il a ensuite atteint un plateau à environ 30 min, après quoi il est resté constant pendant 15 min. Comme prévu, l'activité n'a pas été détectée OGERP lorsque aucun des extraits cellulaires ont été ajoutés à l'essai (figure 2). En outre, cette activité a été abolie en présence de 100 uM de bromure d'octadécyltriméthylammonium, qui est un inhibiteur de L. grands méthyltransférases PE Lmj PEM1 et Lmj PEM2 14. OGERP activité était également la concentration en protéines dependent, et cette activité est linéairement proportionnelle à la quantité de protéine appliquée pour le test enzymatique (Figure 3). Enfin, un essai de OGERP dépendante de la concentration SAM a été réalisée, dans laquelle des concentrations croissantes de SAM ont été testés (Figure 4). OGERP activité a atteint un plateau à une concentration de SAM d'environ 0,5 mm. Tous ensemble, ces quatre essais démontrent que l'activité spécifique est OGERP et peut être mesuré à partir d'extraits de cellules entières, sans la nécessité de purifier l'enzyme (s).
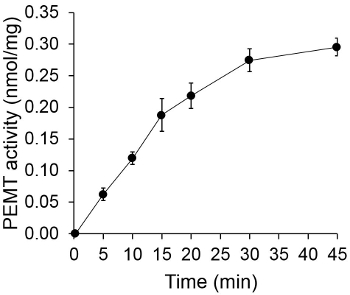
Figure 1. Time OGERP essai de charge. Le dosage enzymatique a été effectuée deux fois en double avec 0,2 mg d'extraits cellulaires entiers de Leishmania en fonction du temps. OGERP activité est représentée en tant que groupes méthyle nmol transférés sur PE par mg de protéine et per h. Pour le temps "O", les extraits cellulaires ont d'abord été mélangés avec 2 ml de solution d'arrêt faite d'un mélange chloroforme / méthanol (1: 1 en volume) avant d'être ajouté à la solution d'essai contenant SAM. Les écarts-types sont présentés.
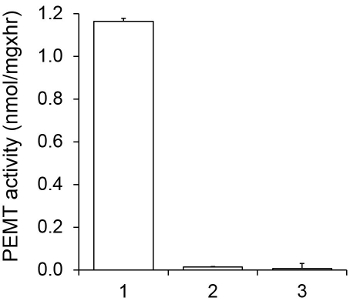
Figure 2. La spécificité de l'activité OGERP. OGERP de dosage a été effectuée deux fois en double exemplaire pendant 15 min en présence de 0,05 mM de SAM. Extrait protéinique 1, 0,2 mg; 2, no extrait cellulaire; 3, 0,2 mg de protéine et 0,1 mM de bromure octadécyltriméthylammonium. Les écarts-types sont présentés.
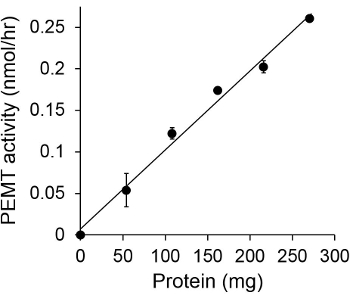
Figure 3. Protéines de dosage OGERP dépendante. Le test enzymatique a été effectuée deux fois dansreproduire en l'absence (le point «O») ou la présence de diverses quantités de protéines Leishmania mM SAM 0,05 pour 15 min. Les écarts-types sont présentés.
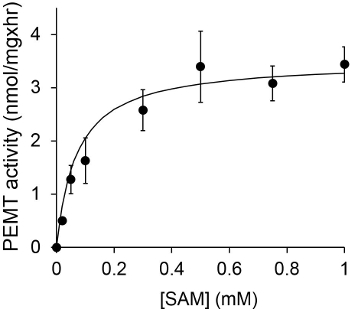
La figure 4. L'analyse de OGERP a été effectuée deux fois en double avec 0,2 mg d'extraits de cellules entières de Leishmania en présence de diverses concentrations de SAM pendant 15 min. Les écarts-types sont présentés.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Ce dosage de OGERP simple, rapide permet la quantification des formes méthylées de PE qui résulte du transfert de groupes méthyle de SAM sur radioactives PE en utilisant un extrait de cellules entières en tant que source de protéine. Il est rapide, sensible, reproductible, et également adapté pour 17 des enzymes purifiées. Monométhylique ou diméthylique-PE peuvent être ajoutés à l'essai si la méthyltransférase d'intérêt est spécifique de ces substrats au lieu de PE 12,13,18,19. Si enzyme purifiée est utilisée OGERP, PE peut être ajouté à l'essai. Une limitation de ce protocole est que le test ne permet pas d'identifier les produits de réaction (monométhylique-PE, diméthyl-PE, ou PC). Cependant, l'identité des produits de réaction (PE-monométhyle, diméthyle-PE, CP) peut en outre être analysé par une Chromatographie en couche mince de dimension tel que décrit dans 20,21. En outre, certains des produits de dégradation tels que la S SAM -adenosylhomocysteine et 5'-méthylthioadénosine peut inhiber l'activité de b OGERPY rétro-inhibition. Cependant, Leishmania possède une hydrolase de -adenosylhomocysteine S 22, S -adenosylhomocysteine qui clive en adénine et S -ribosylhomocysteine, et une méthylthioadénosine phosphorylase, qui produit l'adénine et méthylthioribose-1-phosphate 23. Cependant, on ne sait pas si S -adenosylhomocysteine hydrolase et activités phosphorylase méthylthioadénosine sont suffisamment élevés pour métaboliser efficacement S -adenosylhomocysteine et méthylthioadénosine, respectivement, de sorte qu'aucune inhibition de l'activité de OGERP se produit. Dans le cas -adenosylhomocysteine S hydrolase et / ou 5'-méthylthioadénosine enzyme métabolique sont absents dans la cellule d'intérêt, l'addition des enzymes recombinantes purifiées, respectivement peut être ajouté à l'essai pour soulager la rétro-inhibition par les produits de dégradation SAM 24,25, 26.
Il ya quatre étapes critiques dans ce protocole: i)la poudre inhibiteur de la protéase du cocktail doit être ajoutée au tampon de lyse juste avant l'utilisation (étape 1.1); ii) la cellule entière sont extraits pour être utilisé dans les heures suivantes, après préparation (après l'étape 1.4); iii) au cours des étapes d'extraction des lipides (étapes 4.2 et 4.4), la prudence doit être appliqué à ne pas transférer de l'interphase ou de l'eau phase, qui contient l'excès tritiée SAM, et iv) aliquote S radioactifs - [Methyl- 3 H] adénosyl-L-méthionine et réactif froid lors de la réception que des cycles répétés de gel et de dégel dégrader en 5'-méthylthioadénosine et homosérine lactone suivie par hydrolyse à l'adénine et S -pentosylmethionine 26,27,28, ce qui peut expliquer le manque de activité OGERP mesurable. Extraits de cellules entières Bad peuvent également être responsables pour aucune activité enzymatique. Dans ce cas, la qualité de l'extrait cellulaire peut être évaluée en mesurant une autre activité enzymatique connue. Enfin, le manque d'activité de OGERP peut compter sur le OGERP enenzyme étant spécifique à monométhylique-PE ou diméthyl-PE plutôt que PE. Ajout de monométhyl-PE et / ou diméthyl-PE substrats à l'essai peut restaurer l'activité de OGERP.
Une autre solution, non radioactif SAM méthyltransférase dépendante essai a été mis au point, qui repose sur la quantification enzymatique de l'homocystéine, les produits de transméthylation de 29 SAM. Ce protocole a été appliqué avec succès pour mesurer PE activité méthyltransférase spécifique 16. Malheureusement, cet essai ne convient que pour une enzyme purifiée et non de l'extrait de cellules entières, et ne révèle pas l'identité des produits de réaction non plus.
La présente protocole peut éventuellement être appliquée à tout type de cellule. En outre, ce test peut être utilisé pour tester des médicaments potentiels spécifiques à un OGERP d'intérêt dans le contexte de tester de nouveaux composés anti-microbiens pour lutter contre les infections bactériennes ou de nouvelles thérapies contre l'athérosclérose induite par l'alimentation, l'obésité, Et une résistance à l'insuline.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Aucun conflit d'intérêt déclaré
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



