Introduction
Fosfatidiletanolamina metiltransferase (PEMT) As enzimas catalisam a ligação covalente de um ou mais grupos de metilo utilizando S -adenosylmethionine (SAM) como o doador grupo metilo em PE, PE-monometil ou dimetil-PE para dar monometil-PE, dimetil-PE e / ou fosfatidilcolina (PC). Estas enzimas são quase ubíquo em células animais e de fungos. Eles também podem ser encontrados em algumas plantas 1 e cerca de 10% das bactérias, particularmente aqueles que interagem com 2 eucariotas.
PEMTs são relevantes para a biologia da célula não só por contribuir para a produção de PC, que é a principal classe de lípidos em células animais, mas também por cumprir outras funções celulares importantes. Nos mamíferos, são principalmente PEMTs expressa no fígado, onde eles são necessários para a secreção normal de colesterol de lipoproteína de muito baixa densidade e que também contribuem para a obesidade induzida por dieta 3, 4 aterosclerose, e resistir à insulinaance 5. Além disso, PEMT de mamífero também são expressos em adipócitos, embora a níveis mais baixos, e participar em deposição de gordura 6, 7. PEMT papel no desenvolvimento do cancro 8, 9 a apoptose, e o crescimento celular 10 também foram demonstradas. Em bactérias, as enzimas PEMT têm sido mostrados como sendo importantes para o crescimento normal de células 2, 2 virulência, e a simbiose com a planta hospedeira 11.
O objetivo e razão de ser do presente protocolo é o de medir a atividade PEMT a partir de extratos de células inteiras sem a necessidade de purificar a enzima. Dois protocolos distintos foram desenvolvidos para medir a actividade PEMT. A primeira e mais comum mede-se a transferência do grupo metil tritiada de radioativo SAM em PE, que é o tema deste artigo. Este protocolo foi originalmente desenvolvido para medir a actividade de levedura PEMT 12 e células de mamíferos (fígado) 13 para obter uma understAND de biossíntese PC nestas células, bem como para determinar a especificidade destas enzimas. Mais tarde, esta técnica tem sido aplicada a outros tipos de células tais como bactérias 2 (utilizando um valor de pH de base para o ensaio embora 15) e parasitas protozoários 14. Esta técnica pode ser utilizada com extractos de células completas, bem como de enzima purificada, e, potencialmente, pode ser aplicado a qualquer sistema de extracto celular. Um ensaio não radioactivo também foi projetado que se baseia na quantificação enzimática de S -adenosylhomocysteine, o produto transmetilação de SAM 16. O último ensaio pode ser mais conveniente, uma vez que não envolvem radioactividade, mas é somente apropriado para enzimas purificadas.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Preparação Extracto celular
- Crescer as células de Leishmania em um frasco de plástico estéril, selado com tampa estanque ao ar 26 ° C num meio feito de 1x M199 suplementado com HEPES 20 mM, pH 7,4, 100 U / ml de penicilina, 100 ug / ml de estreptomicina, 5 ug / ml de heme , 0,35 g / L NACo 2H, adenina 0,1 mM, e 2 ug / ml de biopterina sem agitação. Colher as células por centrifugação a 1500 g durante 5 min a 4 ° C quando atingem uma densidade celular de 1-2 x 10 7 / ml.
- Descartar o sobrenadante com uma pipeta e lava-se as células por ressuspensão do sedimento de células com uma pipeta serológica em metade do volume da cultura de solução salina de fosfato tamponada (NaCl 137 mM, KCl 2,7 mM, 10 mM de 2 HPO 4, 1,8 mM de KH 2 PO 4, pH 7,4). Descarte sobrenadantes de células de acordo com as diretrizes de segurança BL2.
- Centrifugar células novamente em 1500 g durante 5 min a 4 ºC. Descartar o sobrenadante com uma pipeta. Prossiga tO passo seguinte ou encaixe congelar o sedimento celular em azoto líquido durante a armazenagem a longo prazo a -80 ° C (até três meses).
- Preparar tampão de lise 2x (0,5 M de sacarose, 0,1 M Tris-HCl, pH 7,5, EDTA 2 mM, e 2x cocktail inibidor de protease) e mantê-lo a 4 ° C em gelo.
- Ressuspender o sedimento de células (frescas ou congeladas) em igual volume de tampão de lise 2x. Adicionar o volume 1x de contas de vidro. Vortex vigorosamente a 4 ºC durante 10 minutos.
- Adicionar 2 volumes de tampão de lise 1x e misturar. Extratos de células centrifugar a 1500 xg a 4 ºC durante 10 minutos para sedimentar as células intactas e núcleos.
- Transferir o sobrenadante com uma pipeta para um tubo de centrífuga de fresco e manter fresco extractos de células em gelo até à conclusão da experiência.
2. Determinar a concentração de proteína do extracto celular utilizando o kit de Estimativa proteína tal como ácido bicinconínico Ensaio
- Prepara-se o ácido bicinconínico solução (BCA) (1 ml / tubo) por mistura do BCA e cobre (II) sulfate numa proporção de 49: 1 (v / v).
- Prepare os padrões de proteína de 0, 10, 20, 30, 40, 50, e 60 ug / ml por diluição de um ml de solução concentrada a 10 mg / albumina de soro bovino (BSA) em aliquotas de 1 mL da solução de BCA.
- Adicionar 2 mL de extractos de células em 1 ml de solução de BCA em duplicado. Incubar as amostras e os padrões de proteína durante 10 minutos num banho de água a 60 ° C pré-aquecido.
- Amostras de transferência de gelo por 3 min. Medir a absorvância dos padrões e das amostras de proteína com um espectrofotómetro a um comprimento de onda de 562 nm.
- Calcula-se a concentração de proteína dos extractos celulares, usando o padrão de BSA, como uma referência, tal como descrito no protocolo do fabricante. Dilui-se extractos de células para uma concentração de proteína de 10 mg / ml com tampão de lise 1x.
3. Ensaio enzimático em 200 ul por tubo
NOTA: Siga os seguintes passos em uma capa química.
- Teste cada amostra em duplicado, a 15 mltubo cónico. Prepare 20 mL 1 M TrisHCl pH 7,5 por tubo e mantê-lo em gelo. Preparar 2 ml de clorofórmio / metanol (1: 1 (v / v); Solução de paragem) à temperatura ambiente para cada um dos tubos.
- Pipetar 20 ul de Tris-HCl 1 M pH 7,5 em cada tubo de 15 ml em gelo.
- Siga as orientações de segurança de radiação a partir daqui. Adicionar o equivalente a 0,06 uM (0,2 uCi) S - [metil-3H] adenosil-L-metionina e 50 uM frio SAM por tubo para um total de 50,06 uM de SAM. Adicionar x ul de água fria, onde x = 200- (20 (para buffer) + 20 (para extratos de células) + volume de frio e radioativo SAM) por tubo.
- Transferir cada tubo cónico para um banho de água a 30 ° C pré-aquecido. Adicionar a cada tubo 20 ul de extractos de células (equivalente a 200 ug de proteína) para iniciar a reacção. Incubar durante o tempo desejado (0 a 45 min).
- Parar a reacção por adição de 2 ml de clorofórmio / metanol (1: 1; v / v; Solução de paragem) para cada tubo. Transferir o tubo cónico para a TA (20-256; C).
4. Lipid Extração
NOTA: Siga os seguintes passos em uma capa química.
- Adicionar 700 uL de água a cada tubo contendo a amostra de reacção enzimática. Vortex vigorosamente por 30 segundos. Centrifugar a 1.500 xg durante 5 min à temperatura ambiente para separar a fase orgânica da fase aquosa.
- Transferir a fase orgânica inferior para um novo tubo cónico de 15 ml com uma pipeta. Adicionar 1 ml de água para cada "fase inferior" contendo tubo e agitar vigorosamente em vórtice durante 30 seg. Centrifugar novamente a 1500 xg durante 5 minutos para separar a fase orgânica da fase aquosa.
- Transferir a fase orgânica inferior para um tubo de cintilação com uma pipeta. As amostras secas sob uma corrente de N2. Elimine as fases de água contendo o SAM radioativo não-incorporados e os tubos cônicos radioativos conforme as diretrizes de radiação.
- Adicione 2 ml / tubo de líquido de cintilação. Medir a radioactividade incorporada com um par de cintilaçãonter acordo com o protocolo dos fabricantes e utilização do instrumento.
- Calcula-se a actividade enzimática em nmol / mg de proteína usando a seguinte equação geral:
valor cpm x 10 x 3 [total (radioativo e frio) concentração de SAM (mM)] x 5 atividade específica de SAM radioativo (Ci / mmol) x [concentração de substâncias radioativas SAM (mM)]
NOTA: o protocolo acima pode ser alterada para uma concentração SAM dependente ou um ensaio PEMT dependente proteína. Para a concentração de SAM ensaio enzimático dependente, o tempo é mantido constante (15 minutos, o que está na gama linear) e várias quantidades de frio SAM são adicionados ao ensaio enquanto que para o ensaio PEMT dependente de proteína, concentração de SAM (escolheu 0,05 mM) e a hora são mantida constante (15 minutos). Além disso, o pH do tampão pode ser alterado conforme necessário, se algumas enzimas PEMT tem um valor de pH óptimo diferente.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
A Figura 1 mostra um ensaio PEMT dependente do tempo, o qual foi realizado com Leishmania extracto de célula completa como uma fonte de enzima utilizando PE endógeno como um substrato. A quantidade de radioactividade na fase orgânica foi quantificada por contagem de cintilação. Os números resultantes foram utilizados para calcular a quantidade de grupos metil tritiados transferidos para PE. A actividade PEMT foi linear durante, aproximadamente, 20 min. Em seguida, ele atingiu um patamar a cerca de 30 minutos, após o que se manteve constante durante mais 15 min. Como esperado, a actividade não foi detectada PEMT quando não há extractos de células foram adicionados ao ensaio (Figura 2). Além disso, esta actividade foi abolida na presença de brometo de octadecyltrimethylammonium 100 uM, o que é um inibidor de L. principais methyltransferases PE LMJ LMJ PEM1 e PEM2 14. Atividade PEMT foi também proteína concentração dependenT, e esta actividade era linearmente proporcional à quantidade de proteína aplicado para o ensaio enzimático (Figura 3). Por último, um ensaio PEMT dependente da concentração de SAM foi realizada, em que as concentrações cada vez maiores de SAM foram testados (Figura 4). PEMT atividade atingiu um patamar no SAM concentração de cerca de 0,5 mM. Todos em conjunto, estes quatro ensaios demonstram que a actividade PEMT é específico e pode ser medida a partir de extractos de células inteiras sem a necessidade de purificar a enzima (s).
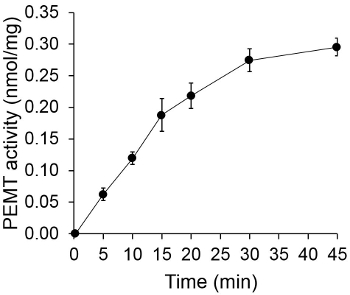
Figura 1. Tempo de ensaio PEMT dependente. O ensaio enzimático foi realizada duas vezes em duplicado com 0,2 mg de extractos de células inteiras de Leishmania como uma função do tempo. Actividade PEMT é representado como grupos metilo nmol transferidos para PE por mg de proteína e Phr er. Por tempo de "O", os extractos celulares foram primeiro misturadas com 2 ml de solução de paragem feito de clorofórmio / metanol (1: 1, em volume) antes de ser adicionada à solução de ensaio contendo SAM. Desvios-padrão são mostrados.
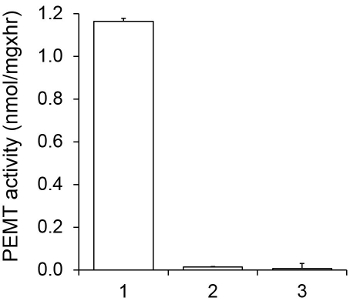
Figura 2. Especificidade da actividade PEMT. PEMT ensaio foi realizado duas vezes, em duplicado, durante 15 min na presença de SAM de 0,05 mM. Extracto de proteína de 1, 0,2 mg; 2, sem extracto celular; 3, 0,2 mg de proteína e 0,1 mM de brometo de octadecyltrimethylammonium. Desvios-padrão são mostrados.
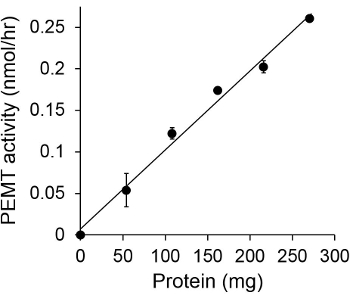
Figura 3. A proteína ensaio PEMT dependente. O ensaio enzimático foi realizada duas vezes emduplicar na ausência (ponto 'O') ou na presença de várias quantidades de proteínas de Leishmania com SAM 0,05 mM para 15 min. Desvios-padrão são mostrados.
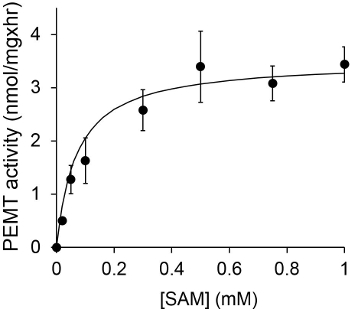
Figura 4. O ensaio PEMT foi realizada duas vezes em duplicado com 0,2 mg de extractos de células inteiras de Leishmania na presença de várias concentrações de SAM, durante 15 min. Os desvios padrão são mostrados.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Este ensaio PEMT simples, rápida permite a quantificação das formas metiladas de PE que resulta da transferência de grupos metilo radioactivas de SAM na PE utilizando o extracto de célula completa como uma fonte de proteína. É rápido, sensível, reprodutível e também adequado para enzimas purificadas 17. Monometil ou dimetil-PE pode ser adicionado ao ensaio se a metiltransferase de interesse é específico para estes substratos em vez de PE 12,13,18,19. Se PEMT enzima purificado é usado, PE pode ser adicionado ao ensaio. Uma limitação deste protocolo é que o ensaio não identifica os produtos Reações (monometil-PE, dimetil-PE, ou PC). No entanto, a identidade dos produtos de reacção (monometil-PE, dimetil-PE, PC) pode ainda ser analisada por uma cromatografia em camada fina como descrito dimensional em 20,21. Além disso, alguns dos produtos de degradação, tais como S SAM -adenosylhomocysteine e 5'-metiltioadenosina pode inibir a actividade de b PEMTy inibição feedback. No entanto, a Leishmania possui uma hidrolase S -adenosylhomocysteine 22, que cliva S -adenosylhomocysteine em adenina e S -ribosylhomocysteine, e um metiltioadenosina-fosforilase, que produz adenina e methylthioribose-1-fosfato-23. No entanto, não se sabe se hidrolase S -adenosylhomocysteine e actividades fosforilase metiltioadenosina são suficientemente altos para metabolizar eficientemente S -adenosylhomocysteine e metiltioadenosina, respectivamente, de modo que não a inibição da actividade PEMT ocorre. No exemplo hidrolase de S -adenosylhomocysteine e / ou 5'-metiltioadenosina enzima metabólica estão ausentes na célula de interesse, além das respectivas enzimas recombinantes purificadas, pode ser adicionado ao ensaio para aliviar a inibição do feedback por sam produtos de degradação 24,25, 26.
Há quatro passos críticos neste protocolo: i)o cocktail inibidor de protease em pó tem de ser adicionado ao tampão de lise imediatamente antes da utilização (passo 1.1); ii) toda a célula extractos são para ser utilizada dentro das seguintes horas após a preparação (após o passo 1.4); iii) durante os passos de extracção de lípidos (os passos 4.2 e 4.4), cuidado deve ser aplicado para não transferir qualquer da fase interfase ou água, que contém o excesso de tritiada SAM, e iv) a alíquota a S radioactivos - [metil- 3 H] adenosil-L-metionina e reagente fria durante o recebimento de ciclos repetidos de congelamento e descongelamento, o degradam em 5'-metiltioadenosina e homo-serina lactona, seguido por hidrólise para adenina e S -pentosylmethionine 26,27,28, o que pode explicar a falta de atividade PEMT mensurável. Extratos de células inteiras ruins também pode ser responsável por nenhuma atividade enzimática. Neste caso, a qualidade do extracto celular pode ser avaliada medindo a outra actividade enzimática conhecida. Por último, a falta de atividade PEMT poderá contar com a PEMT enZyme ser específico para monometil-PE ou dimetil-PE em vez de PE. A adição de monometil-PE e / ou substratos dimetil-PE para o ensaio pode restaurar a actividade PEMT.
Uma alternativa, não radioactivo metiltransferase dependente de SAM ensaio foi desenvolvido, que se baseia na quantificação enzimática de homocisteína, os produtos de transmetilação de SAM 29. Este protocolo foi aplicado com sucesso para medir PE atividade metiltransferase específica 16. Infelizmente, este ensaio só é adequado para a enzima purificada e não de extracto celular total, e não revela a identidade dos produtos da reacção ou.
O presente protocolo podem, eventualmente, ser aplicada a qualquer tipo de célula. Além disso, este ensaio pode ser usado para testar potenciais drogas específicas para um PEMT de interesse no contexto do teste de novos compostos anti-microbianos para combater infecções bacterianas ou terapêuticas contra a aterosclerose induzida por dieta, obesidadeE resistência à insulina.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Declaro não haver conflitos de interesse declarados
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



