성장 곡선: 군집 형성 단위 및 광학 밀도 측정을 사용하여 성장 곡선 생성
English
Share
Overview
출처: 앤드류 J. 반 알스트1,리안논 M. 레베크1,나탈리아 마틴1,빅터 J. 디리타1
1 미생물학 및 분자 유전학학과, 미시간 주립 대학, 이스트 랜싱, 미시간, 미국
성장 곡선은 세균 성장 운동학 및 세포 생리학에 대한 귀중한 정보를 제공합니다. 그들은 우리가 박테리아가 변수 성장 조건에서 반응 하는 방법을 결정 하 고 주어진 된 박테리아에 대 한 최적의 성장 매개 변수를 정의 할 수 있습니다. 고풍스러운 성장 곡선은 지연, 지수, 고정 및 죽음 (1)의 네 단계를 통해 진행됩니다.
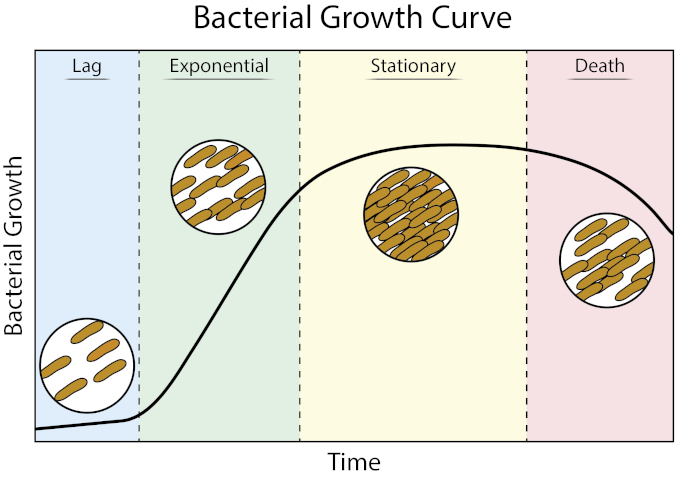
그림 1: 세균 성장 곡선. 배치 배양에서 자란 박테리아는 성장의 4 단계를 통해 진행: 지연, 지수, 고정, 그리고 죽음. 지연 단계는 박테리아가 급속한 세포 성장 및 분열을 할 수있는 생리적 상태에 도달하는 데 걸리는 기간입니다. 지수 단계는 DNA 복제, RNA 전사 및 단백질 생산이 모두 일정하고 빠른 속도로 발생하는 가장 빠른 세포 성장 및 분열의 단계입니다. 고정 된 단계 영양소 제한 및/또는 독성 중간 축적으로 인해 세균 성장의 둔화 및 고원을 특징으로합니다. 죽음의 단계는 세포 리시스가 가혹한 양분 제한결과로 생기는 단계입니다.
지연 단계는 박테리아가 급속한 세포 성장 및 분열을 할 수있는 생리적 상태에 도달하는 데 걸리는 기간입니다. 이 지연은 박테리아가 새로운 환경에 적응하는 데 시간이 걸리기 때문에 발생합니다. 필요한 세포 성분이 지연 단계에서 생성되면 박테리아는 DNA 복제, RNA 전사 및 단백질 생산이 모두 일정하고 빠른 속도(2)로 발생하는 기하급수적 성장 단계에 진입합니다. 기하급수상 동안 급속세포 성장 및 분열의 비율은 생성 시간 또는 두 배로 계산되며, 주어진 조건(1)에서 박테리아가 복제할 수 있는 가장 빠른 속도이다. 두 배로 하는 시간은 세균성 성장에 더 유리한 결정하기 위하여 다른 성장 조건을 비교하기 위하여 이용될 수 있습니다. 기하급수학적 성장 단계는 세균세포 생리학이 전체 인구(3)에 걸쳐 일관되기 때문에 가장 재현가능한 성장 상태입니다. 고정 단계는 세포 성장 고원이 있는 기하급수적 단계를 따릅니다. 고정 된 단계는 영양 고갈 및/또는 독성 중간체의 축적으로 인해 가져온다. 세균세포는 복제 및 세포 분열의 비율이 급격히 감소하더라도, 이 단계에서 살아남기 위하여 계속합니다. 마지막 단계는 심각한 영양소 고갈이 세포의 lysing에 이르게 죽음입니다. 가장 많은 정보를 제공하는 성장 곡선의 특징은 지연 단계의 지속 시간, 두 배의 시간 및 도달 한 최대 셀 밀도를 포함한다.
배치 배양에서 박테리아의 정량화는 식민지 형성 단위 와 광학 밀도 측정을 모두 사용하여 결정될 수 있다. 콜로니 성형 유닛(CFU)에 의한 열거는 세균세포 수의 직접적인 측정을 제공한다. CFU에 대한 측정의 표준 단위는 직렬 희석 및 확산 도금 기술에 의해 결정된 배양 (CFU/mL)의 1 mL 당 존재하는 컬터성 박테리아의 수입니다. 각 타임포인트에 대해, 배치 배양의 1:10 희석 계열이 수행되고 각 희석의 100 μl이 셀 스프레더를 사용하여 도금된다.
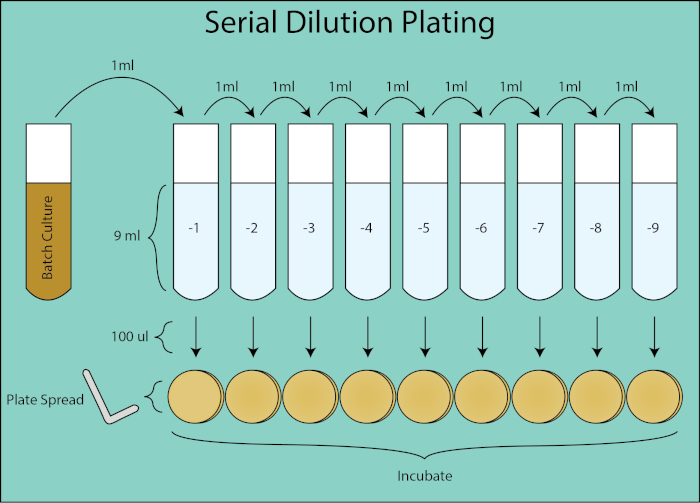
그림 2. 직렬 희석 도금 회로도. 일괄 처리 문화에서 희석 도금에 대한 일반적인 흐름. 배치 배양은 9ml PBS를 포함하는 후속 튜브로 이전 희석의 1mL을 전송하여 1:10으로 연이어 희석된다. 각 희석 관에서, 100 μl은 CFU/mL을 계산할 때 1mL 부피의1/10제부피이기 때문에 1:10의 추가 희석인 플레이트 스프레더를 사용하여 도금된다. 판은 복제 콜로니가 접시에 자면 배양되고 예매됩니다.
플레이트는 하룻밤 동안 배양되고 복제 식민지가 매립됩니다. 30-300콜로니를 성장시킨 희석 플레이트는 주어진 시점(4, 5)에 대한 CFU/mL을 계산하는 데 사용된다. 30 세 미만의 식민지 수의 스토차스 변화는 CFU / mL의 계산에 더 큰 오차가 적용되며 식민지 혼잡및 겹침으로 인해 300 개 이상의 식민지를 세는 것은 과소 평가 될 수 있습니다. 주어진 플레이트에 대한 희석 계수를 사용하여 배치 배양의 CFU는 각 타임포인트에 대해 계산할 수 있습니다.
광학 밀도는 분광광계를 사용하여 측정된 세균 세포 수의 즉각적인 근사치를 제공합니다. 광학 밀도는 1cm의 배양을 통과하고 포토다이오드 센서(6)에 의해 검출되는 광 입자의 흡수를 측정한 값입니다. 배양물의 광학 밀도는 미디어 블랭크와 관련하여 측정되고 세균밀도가 증가함에 따라 증가합니다. 세균 세포의 경우, 600 nm (OD600)의 파장이 일반적으로 광학 밀도 (4)를 측정 할 때 사용됩니다. 콜로니 성형 유닛 및 광학 밀도와 관련된 표준 곡선을 생성함으로써, 광학 밀도 측정은 배치 배양의 세균 세포 수를 쉽게 근사화하는 데 사용될 수 있다. 그러나, 이 관계는 세포가 모양을 바꾸고 CFU (7)와 관련되는 광학 밀도 판독에 영향을 미치는 매체에 세포 외 제품을 축적하기 시작으로 0.3 OD600초기에 악화되기 시작합니다. 이 오류는 고정 및 사망 단계에서 더 두드러집니다.
여기서, 대장균은 30시간(7)의 과정을 통해 루리아-베르타니(LB) 국물에서 37°C에서 재배됩니다. CFU/mL 및 광학 밀도 성장 곡선은 CFU에 대한 광학 밀도와 관련된 표준 곡선뿐만 아니라 생성되었습니다.
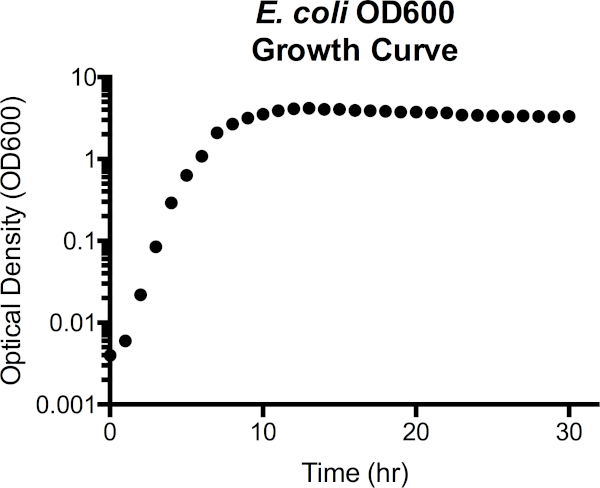
그림 3. 에체리치아 대장균 광학 밀도 600 nm 파장 (OD600) 성장 곡선. 광학 밀도 값은 멸균 LB 매체로 블랭킹한 후 분광계에서 직접 채취하였다. 1.0보다 큰 OD600 값은 100 μl 배양을 900 μl 의 신선한 LB와 결합하여 1:10을 희석하고, 다시 측정한 다음 10을 곱하여 OD600 값을 얻었다. 이 단계는 분광계의 측정정확도가 높은 세포 밀도에서 감소함에 따라 취해질 것이다. 곡선에서 지연 단계는 약 1h의 성장으로 확장되어 2h에서 7h로 기하급수적 단계로 전환한 다음 고원으로 전환하여 고정 단계에 진입합니다. 그러나 15시간 이후에 광학 밀도가 점차 감소하기 시작하면서 사망 단계는 뚜렷한 전환이 아닙니다.

그림 4. 밀리리터당 대장균 콜로니 형성 유닛(CFU/mL) 성장 곡선. 각 시점에 대한 CFU/mL 값은 30-300개의 콜로니를 포함하는 희석 플레이트로부터 계산되었다. 곡선에서 지연 단계는 약 2시간까지 확장되어 2h에서 7h로 기하급수적인 단계로 전환한 다음 고원으로 전환하여 고정 된 단계로 들어갑니다. 그러나 CFU/mL이 30시간 동안 2 x 109의 피크에서 약 5 x 108로 15시간 후에 점차 감소하기 시작하면서 사망 단계는 뚜렷한 전환이 아닙니다.
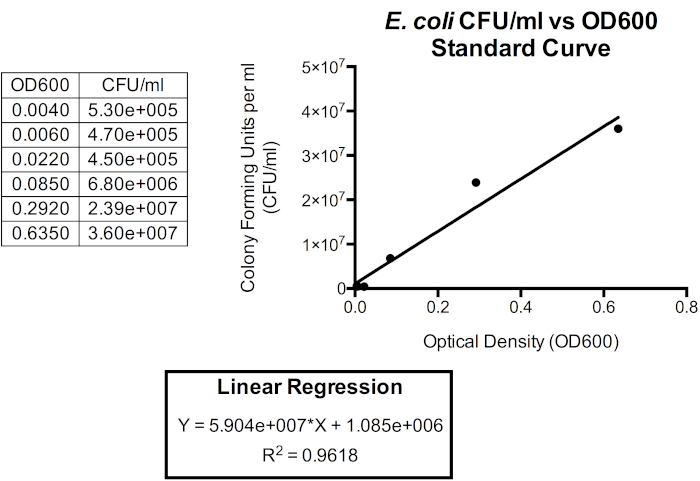
그림 5. CFU/mL 대 OD600의 표준화 곡선. 선형 회귀는 광학 밀도가 세균 세포 밀도를 근사화하는 데 사용될 수 있도록 이러한 단위를 관련시키는 데 사용될 수 있다. 광학 밀도는 배치 배양물의 CFU/mL의 즉각적인 근사치를 제공하고 즉시 근사치하는 데 사용될 수 있다. 여기서, OD600과 CFU/mL 사이의 관계가 1.0 OD600을 넘어서는 것이 덜 정확하기 때문에 처음 6개의 타임포인트만이 1.0 OD600에 도달한 직후 박테리아가 고정된 단계에 진입함에 따라 세포 모양과 세포외 제품이 축적되기 시작하면서 플롯된다. 매체의 세포 모양 및 세포외 제품의 변화는 광학 밀도 판독에 영향을 미치므로 광학 밀도와 배양에 있는 박테리아의 수 사이의 관계도 영향을 미칩니다.
두 배의 시간도 15분 19초로 결정되었습니다. 이 데이터에서, 대장균에 대한 LB의 성장 능력은 시각화되고 다른 미디어 또는 박테리아 사이의 비교에 사용될 수 있다.
Procedure
Results
Plots of colony forming units and optical density are two ways to visualize growth kinetics. By determining the relationship between CFU/mL and OD600, the optical density plot also provides an estimate of CFU/mL over time. Conditions that result in the shortest doubling time are considered optimal for growth of the given bacteria.
Applications and Summary
Growth curves are valuable for understanding the growth kinetics and physiology of bacteria. They allow us to determine how bacteria respond in variable growth conditions as well as define the optimal growth parameters for a given bacterium. Colony forming unit and optical density plots both contain valuable information depicting the duration of lag phase, maximum cell density reached, and allowing for the calculation of bacterial doubling time. Growth curves also allow for comparison between different bacteria under the same growth conditions. Additionally, optical density provides a means of standardizing initial inoculums, enhancing consistency in other experiments.
Determining which approach to use when designing a growth curve experiment requires consideration. As the preferred method for generating growth curves, colony forming unit plots more accurately reflect the viable cell counts in batch culture. Colony forming unit plots also allow for measuring bacterial growth in conditions that would otherwise interfere with an optical density measurement. However, it is a more time consuming process, requiring extensive use of reagents, and must be performed manually. Optical density plots are less accurate and provide only an estimate of the colony forming units, requiring a standard curve to be generated for each unique bacteria. Optical density is primarily used for its convenience as it is far less time consuming and does not require many reagents to perform. What is most attractive to optical density, is that spectrophotometric incubators can automatically generate growth curves, vastly increasing the number of culture conditions that can be tested at once and eliminating the need to constantly attend the culture.
References
- R. E. Buchanan. 1918. Life Phases in a Bacterial Culture. J Infect Dis 23:109-125.
- CAMPBELL A. 1957. Synchronization of cell division. Bacteriol Rev 21:263-72.
- Wang P, Robert L, Pelletier J, Dang WL, Taddei F, Wright A, Jun S. 2010. Robust growth of Escherichia coli. Curr Biol 20:1099-103.
- Goldman E, Green LH. 2015. Practical Handbook of Microbiology, Third Edition. CRC Press.
- Ben-David A, Davidson CE. 2014. Estimation method for serial dilution experiments. J Microbiol Methods 107:214-221.
- Koch AL. 1968. Theory of the angular dependence of light scattered by bacteria and similar-sized biological objects. J Theor Biol 18:133-156.
- Sezonov G, Joseleau-Petit D, D'Ari R. 2007. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol 189:8746-9.
Transcript
Bacteria reproduce through a process called cell division, which results in two identical daughter cells. If the growth conditions are favorable, bacterial populations will grow exponentially.
Bacterial growth curves plot the amount of bacteria in a culture as a function of time. A typical growth curve progresses through four stages: lag phase, exponential phase, stationary phase, and death phase. The lag phase is the time it takes for bacteria to reach a state where they can grow and divide quickly. After this, the bacteria transition to the exponential phase, characterized by rapid cell growth and division. The rate of exponential growth of the bacterial culture during this phase can be expressed as the doubling time, the fastest rate at which bacteria can reproduce under specific conditions. The stationary phase comes next, where bacterial cell growth plateaus and the growth and death rates even out due to environmental nutrient depletion. Finally, the bacteria enter the death phase. This is where bacterial growth declines sharply and severe nutrient depletion leads to the lysing of cells.
Two techniques can be used to quantify the amount of bacteria present in a culture and plot a growth curve. The first of these is via colony forming units, or CFUs. To obtain CFUs a one to ten series of nine dilutions is performed at regular time points. The first of these dilutions, negative one in this example, contains 9mL of PBS and 1mL of the bacterial culture. Resulting in a 1:10 dilution factor. Then, 1mL of this solution is transferred to the next tube, negative two, resulting in a 1:100 dilution factor. This process continues through the last tube, negative nine, resulting in a final dilution factor of 1:1 billion. After this, 100 microliters of each dilution is plated. The plates are then incubated and the clonal colonies are counted. The dilution plate for a given time point that grows between 30 and 300 colonies is used to calculate the CFUs per milliliter for that time point.
The second common method of measuring bacterial concentration is the optical density. The optical density of a culture can be measured instantly, in relation a media blank, with a spectrophotometer. Typically a wave length of 600 nanometers, also referred to as OD600, is used for these measurements, which increase as cell density increases. While optical density is less precise than CFUs, it is convenient because it can be obtained instantaneously and requires relatively few reagents. Both techniques can be used together to create a standard curve that more accurately approximates the bacterial cell count of a culture. In this video, you will learn how to obtain CFUs and OD600 measurements from timed serial dilutions of E. coli. Then, two growth curves using the CFU and OD600 measurements, respectively, will be plotted before being related by a standard curve.
When working with bacteria, it is important to use the appropriate personal protective equipment such as a lab coat and gloves and to observe proper aseptic technique.
After this, sterilize the work station with 70% ethanol. First, prepare the LB broth and LB solid agar media in separate autoclaveable bottles. After partially closing the caps of the bottles, sterilize the media in an autoclave set to 121 degrees Celsius for 35 minutes. Next, allow the agar media to cool in a water bath set to 50 degrees Celsius for 30 minutes. Once cooled, pour 20 to 25 mL into each Petri dish. After this, allow the plates to set for 24 hours at room temperature.
To prepare the single colony isolates that will later be used to produce a liquid bacterial culture, use previously frozen stock and proper streak plating technique to streak E. coli for isolation on LB agar. Incubate the dish at 37 degree Celsius overnight. After this, cool a flame sterilized inoculation loop on the agar before selecting a single colony from the streaked plate. Inoculate 4 mL of liquid media in a 15 mL test tube. Then, grow the E. coli at 37 degrees Celsius overnight with shaking at 210 rpm.
To set up the 1:1000 volume of bacterial culture that will be used in the growth curve, first obtain an autoclaved 500 mL Erlenmeyer flask. Then, use a 50 mL serological pipette to transfer 100 mL of sterile media to the flask. Next, label nine 15 ml test tubes consecutively as one through nine. These numbers correspond to the dilution factor that will be used to calculate the colony forming unit, or CFU. Then, add 9 mL of 1X PBS to each tube. After this, label the prepared agar plates with the corresponding time points and dilution factors that will be grown. In this example with E. coli, after the starting time point, time points are taken once every hour. Using the previously prepared overnight liquid E. coli culture, inoculate the media in the autoclave 500 mL Erlenmeyer flask with 1:1000 volume of culture. Swirl the media to evenly distribute the bacteria.
After blanking a spectrophotometer, clean the cuvette with a lint-free wipe. Next, dispense 1 mL of the culture into the cuvette and place it into the spectrophotometer to obtain the optical density of the culture at time point zero. Then, grow the E. coli at 37 degrees Celsius with shaking at 210 rpm. At each time point after time point zero, withdraw another 1 mL of bacterial culture from the flask and repeat the optical density measurement. If the optical density reading is greater than 1.0, dilute 100 microliters of bacterial culture with 900 microliters of fresh media and then measure the optical density once more. This value can be multiplied by 10 for the OD 600 measurement.
To obtain the colony forming unit measurement for each time point, withdraw an additional 1 mL of bacterial culture from the flask at each time point. Dispense the bacterial culture into the negative one test tube and vortex to mix. Then, perform the dilution series by first transferring 1 mL from the negative one tube into the negative two tube and vortex to mix. Transfer 1 mL from the negative two tube into the negative three tube and vortex to mix. Continue this serial transfer down all the dilution tubes to the negative nine tube. Dispense 100 microliters of cell suspension onto the correspondingly labeled plate for each dilution. For every dilution, sterilize a cell spreader in ethanol, pass it through a Bunsen burner flame, and cool it by touching the surface of the agar away from the inoculate. Then, use the cell spreader to spread the cell suspension until the surface of the agar plate becomes dry. Incubate the plates upside down at 37 degrees Celsius. Once visible colonies arise, count the number of bacterial colonies on each plate. Record these values and their associated dilution factors for each plate at each time point.
To create an OD 600 growth curve, after ensuring all the data points are entered correctly into a table, select all of the time points and their corresponding data. To generate a colony forming unit growth curve plot, choose the dilution plate where the colony counts fell within the range 30 to 300 bacteria for each time point. Multiply the colony count number by the dilution factor, and then by ten. This is because the 100 microliters spread is considered an additional 1:10 dilution when calculating colony forming units per milliliter. After this, plot the colony forming units versus time on a semi-log scale.
These plots produced with OD 600 and CFU measurements, respectively, can provide valuable information on E. coli growth kinetics. The optical density and colony forming units can be related, so that CFUs per milliliter can be estimated from OD 600 measurements, saving time and materials in future experiments.
To do this, plot the colony forming units against the optical density on a linear scale for OD 600 readings less than or equal to 1. 0. After this, generate a linear regression trend line in Y = MX + B format, where M is the slope and B is the y-intercept. Right click on the data points and select add trend line and linear. Then, check the box to display the equation on the chart and display the R squared value on the chart. The R squared value is the statistical measurement of how closely the data matched the fitted regression line. In this example, the first 6 time points are plotted with OD 600 on the x axis and CFUs per milliliter on the y axis. In future experiments with the same growth conditions, these slope and y-intercept values can be plugged into this equation to estimate CFUs from OD 600 readings. Next, look at the colony forming unit growth curve plot. During the exponential phase, identify two time points with the steepest slope between them. To calculate the doubling time, first calculate the change in time between the selected time points. Then, calculate the change in generations using the equation shown here. Here, lower case b is the number of bacteria at time point three and upper case B is the number of bacteria at time point two. Finally, divide the change in time by the change in generations. In this example, the doubling time is 0. 26 hours or 15 minutes and 19 seconds. Comparing doubling times across different experimental treatment allows us to identify the best growth conditions for a certain bacterial species. Therefore, the treatment with the lowest doubling time will be most optimal of the conditions tested.
