La trasduzione batterica tramite fagi: un metodo per trasferire la resistenza all'ampicillina da una cellula donatore di E. coli ad una ricevente
English
Share
Overview
Fonte: Alexander S. Gold1, Tonya M. Colpitts1
1 Dipartimento di Microbiologia, Boston University School of Medicine, National Emerging Infections Diseases Laboratories, Boston, MA
La trasduzione è una forma di scambio genetico tra batteri che utilizza batteriofagi, o fagi, una classe di virus che infetta esclusivamente organismi procariotici. Questa forma di trasferimento del DNA, da un batterio all’altro attraverso un fago, fu scoperta nel 1951 da Norton Zinder e Joshua Ledererg, che definirono il processo “trasduzione” (1). I batteriofagi furono scoperti per la prima volta nel 1915 dal batteriologo britannico Frederick Twort, poi scoperti indipendentemente nel 1917 dal microbiologo franco-canadese Felix d’Herelle (2). Da allora, la struttura e la funzione di questi fagi sono state ampiamente caratterizzate (3), dividendo questi fagi in due classi. La prima di queste classi sono i fagi litici che al momento dell’infezione si moltiplicano all’interno del batterio ospite, interrompendo il metabolismo batterico, lisi della cellula e rilasciando fagi di progenie (4). Come risultato di questa attività antibatterica e della crescente prevalenza di batteri resistenti agli antibiotici, questi fagi litici si sono recentemente dimostrati utili come trattamento sostitutivo degli antibiotici. La seconda di queste classi sono i fagi lisogenici che possono moltiplicarsi all’interno dell’ospite attraverso il ciclo litico o entrare in uno stato quiescente in cui il loro genoma è integrato in quello dell’ospite (Figura 1), un processo noto come lisogenesi, con la capacità di indotta dalla produzione di fagi in più generazioni successive (4).
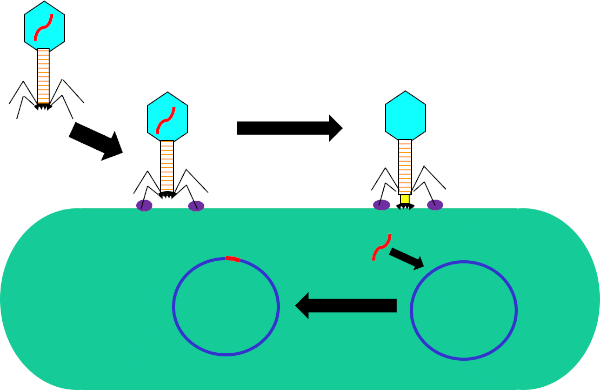
Figura 1: Infezione della cellula ospite da parte del batteriofago. Adsorbimento da parte del fago alla parete cellulare batterica attraverso interazioni tra le fibre della coda e il recettore (viola). Una volta sulla superficie cellulare, il fago viene irreversibilmente attaccato alla cellula batterica usando la piastra di base (nera) che viene spostata sulla parete cellulare dalla guaine contrattile (gialla). Il genoma dei fagi (rosso) entra quindi nella cellula e si integra nel genoma della cellula ospite.
Mentre la trasduzione batterica è un processo naturale, utilizzando la tecnologia moderna è stata manipolata per il trasferimento di geni nei batteri in laboratorio. Inserendo geni di interesse nel genoma di un fago lisogenico, come il fago, si è in grado di trasferire questi geni nei genomi dei batteri e di conseguenza esprimerli all’interno di queste cellule. Mentre altri metodi di trasferimento genico, come la trasformazione, utilizzano un plasmide per il trasferimento e l’espressione genica, l’inserimento del genoma del fago in quello del batterio ricevente non solo ha il potenziale per conferire nuovi tratti a questo batterio, ma consente anche mutazioni naturali e altri fattori dell’ambiente cellulare per alterare la funzione del gene trasferito.
Rispetto ad altri metodi di trasferimento genico orizzontale, come la coniugazione, la trasduzione è abbastanza flessibile nei criteri richiesti per le cellule donatrici e riceventi. Qualsiasi elemento genetico che può adattarsi all’interno del genoma del fago utilizzato può essere trasferito da qualsiasi ceppo di batteri donatori a qualsiasi ceppo di batteri riceventi purché entrambi siano permissivi al fago, richiedendo l’espressione dei necessari recettori fagici sulle superfici cellulari. Una volta che questo gene viene spostato fuori dal genoma del donatore e confezionato nel fago, può essere trasferito al ricevente. Dopo la trasduzione, è necessario selezionare per i batteri riceventi che contengono il gene di interesse devono essere selezionati per. Questo potrebbe essere fatto utilizzando un marcatore genetico, come un FLAG-tag o un polyhistidine-tag, per contrassegnare il gene di interesse, o la funzione intrinseca del gene, nel caso di geni che codificano per la resistenza agli antibiotici. Inoltre, la PCR potrebbe essere utilizzata per confermare ulteriormente il successo della trasduzione. Utilizzando primer per una regione all’interno del gene di interesse e confrontando il segnale con un controllo positivo, batteri che hanno il gene di interesse e un controllo negativo, batteri che hanno subito gli stessi passaggi della reazione di trasduzione senza fagi. Mentre la trasduzione batterica è uno strumento utile in biologia molecolare, ha e continua a svolgere un ruolo importante nell’evoluzione dei batteri, in particolare per quanto riguarda il recente aumento della resistenza agli antibiotici.
In questo esperimento, la trasduzione batterica è stata utilizzata per trasferire il gene che codifica per la resistenza all’ampicillina antibiotica dal ceppo W3110 di E.coli al ceppo J53 attraverso il batteriofago P1 (5). Questo esperimento consisteva in due fasi principali. In primo luogo, la preparazione del fago P1 contenente il gene di resistenza all’ampicillina dal ceppo donatore. In secondo luogo, il trasferimento di questo gene al ceppo ricevente per trasduzione con il fago P1 (Figura 1). Una volta effettuato, il successo del trasferimento del gene di resistenza all’ampicillina potrebbe essere determinato dalla qPCR (Figura 2). Se la trasduzione ha successo, il ceppo J53 di E. coli sarebbe resistente all’ampicillina e il gene che conferisce questa resistenza rilevabile dalla qPCR. In caso di insuccesta, non ci sarebbe alcun rilevamento del gene di resistenza all’ampicillina e l’ampicillina funzionerebbe ancora come un antibiotico efficace contro il ceppo J53.
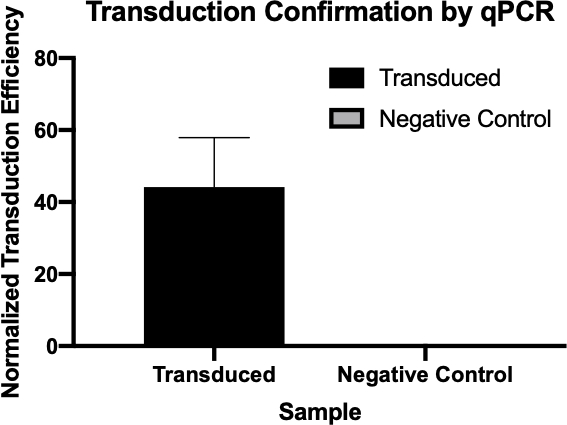
Figura 2: La conferma del successo della trasduzione da parte della qPCR. Confrontando i valori di Cq rilevati per il gene di interesse dalla reazione di trasduzione e dalla reazione di controllo negativa e normalizzando questi valori con un gene di pulizia, si è stati in grado di confermare che la trasduzione batterica ha avuto successo.
Procedure
Applications and Summary
The transfer of genes to and from bacteria by bacteriophage, while a natural process, has proved extremely useful for a multitude of research purposes. While other methods of gene transfer such as transformation and conjugation are possible, transduction uniquely uses bacteriophages; not only allowing for gene integration into the host genome, but also for gene delivery to multiple bacteria that are not susceptible to other methods. This process, while especially useful in the laboratory, has also been used in the recently emerging field of gene therapy, more specifically in alternative gene therapy, a therapeutic strategy that utilizes bacteria to deliver therapeutics to target tissues, many of which are not susceptible to other delivery methods and have much clinical relevance (8,9).
References
- Lederberg J, Lederberg E.M., Zinder, N.D., et al. Recombination analysis of bacterial heredity. Cold Spring Harbor symposia Quantitative Biol. 1951;16:413-43.
- Duckworth DH. "Who Discovered Bacteriophage?". Bacteriology Reviews. 1976;40:793-802.
- Yap ML, Rossman, M.G. Structure and Function of Bacteriophage T4. Future Microbiol. 2014;9:1319-27.
- Sulakvelidze A, Alavidze, Z., Morris, J. G. Bacteriophage Therapy Antimicrobial Agents and Chemotherapy 2001;45(3):649-59.
- Moore S. Sauer:P1vir phage transduction 2010 [Available from: https://openwetware.org/wiki/Sauer:P1vir_phage_transduction].
- Kobayashi A, et al. Growth Phase-Dependent Expression of Drug Exporters in
- Escherichia coli and Its Contribution to Drug Tolerance. Journal of Bacteriology. 2006;188(16):5693-703.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.
- Pálffy R. et al. Bacteria in gene therapy: bactofection versus alternative gene therapy. Gene Ther. 2006 13:101-5.
- O'Neill JM, et al. Intestinal delivery of non-viral gene therapeutics: physiological barriers and preclinical models. Drug Discovery Today. 2011;16:203-2018.
Transcript
Bacteria can adapt quickly to a fast-changing environment by exchanging genetic material and one way they can do this is via transduction, the exchange of genetic material mediated by bacterial viruses. A bacteriophage, often abbreviated to phage, is a type of virus that infects bacteria by first attaching to the surface of the host and then injecting its DNA into the bacterial cell. It then degrades the host cell’s own DNA and replicates its viral genome, whilst hijacking the cell’s machinery to synthesize many copies of its proteins. These phage proteins then self-assemble and package the phage genomes to form multiple progeny. However, due to the low fidelity of the DNA packaging mechanism, occasionally, the phage packages fragments of bacterial DNA into the phage capsid. After inducing the lysis of the host, the phage progeny are released and, once such a phage infects another host cell, it transfers the DNA fragment of its previous host. This can then recombine and become permanently incorporated into the new host’s chromosome, thereby mediating gene transfer between the two bacteria.
To carry out phage transduction in the laboratory requires a donor strain that contains a gene of interest, a recipient strain that lacks it, a phage that can infect both the strains, and a method to select the transduced bacteria. In most cases, this will be a selective solid growth media that supports the growth of transduced bacteria but inhibits the growth of non-transduced ones. To begin, the donor strain that contains the gene of interest is cultured in a liquid growth medium. When all the bacteria are actively dividing in the log phase of their growth, the culture is inoculated with the target phage. After three to four hours of incubation, when nearly all the bacteria have lysed and released the phage particles, the donor phage lysate is inoculated into a freshly grown culture of the recipient bacterial strain. After a brief incubation of one hour, the culture should now contain a mixture of transduced and non-transduced bacterial cells and this is screened for the transduced cells by spreading a fraction of the suspension onto an appropriate selective solid growth media. Upon further incubation, the transduced cells should grow and multiply to yield visible colonies. These colonies can then be selected for further analysis using a variety of methods to further confirm successful transduction, such as colony PCR, DNA sequencing, or quantitative PCR.
Before starting the procedure, put on any appropriate personal protective equipment, including a lab coat and gloves. Next, sterilize the workspace with 70% ethanol and wipe down the surface.
After this, prepare three one-milliliter aliquots of LB salt solution. Now, prepare a donor strain culture by adding 100 microliters of E. coli to a 15 milliliter conical vial containing five milliliters of LB growth medium with 500 micrograms of ampicillin. Then, grow the culture overnight at 37 degrees Celsius with aeration and shaking at 220 rpm. The next day, wipe down the bench top with 70% ethanol before removing the culture from the shaking incubator. Next, dilute the overnight culture one to 100 by adding 10 microliters of donor strain to 990 microliters of fresh LB supplemented with salt solution.
Allow the bacterial dilution to grow at 37 degrees Celsius for two hours with aeration and shaking at 220 rpm. Once the cells have reached early log phase, remove the culture from the incubator, add 40 microliters of P1 phage to the culture and incubate again. Continue to monitor the cells for one to three hours until the culture has lysed. Next, add 50 to 100 microliters of chloroform to the lysate and mix by vortexing. Then, centrifuge the lysate to remove debris and transfer the supernatant to a fresh tube. Add a few drops of chloroform to the supernatant and store it at four degrees Celsius for no more than one day.
To begin the transduction procedure, obtain a one milliliter culture of recipient strain. Next, transfer 100 microliters of donor phage lysate into a 1.5 milliliter microcentrifuge tube and incubate it at 37 degrees Celsius with the cap open for 30 minutes to allow any remaining chloroform to evaporate. While the donor phage lysate incubates, pellet the recipient strain cells via gentle centrifugation. Discard the supernatant and resuspend the cell pellet in 300 microliters of fresh LB containing 100 millimolar magnesium sulfate and five millimolar calcium chloride.
Next, set up the transduction reaction by combining 100 microliters of the recipient strain and 100 microliters of the donor phage lysate in a microcentrifuge tube. Then, set up the negative control by combining 100 microliters of the recipient strain and 100 microliters of the LB with magnesium sulfate and calcium chloride. After incubation, add 200 microliters of one molar sodium citrate and one milliliter of LB to both tubes, and mix by gently pipetting up and down. Then, after the tubes have been incubated for an hour, gently pellet the cells via centrifugation.
After centrifuging, discard the supernatant and resuspend the pelleted cells in 100 microliters of LB with 100 millimolar sodium citrate. Vortex the solutions and pipette the entire transduced sample onto an LB agar plate with 1X ampicillin. Finally, pipette the entire volume of the negative control cell mixture onto an LB agar plate without ampicillin. After incubating the plates overnight at 37 degrees Celsius, use a sterile pipette tip to pick three to four colonies from the transduction plate and streak them onto a new LB agar plate containing 1X ampicillin and 100 microliters of one molar sodium citrate. Repeat this plating method for the negative control on another LB agar plate containing only 100 microliters of one molar sodium citrate. Then, incubate the plates at 37 degrees Celsius overnight to allow colonies free of phage to grow.
The next day, wipe down the bench top with 70% ethanol before removing your plates from the incubator. Using a sterile pipette tip, pick three colonies from the transduction plate and add them each to a separate tube containing five milliliters of LB media. Then, select three colonies from the negative control plate and add them to another tube containing five milliliters of LB media. Grow the cultures overnight at 37 degrees Celsius with aeration and shaking at 220 rpm. After sterilizing the bench top as previously demonstrated, use a DNA miniprep kit to isolate DNA from 4.5 milliliters of each culture according to the manufacturer’s instructions. Then, elute the DNA with 35 microliters of nuclease-free water and measure the resulting concentration by lab spectrophotometer. Finally, prepare glycerol stocks by adding the remaining 0.5 milliliters of both bacterial solutions to 0.5 milliliters of 100% glycerol.
To confirm transduction, first prepare two qPCR master mixes for 24 qPCR reactions. For the first master mix, add 150 microliters of qPCR buffer mix to a microcentrifuge tube and 12 microliters each of a forward and reverse primer designed to amplify the ampicillin resistance gene. Next, prepare a second qPCR master mix by adding 150 microliters of qPCR master mix to a microcentrifuge tube and then adding 12 microliters each of a forward primer and reverse primer designed to amplify a housekeeping gene.
For each qPCR reaction, combine 100 micrograms of experimental DNA from each reaction with 14.5 microliters of qPCR master mix. Now, prepare the remaining reactions as previously demonstrated. Transfer the reactions to a thermocycler preheated to 94 degrees Celsius and then initiate the program. Finally, use the cycle quantification, or Cq, values generated by qPCR to calculate the normalized transduction efficiency of the ampicillin resistance gene.
The cycle quantitation, or Cq, values for the genes of interest were tabulated for each of the negative controls and transduced samples. Low Cq values, typically below 29 cycles, like the transduced samples in this example indicate high amounts of the target sequence.
A housekeeping gene, also tabulated here, is used as a loading control to normalize the amount of DNA in each reaction and as a positive control to ensure the qPCR is working. Provided the same amounts of the housekeeping gene are loaded, it is found at relatively the same rate in each sample.
Next, to calculate the delta Cq value for each sample, subtract the Cq value of the housekeeping gene for each sample from the Cq value of its corresponding target gene. For example, the delta Cq of the first negative control is 13.54. Then, use this value to calculate the normalized transduction efficiency of each sample using the formula shown here. Finally, the average normalized transduction efficiency for each sample group can be calculated.
