Summary
合成生物学使蛋白质的工程具有空前的特性, 使用非规范氨基酸的共平移插入。在这里, 我们介绍了如何一个光谱红色转移变种的 GFP 型荧光与新的荧光光谱性质, 称为 "金" 荧光蛋白 (GdFP), 是产生于大肠杆菌通过选择性压力成立 (SPI)。
Abstract
荧光蛋白是生命科学的基本工具, 特别是活体细胞的荧光显微镜。虽然从水母维多利亚(avGFP) 和其他物种同系物的绿色荧光蛋白的野生型和工程化变种已经覆盖了大部分的光学光谱, 但在近红外区域仍然存在光谱缝隙,哪些 avGFP 的显影是不可用的。红移荧光蛋白 (FP) 变体将极大地扩大多分子物种光谱分解的工具箱, 但从珊瑚或海葵衍生出来的自然发生的红移 FPs 具有较低的荧光量子产量和比 avGFP 变体差的照片稳定性。生色的共轭系统对远红色光谱区域的进一步操纵和可能的扩展也被基因代码规定的20标准氨基酸的汇辑所限制。为了克服这些局限性, 合成生物学可以通过将非规范氨基酸插入生色三合会, 进一步实现光谱红移。我们描述了 SPI 在设计具有新的光谱性质的 avGFP 变型的应用。蛋白质表达在色氨酸-auxotrophic 的大肠杆菌菌株中进行, 并通过适当的吲哚前体补充生长培养基。在细胞内, 这些前体被转化为相应的色氨酸类似物, 并被核糖体机械纳入蛋白质中, 以响应该密码子。avGFP (ECFP) 的增强的 "青色" 变体中的 Trp-66 替换为电子捐赠的 4-aminotryptophan 结果 GdFP 具有 108 nm 转移和强烈红移发射最大值 (574 nm), 而热力学上更稳定比它的前辈 ECFP。用质谱法分析了非规范氨基酸的残留特异性。GdFP 的光谱特性以时间分辨荧光光谱为特征, 是基因编码 FPs 在生命科学中的重要应用之一。
Introduction
自从 1962年1和 1994年2在其他真核细胞中的第一个异基因表达在水母水母维多利亚(avGFP) 中发现绿色荧光蛋白, GFP 家族的荧光蛋白就成为生命科学中非常有价值的工具和目标。广泛的遗传和分子工程学包括调整特定种类的密码子使用, 加速折叠, 改进成熟, 提高亮度, 防止齐聚和调整光谱和光化学性质包括可逆 photoswitch3,4,5,6的能力。GFP 的荧光来自其 4-(p-hydroxybenzylidene) imidazolidin-5-一 (HBDI) 生色。后者是 autocatalytically 形成的所谓的生色三联氨基酸 (Ser-65/Tyr-66/Gly-67 在 avGFP) 后形成了额外的共价键在肽骨干内的影响下的分子氧7。共振稳定共轭系统动态地与它的分子环境相互作用, 允许吸收在可见光范围和特征绿色荧光的这些蛋白质。
在生色三合会内, 芳香氨基酸的存在是强制性的。然而, 标准氨基酸汇辑仅包括四芳香残留物 (他的, Tyr, 激进党和的)。这限制了传统的诱变方法, 实现了相对于最红移的自然 FPs (如 DsRed8 ) 从Discosoma 白芨coralimorphs 或 mKate/mNeptune9到更大程度上更红移的 avGFP 变体。海葵Entacmaea quadricolor。因此, 600 纳米以上的光学光谱的远红色和近红外部分是由 GFP 变体所覆盖的。当然, 这是一个严重的限制, 荧光显微方法需要的光谱解复用的几个荧光物种的同时。例如, 长波长标记也是必要的, 利用皮肤组织的低吸收制度在 700-1, 000 毫微米在设置为深部组织成像10。
从 avGFP 中提取的荧光蛋白根据其染色体11的光谱性质和化学性质分为若干类。与它的三合会 Ser-65/Tyr-66/Gly-67, 狂放的类型生色存在作为平衡混合物在中性, 酚醛形式之间 (λmax = 395 毫微米, ε = 2.1万 M-1cm-1) 和阴离子苯酚形式 (λ最大= 475 毫微米, ε = 7100 M-1cm-1), 发射光谱呈 508 nm 的单一峰值。Ser-65 的羟基是至关重要的, 因为它向生色附近 (距离: 3.7 Å) 的 Glu-222 捐赠了一个 H 键, 促进了这种羧酸的电离。类 I 的特点是阴离子苯酚生色, 如在 EGFP (Phe-64-Leu/Ser-65-Thr; λmax = 488 毫微米, ε = 35600 M-1cm-1, λem = 509 nm)。由于 65 (甘) 取代, 中性苯酚的 395 nm 激发峰值被抑制, 阴离子苯酚的 470-475 nm 峰值为五-六倍增强, 并转移到 490 nm。第二类是含有中性酚生色的蛋白质, 如蓝宝石-GFP。在这里, Thr-203-Ile 替代几乎完全压制 475 nm 激发, 只留下峰值在 399 nm。由于阴离子生色不能正确溶解, 所以它的中性形式受到青睐。第三类包括 "黄色" 荧光变体 (EYFP;Ser-65-Gly/Val-68-Leu/Ser-72-Ala/Thr-203-Tyr;λmax ε = 514 nm, ε = 84600 M-1cm-1, λem = 527 毫微米) 与一个芳香的边链子和苯酚的π堆叠相互作用, 由 203-他 (激进党,, Tyr) 替换带来, 导致20毫微米红色转移的发射极大值 (Thr-203-Tyr)。进一步替换 (Gln-69-Lys) 导致另外1-2 毫微米红色转移到529毫微米, 最红色被转移的 avGFP 变形已知11。酚的交换为吲哚 (Tyr-66-Trp) 创造 IV 类, 如在青色荧光 ECFP (Ser-65-Thr/Tyr-66-Trp; λmax1 = 434 毫微米, ε = 24800 M-1cm-1; λmax2 = 452 毫微米, ε = 23600 M-1cm-1;λem1 = 477 nm, λem2 = 504 nm)。大量的吲哚的容纳可能是由其他补偿性突变所启用的。ECFP 的激发和发射最大值下降 inbetween 蛋白质的中性或阴离子染色体。V 类蛋白将咪唑置于苯酚 (Tyr-66-His) 的位置, 如 EBFP 的蓝色荧光蛋白. VI 类是由苯酚-苯基交换产生的, 只偏向中性生色形式, 从而导致最蓝移的激发和发射峰值位置 (分别为 360 nm 和 442 nm)。
经典的站点定向诱变特别适合生产新的 avGFP 生色变种, 由65-67 三肽的置换和相互作用的残留物在20规范氨基酸框架。当在核糖体蛋白合成过程中引入非典型的芳香氨基酸变体时, 这些可能性可以进一步扩大12。原则上, 有两种方法来实现这一点。第一种策略依赖于蛋白质翻译机械的基体耐受性, 特别是氨酰-tRNA 合成酶系 (aaRSs) 对相关氨基酸类似物的依赖性。为了高效地实现这一目的, auxotrophic 的大肠杆菌表达菌株无法合成相应的天然氨基酸。这可以通过在培养基中加入适当的非规范氨基酸 (ncAAs) 或前体来替代后者。这个策略, 也称为选择性压力合并 (SPI)13,14, 启用了特定于残滓的替换, 这导致了 ncAA 的全球合并。第二个策略使用停止密码子抑制 tRNAs, 这是由设计的 aaRS 酶充电的 ncAA。这就导致了帧内停止密码子的系通读, 并允许特定于站点的 ncAA 合并。因此, 这种停止密码子抑制方法 (SCS) 导致了遗传代码15的扩展。通过突变, 一个停止密码子被放置到目标基因在所需的地点。在原则上, SPI 也可以用来创建具有独特的 ncAA 安装的重组肽和蛋白质, 考虑到罕见的标准氨基酸, 如满足或跨国激进分子选择替代。通过跨国激进党, SPI 方法已被证明与大量的类似物, 包括 4 F, 5-f 和 6 f-跨国激进党, 7-aza-跨国激进党, 4-oh 和 5-oh 跨国激进党, 以及 4, 和 5-NH2-跨国激进党或甚至β (thienopyrrolyl) 丙氨酸衍生物16 ,17,18,19,20。因此, SPI 可能是非常有利的取代芳香氨基酸的 GFP 染色体的非规范变体, 以探索进一步调整光谱和斯托克斯转移这些 FPs 的可能性。对于所有蛋白质序列的修改, 必须对与 FP 折叠和生色成熟的相容性进行实验测试。
在这项工作中, 我们利用 IV 类 ECFP21, 它运载而不是野生类型的 avGFP Tyr, 在其生色三合会内的一个激进分子残留物。使用 SPI, 这种 Trp-66 (和 Trp-57, ECFP 中唯一的其他的激进党残留物) 被 4-氨基跨国激进党取代。在生色中, 4-氨基跨国激进党的电子捐赠氨基酸群的存在, 有利于一个远红色转移的激发态质子转移 (ESPT) 的共振镇定, 赋予了 108 nm 转移。这种 "金" 荧光蛋白 (GdFP) 构成的变种与最大的红色转移的荧光最大值 (574 毫微米) 之间的所有 avGFP 衍生蛋白。我们描述了 SPI GdFP 蛋白生产的方法, 并提供了通过质谱对产生的修饰蛋白进行强制性分析的协议。此外, 我们还展示了如何利用和分析 GdFP 的时间分辨荧光光谱方法。
Protocol
1. 跨国激进党的 auxotrophic大肠杆菌的转换
- 转换化学或 electrocompetent 细胞 (50 µL) 的激进党 auxotrophic大肠杆菌菌株, e. g。美洲金枪鱼委员会 49980 (WP2, 突变体来自大肠杆菌应变 B/R22), 分别采用热休克或电穿孔的 pQE-80L His6-ECFP 质粒的 1 ng/µL 水溶液的1µL。有关详细信息, 请参阅朱庇特科学教育数据库23,24 。
注意: 表达式向量 pQE-80L His6-ECFP 编码一个 N 末期6x 他标记的 ECFP21 , 由细菌 T5 启动子与紫胶运算符驱动。它还带有一个 AmpR选择标记和一个 colE1 的复制源 (pQE-80L 向量主干序列可以在上找到: https://www.qiagen.com/mx/resources/resourcedetail?id=c3b71572-4d82-4671-a79b-96357fe926d1 & lang = en& autoSuggest = true)。His6-ECFG 野生型蛋白 (生色成熟后25) 的理论分子量为 28303.92 Da。翻译的目标蛋白质序列如下 (他的标记带下划线, 向量派生序列以粗体表示): MRGSHHHHHHGSMVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTWGVQCFSRYPDHMK
QHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYISHNVYITADKQKNGIKANFKIRHNIEDGS
VQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK。 - 在 LB 琼脂板上的板变换细胞 (表 1) 辅以10克/升葡萄糖, 100 µg/毫升氨苄西林, 并在37摄氏度一夜之间孵化板材。
2. 重组蛋白表达
-
隔夜文化的大肠杆菌ATCC 49980 pQE-80L His6-ECFP
- 准备5毫升的 LB 培养基 (表 1; 补充10克/升葡萄糖, 100 µg/毫升氨苄西林) 在一个无菌的14毫升聚苯乙烯培养管有氧生长和接种与单一殖民地从琼脂板使用不育的吸管尖端或接种循环。
注意: 建议使用新转换细胞的菌落。有细菌菌落的板块 (从步骤 1.2) 可以储存在4摄氏度, 数天。 - 在200-250 转一夜的轨道振动筛中孵化37摄氏度的细胞。
- 准备5毫升的 LB 培养基 (表 1; 补充10克/升葡萄糖, 100 µg/毫升氨苄西林) 在一个无菌的14毫升聚苯乙烯培养管有氧生长和接种与单一殖民地从琼脂板使用不育的吸管尖端或接种循环。
-
野生型 ECFP 的表达
- 接种10毫升新鲜 LB 培养基 (表 1; 辅以10克/升葡萄糖, 100 µg/毫升氨苄西林), 100 毫升锥形瓶中的隔夜文化µL。在 200 rpm 的轨道振动筛中孵化37摄氏度的烧瓶。
注: 可选, 此步骤可以在10毫升 NMM19 培养基 (表 1) 中进行, 辅以100µg/升氨苄西林和0.5 毫米 l-色氨酸 (或者, 可以使用吲哚)。 - 每20分钟测量600毫微米 (od600) 的光学密度. 优先测量细胞密度, 方法是使用具有600厘米路径长度的试管, 在分光光度计中测定 600 nm (od1) 的灭绝. 始终执行引用使用相应的培养基进行测量。稀释样品并混合样品, 以获得 0.1-0.8 的测量值, 然后使用稀释因子计算 OD600 。有关详细信息, 请参阅以前的出版物26。
- 当到达 OD600值 0.5-0.8 (大约 2-3 h 在接种以后), 采取样品 "在归纳之前" 为 SDS 页 (月桂酸钠聚丙烯酰胺凝胶电泳, 步 4)。
- 通过调整液体培养到0.5 毫米 IPTG (异丙基β-1-thiogalactopyranoside, 从 1 M 库存溶液) 中诱导靶蛋白表达, 并在30摄氏度的轨道振动筛中孵化, 200 rpm 为4-8 小时。
注: 青色荧光蛋白通常在温度低于37摄氏度27时表达。 - 为 SDS 页 (步骤 4) 取样 "后表达式"。
- 在 5000 x g 和4摄氏度处离心10分钟, 以收获细菌细胞。
- 在-20 摄氏度或-80 摄氏度之前, 用醒酒和冷冻细胞颗粒, 直到目标蛋白质纯化。
- 接种10毫升新鲜 LB 培养基 (表 1; 辅以10克/升葡萄糖, 100 µg/毫升氨苄西林), 100 毫升锥形瓶中的隔夜文化µL。在 200 rpm 的轨道振动筛中孵化37摄氏度的烧瓶。
-
生产 GdFP 的 SPI
- 接种10毫升的 NMM19 培养基 (表 1) 补充100µg/毫升氨苄西林, 15 µM 色氨酸和10µL 隔夜文化在100毫升锥形瓶和孵化文化瓶隔夜在30°c 在一个轨道振动筛在 200 rpm。
注: 有多种化学定义的培养基可用于培养大肠杆菌和 SPI。除此处使用的 NMM 外, 拖把介质28、葡萄糖矿物盐介质29、戴维斯最小介质30、M9 最小中31或 GMML32都可以使用。 - 第二天, 每30分钟测量600 OD, 直到该值仅在30分钟内小于0.05。高原值应约为1。
注: 偏差0.3 单位是可以接受的。根据细菌的应变和培养基使用, 最初的色氨酸浓度 (2.3.1) 可能需要调整。 - 在 SDS 页 (步骤 4) 中取样 "感应前"。
- 在 5000 x g 和4摄氏度处离心10分钟, 以收获细菌细胞。用醒酒丢弃上清液。
- 并用重悬10毫升的 NMM19 培养基中的细胞, 100 µg/毫升氨苄西林成100毫升锥形瓶, 并添加4氨基吲哚到最后浓度1毫米使用50毫米的股票溶液。在 200 rpm 的轨道振动筛中继续孵化30分钟, 30 摄氏度。
注意: 建议这一步骤是因为氨苄西林化学稳定性低, 确保细胞摄取 4-氨基吲哚。 - 通过添加 IPTG 到最终浓度为0.5 毫米的目标蛋白表达, 使用1米的股票, 并在 200 rpm 的轨道振动筛中以30摄氏度在一夜之间孵化样品。
注: 青色荧光蛋白通常在温度低于37摄氏度27时表达。 - 第二天, 测量 OD600。
- 为 SDS 页 (步骤 4) 取样 "后表达式"。
- 通过离心10分钟, 在 5000 x g 和4摄氏度的情况下, 通过醒酒来去除细菌细胞。
- 如果此类容器不用于离心, 则使用刮刀将细胞颗粒转化为50毫升锥形聚苯乙烯管。冷冻细胞颗粒在-20 °c 或-80 °c, 直到目标蛋白质纯化。
- 接种10毫升的 NMM19 培养基 (表 1) 补充100µg/毫升氨苄西林, 15 µM 色氨酸和10µL 隔夜文化在100毫升锥形瓶和孵化文化瓶隔夜在30°c 在一个轨道振动筛在 200 rpm。
3. 通过固定化金属离子亲和层析 (IMAC) 纯化靶蛋白
-
细菌细胞裂解
- 在冰上解冻细胞颗粒10-20 分钟。
- 并用重悬在50毫升圆锥聚苯乙烯管中使用5毫升的冰冷结合缓冲器(表 1) 在冰上的细胞颗粒。
- 添加20µL 50 毫克/毫升溶菌酶, 20 µL 1 毫克/毫升 DNase I, 20 毫克/毫升 RNase a. 关闭管, 轻轻混合, 反转1次, 并保持在冰上5分钟。
注: 部分细胞紊乱发生于溶菌酶催化。 - 溶解发音使用超声均质器尖端, 使用三循环3分钟, 在15毫升的聚苯乙烯管冷却, 2 秒的脉冲, 4 s 的暂停和45% 振幅。
注: 另外, 可以使用高压均质, e. g, 20 循环 1.4万 psi。如有必要, 稀释使用装订缓冲器达到最小仪器体积。此外, 蛋白质提取试剂可用于细胞分裂。请参阅材料表中的示例。 - 离心样品为30分钟在 1500万 x g, 4 °c。
- 将上清液转移到新鲜的管子中, 并记下液体体积。
- 使用5毫升塑料鲁尔锁注射器和聚偏氟乙烯 (PVDF) 注射器过滤器过滤溶液, 通过0.45 µm 注射器过滤器。
- 为 SDS 页 (步骤 4) 抽取样本 "裂解"。
- 并用重悬细胞碎片颗粒在ddH2 O (相等的容量作为前裂解物)。
- 为 SDS 页取样 "颗粒" (步骤 4)。
-
IMAC 净化
- 根据制造商的说明使用1毫升预或自包装的 IMAC FPLC (快速蛋白质液相色谱) 柱。使用绑定缓冲区 (表 1) 进行列平衡以及在将单元裂解器应用于该列之后的洗涤步骤。
- 收集和池洗脱液分数与 GdFP, 可以识别的可见光金黄色的颜色。
注: 可以选择使用自动 FPLC 系统, 使用线性咪唑梯度 (0-250 毫米) 洗脱靶蛋白。 - 在 466 nm (ɛ466 nm = 23700 M-1 cm-1)33中, 用洗脱缓冲器作为参考, 确定蛋白质浓度的文献价值。有关该过程的详细信息, 请参阅以前的出版物26。
- 取样本 "洗脱液" 为 SDS 页, 并使用1-10 µg 的蛋白质每条车道的情况下, 考马斯染色。
注: SDS 样本量根据染色方法和染料灵敏度而定。 - 透析使用一个分子量截止 (截留分子量) 为500万的膜, 对透析缓冲液或 MS 缓冲器整除洗脱液分数。根据制造商的指示准备透析膜。透析1毫升样品三次对100毫升缓冲器至少 2 h。有关此过程的详细信息, 请参阅以前的出版物34。
- 贮存时, 在透析缓冲液中冷冻蛋白样本-80 摄氏度。
注: 整除数应稳定至少6月。
4. SDS 页样品制备大肠杆菌全细胞提取物
- 将细胞悬浮液等效于1毫升 od600 = 1 悬浮 (e. g500 µL 的 od600= 2) 成1.5 毫升离心管。
- 在 5000 x g, 室温下离心10分钟来收割细胞。用吹打丢弃上清液。
- 添加80µL 的 ddH2O 和20µL 的 5x SDS 加载染料缓冲区 (表 1) 到细胞颗粒和混合吹打。
- 变性在水浴或热块中加热至95摄氏度以5分钟的温度。随后, 将样品冷却到室温。
- 根据以前的出版物35, 使用10µL 考马斯着色的 SDS 页面。
注: SDS 样本量根据染色方法和染料灵敏度而定。
5. 用高效液相色谱 (HPLC) 耦合到电喷雾电离飞行时间质谱 (LC-ESI-MS) 的完整蛋白质质量分析
注: 高效液相色谱梯度, 设置和缓冲可能会因分离柱和仪器使用而异。参见材料表为模范设备。
- 根据上述 (步骤 3.2.3) 使用 ms 缓冲器 (参见材料表), 确定从样品透析到 ms 缓冲器的蛋白质浓度。
- 用 ms 缓冲器将蛋白质样品稀释为0.1 毫克/毫升, 最后体积为80µL, 通过仔细的吹打混合, 将溶液转化为 autosampler 的小瓶, 并用瓶盖将其关闭。通过弹瓶子去除气泡。
- 用1毫升 MS 缓冲器填充第二 autosampler 瓶, 不带玻璃插入 (缓冲器空白)。
- 让仪器预热。执行仪器校准。请确保有足够数量的液相色谱级溶剂 (> 100 毫升)。
- 程序一个线性20分钟 HPLC 梯度从5% 到80% 缓冲器 a (0.1% 甲酸在 ddH2O), 结合缓冲 B (0.1% 甲酸在乙腈)。
- 以0.3 毫升/分钟的流量开始高效液相色谱, 等待柱压力稳定。
- 为 LC-ESI-MS 方法设置一个 autosampler 注入量5µL, 为空白运行创建一个工作列表中, 然后执行一个示例运行, 并分配相应的 autosampler 小瓶位置。运行工作列表中。
- 完成工作列表中后, 打开生成的示例数据文件。在总离子电流 (TIC) 图中选择一个范围, 用于反褶积, 并利用最大熵反褶积算法 deconvolute MS 谱。
注意: 根据实验条件, 额外的物种可能发生在非成熟的 FP 或缓冲离子加合物。
6. GdFP 的荧光寿命测量和衰变相关谱 (DAS)
注: 用于仪器的时间分辨荧光光谱学, 请参阅材料表的模范设备。荧光蛋白的吸光度以及荧光激发和发射光谱也可以用实验室紫外/可见光和荧光光度计记录。
-
GdFP 波长分辨荧光寿命的测定
- 在 pH 7 上稀释成 PBS 缓冲器 (表 1), 准备2毫升1µM 溶液 GdFP。将溶液填入1厘米的石英试管。
- 安装 ps 脉冲 470 nm 激光用于样品励磁和 488 nm 长通发射过滤器, 并调整600升/毫米光栅的时间和波长相关的单光子计数36 (TWCSPC) 探测器的波长机制的获取500-700 毫微米。
- 获得荧光发射的计数率约 200 x 103光子/s, 直到约 103计数累积在获取最大的荧光衰减曲线与单光子计数软件。
-
仪器响应函数的测量37 (公路联合会)
- 将样品试管用1厘米石英试管填充, 在 PBS 缓冲器中用1克/升胶体二氧化硅 (~ 220 米2/克) 来代替 pH 值7。
注: 硅胶悬浮液采用400克/升水悬浮液制备。 - 删除 488 nm 长通发射过滤器并插入灰色滤镜, 将 TWCSPC 探测器的计数速率调整到低于 100 x 103计数/秒。
- 在16通道 TWCSPC 探测器的通道8中, 调整光栅以获取 470 nm 光子。
- 获取公路联合会, 直到约 10 x 103计数在发射最大值中累计。
- 使用全局管接头38程序将荧光衰减曲线和公路联合会转换为 ASCII 数据文件。
- 根据与生存期为链接参数的三指数分量的总和的模型进行全局拟合。
- 在波长与数据分析软件的依赖性下, 将衰变相关谱 (DAS) 作为单个衰变分量的振幅分布。
- 将样品试管用1厘米石英试管填充, 在 PBS 缓冲器中用1克/升胶体二氧化硅 (~ 220 米2/克) 来代替 pH 值7。
Representative Results
采用选择性压力组合技术, Trp-66 在 ECFP (Trp-57) 生色三合会中, ECFP 中唯一的其他跨国激进分子残留物可由4氨基跨国激进分子取代, 从而产生具有不同光谱性质的红移 GdFP。质谱法必须用于证明所期望的非规范氨基酸的化学计量集成到蛋白质中, 结果显示在图 1中。之后, 我们提供的数据从显微镜, 紫外-可见光吸收光谱以及稳态和时间和波长分辨荧光光谱, 以表征 GdFP 荧光的性质, 重点是 pH 依赖性光谱。
为了确认 4-氨基跨国激进党在 ECFP 中两种激进的残留物的交换, 进行了质谱分析。图 1显示了 GdFP 的代表性 deconvoluted ESI-MS 频谱。野生型 ECFP 在生色成熟后有 28283.9 da 的计算蛋白质质量, 相应的 GdFP 质量为 28313.9 da。GdFP 的 deconvoluted ESI 谱显示一个主要质量峰值在 28314.1 0.1 Da, 它偏离理论值小于 10 ppm。在这类分析的典型精确度范围内25, 这证实了 ncAA 通过 SPI (实验值为狂放的类型 ECFP: 28283.7 Da) 的采纳。
图 2显示了 ECFP、EGFP、EYFP 和 GdFP 的细菌细胞共焦荧光成像显微术 (CFIM) 图像, 它表达了 PBS 缓冲器中的细菌泥沙。所有的图像都是在显微镜下获得的, 它具有紫外线目标和激光激发, 每个样品的能量大约相同。
图 3A显示了大肠杆菌细菌的 CFIM 图像的叠加, 它们表示各种 FPs, 包括 GdFP, 总是用非常相似的激发能量 (波长如 图 2) 进行监视。图 3B显示了所显示的 FP 变体的生色结构。关于 GdFP 的亮度比 ECFP (荧光量子屈服φ = 0.4), EGFP (φ = 0.6) 和 EYFP (φ = 0.6) 重要的是要注意的是, 对于 GdFP, 荧光光 (30 nm) 的更广的采集范围与用于所有其他 spe 的 20 nm 对比。为了将图像的强度调整到相似的值。由于独特的物理特性, 由于消光系数略低, 量子屈服率降低, GdFP 的亮度与其他 FPs 相比更低。
ECFP 的吸收光谱 (图 3C) 有两个特征极大值, 在434毫微米和452毫微米。相比之下, GdFP 的特点是一个广泛的红色转移吸收带最大值在 466 nm。EGFP 的吸收是进一步红色转移到488毫微米。然而, 由于 GdFP (108 毫微米) 比 ECFP (41 nm) 和 EGFP (20 nm) 大得多的斯托克斯转移, GdFP 的发射光谱是所有三种 GFP 衍生物中最红移的 (图 3D)。当 ECFP 的荧光发射显示两个特征极大值在475毫微米和505毫微米, EGFP 有一个宽广的主要发射带峰值在508毫微米 (λmax) 与轻微的肩膀在540毫微米。GdFP 的荧光显示约 565 nm (λmax).(图 3D)。它的发射光谱包含野生型 ECFP 的小贡献, 在 475 nm 的小肩上也可见。此小 ECFP 分数是在 SPI 过程中归纳之前合成的, 如33所述。
图 3E显示了 GdFP 的吸收谱中 pH 值相关的变化。对于 pH 值的变化从8到 5, 排放的最大变化轻微的红色和轻微的扩大吸收带被观察到。然而, 在 ph 值8和 ph 值5之间, 吸收振幅的减少仅为10% 左右, 这表明 GdFP 生色的基态性质受到 ph 值的微弱修正。
用单光子计数监测的时间分辨荧光发射量显示在图 4中。在以550毫微米和600毫微米为中心的光谱通道中监测到的衰变曲线 (图 4A) 在600毫微米上显示了稍快的荧光衰变, 而衰变在 550 nm。具有两个指数分量的荧光衰减曲线的全局拟合结果在两个光谱上区分的荧光衰变组件, 其时间常数为 1.0 ns 和 3.3 ns (图 4C和 D)。
GdFP 的荧光发射强烈依赖于 pH 值, 因为它是 GFP 家族许多荧光蛋白变种的典型。图 4B比较了 ph 值5和 ph 值8之间的 GdFP 的荧光发射, 这清楚地表明低 pH 值的荧光强度下降, 而光谱特性保持不变。
GdFP 的衰减相关谱 (DAS)39 (图 4C和 D) 的特征是两个不同的发射波段。慢 3.3 ns 组分的贡献是更加明显的在短波长范围大约550毫微米 (60%) 以较快的组分 (40%) 的次要贡献。在600毫微米, 两个组件有大约相同的振幅。从 ph 值 7 (图 4C) 到 ph 值 6 (图 4D) 的转变后, das 的频谱特性几乎没有变化, 并且来自全局拟合例程的时间常数也相同 (das 时间常数的准确性约为 0.15 ns)。然而, 两个 DAS 组件的绝对振幅的差异显然是明显的, 这充分说明了在图 4B中相同 pH 值变化时荧光发射振幅的降低。
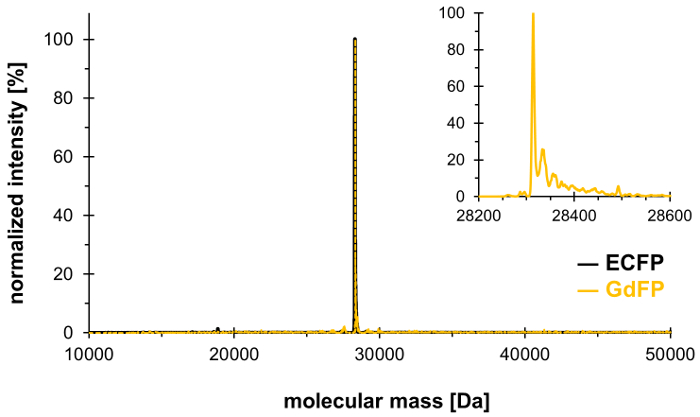
图 1: 代表性 deconvoluted ESI-GdFP 的 MS 频谱.GdFP 的 ESI-MS 谱 (金色, 放大图显示为嵌入) 显示一个主要峰值在 28314.1 da (计算值 28313.9 da)。野生型 ECFP 的光谱呈黑色。请单击此处查看此图的较大版本.

图 2: 共聚焦荧光显微图像, 来自细菌群表达各种 FPs.以下波长设置用于图像采集: ECFP (λex = 457 nm, 检测: 461-480 nm), EGFP (λex = 488 nm, 检测: 495-515 nm), GDFP (λex = 476 nm, 检测: 560-590 nm), EYFP (λ前= 514 nm,检测: 520-530 nm)。请单击此处查看此图的较大版本.

图 3: GdFP 的光谱属性.(A) CFIM 在 PBS 缓冲区中的细菌泥沙后表达 ECFP、EGFP 和 GdFP 的细菌细胞的混合图像。(B) 生色结构 GdFP (用4氨基跨国激进党代替残留物 66), 父母 ECFP (与在位置66的跨国激进党) 和 EFGP (与 Tyr 在位置 66)。(C) 对 GdFP、ECFP 和 EGFP 的归一化吸收光谱进行比较, 而在 (D) 中, 将 ECFP 的归一化荧光发射谱 (430 nm 的激发) 与 EGFP 和 GdFP 的荧光发射光谱进行比较 (两者均兴奋在 450 nm)。(E) 吸收谱的 pH 依赖性 (归一化吸收在 280 nm)。请单击此处查看此图的较大版本.

图 4: GdFP 的时间分辨荧光.(A) 在 550 nm 和 600 nm (12.5 nm) 的光谱信道中, 以 470 nm 激光脉冲为中心, 用时间和波长分辨的单光子计数监测 GdFP 的荧光衰减。工具响应函数 (公路联合会) 提供有关使用的安装程序的时间分辨率的信息。(B) 依赖于 pH 值 (460 nm 的激发) 的 GdFP 的发射谱的变化。(C、D)GdFP 在 ph 值为 7 (C) 和 ph 值 6 (D) 上的衰变相关谱 (DAS) 是在时间和波长分辨荧光衰变的反褶积和所有通道中的衰变的全局拟合, 由一个全局集合的两个指数函数与链接的时间常数。请单击此处查看此图的较大版本.

图 5: ECFP (黑色) 和 GdFP (金) 染色体的分子内电荷转移结构.生色系统的大小增加由一个氨基酸的良好的电子捐助者作为 ncAA 的一部分, 使形成更多的中介结构, 以实现共振稳定的兴奋状态。与 FP 脚手架的连接点显示为半圆。请单击此处查看此图的较大版本.
| 库存解决方案 | 浓缩、溶剂 | 注意 | |
| 20% d-葡萄糖 | 200克/升 d-葡萄糖在 ddH2O | 通过0.45 µm 孔大小注射器过滤器进行杀菌消毒 | |
| 吲 哚 | 50毫米在异丙醇 | ||
| 4-氨基吲哚 | 50毫米在20% 乙醇 (20 毫升乙醇在最后容量100毫升填装与 ddH2O) | ||
| IPTG | 1 M ddH2O | ||
| l-色氨酸 | 15毫米溶解在 ddH2O 使用 1 M HCl (添加 hcl 滴在搅拌, 直到粉末 dissoved) | ||
| 溶 菌 酶 | 50毫克/毫升在 ddH2O | ||
| DNase 我 | 1毫克/毫升在 ddH2O | ||
| RNase A | 1毫克/毫升在 ddH2O | ||
| Amp100 | 100毫克/毫升氨苄西林在 ddH2O | ||
| 钠硫酸钠 (SDS) | ddH2O 中的200克/升 | ||
| 硫酸铵 (NH4)24) | 1 M ddH2O | 热处理消毒 | |
| 磷酸二氢钾 (2PO4) | 1 M ddH2O | 热处理消毒 | |
| 二磷酸氢钾 (K2HPO4) | 1 M ddH2O | 热处理消毒 | |
| 硫酸镁 (MgSO4) | 1 M ddH2O | 热处理消毒 | |
| d-葡萄糖 | 1 M ddH2O | 通过0.45 µm 孔大小注射器过滤器进行杀菌消毒 | |
| 氯化钠 (氯化钠) | 5 M ddH2O | 热处理消毒 | |
| 氯化钙 (CaCl2) | 1克/升 | 通过0.45 µm 孔大小注射器过滤器进行杀菌消毒 | |
| 氯化铁 (FeCl2) | 1克/升 | 通过0.45 µm 孔大小注射器过滤器进行杀菌消毒 | |
| 硫胺素 | 10克/升 | 通过0.45 µm 孔大小注射器过滤器进行杀菌消毒 | |
| 素 | 10克/升 | 通过0.45 µm 孔大小注射器过滤器进行杀菌消毒 | |
| 跟踪元素组合 | 硫酸铜 (丘索4)、氯化锌 (ZnCl2)、氯化锰 (MnCl2)、钼酸铵 ((NH4)2MoO4);ddH2O 中的每1毫克/升 | 通过0.45 µm 孔大小注射器过滤器进行杀菌消毒 | |
| 19氨基酸混合 | 1.) 将0.5 克 l-苯丙氨酸和0.5 克 l-酪氨酸溶于100毫升 ddH2O, 并在搅拌后滴状添加1米 HCl, 直至粉末溶解。 | ||
| 2.) 对其余的 l-氨基酸 (l-色氨酸除外) 的0.5 克进行称量。与22毫升1米的2PO4和48毫升 1 M K2HPO4混合。将 ddH2O 添加到大约800毫升。搅拌, 直到解决方案变得清晰。 | |||
| 3.) 在步骤1中加入溶解的 l-苯丙氨酸和 l-酪氨酸。用 ddH2O 将音量调整为1升。 | |||
| 4. 用瓶顶过滤装置对氨基酸混合物进行真空过滤消毒。 | |||
| 缓冲区和媒体 | 组成/准备 | ||
| 负载染料缓冲液, 5x 浓缩 | 0.25 米三 pH 值 6.8, 50% 伏/v 甘油, 0.25% 瓦特/v 溴酚蓝色, 0.5 M didhiothreitol (德勤; 或者5% β-巯基乙醇), 10% 瓦特/v 钠硫酸钠 (SDS) | ||
| 绑定缓冲区 | 50毫米 dihydrogenphosphate 钠 (NaH2PO4), 500 毫米氯化钠, 10 毫米咪唑, pH 8 | ||
| 洗脱缓冲器 | 50毫米 dihydrogenphosphate 钠 (NaH2PO4), 500 毫米氯化钠, 250 毫米咪唑, pH 8 | ||
| 透析缓冲液 | 50毫米 dihydrogenphosphate 钠 (NaH2PO4), 150 毫米氯化钠, 100 毫升/升甘油, pH 8 | ||
| MS 缓冲器 | 10毫米三盐酸, pH 值8 | ||
| 除 l-色氨酸 (NMM19) 外含有19种 l-氨基酸的新型最小培养基 | 混合所有库存解决方案以获得以下最终浓度: 7.5 毫米 (NH4)2所以4, 1.7 毫米氯化钠, 22 毫米2PO4, 50 毫米 K2HPO4, 1 毫米 MgSO4, 20 毫米 d-葡萄糖, 50 毫克/升19氨基酸混合, 1 µg/升 CaCl2, 1 µg/升 FeCl2, 10 µg/升硫胺, 10 毫克/升生物素, 0.01 毫克/升微量元素混合 | ||
| LB 介质 | 成份:10 克/升胰蛋白胨, 5 克/升酵母萃取物, 10 克/升氯化钠, pH 7.0 在 ddH 2 O | ||
| 制备: | |||
| 1.) 称50克胰蛋白胨, 25 克酵母提取物, 5 克氯化钠为1升玻璃瓶。 | |||
| 2. 增加 ddH2O 至800毫升, 并在搅拌下溶解元件。 | |||
| 3.) 测量 ph 值, 并在必要时通过滴状添加1米 HCl 或1米氢氧化钠来调整 ph 值7。添加 ddH2O 高达1升。 | |||
| 4.) 热处理消毒, 事后检查容积损失, 并添加无菌 ddH2O 以补偿必要的情况。存储在4°c, 直到使用。 | |||
| LB 琼脂板 | 成份:10 克/升胰蛋白胨, 5 克/升酵母萃取物, 10 克/升氯化钠, 15 克/升琼脂, pH 7.0 在 ddH 2 O | ||
| 制备: | |||
| 1.) 称50克胰蛋白胨, 25 克酵母提取物, 5 克氯化钠, 7.5 克琼脂琼脂为1升玻璃瓶。 | |||
| 2.) 添加 ddH2O 最多500毫升, 并在搅拌下溶解元件。 | |||
| 3.) 测量 ph 值, 并在必要时通过滴状添加1米 HCl 或1米氢氧化钠来调整 ph 值7。添加 ddH2O 高达1升。 | |||
| 4.) 热处理消毒, 事后检查容积损失, 并添加无菌 ddH2O, 如有必要, 进行补偿。(注: lb 琼脂可储存在4摄氏度, 直到用于制备 LB 琼脂板。用微波炉精心熔融固化琼脂) | |||
| 5.) 当溶液仍然温暖 (30-40 摄氏度) 时, 将氨苄西林添加到最后浓度为100µg/毫升 | |||
| 6.) 从步骤5中倒入大约15毫升液体。在无菌条件下进入无菌的10厘米培养皿。当琼脂凝固时, 盘子可以储存1周在4摄氏度, 直到使用。 | |||
| 磷酸盐缓冲盐水 (PBS) | 成份: 137 毫米氯化钠, 2.7 毫米氯化钾, 10 毫米 Na2HPO4, 1.8 毫米 2 PO 4, 1 毫米 CaCl 2, 0.5 毫米氯化镁 2, pH 7.热处理或过滤消毒。 | ||
表 1: 库存解决方案和缓冲。
Discussion
为了实现非常高的 ncAA 整合效率, 基于 auxotrophy 的 SPI 方法依赖于新陈代谢工程的宿主细胞, 它们无法合成 ncaa 的相应自然对应物。对于大肠杆菌, 这种菌株是容易获得的。使用 multiauxotrophic 菌株, 即使同时将多种 ncAAs 加入同一蛋白质中也是可行的。残留物的替代模式和化学汇限量被限制在类似的化学类比可以被视为缺点。然而, 由于天然细菌翻译器具容忍许多氨基酸类似物, 大量的蛋白质变异可以被生产。例如, 使用体外翻译可以将超过 50 ncAAs 合并到蛋白质中, 将遗传代码的所有密码子中约73% 用于重新分配40。此外, SPI 还可以允许有效的多站点标记目标蛋白41。原则上, SPI 方法不限于大肠杆菌, 但可以在任何其他宿主和标准的20氨基酸中工作, 前提是 auxotrophic 菌株和定义的培养培养基可用。例如, 两个蛋氨酸类似物, azidohomoalanine (哈) 和 homopropargylglycine (Hpg), 是商业上可用的, 用于标记蛋白质和蛋白质组在不同的有机体。此外, 阿哈可生产 intracellularly, 并随后纳入蛋白质42。这是特别适合 bioorthogonal 动词, 如单击化学开发的 Tirrel 和同事: 例如, 在植物组织的拟南芥, 在蚕森幼虫43,果蝇单元格44, 幼虫斑马鱼45并且哺乳动物细胞包括神经元46, 蛋白质可以被标记与阿哈47,48。类似地, 在跨国激进分子-auxotrophic球菌球菌菌株49中成功地将多党类似物纳入抗菌肽。SPI 对于 Xenobiology50、51的字段也很有用, 它探索了生命基本化学组成的替代方法。例如, 基于以前在大肠杆菌52和B. 枯草53上的工作, 一个大肠杆菌菌株是最近开发的一种具有选择性压力的进化策略, 利用 thienopyrrole 而不是吲哚, 导致蛋白质组广泛取代色氨酸由 thienopyrrole-丙氨酸在遗传代码54。通常, 由单三重编码的标准氨基酸跨国激进分子, 由于吲哚化学的丰富方面, 为蛋白质工程提供了一个有希望的目标, 它具有多种化学变异。最近, 作为一个替代 SPI 为基础的合并, 一个新的 SCS 平台, 能够结合跨国激进的类似网站-特别是在细菌和真核主机已报告了55。这进一步拓宽了在体内基于 ncAA 的蛋白质工程的工具箱, 包括光谱特性的改变。
除了使用 auxotrophic 表达主机, SPI 协议要求严格的发酵条件, 无论是在目标表达时间和培养基的组成, 以达到高 ncAA 的整合效率和目标蛋白质产量56. 耕种是使用化学定义的极小的媒介, 根本上包含除主要盐以外的来源为氮气 (铵盐) 和碳 (d-葡萄糖), 维生素和微量元素。虽然没有严格要求在没有进一步的 auxotrophies, 剩余的氨基酸 (20-n, 如果n氨基酸将被替换) 通常被增加促进细菌成长57。在诱导目标蛋白表达前的初始生长阶段, 将被替换的n规范氨基酸添加到限制浓度中。细胞生长一直持续到被靶向的必需氨基酸耗尽为止, 这是一个固定 OD600所表明的实验结果。随后, 培养基被新鲜培养基所取代, 缺乏枯竭的氨基酸, 并包含了全国体育协会在丰富的浓度。对于色氨酸类似物的核糖体合并, 如本协议所示, 一个吲哚模拟被喂食, 成为 intracellularly 转化为相应的色氨酸合成酶58。其次, 诱导靶蛋白表达。在这个阶段, 细胞接近于对数生长的末端, 作为总细胞数和健身之间的平衡。由于标准氨基的存在和加入会导致野生型蛋白的产生, 所以在诱导前必须保证必需氨基酸完全耗尽是至关重要的。同样, 它是强制性的检查的效率, ncAA 纳入目标蛋白, 通常通过质谱。在典型氨基酸的大量存在情况下, 需要对培养条件进行调整,例如, 通过改变基本氨基酸在初始生长阶段的浓度或后者的持续时间。如果对 ncaa 的 aaRS 活动很低, 则可以进行59, 以表达不同 aaRS 的内生酶或共同表达, 这对 ncaa 更积极。
典型氨基酸多党具有三显著特征: (i) 其蛋白质的自然丰度低;(二) 其生物物理和化学性质是独一无二的 (e. g), 它通常是蛋白质和肽的内在荧光的主要来源, (iii) 它有助于各种生物化学的相互作用和功能, 包括π叠加, H 键合和阳离子π相互作用。所有这些特征都发生了根本性的变化, 在跨国激进党→ 4-氨基跨国激进党替代 GdFP. 毫无疑问, "金" 类 avGFPs 的设计是工程定制 autofluorescent 蛋白的一个显著的例子。有了独特的光谱特性, FPs 可以通过诱变和 ncAA 的结合, 对某些光谱窗口进行调谐。在 GdFP 的情况下, 这是由一个简单的化学交换 H → NH2在 ECFP 生色三合会内的吲哚环的框架内完成的。图 5显示了 ncAA 在生色中的合并效果。引入由 4-氨基吲哚 (intracellularly 转化为 4-氨基跨国激进党) 的电子捐赠组, 使各种中介结构能够解释稳定的激发态。Spectroscopically, 其扩大的斯托克斯转移和红色转移荧光发射结果从这些明显的性质的延伸共轭系统。如前所述, 在 GdFP 生色内的增强内分子电荷转移本质上是对 pH 值 (图 4B) 的敏感, 并且伴随着 s0地面和 s1兴奋状态之间偶极矩的较大变化。相对于 ECFP33。作为替代的电子捐赠组, 在与模型蛋白 barstar41的比较研究中, 可以使用含羟基团取代的吲哚环的色氨酸类似物。
与 ECFP 和 EGFP (图 3C和 D) 相比, GdFP 的吸收和荧光光谱被拓宽。吸收和荧光带的均匀加宽一般是由生色中的振动模式引起的, 另外, 通过生色与蛋白质60中存在的进一步振动模式耦合。与局部蛋白质环境的耦合是由生色上的电荷所支持的。由于蛋白质结构的不均匀性导致了 vibronic 谱的局部变化, 生色的 vibronic 谱与蛋白质的其余部分之间的耦合是由电荷服务业和中介状态所表示的, 如图 5。这种耦合也支持大斯托克斯移位, 并必然降低荧光量子收益率。与其他红移 FPs 相比, GdFP 甚至表现出改进的蛋白质稳定性和低聚合趋势33,61,62。它不仅不同于其他 FP 变体的颜色, 而且还展示了极大增加的热稳定性和增强的协同折叠33。在加热到60摄氏度时, 其荧光强度至少保持 90%, 而 ECFP 荧光量减少到约30%。在蛋白质中, 芳香氨基酸常常有助于相互作用的侧链网络, 这通常对蛋白质的三级结构有稳定的影响。avGFP 拥有这样一个侧链网络, 包括生色本身, 以及 Phe-165、His-148 和 Tyr-145。这些侧链在 GdFP 结构中不仅相当刚性33, 但重要的是, 它们与生色形成疏水性接触。GdFP 中发现的最突出的新特征是合成生色更接近 Phe-165。此交互是其他已知 avGFPs 中未观察到的功能。由于这两种残留物分别为 3.2-4.5 Å, 因此也可能存在氨基芳香的相互作用。与胺化诱导的生色共振稳定, 这些最有可能稳定这种疏水性氨基酸网络的合作方式。相对于生色的基态, 在激发状态下, 可以支持更有效的内分子电荷转移, 并且至少部分地占 108 nm 移位33,62.
在荧光性能的合理设计中, 预测移位π系统的尺寸增大, 从而产生红移激发波长。这个拇指规则服从于位置66的氨基酸系列导致中性染色体: (λmax = 355 nm) < 他的 (λma x= 386 nm) < Tyr (λ最大= 395 nm) < 跨国激进党 (λ最大= 436 nm)63。在自然界中, 生色共轭系π键的延伸是通过不同的策略来实现的。对于来自Discosoma 白芨的 DsRed, 它由附加氨基酸的集成扩展, 从而将λmax转换为 573 nm64。Anemonia 苏卡达中的 asFP595 (λmax = 595 nm) 的生色是由亚氨基组扩展的, 其π系统65扩大.由于 GdFP 和其他 avFPs 的生色大小相同, 不同的原则必须在扩大的 DsRed 和 asFP595 染色体的范围内产生发射波长。108 nm 的深刻斯托克斯转移归因于 GdFP 生色的独特结构, 揭示了 autofluorescent 蛋白设计中的一个新的物理原理。初步计算 (如62中所报告) 表明, GdFP 的激发态生色的偶极矩大于基态, 与 ECFP 的各自值形成对比。而 GdFP 的偶极矩从 s0状态的 ~ 3 d (德拜) 增加到 s1中的 ~ 15 d, ECFP 生色的变化相当温和 (从 ~ 4 d 到 ~ 6 d)。因此, GdFP 的独特的金黄色荧光是由生色内的大量分子内电荷转移引起的, 它增加了可能的中介结构的多样性 (参见图 5), 允许共振稳定。这减少了排放发生的能量水平。由于偶极矩在励磁作用下的深刻变化, 分子内电荷分离是生色环境静电势变化的主要原因。生色激发后, 周围蛋白质基质依次调整到电荷分布的变化。随后的结构弛豫降低了激发生色的能级, 因为它的电荷传递特性将荧光光谱转移到红色。出于同样的原因, 由于大的斯托克斯移位和 radiationless 过程的提高率, GdFP 的荧光量子产量比 ECFP33减少。
ECFP 和 EGFP 的高量子产量和小斯托克斯转移通常归因于生色的刚性蛋白质环境, 从而降低了自由度, 从而使内部转换有利于激发态的辐射松弛。66. 因此, 更严格的嵌入染色体与剩余蛋白质基质的耦合的分子设计可以作为一个指南, 以产生更远的红色转移的 GFP 衍生物高荧光量子产量。因此, 为了进一步的工程方法, 以产生红移 autofluorescent 蛋白, 扩大的π电子系统和刚性生色结构与弱耦合的蛋白质环境是非常可取的。这种修改也可以直接引入 GFP 为基础的染色体或在生色附近放置所需的 ncAAs。
Disclosures
作者声明他们没有竞争的财政利益。
Acknowledgments
这项工作得到了德国研究基金会 ("统一的概念在催化) T.F. 和附注, 并由联邦教育和科学部 (BMBF 项目" 热休克 2020 ", WIMIplus 项目 SynTUBio) 支持 F。
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| 4-aminoindole | Sigma-Aldrich | 525022 | |
| acetonitrile | VWR | HiPerSolv CHROMANORM ULTRA for LC-MS, 83642 | LC-MS grade required |
| agar-agar | Carl Roth | 5210 | |
| ammonium molybdate ((NH4)2MoO4) | Sigma-Aldrich | 277908 | |
| ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| ampicillin sodium salt | Carl Roth | K029 | |
| biotin | Sigma-Aldrich | B4501 | |
| bromophenol blue | Sigma-Aldrich | B0126 | |
| calcium chloride (CaCl2) | Sigma-Aldrich | C5670 | |
| colloidal silica | Sigma-Aldrich | Ludox HS-40, 420816 | |
| Coomassie Brillant Blue R 250 | Carl Roth | 3862 | |
| copper sulfate (CuSO4) | Carl Roth | CP86.1 | |
| D-glucose | Carl Roth | 6780 | |
| di-sodium hydrogen phosphate (Na2HPO4) | Carl Roth | X987 | |
| di-potassium hydrogen phosphate (K2HPO4) | Carl Roth | P749.1 | |
| 1,4-dithiothreitol (DTT) | Carl Roth | 6908 | |
| DNase I | Sigma-Aldrich | D5025 | |
| ethanol | Carl Roth | 9065.1 | |
| formic acid | VWR | HiPerSolv CHROMANORM for LC-MS, 84865 | LC-MS grade required |
| glycerol | Carl Roth | 3783 | |
| imidazole | Carl Roth | X998 | |
| indole | Sigma-Aldrich | I3408 | |
| iron(II) chloride (FeCl2) | Sigma-Aldrich | 380024 | |
| isopropanol | Carl Roth | AE73.1 | |
| isopropyl β-D-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I6758 | |
| lysozyme | Sigma-Aldrich | L6876 | |
| magnesium chloride (MgCl2) | Carl Roth | KK36.1 | |
| magnesium sulfate (MgSO4) | Carl Roth | 8283.2 | |
| manganese chloride (MnCl2) | Sigma-Aldrich | 63535 | |
| β-mercaptoethanol | Carl Roth | 4227.3 | |
| potassium chloride (KCl) | Carl Roth | 6781.3 | |
| potassium dihydrogen phosphate (KH2PO4) | Sigma-Aldrich | P5655 | |
| RNase A | Carl Roth | 7156 | |
| sodium chloride (NaCl) | Carl Roth | P029 | |
| sodium dihydrogen phosphate (NaH2PO4) | Carl Roth | T879 | |
| sodium dodecyl sulphate (NaC12H25SO4) | Carl Roth | 0183 | |
| thiamine | Sigma-Aldrich | T4625 | |
| Tris(hydroxymethyl)-aminomethane (Tris) | Carl Roth | 5429 | |
| Tris hydrochloride (Tris-HCl) | Sigma-Aldrich | 857645 | |
| tryptone | Carl Roth | 8952 | |
| yeast extract | Carl Roth | 2363 | |
| zinc chloride (ZnCl2) | Sigma-Aldrich | 229997 | |
| Name | Company | Catalog Number | Comments |
| amino acids | |||
| L-alanine | Sigma-Aldrich | A7627 | |
| L-arginine | Sigma-Aldrich | A5006 | |
| L-asparagine | Sigma-Aldrich | A8381 | |
| L-aspartic acid | Sigma-Aldrich | A0884 | |
| L-cysteine | Sigma-Aldrich | C7352 | |
| L-glutamic acid | Sigma-Aldrich | G2128 | |
| L-glutamine | Sigma-Aldrich | G3126 | |
| L-glycine | Sigma-Aldrich | G7126 | |
| L-histidine | Sigma-Aldrich | H8000 | |
| L-isoleucine | Sigma-Aldrich | I2752 | |
| L-leucine | Sigma-Aldrich | L8000 | |
| L-lysine | Sigma-Aldrich | L5501 | |
| L-methionine | Sigma-Aldrich | M9625 | |
| L-proline | Sigma-Aldrich | P0380 | |
| L-phenylalanine | Sigma-Aldrich | P2126 | |
| L-serine | Sigma-Aldrich | S4500 | |
| L-threonine | Sigma-Aldrich | T8625 | |
| L-tryptophan | Sigma-Aldrich | T0254 | |
| L-tyrosine | Sigma-Aldrich | T3754 | |
| L-valine | Sigma-Aldrich | V0500 | |
| Name | Company | Catalog Number | Comments |
| Lab materials | |||
| 0.45 µm syringe filter with PVDF membrane | Carl Roth | CCY1.1 | |
| 1.5 mL microcentrifuge tubes | Eppendorf | 30120086 | |
| conical polystyrene (Falcon) tubes, 50 mL | Fisher Scientific | 14-432-22 | |
| Luer-Lock syringe 5 mL | Carl Roth | EP96.1 | |
| dialysis membrane, Molecular Weight Cut-Off (MWCO) 5,000 | Spectrum Medical Industries | Spectra/Por MWCO 5000 dialysis membrane, 133198 | |
| Immobilized Metal ion Affinity Chromatography (IMAC) column 1 mL, Ni-NTA | Macherey Nagel | Protino series, 745410.5 | |
| petri dishes (polystyrene, sterile) | Carl Roth | TA19 | |
| pQE-80L plasmid vector | Qiagen | no longer available | replaced by N-terminus pQE Vector set Cat No./ID: 32915 |
| protein extraction reagent BugBuster | EMB Millipore | 70921-4 | |
| round-bottom polystyrene tubes, 14 mL | Fisher Scientific | Corning Falcon, 14-959-1B | |
| Trp-auxotrophic E. coli strain | ATCC | ATCC 49980 | Bridges BA et al., Chem Biol Interact., 1972, 5(2):77-84; see main text for alternatives |
| Name | Company | Catalog Number | Comments |
| Mass Spectrometry equipment | |||
| mass spectrometer for LC-ESI-TOF-MS | Agilent | Agilent 6530 Accurate-Mass QTOF | coupled with Infinity LC system |
| mass spectrometry data analysis software | Agilent | MassHunter Qualitative Analysis software v. B.06.00 | |
| High-Performance Liquid Chromatography (HPLC) column for LC-ESI-TOF-MS | Sigma-Aldrich | Supelco Discovery BIO Wide Pore C5 HPLC column, 3 µm particle size, 10 cm x 2.1 mm | |
| HPLC autosampler vials 1.5 mL | Sigma-Aldrich | Supelco 854165 | with conical 0.1 mL glass inserts, screw caps and septa |
| Name | Company | Catalog Number | Comments |
| General equipment | |||
| benchtop centrifuge for 1.5 mL Eppendorf tubes | Eppendorf | 5427 R | |
| cooling centrifuge for 50 mL Falcon tubes | Eppendorf | 5810 R | |
| high pressure microfluidizer for bacterial cell disruption | Microfluidics | LM series with “Z” type chamber | |
| peristaltic pump for LC | GE Healthcare | P-1 | |
| Fast Protein Liquid Chromatography (FPLC) system | GE Healthcare | ÄKTA pure 25 L | |
| orbital shaker for bacterial cultivation | Infors HT | Minitron | |
| UV/Vis spectrophotometer | Biochrom | ULTROSPEC 2100 | |
| ultrasonic homogenizer for bacterial cell disruption | Omnilab | Bandelin SONOPULS HD 3200, 5650182 | with MS72 sonifier tip |
| Name | Company | Catalog Number | Comments |
| Fluorescence spectroscopy equipment | |||
| ps-pulsed laser 470 nm | Picoquant GmbH | PDL-470 | |
| time- and wavelength-correlated single photon counting (TWSPC) acquisition software | Picoquant GmbH | SymPhoTime 64 | |
| time- and wavelength-correlated single photon counting (TWSPC) detector | Picoquant GmbH | PML-16C | 16 spectral channels, to be selected by grating settings |
| single photon counting software | Picoquant GmbH | SPCM 9.75 | |
| global fitting software | Picoquant GmbH | SPC2Glo(R) | |
| fluorescence decay data analysis software | Picoquant GmbH | FluoFit program | |
| data analysis software | OriginLab Inc. | Origin 9.2 | |
| neutral density filter set | Schott | NG1 to NG11 | (400 - 650 nm, transmission 50 %, 20%, 10 %, 5 %) |
| 488 nm long-pass emission filter | AHF Analysentechnik | AHF-488 | |
| quartz cuvette | Thorlabs GmbH | CV10Q1400 | 1 cm pathlength |
References
- Shimomura, O., Johnson, F. H., Saiga, Y. Extraction, Purification and Properties of Aequorin, a Bioluminescent Protein from the Luminous Hydromedusan, Aequorea. J Cell Compar Physl. 59 (3), 223-239 (1962).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W. W., Prasher, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Andresen, M., et al. Structure and mechanism of the reversible photoswitch of a fluorescent protein. P Natl Acad Sci USA. 102 (37), 13070-13074 (2005).
- Andresen, M., et al. Structural basis for reversible photoswitching in Dronpa. P Natl Acad Sci USA. 104 (32), 13005-13009 (2007).
- Brakemann, T., et al. A reversibly photoswitchable GFP-like protein with fluorescence excitation decoupled from switching. Nat Biotechnol. 29 (10), 942-947 (2011).
- Kremers, G. -J., Gilbert, S. G., Cranfill, P. J., Davidson, M. W., Piston, D. W. Fluorescent proteins at a glance. J Cell Sci. 124 (Pt 2), 157-160 (2011).
- Shimomura, O. Structure of the chromophore of aequorea 0. shimomura green fluorescent protein. FEBS Lett. 104 (2), 220-222 (1979).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N. G., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Shcherbo, D., et al. Bright far-red fluorescent protein for whole-body imaging. Nat Methods. 4 (9), 741-746 (2007).
- Shcherbakova, D. M., Subach, O. M., Verkhusha, V. V. Red fluorescent proteins: advanced imaging applications and future design. Angew Chem Int Edit. 51 (43), 10724-10738 (2012).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Curr Protein Pept Sc. 9 (4), 338-369 (2008).
- Wang, L., Xie, J., Deniz, A. A., Schultz, P. G. Unnatural amino acid mutagenesis of green fluorescent protein. J Org Chem. 68 (1), 174-176 (2003).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Sharma, N., Furter, R., Kast, P., Tirrell, D. A. Efficient introduction of aryl bromide functionality into proteins in vivo. FEBS Lett. 467 (1), 37-40 (2000).
- Liu, C. C., Schultz, P. G. Adding new chemistries to the genetic code. Annu Rev Biochem. 79, 413-444 (2010).
- Twine, S. M., Murphy, L., Phillips, R. S., Callis, P., Cash, M. T., Szabo, A. G. The Photophysical Properties of 6-Azaindole. J Phys Chem B. 107 (2), 637-645 (2003).
- Lepthien, S., Hoesl, M. G., Merkel, L., Budisa, N. Azatryptophans endow proteins with intrinsic blue fluorescence. P Natl Acad Sci USA. 105 (42), 16095-16100 (2008).
- Budisa, N., et al. Probing the role of tryptophans in Aequorea victoria green fluorescent proteins with an expanded genetic code. Biol Chem. 385 (2), 191-202 (2004).
- Ross, J. B., et al. Spectral enhancement of proteins: biological incorporation and fluorescence characterization of 5-hydroxytryptophan in bacteriophage lambda cI repressor. P Natl Acad Sci USA. 89 (24), 12023-12027 (1992).
- Soumillion, P., Jespers, L., Vervoort, J., Fastrez, J. Biosynthetic incorporation of 7-azatryptophan into the phage lambda lysozyme: Estimation of tryptophan accessibility, effect on enzymatic activity and protein stability. Protein Eng Des Sel. 8 (5), 451-456 (1995).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Bridges, B. A., Mottershead, R. P., Rothwell, M. A., Green, M. H. L. Repair-deficient bacterial strains suitable for mutagenicity screening: tests with the fungicide captain. Chem Biol Interact. 5 (2), 77-84 (1972).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: The Heat Shock Method. J Vis Exp. , (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: Electroporation. J Vis Exp. , (2017).
- Grigorenko, B. L., Krylov, A. I., Nemukhin, A. V. Molecular modeling clarifies the mechanism of chromophore maturation in the green fluorescent protein. J Am Chem Soc. , (2017).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Spectrophotometer. J Vis Exp. , (2017).
- Goedhart, J., et al. Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93%. Nat Commun. 3, 751 (2012).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J Bacteriol. 119 (3), 736-747 (1974).
- Hörnsten, E. G. On culturing Escherichia coli on a mineral salts medium during anaerobic conditions. Bioprocess Eng. 12 (3), 157-162 (1995).
- Davis, B. D. The Isolation of Biochemically Deficient Mutants of Bacteria by Means of Penicillin. P Natl Acad Sci USA. 35 (1), 1-10 (1949).
- Sambrook, J., Russell, D. W. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY, USA. (2001).
- Wang, Y. -S., et al. The de novo engineering of pyrrolysyl-tRNA synthetase for genetic incorporation of L-phenylalanine and its derivatives. Mol Biosyst. 7 (3), 714-717 (2011).
- Bae, J. H., et al. Expansion of the genetic code enables design of a novel "gold" class of green fluorescent proteins. J Mol Biol. 328 (5), 1071-1081 (2003).
- JoVE Science Education Database. Dialysis: Diffusion Based Separation. J Vis Exp. , Cambridge, MA. (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Separating Protein with SDS-PAGE. J Vis Exp. , (2017).
- Petrásek, Z., et al. Excitation energy transfer from phycobiliprotein to chlorophyll d in intact cells of Acaryochloris marina studied by time- and wavelength-resolved fluorescence spectroscopy. Photoch Photobio Sci. 4 (12), 1016-1022 (2005).
- Kolber, Z. S., Barkley, M. D. Comparison of approaches to the instrumental response function in fluorescence decay measurements. Anal Biochem. 152 (1), 6-21 (1986).
- Pelet, S., Previte, M. J. R., Laiho, L. H., So, P. T. C. A fast global fitting algorithm for fluorescence lifetime imaging microscopy based on image segmentation. Biophys J. 87 (4), 2807-2817 (2004).
- Loefroth, J. E. Time-resolved emission spectra, decay-associated spectra, and species-associated spectra. J Phys Chem. 90 (6), 1160-1168 (1986).
- Hartman, M. C. T., Josephson, K., Lin, C. -W., Szostak, J. W. An expanded set of amino acid analogs for the ribosomal translation of unnatural peptides. PLoS One. 2 (10), e972 (2007).
- Budisa, N., et al. Global replacement of tryptophan with aminotryptophans generates non-invasive protein-based optical pH sensors. Angew Chem Int Edit. 41 (21), 4066-4069 (2002).
- Ma, Y., Biava, H., Contestabile, R., Budisa, N., di Salvo, M. L. Coupling bioorthogonal chemistries with artificial metabolism: intracellular biosynthesis of azidohomoalanine and its incorporation into recombinant proteins. Molecules. 19 (1), 1004-1022 (2014).
- Teramoto, H., Kojima, K. Incorporation of Methionine Analogues Into Bombyx mori Silk Fibroin for Click Modifications. Macromol Biosci. 15 (5), 719-727 (2015).
- Deal, R. B., Henikoff, J. G., Henikoff, S. Genome-wide kinetics of nucleosome turnover determined by metabolic labeling of histones. Science. 328 (5982), 1161-1164 (2010).
- Hinz, F. I., Dieterich, D. C., Tirrell, D. A., Schuman, E. M. Non-canonical amino acid labeling in vivo to visualize and affinity purify newly synthesized proteins in larval zebrafish. ACS Chem Neurosci. 3 (1), 40-49 (2012).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat Neurosci. 13 (7), 897-905 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). P Natl Acad Sci USA. 103 (25), 9482-9487 (2006).
- Glenn, W. S., et al. Bioorthogonal Noncanonical Amino Acid Tagging (BONCAT) Enables Time-Resolved Analysis of Protein Synthesis in Native Plant Tissue. Plant Physiol. 173 (3), 1543-1553 (2017).
- Zhou, L., et al. Incorporation of tryptophan analogues into the lantibiotic nisin. Amino Acids. 48 (5), 1309-1318 (2016).
- Acevedo-Rocha, C. G., Budisa, N. Xenomicrobiology: a roadmap for genetic code engineering. Microb Biotechnol. 9 (5), 666-676 (2016).
- Agostini, F., Völler, J. -S., Koksch, B., Acevedo-Rocha, C. G., Kubyshkin, V., Budisa, N. Biocatalysis with Unnatural Amino Acids: Enzymology Meets Xenobiology. Angew Chem Int Edit. 56 (33), 9680-9703 (2017).
- Bacher, J. M., Ellington, A. D. Selection and characterization of Escherichia coli variants capable of growth on an otherwise toxic tryptophan analogue. J Bacteriol. 183 (18), 5414-5425 (2001).
- Wong, J. T. Membership mutation of the genetic code: loss of fitness by tryptophan. Pc Natl Acad Sci USA. 80 (20), 6303-6306 (1983).
- Hoesl, M. G., et al. Chemical Evolution of a Bacterial Proteome. Angew Chem Int Edit. 54 (34), 10030-10034 (2015).
- Italia, J. S., et al. An orthogonalized platform for genetic code expansion in both bacteria and eukaryotes. Nat Chem Biol. 13 (4), 446-450 (2017).
- Völler, J. -S., Thi To, T. M., Biava, H., Koksch, B., Budisa, N. Global substitution of hemeproteins with noncanonical amino acids in Escherichia coli with intact cofactor maturation machinery. Enzyme Microb Tech. 106, 55-59 (2017).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Völler, J. -S., Budisa, N. Coupling genetic code expansion and metabolic engineering for synthetic cells. Curr Opin Biotech. 48, 1-7 (2017).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr Opin Chem Biol. 14 (6), 774-780 (2010).
- Somsen, O. J., van Grondelle, R., van Amerongen, H. Spectral broadening of interacting pigments: polarized absorption by photosynthetic proteins. Biophys J. 71 (4), 1934-1951 (1996).
- Kurschus, F. C., Pal, P. P., Bäumler, P., Jenne, D. E., Wiltschi, B., Budisa, N. Gold fluorescent annexin A5 as a novel apoptosis detection tool. Cytom Part A. 75 (7), 626-633 (2009).
- Lepthien, S., Wiltschi, B., Bolic, B., Budisa, N. In vivo engineering of proteins with nitrogen-containing tryptophan analogs. Appl Microbiol Biot. 73 (4), 740-754 (2006).
- Wachter, R. M., Elsliger, M. -A., Kallio, K., Hanson, G. T., Remington, S. J. Structural basis of spectral shifts in the yellow-emission variants of green fluorescent protein. Structure. 6 (10), 1267-1277 (1998).
- Verkhusha, V. V., Lukyanov, K. A. The molecular properties and applications of Anthozoa fluorescent proteins and chromoproteins. Nat Biotechnol. 22 (3), 289-296 (2004).
- Martynov, V. I., Savitsky, A. P., Martynova, N. Y., Savitsky, P. A., Lukyanov, K. A., Lukyanov, S. A. Alternative cyclization in GFP-like proteins family. The formation and structure of the chromophore of a purple chromoprotein from Anemonia sulcata. J Biol Chem. 276 (24), 21012-21016 (2001).
- Piatkevich, K. D., Malashkevich, V. N., Morozova, K. S., Nemkovich, N. A., Almo, S. C., Verkhusha, V. V. Extended Stokes shift in fluorescent proteins: chromophore-protein interactions in a near-infrared TagRFP675 variant. Sci Rep. 3 (1), 1847 (2013).



