Summary
Syntetisk biologi giver mulighed for manipulation af proteiner med hidtil uset egenskaber ved hjælp af co-translational indsættelse af ikke-kanoniske aminosyrer. Vi præsenteres her, hvordan en spektralt rød-forskudt variant af en normal god landbrugspraksis-type fluorophore med romanen fluorescens spektroskopiske egenskaber, betegnes "guld" fluorescerende proteiner (GdFP), er produceret i E. coli via selektiv pres indarbejdelse (SPI).
Abstract
Fluorescerende proteiner er grundlæggende instrumenter for biovidenskab, navnlig til fluorescens mikroskopi af levende celler. Mens vildtype og manipuleret varianter af den grønne fluorescerende proteiner fra Aequorea victoria (avGFP) samt homologs fra andre arter allerede dækker store dele af den optiske spektrum, forbliver et spektrale hul i regionen nær-infrarødt for der findes ikke som avGFP-baseret fluorophores. Rød-forskudt fluorescerende proteiner (FP) varianter ville væsentligt udvide toolkit for spektral urtåge af flere molekylære arter, men naturligt forekommende rød-forskudt FPs afledt af koraller eller søanemoner har lavere fluorescens kvanteudbytte og ringere foto-stabilitet i forhold til avGFP varianterne. Yderligere manipulation og eventuel udvidelse af farvelegemes konjugeret system mod far-red spektrale regionen er også begrænset af repertoire af 20 kanoniske aminosyrer ordineret af den genetiske kode. For at overvinde disse begrænsninger, kan syntetisk biologi opnå yderligere spektrale rød-shifting via indsættelse af ikke-kanoniske aminosyrer i den farvelegeme triade. Vi beskriver anvendelsen af SPI til ingeniør avGFP varianter med romanen spektrale egenskaber. Protein udtryk er udført i en tryptophan-auxotrophic E. coli stamme og ved at supplere vækst medier med passende indol prækursorer. Inde i cellerne, er disse prækursorer konverteres til den tilsvarende tryptophan analoger og indarbejdet i proteiner af de ribosomale maskiner som svar på UGG kodon. Udskiftning af Trp-66 i den forbedret "cyan" variant af avGFP (ECFP) af en elektron-donere 4-aminotryptophan resulterer i GdFP byder på en 108 nm Stokes Skift og en maksimal stærkt rød-forskudt emission (574 nm), mens er termodynamisk mere stabile end forgængeren ECFP. Rest-specifikke indarbejdelse af den ikke-kanoniske aminosyre er analyseret af massespektrometri. De spektroskopiske egenskaber af GdFP er karakteriseret ved tidsopløst fluorescens spektroskopi som en af de værdifulde anvendelser af genetisk kodet FPs i biovidenskab.
Introduction
Siden opdagelsen af den grønne fluorescerende proteiner i vandmand Aequorea victoria (avGFP) i 19621 og den første Heterolog ekspression i 19942 i andre eukaryote celler, er fluorescerende proteiner af familien normal god landbrugspraksis blevet værdifulde værktøjer og mål inden for biovidenskab. Omfattende genetiske og molekylære engineering inkluderet justering af artsspecifikke codon skik, acceleration af folde, forbedret modning, øgede lysstyrke, forebyggelse af oligomerisering og skræddersy spektrale og fotokemiske egenskaber herunder evnen til at reversibelt photoswitch3,4,5,6. Normal god landbrugspraksis skylder sin fluorescens fra sin 4-(p- hydroxybenzylidene) imidazolidin-5-en (HBDI) farvelegeme. Sidstnævnte er autocatalytically dannet fra den såkaldte farvelegeme triade af aminosyrer (Ser-65/Tyr-66/Gly-67 i avGFP) efter dannelsen af en yderligere kovalente bånd inden for peptid rygraden under indflydelse af Molekylær ilt7. Den resonantly stabiliseret konjugeret system interagerer dynamisk med sin molekylære miljø, giver mulighed for absorption i det synlige spektrum og karakteristiske grønne fluorescens af disse proteiner.
Inden for den farvelegeme triade er tilstedeværelsen af en aromatisk aminosyre obligatorisk. Standard aminosyre repertoire omfatter dog kun fire aromatiske restkoncentrationer (hans, Phe, Trp og Tyr). Dette begrænser konventionelle mutagenese tilgange for at opnå betydeligt mere rød-forskudt avGFP varianter i forhold til de mest rød-forskudt naturlige FPs DsRed8 fra Discosoma striata coralimorphs eller mKate/mNeptune9 fra søanemone Entacmaea quadricolor. Derfor, den far-red og nær-infrarøde del af den optiske spektrum over 600 nm er sparsomt omfattet af normal god landbrugspraksis varianter. Dette er naturligvis en alvorlig begrænsning for fluorescens mikroskopiske tilgange, der kræver spektral demultiplexing af flere arter, fluorophore på samme tid. For eksempel, lang bølgelængde markører er også nødvendigt at gøre brug af den lave absorption regime af hudvæv mellem 700-1000 nm i indstillinger for dybe væv imaging10.
Fluorescerende proteiner fra avGFP er opdelt i flere klasser baseret på spektroskopiske egenskaber og kemisk karakter af deres lysopfangende11. Med sin treklang Ser-65/Tyr-66/Gly-67, wild-type farvelegeme eksisterer som et afbalancerede blanding mellem formen neutral, phenol (λmax = 395 nm, ε = 21.000 M-1cm-1) og formen anioniske phenolate (λmax = 475 nm, ε = 7,100 M -1cm-1), og emission spektrum udstiller en enkelt peak på 508 nm. Hydroxylgruppe Ser-65 er af afgørende betydning, som det donerer en H-obligation til Glu-222 i farvelegeme nærhed (afstand: 3.7 Å), som fremmer ionisering af denne carboxylat. Klasse jeg er karakteriseret ved en anioniske phenolate farvelegeme, som i EGFP (Phe-64-Leu/Ser-65-Thr; λmax = 488 nm, ε = 35,600 M-1cm-1, λem = 509 nm). På grund af Ser-65-Thr(Ala,Gly) substitution, 395 nm excitation toppen af formen neutral phenol er undertrykt og 470-475 nm toppen af de anioniske phenolate er fem til seks gange forbedret og flyttet til 490 nm. Klasse II består af proteiner med en neutral phenol farvelegeme, som i safir-normal god landbrugspraksis. Her, Thr-203-Ile substitution næsten helt undertrykker 475 nm excitation, forlader kun peak på 399 nm. Da de anioniske farvelegeme ikke kan være korrekt solvated, er sin neutrale form begunstiget. Klasse III omfatter de "gule" fluorescerende varianter (EYFP; Ser-65-Gly/Val-68-leu/ser-72-ALA/thr-203-tyr; Λmax ε = 514 nm, ε = 84, 600 M-1cm-1, λem = 527 nm) med π-stacking samspil af en aromatisk sidekæde og phenolate, som er fremkaldt af Thr-203-His(Trp,Phe,Tyr) udskiftninger, som fører til op til 20 nm rød-forskudt emission maxima (Thr-203-Tyr). Yderligere substitution (Localisation-69-Lys) resulterer i en anden 1-2 nm rød Skift til 529 nm, den mest rød-forskudt avGFP variant kendt11. Udveksling af phenol for en indol (Tyr-66-Trp) skaber klasse IV, som i den cyan-fluorescerende ECFP (Ser-65-Thr/Tyr-66-Trp; λmax1 = 434 nm, ε = 24.800 M-1cm-1, λmax2 = 452 nm, ε = 23,600 M-1cm-1 ; Λem1 = 477 nm, λem2 = 504 nm). Overnatning omfangsrig indol er sandsynligvis aktiveret af andre, kompenserende mutationer. ECFP excitations- og maxima falder inbetween dem af proteiner med neutral eller anioniske lysopfangende. Klasse V proteiner havnen en imidazol i stedet for phenol (Tyr-66-hans), fx., blå-fluorescerende proteiner som EBFP. Klasse VI er produceret af en phenol-til-phenyl exchange favorisere formen neutral farvelegeme udelukkende, som dermed fører til de mest blå-skiftet excitations- og peak positioner (360 nm og 442 nm, henholdsvis).
Klassisk site-directed mutagenese er særligt velegnet til produktion af romanen avGFP farvelegeme varianter af permutation af 65-67 tripeptid og interagerende restkoncentrationer i rammen af de 20 kanoniske aminosyrer. Disse muligheder kan udvides yderligere, når ikke-kanoniske varianter af aromatiske aminosyrer er indført under ribosomale protein syntese12. I princippet er der to måder at opnå dette. Den første strategi er afhængig af underlaget tolerance af protein oversættelse maskiner, især af aminoacyl-tRNA synthetases (aaRSs) mod relaterede aminosyre analoger. At opnå dette med høj effektivitet, auxotrophic E. coli udtryk stammer er ansat som er i stand til at syntetisere den tilsvarende naturlige aminosyre. Dette giver mulighed for udskiftning af sidstnævnte ved at tilføje passende ikke-kanoniske aminosyrer (ncAAs) eller forstadier dertil næringssubstratet. Denne strategi, også kendt som selektiv pres indarbejdelse (SPI)13,14, gør det muligt for rest-specifikke udskiftninger, hvilket resultere i globale indarbejdelse af ncAA. Den anden strategi bruger stop codon suppressor tRNAer, som er anklaget for ncAA af manipuleret aaRS enzymer. Dette resulterer i readthrough af i-frame stop kodon og tillader lokationsspecifikke ncAA indarbejdelse. Derfor fører denne metode af stop-codon suppression (SCS) til en udvidelse af den genetiske kode15. Via mutagenese placeres en stop codon i target-gen på det ønskede sted. I princippet kan SPI også bruges til at oprette rekombinant peptider og proteiner forsynet med en unik ncAA installation, at sjældne kanoniske aminosyrer som markedsøkonomisk eller Trp er valgt til Ressourceerstatning. Med Trp, SPI tilgange har vist sig at arbejde med et stort udvalg af analoger herunder 4 - F-, 5 - F - og 6-F-Trp, 7-aza-Trp, 4-OH - og 5-OH-Trp, samt 4- og 5-NH2- Trp eller endda β (thienopyrrolyl) alanin derivater16 ,17,18,19,20. SPI kunne således meget fordelagtige for udskiftning af aromatiske aminosyrer af normal god landbrugspraksis lysopfangende af ikke-kanoniske varianter at undersøge muligheden for at yderligere skræddersy spectra og Stokes skift af disse FPs. Med hensyn til alle ændringer i protein sekvens, skal kompatibilitet med FP folde og farvelegeme modning afprøves eksperimentelt.
I dette arbejde udnytte vi klasse IV ECFP21, der bærer i stedet for vildtype avGFP Tyr, en Trp rest i sine farvelegeme triade. Ved hjælp af SPI, er denne Trp-66 (og Trp-57, kun andre Trp rester i ECFP) erstattet af 4-amino-Trp. Tilstedeværelsen af den elektron-donere amino gruppe af 4-amino-Trp inden for farvelegeme favoriserer resonans stabilisering af et langt rød-forskudt exciterede tilstand proton overførsel (ESPT) udstyret med en 108 nm Stokes Skift. Denne "guld" fluorescerende proteiner (GdFP) udgør varianten med de største rød skift af fluorescens maksimale (574 nm) blandt alle avGFP-afledte proteiner. Vi beskrive metoden for GdFP protein produktion af SPI og give protokollerne for den obligatoriske analyse af de resulterende modificerede proteiner af masse spektroskopi. Yderligere, viser vi hvordan GdFP kan udnyttes og analyseret i tidsopløst fluorescens spektroskopi tilgange.
Protocol
1. forvandling af Trp-auxotrophic E. coli
- Omdanne kemisk eller electrocompetent celler (50 µL) af en Trp-auxotrophic E. coli -stamme, fx. ATCC 49980 (WP2, mutant stammer fra E. coli stamme B/R22), med 1 µL af en 1 ng/µL vandig opløsning af pQE - 80 L His6-ECFP plasmid med heat shock eller elektroporation, hhv. Henvises til JoVE videnskab uddannelse Database23,24 for detaljer.
NOTE: Udtrykket vektor pQE - 80L His6-ECFP koder en N-terminalt 6 x hans markeret ECFP21 drevet af en bakteriel T5 promotor med lac operatør. Det bærer yderligere en AmpR udvalg markør og en colE1 oprindelse af replikering (pQE - 80 L vektor rygraden rækkefølge kan findes på: https://www.qiagen.com/mx/resources/resourcedetail?id=c3b71572-4d82-4671-a79b-96357fe926d1&lang=en & autoSuggest = true). Den teoretiske molekylvægt af vildtypeprotein His6-ECFG (efter farvelegeme modning25) er 28303.92 Da. Oversatte target protein sekvens er som følger (hans-tag understreget, vektor-afledte sekvenser i fed): MRGSJANNICKGSMVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTWGVQCFSRYPDHMK
QHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYISHNVYITADKQKNGIKANFKIRHNIEDGS
VQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK. - Plade transformerede celler på LB-agar plader (tabel 1) suppleret med 10 g/L glukose, 100 µg/mL ampicillin og inkuberes plader ved 37 ° C natten over.
2. rekombinante Protein udtryk
-
Overnight kultur af E. coli ATCC 49980 pQE - 80 L His6-ECFP
- Forberede 5 mL af LB medium (tabel 1, suppleret med 10 g/L glukose, 100 µg/mL ampicillin) i en steril 14 mL polystyren kultur rør for aerob vækst og podes med en enkeltvis koloni fra en agar plade med en steril pipette tip eller podning løkke.
Bemærk: Ved hjælp af kolonier fra frisk transformerede celler anbefales. Plader med bakteriel kolonier (fra trin 1.2.) kan opbevares ved 4 ° C i flere dage. - Inkuber celler ved 37 ° C i en orbitalryster på 200-250 rpm natten over.
- Forberede 5 mL af LB medium (tabel 1, suppleret med 10 g/L glukose, 100 µg/mL ampicillin) i en steril 14 mL polystyren kultur rør for aerob vækst og podes med en enkeltvis koloni fra en agar plade med en steril pipette tip eller podning løkke.
-
Udtryk for wild-type ECFP
- Podes 10 mL frisk LB medium (tabel 1, suppleret med 10 g/L glukose, 100 µg/mL ampicillin) med 100 µL af overnight kultur i en 100 mL Erlenmeyerkolbe. Inkuber i kolben ved 37 ° C i en orbitalryster på 200 rpm.
Bemærk: Du kan vælge dette trin kan udføres i 10 mL NMM19 medium (tabel 1) suppleret med 100 µg/L ampicillin og 0,5 mM L-tryptophan (alternativt indol kan bruges). - Måle den optisk tæthed på 600 nm (OD600) hvert 20 min. fortrinsvis måle celle tæthed ved at bestemme uddø på 600 nm (OD600) i et spektrofotometer ved hjælp af en kuvette med en lysvej på 1 cm. altid udføre en henvisning måling ved hjælp af det tilsvarende næringssubstratet. Fortynd prøver og bland prøverne godt for at få en måleværdi af 0.1-0.8, så beregne OD600 ved hjælp af fortyndingsfaktoren. For detaljer henvises til tidligere offentliggørelse 26.
- Ved ankomsten til en OD600 værdi på 0,5-0,8 (ca. 2-3 h efter podning), skal du tage prøve "før induktion" for SDS-PAGE (sodium dodecyl sulfat polyacrylamid gelelektroforese, trin 4).
- Fremkalde target protein udtryk ved at justere den flydende kultur til 0,5 mM IPTG (isopropyl β-D-1-thiogalactopyranoside, fra 1 M stamopløsning) og der inkuberes ved 30 ° C i en orbitalryster på 200 rpm i 4-8 timer.
Bemærk: Cyan fluorescerende proteiner er almindeligvis udtrykt ved temperaturer under 37 ° C27. - Tage prøve "efter udtryk" for SDS-PAGE (skridt 4.).
- Høste de bakterieceller ved centrifugering i 10 min på 5.000 x g og 4 ° C.
- Supernatanten ved dekantering og fryse celle pellets på-20 ° C eller-80 ° C indtil målet protein oprensning.
- Podes 10 mL frisk LB medium (tabel 1, suppleret med 10 g/L glukose, 100 µg/mL ampicillin) med 100 µL af overnight kultur i en 100 mL Erlenmeyerkolbe. Inkuber i kolben ved 37 ° C i en orbitalryster på 200 rpm.
-
SPI til fremstilling af GdFP
- Podes 10 mL af NMM19 medium (tabel 1) suppleret med 100 µg/mL ampicillin, 15 µM tryptophan og 10 µL af overnight kultur i en 100 mL Erlenmeyerkolbe og der inkuberes kultur kolben natten over ved 30 ° C i en orbitalryster på 200 rpm.
Bemærk: En række kemisk definerede medier for dyrkning af E. coli og SPI er tilgængelig. Ud over NMM bruges heri, MOPPER medium28, glucose-mineralske salte medium29, Davis minimal medium30, M9 minimal medium31eller GMML32 kan bruges. - Den næste dag, måle OD600 hvert 30 min, indtil værdien skifter kun mindre end 0,05 over 30 min. Plateau værdien bør være ca. 1.
Bemærk: Afvigelser af ± 0,3 enheder er acceptabel. Afhængig af bakteriel stamme og anvendte medium, muligvis den første tryptophan koncentration (trin. 2.3.1) justering. - Tage prøve "før induktion" for SDS-PAGE (skridt 4.).
- Høste de bakterieceller ved centrifugering i 10 min på 5.000 x g og 4 ° C. Supernatanten ved dekantering.
- Resuspend celler i 10 mL af NMM19 medium med 100 µg/mL ampicillin i en 100 mL Erlenmeyerkolbe og tilføje 4-amino-indol til en endelig koncentration på 1 mM med 50 mM stamopløsning. Fortsætte inkubation i 30 min. ved 30 ° C i en orbitalryster på 200 rpm.
Bemærk: Dette trin anbefales på grund af den lave kemiske stabilitet af ampicillin og sikrer cellulære optagelse af 4-amino-indol. - Fremkalde target protein udtryk ved at føje IPTG til en endelig koncentration på 0,5 mM med 1 M lager og inkuberes prøve natten over ved 30 ° C i en orbitalryster på 200 rpm.
Bemærk: Cyan fluorescerende proteiner er almindeligvis udtrykt ved temperaturer under 37 ° C27. - Den næste dag, måle OD600.
- Tage prøve "efter udtryk" for SDS-PAGE (skridt 4.).
- Høste de bakterieceller ved centrifugering i 10 min på 5.000 x g og 4 ° C og kassér supernatanten ved dekantering.
- I tilfælde sådant skib ikke blev brugt til centrifugering, overføre celle pellet ind i en 50 mL konisk polystyren rør ved hjælp af en spatel. Fryse celle pellet ved-20 ° C eller-80 ° C indtil målet protein oprensning.
- Podes 10 mL af NMM19 medium (tabel 1) suppleret med 100 µg/mL ampicillin, 15 µM tryptophan og 10 µL af overnight kultur i en 100 mL Erlenmeyerkolbe og der inkuberes kultur kolben natten over ved 30 ° C i en orbitalryster på 200 rpm.
3. mål Protein oprensning via immobiliseret Metal Ion affinitet kromatografi (IMAC)
-
Bakteriel celle lysis
- Tø celle pellet i isbad i 10-20 min.
- Resuspenderes celle i en 50 mL konisk polystyren reagensglas med 5 mL iskold binding buffer (tabel 1) på is.
- Tilføj 20 µL af 50 mg/mL Lysozym, 20 µL af 1 mg/mL DNase jeg 20 µL af 1 mg/mL RNase A. lukke røret, blandes forsigtigt ved at invertere 5 gange, og holde det på is i 30 min.
Bemærk: Delvis celle forstyrrelser opstår som katalyseret af lysozym. - Lyse celler af sonification ved hjælp af en ultralyd homogeniseringsapparat tip ved hjælp af tre cyklusser på 3 min. i en 15 mL polystyren tube køles af slush ice med 2 s af puls, 4 s af pause og 45% amplitude.
BEMÆRKNING: Alternativt, højtryk homogenisering kan anvendes, fx., 20 cyklusser på 14.000 psi. Om nødvendigt, fortyndes ved hjælp af bindende buffer til at nå den minimale instrument volumen. Desuden kan protein udvinding reagenser bruges til celle forstyrrelser. Se materialer tabel for eksempler. - Der centrifugeres prøven i 30 min. ved 15.000-18.000 x g, 4 ° C.
- Overføre supernatanten til en frisk rør og optegne den flydende volumen.
- Opløsningen filtreres gennem et 0,45 µm sprøjte filter ved hjælp af en 5 mL plastik Luer lock sprøjte og en polyvinylidene fluorid (PVDF) sprøjte filter.
- Tage prøve "lysate" for SDS-PAGE (skridt 4.).
- Resuspend celle debris pellet i ddH2O (tilsvarende mængde som tidligere lysate).
- Tage prøven "pille" for SDS-PAGE (skridt 4.).
-
IMAC rensning
- Bruge en 1 mL færdigpakkede eller selv pakket IMAC FPLC (hurtig protein væskekromatografi) kolonne ifølge producentens anvisninger. Bruge binding buffer (tabel 1) for kolonne ækvilibrering såvel som for trinnet vask, som følger efter den lysate celle er blevet anvendt til kolonnen.
- Indsamle og pool eluatet fraktioner med GdFP, der kan identificeres af synligt lys gylden farve.
Bemærk: Du kan eventuelt target proteinet kan være elueres ved hjælp af en lineær imidazol gradient (0-250 mM) ved hjælp af en automatiseret FPLC system. - Protein koncentrationen ved hjælp af litteratur værdi for udryddelse koefficient på 466 nm (ɛ466 nm = 23,700 M-1 cm-1)33 med eluering buffer som reference. For detaljer om proceduren, henvises til tidligere offentliggørelse26.
- Tag prøven "eluat" for SDS-PAGE, og brug 1-10 µg af protein pr. vognbane i tilfælde af Coomassie farvning.
Bemærk: SDS prøve beløb kan variere afhængigt af farvning metode og farvestof følsomhed. - Dialyze en alikvot af eluatet fraktioner mod dialyse buffer eller MS buffer ved hjælp af en membran med en molekylevægt på cutoff (MWCO) af 5.000-10.000. Forberede dialyse membran ifølge producentens anvisninger. Dialyze en 1 mL prøve tre gange mod 100 mL buffer i mindst 2 timer. Yderligere oplysninger om denne procedure, henvises til tidligere publikation34.
- Til opbevaring, fryse protein prøven i dialyse buffer ved-80 ° C.
Bemærk: Delprøver skal være stabil i mindst 6 måneder.
4. SDS-PAGE prøveforberedelse E. coli hele cellen ekstrakt
- Overføre en svarende til 1 mL af OD600 cellesuspension = 1 suspension (f.eks. 500 µL af OD600= 2) ind i en 1,5 mL microcentrifuge rør.
- Høste cellerne ved centrifugering i 10 min på 5000 x g, stuetemperatur. Supernatanten af pipettering.
- Tilsættes 80 µL af Hedeselskabet2O og 20 µL af 5 x SDS lastning farvestof buffer (tabel 1) til celle pellet og mix af pipettering.
- Denaturere cellerne ved opvarmning til 95 ° C i 5 min i et vand bad eller varme blok. Efterfølgende, cool prøver for stuetemperatur.
- Anvendes 10 µL til Coomassie farvede SDS-PAGE ifølge tidligere publikation35.
Bemærk: SDS prøve beløb kan variere afhængigt af farvning metode og farvestof følsomhed.
5. intakt Protein masse analyse af High-performance væskekromatografi (HPLC) koblet til Electrospray Ionisation Time of flight massespektrometri (LC-ESI-TOF-MS)
Bemærk: HPLC gradient, indstillinger og buffere kan variere afhængigt af adskillelse kolonne og instrument, der anvendes. Se materialer tabel for eksemplarisk udstyr.
- Bestemme proteinkoncentration fra en stikprøve dialysebehandling mod MS buffer som beskrevet ovenfor (trin 3.2.3.) ved hjælp af MS buffer (Se materialer tabel) som reference.
- Fortynd protein prøven til 0,1 mg/mL ved hjælp af MS buffer for en endelige mængden af 80 µL, blandes ved omhyggelig pipettering, opløsningen overføres til en MS autosampler hætteglas med glasindsats og lukker det med en fælles landbrugspolitik. Fjerne luftbobler ved at bladre i hætteglasset.
- Fyld en anden autosampler hætteglas uden glasindsats (buffer blank) med 1 mL af MS buffer.
- Tillad instrument til at varme op. Udføre instrument kalibrering. Sørg for, at der findes tilstrækkelige mængder af liquid chromatography-grade opløsningsmidler (> 100 mL).
- Programmere en lineær 20 min HPLC gradient fra 5% til 80% buffer en (0,1% myresyre i ddH2O), kombineret med buffer B (0,1% myresyre i acetonitril).
- Start HPLC på en strøm af 0,3 mL/min og vente, indtil kolonnen pres er stabil.
- Angive en autosampler Injektionsvolumen af 5 µL for LC-ESI-TOF-MS metoden, oprette en værklisten for en tom køre efterfulgt af en prøve køre og tildele de tilsvarende autosampler hætteglas positioner. Køre værklisten.
- Efter færdiggørelsen af worklist, åbne den genererede eksempeldatafil. Marker et område i den samlede ion aktuelle (TIC) plot for deconvolution og deconvolute MS spektrum ved hjælp af maksimal entropi deconvolution algoritme.
Bemærk: Afhængigt af forsøgsbetingelser, yderligere arter kan forekomme fra ikke-modnet FP eller buffer ion adukter.
6. fluorescens levetid målinger og henfald-associerede spektre (DAS) af GdFP
Bemærk: Instrumentation af tidsopløst fluorescens spektroskopi, henvises til Tabel af materialer for eksemplarisk udstyr. Absorbansen samt fluorescens excitation og emission spektre af fluorescerende proteiner kan også registreres ved hjælp af laboratoriet UV/synligt lys og fluorescens spektrofotometre.
-
Bølgelængde-løst fluorescens levetid måling af GdFP
- Forberede 2 mL af en 1 µM løsning af GdFP ved fortynding i PBS buffer (tabel 1) ved pH 7. Fylde løsningen i en 1 cm kvarts kuvette.
- Installere ps-pulserende 470 nm laser til prøven excitation og filteret 488 nm long-pass emission, og justere 600 L/mm rist af korreleret tid og bølgelængde enkelt foton tælle36 (TWCSPC) detektor for erhvervelse af ordningen bølgelængde 500-700 nm.
- Erhverve fluorescens emission med en tælle hastighed på omkring 200 x 103 fotoner/s, indtil omkring 103 tæller er akkumuleret i erhvervelse maksimalt fluorescens henfald kurver med single photon tælle software.
-
Måling af instrumentale svar funktion37 (IRF)
- Erstatte prøve kuvette med en 1 cm kvarts kuvette fyldt med 1 g/L kolloid silica (~ 220 m2/g) i PBS buffer pH-værdi på 7.
Bemærk: Silica suspension er udarbejdet ved hjælp af en 400 g/L vandig suspension. - Fjerne 488 nm long-pass emission filter og Indsæt grå filtre til at justere tælle hyppigheden af TWCSPC-detektor til under 100 x 103 tæller/s.
- Justere rist til erhvervelse af 470 nm fotoner i kanal 8 af 16-kanals TWCSPC detektor.
- Erhverve IRF, indtil omkring 10 x 103 tæller er akkumuleret i den maksimale emission.
- Konverter fluorescens henfald kurver og IRF til ASCII datafiler med globale montering38 program.
- Adfærd Global passer efter en model for en sum af tre eksponentiel komponenter med levetider som sammenkædet parametre.
- Plot henfald forbundet spektre (DAS) som amplitude distributioner af individuelle henfald komponenter i afhængighed af bølgelængde med data analyse software.
- Erstatte prøve kuvette med en 1 cm kvarts kuvette fyldt med 1 g/L kolloid silica (~ 220 m2/g) i PBS buffer pH-værdi på 7.
Representative Results
Ved hjælp af teknikken selektivt pres vedtægter, kan Trp-66 i den farvelegeme triade af ECFP (og Trp-57, kun andre Trp rester i ECFP) erstattes af 4-amino-Trp, derved skabe den rød-forskudt GdFP med særskilte spektrale egenskaber. Massespektrometri skal bruges til at demonstrere den ønskede støkiometriske integration af ikke-kanoniske aminosyren i protein, med resultaterne vist i figur 1. Bagefter kan vi levere data fra mikroskopi samt UV-Vis absorption spektroskopi og steady-state og og bølgelængde-tidsopløst fluorescens spektroskopi til at karakterisere egenskaber af GdFP fluorophore med fokus på pH afhængighed af den spektre.
For at bekræfte udveksling af de to Trp restkoncentrationer i ECFP af 4-amino-Trp, udføres massespektrometrisk analyse. Figur 1 viser et repræsentativt deconvoluted ESI-MS spektrum af GdFP. Mens vildtype ECFP har en beregnet protein massen af 28,283.9 Da efter farvelegeme modning, er den tilsvarende masse af GdFP 28,313.9 Da. GdFP deconvoluted ESI-MS spektrum udstiller en største masse peak på 28,314.1 ± 0,1 Da, som afviger fra den teoretiske værdi af under 10 ppm. At være inden for området typiske nøjagtighed for denne type analyse25, dette bekræfter indarbejdelse af ncAA via SPI (eksperimentelle værdi for wild-type ECFP: 28,283.7 Da).
Figur 2 viser Konfokal fluorescens imaging mikroskopi (CFIM) billeder af bakterieceller udtrykker ECFP, EGFP, EYFP og GdFP ved ophvirvling af bakterier i PBS buffer. Alle billeder blev erhvervet på et mikroskop udstyret med en UV mål og laser excitation på om den samme energi for hver prøve.
Figur 3A viser en overlejring af CFIM billeder af E. coli bakterier at udtrykke forskellige FPs herunder GdFP, altid overvåges med meget lignende excitation energi (bølgelængder som i figur 2). Figur 3B viser farvelegeme strukturer af FP varianter vist. Med hensyn til lysstyrke af GdFP i forhold til ECFP (Fluorescens quantum udbytte φ = 0,4), EGFP (φ = 0,6) og EYFP (φ = 0,6) det er vigtigt at bemærke, at for GdFP, en bredere erhvervelse række fluorescens lys (30 nm) blev brugt i modsætning til 20 nm anvendes til alle andre spe Cies, for at justere intensiteten af billeder til lignende værdier. Med en lidt lavere extinction koefficient og en reduceret kvanteudbytte som følge af unikke photophysical egenskaber sammenlignet lysstyrken af GdFP lavere med andre FPs vist.
Absorptionsspektrum af ECFP (figur 3 c) har to karakteristiske maxima på 434 nm og 452 nm. Derimod GdFP er kendetegnet ved en bred rød-forskudt absorption band med maksimum på 466 nm. Absorption af EGFP er yderligere rød-forskudt til 488 nm. Men på grund af den meget større Stokes skift af GdFP (108 nm) i forhold til ECFP (41 nm) og EGFP (20 nm), emission spektrum af GdFP er den mest rød-forskudt af alle tre normal god landbrugspraksis derivater undersøgt her (figur 3D). Mens fluorescens emission af ECFP viser to karakteristiske maxima på 475 nm og 505 nm, EGFP har en bred vigtigste emission band toppede ved 508 nm (λmax) med en lille skulder ved 540 nm. Fluorescens af GdFP vises på omkring 565 nm (λMaks.) (Figur 3D). Dens emission spektrum indeholder et lille bidrag af wild-type ECFP, der kan også ses som en lille skulder på 475 nm. Denne lille ECFP fraktion er syntetiseret før induktion under proceduren SPI som beskrevet33.
Figur 3E viser pH-afhængige ændringer i absorptionsspektrum af GdFP. For en pH ændringen fra 8-5, den maksimale emission skifter lidt til røde og en lille udvidelse af absorption band er observeret. Reduktion af absorption amplitude er imidlertid kun omkring 10% mellem pH 8 og pH 5, der angiver, at grundtilstand egenskaberne af GdFP farvelegeme er meget svagt modificeret af pH.
Tiden løst fluorescens emission overvåget ved optælling af single photon er vist i figur 4. Henfald kurver overvåges i de spektrale kanaler centreret på 550 nm og 600 nm (figur 4A) udviser en lidt hurtigere fluorescens forfald på 600 nm i forhold til forfald ved 550 nm. Resultaterne af en global pasform af fluorescens henfald kurver med to eksponentiel komponenter resulterer i to spektralt skelnes fluorescens henfald komponenter med tiden konstanter af 1.0 ns og 3.3 ns (figur 4 c og D).
Fluorescens emission af GdFP afhænger stærkt af pH, som det er typisk for mange fluorescerende proteiner varianter af familien normal god landbrugspraksis. Figur 4B sammenligner fluorescens emission af GdFP mellem pH 5 og pH 8, som klart viser et fald i fluorescens-intensiteten ved lavere pH, mens spectral Karakteristik forblive konstant.
Decay-associerede spektre (DAS)39 af GdFP (fig. 4 c og D) er karakteriseret ved to særskilte emission bands. Bidrag af de langsomme 3.3 ns komponent er mere udtalt i området kort bølgelængde omkring 550 nm (60%) med mindre bidrag af komponenten hurtigere (40%). På 600 nm, begge komponenter har om den samme amplitude. Ved et skift fra pH 7 (figur 4 c) til pH 6 (figur 4D), spectral Karakteristik af DAS ændre næppe og tid konstanterne fra den globale montering rutine er også den samme (nøjagtigheden af DAS tid konstanter er om ± 0,15 ns). Men forskellen i de absolutte amplituder af de to DAS-komponenterne er klart synlige, som fuldt regnskab for reduceret fluorescens emission amplitude ved samme pH skift i figur 4B.
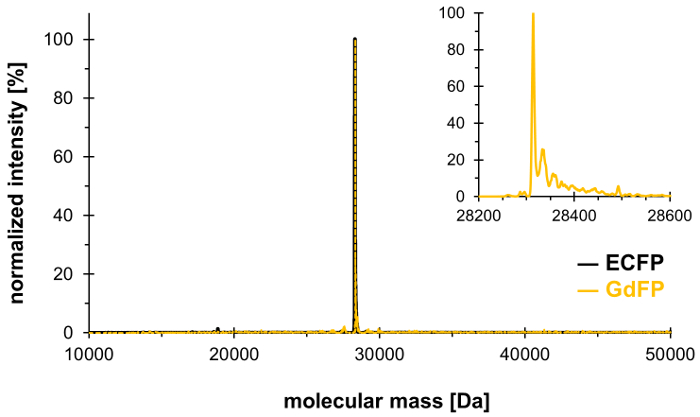
Figur 1: repræsentative deconvoluted ESI-MS spektrum af GdFP. ESI-MS spektret af GdFP (guld farve, forstørret plot vist som inset) viser en vigtigste peak på 28314.1 Da (beregnet værdi 28313.9 Da). Spektrum for wild-type ECFP er vist i sort. Venligst klik her for at se en større version af dette tal.

Figur 2: Konfokal Fluorescens mikroskopi billeder fra bakteriel befolkninger udtrykker forskellige FPs. Følgende indstillinger for bølgelængde blev brugt til billede erhvervelse: ECFP (λex = 457 nm, afsløring: 461-480 nm), EGFP (λex = 488 nm, afsløring: 495-515 nm), GDFP (λex = 476 nm, afsløring: 560-590 nm), EYFP (λex = 514 nm, afsløring: 520-530 nm). Venligst klik her for at se en større version af dette tal.

Figur 3: spektrale egenskaber af GdFP. (A) CFIM billede af en blanding af bakterieceller udtrykker ECFP, EGFP og GdFP efter ophvirvling af bakterier i PBS buffer. (B) farvelegeme strukturer af GdFP (med 4-amino-Trp i stedet for restkoncentrationer 66), forældrenes ECFP (med Trp på position 66) og EFGP (med Tyr på position 66). (C) sammenligningen af de normaliserede absorptionsspektre af GdFP, ECFP og EGFP, mens i (D), den normaliserede fluorescens emission spektrum af ECFP (excitation på 430 nm) er i forhold til fluorescens emission spektre af EGFP og GdFP (begge glade ved 450 nm). (E) pH-afhængighed af Absorptionsspektra (normaliseret absorption på 280 nm). Venligst klik her for at se en større version af dette tal.

Figur 4: tidsopløst fluorescens af GdFP. (A) Fluorescens henfald af GdFP overvåges af tid - og bølgelængde-løst enkelt foton tælle i de spektrale kanaler centreret på 550 nm og 600 nm (± 12,5 nm) efter excitation med 470 nm laserpulser. Funktionen instrumental svar (IRF) indeholder oplysninger om tidsopløsning af den anvendte setup. (B) Variation af emission spektrum af GdFP afhænger af pH (excitation på 460 nm). (C, D) Decay-associerede spektre (DAS) af GdFP ved pH 7 (C) og pH 6 (U) bestemmes efter deconvolution af og bølgelængde-tidsopløst fluorescens henfalder og globale montering af henfalder i alle kanaler af en global sæt af to eksponentielle funktioner med sammenkædede tid konstanter. Venligst klik her for at se en større version af dette tal.

Figur 5: strukturer af intramolekylære afgift overførsel af ECFP (sort) og GdFP (guld) lysopfangende. Stigning i størrelsen af det farvelegeme system af god elektron donor af en amino gruppe som en del af ncAA muliggør dannelsen af mere mesomeric strukturer at opnå resonans stabilisering af den eksiterede tilstand. Forbindelsespunkter til FP stillads er vist som halvcirkler. Venligst klik her for at se en større version af dette tal.
| Stamopløsningen | koncentration, opløsningsmidler | Bemærk | |
| 20% D-glukose | 200 g/L D-glucose i ddH2O | sterilisere ved filtrering gennem et 0,45 µm pore størrelse sprøjte filter | |
| indol | 50 mM i isopropanol | ||
| 4-amino-indol | 50 mM i 20% ethanol (20 mL ethanol i en endelige rumfang på 100 mL fyldt med ddH2O) | ||
| IPTG | 1 M i ddH2O | ||
| L-tryptophan | 15 mM opløst i ddH2O ved hjælp af 1 M HCl (tilføje HCl dråbevis under omrøring indtil pulveret er dissoved) | ||
| Lysozym | 50 mg/mL i ddH2O | ||
| DNase jeg | 1 mg/mL i ddH2O | ||
| RNase A | 1 mg/mL i ddH2O | ||
| Amp100 | 100 mg/mL ampicillin i ddH2O | ||
| natrium-dodecylsulfate (SDS) | 200 g/L i ddH2O | ||
| ammonium sulfat ((NH4)2SO4) | 1 M i ddH2O | steriliseres ved autoklavering | |
| kalium dihydrogen fosfat (KH2PO4) | 1 M i ddH2O | steriliseres ved autoklavering | |
| di-kalium hydrogen fosfat (K2HPO4) | 1 M i ddH2O | steriliseres ved autoklavering | |
| magnesium-sulfat (MgSO4) | 1 M i ddH2O | steriliseres ved autoklavering | |
| D-glukose | 1 M i ddH2O | sterilisere ved filtrering gennem et 0,45 µm pore størrelse sprøjte filter | |
| natriumchlorid (NaCl) | 5 M i ddH2O | steriliseres ved autoklavering | |
| calciumchlorid (CaCl2) | 1 g/L | sterilisere ved filtrering gennem et 0,45 µm pore størrelse sprøjte filter | |
| Aluminiumacetat chlorid (FeCl2) | 1 g/L | sterilisere ved filtrering gennem et 0,45 µm pore størrelse sprøjte filter | |
| thiamin | 10 g/L | sterilisere ved filtrering gennem et 0,45 µm pore størrelse sprøjte filter | |
| Biotin | 10 g/L | sterilisere ved filtrering gennem et 0,45 µm pore størrelse sprøjte filter | |
| sporstoffer mix | kobber sulfat (CuSO4), zink chlorid (ZnCl2), mangan chlorid (frihedsbevægelse2), ammoniummolybdat ((NH4)2MoO4); hver 1 mg/L i ddH2O | sterilisere ved filtrering gennem et 0,45 µm pore størrelse sprøjte filter | |
| 19 aminosyrer mix | 1.) opløses 0,5 g L-phenylalanin og 0,5 g L-tyrosin i 100 ml ddH2O med dråbevis tilsætning af 1 M HCl under omrøring indtil pulveret er opløst. | ||
| 2.) vejes ud 0,5 g af hver af de resterende L-amino syrer (undtagen L-tryptophan). Bland med 22 mL fo 1 M KH2PO4 og 48 mL 1 M K2HPO4. Tilføje ddH2O til omkring 800 mL. Rør indtil opløsningen er klar. | |||
| 3.) Tilføj den opløste L-phenylalanin og L-tyrosin fra trin 1.) og justere lydstyrken til 1 L med ddH2O. | |||
| 4.) Sterilize aminosyre blanding af vakuum filtrering med en flaske top filterenhed. | |||
| Buffere og medier | Sammensætning/forberedelse | ||
| SDS lastning farvestof buffer, 5 x koncentreret | 0,25 M Tris pH 6,8, 50% v/v glycerol, 0,25% w/v bromphenol blå, 0,5 M didhiothreitol (DTT, alternativt 5% β-mercaptoethanol), 10% w/v natrium-dodecylsulfate (SDS) | ||
| binding buffer | 50 mM natrium dihydrogenphosphate (NaH2PO4), 500 mM NaCl, 10 mM imidazol, pH 8 | ||
| eluering buffer | 50 mM natrium dihydrogenphosphate (NaH2PO4), 500 mM NaCl, 250 mM imidazol, pH 8 | ||
| dialyse buffer | 50 mM natrium dihydrogenphosphate (NaH2PO4), 150 mM NaCl, 100 mL/L glycerol, pH 8 | ||
| MS buffer | 10 mM Tris-HCl, pH 8 | ||
| nye minimal medie som indeholder 19 L-amino syrer undtagen L-tryptophan (NMM19) | Bland alle stock løsninger for at opnå følgende endelige koncentrationer: 7.5 mM (NH4)2SO4, 1,7 mM NaCl, 22 mM KH2PO4, 50 mM K2HPO4, 1 mM MgSO4, 20 mM D-glucose, 50 mg/L 19 aminosyrer mix, 1 µg/L CaCl2, 1 µg/L FeCl2, 10 µg/L thiamin, 10 mg/L biotin, 0,01 mg/L sporstoffer mix | ||
| LB medium | Sammensætning: 10 g/L trypton, 5 g/L gær extract, 10 g/L NaCl, pH 7,0 i ddH2O | ||
| Forberedelse: | |||
| 1.) vejes ud af 50 g trypton, 25 g gær extract, 5 g NaCl i en 1 L glasflaske. | |||
| 2.) Tilføj ddH2O op til ~ 800 mL og opløse komponenter under omrøring. | |||
| 3.) måle pH og justeres til pH 7 ved dråbevis tilsætning af 1 M HCl eller 1 M NaOH, om nødvendigt. Tilføj ddH2O op til 1 L. | |||
| 4.) Sterilize ved autoklavering, check for volumen tab bagefter og tilføje sterile ddH2O at kompensere hvis nødvendigt. Opbevares ved 4 ° C indtil brug. | |||
| LB agar plader | Sammensætning: 10 g/L trypton, 5 g/L gær extract, 10 g/L NaCl, 15 g/L agar-agar, pH 7,0 i ddH2O | ||
| Forberedelse: | |||
| 1.) vejes ud af 50 g trypton, 25 g gær extract, 5 g NaCl, 7,5 g agar-agar i en 1 L glasflaske. | |||
| 2.) Tilføj ddH2O op til 500 mL og opløse komponenter under omrøring. | |||
| 3.) måle pH og justeres til pH 7 ved dråbevis tilsætning af 1 M HCl eller 1 M NaOH, om nødvendigt. Tilføj ddH2O op til 1 L. | |||
| 4.) Sterilize ved autoklavering, check for volumen tab bagefter og tilføje sterile ddH2O at kompensere, hvis nødvendigt. (Bemærk: LB agar kan opbevares ved 4 ° C indtil brug for forberedelse af LB agar plader. Omhyggeligt smelte størknede agar ved hjælp af en mikroovn) | |||
| 5.) når løsningen er stadig varm (30-40 ° C), tilføjes en endelig koncentration på 100 µg/mL ampicillin | |||
| 6.) hældes ca. 15 mL væske fra skridt 5.) i en steril 10 cm petriskål under sterile forhold. Da agaren er størknet, kan pladerne opbevares i 1 uge på 4 ° C indtil brug. | |||
| fosfatbufferet saltopløsning (PBS) | Sammensætning: 137 mM NaCl, 2,7 mM KCl, 10 mM Na2HPO4, 1,8 mM KH2PO4, 1 mM CaCl2, 0,5 mM MgCl2, pH 7. Steriliseres ved autoklavering eller filtrering. | ||
Tabel 1: Materiel løsning og buffer.
Discussion
For at opnå meget høje ncAA indarbejdelse effektivitetsgevinster, metoden auxotrophy-baseret SPI er baseret på brugen af metabolisk manipuleret værtsceller, som ikke er i stand til at syntetisere det tilsvarende naturlige modstykke til ncAA. For E. colier sådan stammer let tilgængelige. Selv samtidige inddragelse af flere ncAAs i den samme protein er muligt ved hjælp af multiauxotrophic stammer. Rest-specifikke tilstand af udskiftning og de kemiske repertoire at være begrænset til lignende kemiske analoger kan ses som ulemper. Ikke desto mindre kan et stort antal protein varianter produceres som naturlige bakterielle oversættelse apparater tolererer talrige aminosyre analoger. For eksempel, kunne mere end 50 ncAAs blive indarbejdet i proteiner ved hjælp af in vitro- oversættelse, tegner sig for omkring 73% af alle kodon af den genetiske kode til rådighed for omplacering40. Derudover kan SPI også giver mulighed for effektiv flere lokationer mærkning af target protein41. I princippet, SPI metode er ikke begrænset til E. coli, men kan arbejde i alle andre værter og for hver af de kanoniske 20 aminosyrer, forudsat at auxotrophic stammer og definerede dyrkning medier er tilgængelige. For eksempel, er to methionin analoger, azidohomoalanine (Aha) og homopropargylglycine (hypothalamus), kommercielt tilgængelige og anvendes til mærkning af proteiner og proteomes i forskellige organismer. Aha kan derudover produceres intracellulært og efterfølgende indarbejdet i protein42. Denne ncAA er specielt velegnet til bioorthogonal bøjninger såsom Klik kemi som udviklet af Tirrel og kolleger: For eksempel i plante væv fra Arabidopsis thaliana, Bombyx mori larver43, Drosophila celler44, larve zebrafisk45 samt pattedyrceller herunder neuroner46, proteiner kan mærkes med Aha47,48. På samme måde, Trp analoger blev med held indarbejdet i antimikrobielle peptider i Trp-auxotrophic Lactococcus lactis stammer49. SPI er også nyttigt for feltet af Xenobiology50,51, som udforsker alternativer til de grundlæggende kemiske make-up af liv. For eksempel, blev baseret på tidligere værker på E. coli52 og B. subtilis53, en E. coli -stamme udviklet for nylig af en evolutionær strategi med selektiv pres for at udnytte thienopyrrole i stedet for indol, hvilket resulterer i proteom-wide substitution af tryptophan af thienopyrrole-alanin i den genetiske kode54. Generelt, de kanoniske aminosyre Trp, der er kodet af en enkelt triplet (UGG), præsenterer et lovende mål for protein engineering på grund af de rige facetter af indol kemi, som tilbyder mange kemiske variationer. For nylig, og som et alternativ til SPI-baseret iblanding, rapporteret en roman SCS platform i stand til at indarbejde Trp analoger site-specifically i både bakterier og eukaryote værter har været55. Dette udvider yderligere værktøjskasse i vivo ncAA-baseret protein engineering, herunder ændring af spektrale egenskaber.
Foruden brug af auxotrophic udtryk værter kræver SPI-protokollen strenge gæring betingelser, både hvad angår målet udtryk timing og sammensætning af medium for at nå høje ncAA indarbejdelse effektivitet og målet protein udbytte 56. dyrkning er foretaget ved hjælp af kemisk definerede minimal medier, som hovedsagelig indeholder udover store salte kilder til kvælstof (ammoniumsalt) og kulstof (D-glucose), vitaminer og sporstoffer. Selvom det ikke strengt nødvendigt i mangel af yderligere auxotrophies, de resterende aminosyrer (20 -Nielsen, hvis n aminosyrer er at blive erstattet) er ofte føjet til fremme bakterievækst57. Under en indledende vækstfase før induktion af target protein udtryk føjes de n kanoniske aminosyrer erstattes begrænse koncentrationer. Cellulære vækst fortsætter indtil de målrettede essentielle aminosyrer er udtømte, som eksperimentelt angivet ved en stationær OD600. Efterfølgende erstattes næringssubstratet af frisk medium, der mangler forarmet aminosyren og indeholder ncAA i rigelige koncentrationer. For de ribosomale indlemmelse af tryptophan analoger som vist i denne protokol, er en analog indol fodret, som bliver intracellulært konverteres til den tilsvarende tryptophan derivat af tryptophan syntase58. Næste, target protein udtryk er induceret. På dette stadium er cellerne tæt på slutningen af logaritmisk vækst, som en balance mellem cellen total antal og fitness. Da tilstedeværelse og indarbejdelse af den kanoniske amino ville føre til wild-type protein produktion, er det afgørende at sikre, at den essentielle aminosyre er fuldt opbrugt før induktion. Ligeledes er det obligatorisk at undersøge effektiviteten af ncAA indarbejdelse i target proteinet, almindeligvis af massespektrometri. I tilfælde af betydelig tilstedeværelse af den kanoniske aminosyre, dyrkning betingelser skal justeres, fxved at ændre koncentrationen af den væsentlige amino acid(s) for den indledende vækstfase eller varigheden af sidstnævnte. I tilfælde af lav aaRS aktivitet mod ncAA, kan overekspression af endogene enzym eller co udtryk for en anden aaRS, som er mere aktive mod ncAA, være udført59.
Den kanoniske aminosyre Trp er udstyret med tre bemærkelsesværdige funktioner: (i) sin naturlige overflod i proteiner er lav; (ii) dens biofysiske og kemiske egenskaber er enestående (fx., det er normalt den dominerende oprindelsen af den iboende fluorescens af proteiner og peptider), og (iii) det bidrager til en lang række biokemiske samspil og funktioner, herunder Π-stabling, H-bonding og kation-π interaktioner. Alle disse funktioner er radikalt ændret ved Trp → 4-amino-Trp substitution GdFP. Beyond tvivlrådig, design af en "guld" klasse af avGFPs er et bemærkelsesværdigt eksempel for engineering skræddersyede autofluorescent proteiner. Med særskilte spektrale egenskaber, kan blive tunet FPs mod visse spektrale windows via mutagenese og ncAA indarbejdelse. I tilfælde af GdFP opnås dette ved en simpel kemiske udveksling H → NH2 i rammen af indol ring i ECFP farvelegeme treklang. Figur 5 viser virkningerne af ncAA indarbejdelse i farvelegeme. Indførelsen af elektron-donere gruppen med oprindelse fra 4-amino-indol (intracellulært konverteret til 4-amino-Trp) giver mulighed for en bred vifte af mesomeric strukturer, der kan forklare en stabiliseret exciterede tilstand. Hjælp, dens udvidede Stokes Skift og rød-forskudt fluorescens emission skyldes disse forskellige egenskaber af det udvidede konjugerede system. Som rapporteret tidligere, forbedret intramolekylære afgift overførsel inden for GdFP farvelegeme er i sagens natur følsomme pH (figur 4B) og ledsaget af en større ændring i dipolmoment mellem S0 jorden og S1 ophidset tilstand i forhold til ECFP33. Som alternativ elektron-donere grupper, kunne tryptophan analoger forsynet med en indol ring substitueret med hydroxy grupper bruges, som rapporteret i en sammenlignende undersøgelse med model protein barstar41.
Absorption og fluorescens-spektre af GdFP er udvidet i forhold til ECFP og EGFP (figur 3 c og D). Homogen udvidelse af absorption og fluorescens bands er normalt forårsaget af vibrationelle modes i farvelegeme og derudover af kobling af farvelegeme til yderligere vibrationelle tilstande i protein60. Kobling til den lokale protein miljø understøttes af afgifter er lokaliseret på farvelegeme. Som de strukturelle uensartethed af protein fører til lokale variationer af den vibronic spektrum, sådan kobling mellem de vibronic spektre af farvelegeme og resten af proteinet understøttes af afgift virksomhedsflytninger og mesomeric stater som anført i Figur 5. Denne kobling understøtter også den store Stokes Skift og reducerer nødvendigvis kvanteudbytte fluorescens. I forhold til andre rød-forskudt FPs udstiller GdFP endda forbedret protein stabilitet og en lav tendens til sammenlægning33,61,62. Det ikke kun adskiller sig i farve fra andre FP varianter men også udstiller et betydeligt øget thermostability og øget cooperative folde33. Fluorescens intensiteten er mindst 90% bevaret ved opvarmning til 60 ° C, mens ECFP Fluorescens er reduceret til ca. 30%. I proteiner bidrage ofte aromatiske aminosyrer til net af vekselvirkende sidekæder, der almindeligvis har en stabiliserende virkning på proteinets tertiære struktur. avGFP havne sådan en side kæde netværk, som består af farvelegeme selv, som godt som Phe-165, hans-148 og Tyr-145. Disse sidekæder er ikke kun helt stiv i GdFP struktur33, men vigtigst, de danner hydrofobe kontakter med farvelegeme. Det mest fremtrædende roman træk identificeret i GdFP er, at aminated farvelegeme er mere proksimalt for Phe-165. Denne interaktion er en funktion, der er ikke observeret i andre kendte avGFPs. Som de to restprodukter 3.2-4.5 Å apart, kan amino-aromatiske interaktioner findes også. Sammen med undersøgelsesområde-induceret resonans stabilisering af farvelegeme stabilisere disse sandsynligvis denne hydrofobe netværk af aminosyrer i en kooperativ måde. En mere effektiv intramolekylære afgift overførsel kan støttes af disse interaktioner i den eksiterede tilstand i forhold til grundtilstanden af farvelegeme, og det i det mindste delvis tegner sig for 108 nm Stokes Skift33,62 .
I rationelt design af fluorophore egenskaber forventes en stigning i størrelsen af delocalized π-systemet for at resultere i en rød-forskudt excitation bølgelængde. Denne tommelfingerregel er overholdt af rækken af aminosyrer i position 66 fører til neutral lysopfangende: Phe (λmax = 355 nm) < hans (λma x= 386 nm) < Tyr (λmax = 395 nm) < Trp (λmax = 436 nm)63. I naturen, er denne udvidelse af farvelegemes konjugeret system af π-bindinger opnået ved forskellige strategier. For DsRed fra Discosoma striata, er det udvidet med integration af en yderligere aminosyre, således at flytte λmax til 573 nm64. Farvelegeme af asFP595 (λmax = 595 nm) fra Anemonia sulcata blev udvidet med en imino gruppe, udvide sin π-systemet65. Da farvelegeme af GdFP og andre avFPs er af samme størrelse, skal et andet princip medføre en emission bølgelængde i rækken af udvidet DsRed og asFP595 lysopfangende. En dyb Stokes forskydning af 108 nm er tilskrevet forskellige strukturen af GdFP farvelegeme, der afslører en ny photophysical princippet i udformningen af autofluorescent proteiner. Foreløbige beregninger (som rapporteret i 62) har vist, at dipolmoment af ophidset tilstand farvelegeme af GdFP er væsentligt større end i grundtilstand, i modsætning til de respektive værdier af ECFP. Mens dipolmoment af GdFP øger fra ~ 3 D (Debye) i tilstanden S0 til ~ 15 D i S1, ændringen for ECFP farvelegeme var temmelig moderat (fra ~ 4 D til ~ 6 D). Således, den unikke gyldne fluorescens af GdFP er forårsaget af betydelige intramolekylære afgift overførsel inden for farvelegeme, hvilket øger vifte af mulige mesomeric strukturer (Se figur 5), der tillader for resonans stabilisering. Dette reducerer den energi niveau som emission opstår. Som følge af de dybtgående ændringer i dipolmoment ved excitation er intramolekylære afgift adskillelse den vigtigste årsag til ændringer i farvelegeme miljø elektrostatisk potentiale. Matrixen omkringliggende protein justerer til gengæld at ændringer i afgift fordelingen efter farvelegeme excitation. Den efterfølgende strukturelle afslapning sænker energi niveau af den glade farvelegeme, som skifter fluorescens spektret til rød på grund af sin afgift overførsel karakter. Af samme grund, som følge af den store Stokes Skift og forbedret satserne for radiationless processer, fluorescens quantum udbyttet af GdFP er reduceret i forhold til ECFP33.
Høj kvanteudbytte og små Stokes skift af ECFP og EGFP er normalt tilskrives en stiv protein miljø af farvelegeme, hvilket reducerer frihedsgrader og dermed interne konvertering til at favorisere den eksiterede tilstand radiative afslapning 66. derfor den molekylære design af mere stift indkapslet lysopfangende med reduceret kobling til de resterende protein matrix kan tjene som en guide til at producere længere rød-forskudt normal god landbrugspraksis derivater med høj fluorescens kvanteudbytte. Udvidelsen af ordningen med π-elektron og en stiv farvelegeme struktur med svag kobling til protein miljø er derfor for yderligere engineering metoder til at fremstille rød-forskudt autofluorescent proteiner, særdeles ønskeligt. Sådanne ændringer vil også kunne indføres, enten direkte i normal god landbrugspraksis-baserede lysopfangende eller af placering af ønskede ncAAs i farvelegeme nærhed.
Disclosures
Forfatterne erklærer, at de har ingen konkurrerende finansielle interesser.
Acknowledgments
Dette arbejde blev støttet af den tyske Forskningsfonds (Cluster of Excellence "Unifying begreber i katalyse) T.F. og NB og af Forbundsministeriet for uddannelse og videnskab (BMBF Program"HSP 2020", TU-WIMIplus projekt SynTUBio) til F.-J.S.
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| 4-aminoindole | Sigma-Aldrich | 525022 | |
| acetonitrile | VWR | HiPerSolv CHROMANORM ULTRA for LC-MS, 83642 | LC-MS grade required |
| agar-agar | Carl Roth | 5210 | |
| ammonium molybdate ((NH4)2MoO4) | Sigma-Aldrich | 277908 | |
| ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| ampicillin sodium salt | Carl Roth | K029 | |
| biotin | Sigma-Aldrich | B4501 | |
| bromophenol blue | Sigma-Aldrich | B0126 | |
| calcium chloride (CaCl2) | Sigma-Aldrich | C5670 | |
| colloidal silica | Sigma-Aldrich | Ludox HS-40, 420816 | |
| Coomassie Brillant Blue R 250 | Carl Roth | 3862 | |
| copper sulfate (CuSO4) | Carl Roth | CP86.1 | |
| D-glucose | Carl Roth | 6780 | |
| di-sodium hydrogen phosphate (Na2HPO4) | Carl Roth | X987 | |
| di-potassium hydrogen phosphate (K2HPO4) | Carl Roth | P749.1 | |
| 1,4-dithiothreitol (DTT) | Carl Roth | 6908 | |
| DNase I | Sigma-Aldrich | D5025 | |
| ethanol | Carl Roth | 9065.1 | |
| formic acid | VWR | HiPerSolv CHROMANORM for LC-MS, 84865 | LC-MS grade required |
| glycerol | Carl Roth | 3783 | |
| imidazole | Carl Roth | X998 | |
| indole | Sigma-Aldrich | I3408 | |
| iron(II) chloride (FeCl2) | Sigma-Aldrich | 380024 | |
| isopropanol | Carl Roth | AE73.1 | |
| isopropyl β-D-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I6758 | |
| lysozyme | Sigma-Aldrich | L6876 | |
| magnesium chloride (MgCl2) | Carl Roth | KK36.1 | |
| magnesium sulfate (MgSO4) | Carl Roth | 8283.2 | |
| manganese chloride (MnCl2) | Sigma-Aldrich | 63535 | |
| β-mercaptoethanol | Carl Roth | 4227.3 | |
| potassium chloride (KCl) | Carl Roth | 6781.3 | |
| potassium dihydrogen phosphate (KH2PO4) | Sigma-Aldrich | P5655 | |
| RNase A | Carl Roth | 7156 | |
| sodium chloride (NaCl) | Carl Roth | P029 | |
| sodium dihydrogen phosphate (NaH2PO4) | Carl Roth | T879 | |
| sodium dodecyl sulphate (NaC12H25SO4) | Carl Roth | 0183 | |
| thiamine | Sigma-Aldrich | T4625 | |
| Tris(hydroxymethyl)-aminomethane (Tris) | Carl Roth | 5429 | |
| Tris hydrochloride (Tris-HCl) | Sigma-Aldrich | 857645 | |
| tryptone | Carl Roth | 8952 | |
| yeast extract | Carl Roth | 2363 | |
| zinc chloride (ZnCl2) | Sigma-Aldrich | 229997 | |
| Name | Company | Catalog Number | Comments |
| amino acids | |||
| L-alanine | Sigma-Aldrich | A7627 | |
| L-arginine | Sigma-Aldrich | A5006 | |
| L-asparagine | Sigma-Aldrich | A8381 | |
| L-aspartic acid | Sigma-Aldrich | A0884 | |
| L-cysteine | Sigma-Aldrich | C7352 | |
| L-glutamic acid | Sigma-Aldrich | G2128 | |
| L-glutamine | Sigma-Aldrich | G3126 | |
| L-glycine | Sigma-Aldrich | G7126 | |
| L-histidine | Sigma-Aldrich | H8000 | |
| L-isoleucine | Sigma-Aldrich | I2752 | |
| L-leucine | Sigma-Aldrich | L8000 | |
| L-lysine | Sigma-Aldrich | L5501 | |
| L-methionine | Sigma-Aldrich | M9625 | |
| L-proline | Sigma-Aldrich | P0380 | |
| L-phenylalanine | Sigma-Aldrich | P2126 | |
| L-serine | Sigma-Aldrich | S4500 | |
| L-threonine | Sigma-Aldrich | T8625 | |
| L-tryptophan | Sigma-Aldrich | T0254 | |
| L-tyrosine | Sigma-Aldrich | T3754 | |
| L-valine | Sigma-Aldrich | V0500 | |
| Name | Company | Catalog Number | Comments |
| Lab materials | |||
| 0.45 µm syringe filter with PVDF membrane | Carl Roth | CCY1.1 | |
| 1.5 mL microcentrifuge tubes | Eppendorf | 30120086 | |
| conical polystyrene (Falcon) tubes, 50 mL | Fisher Scientific | 14-432-22 | |
| Luer-Lock syringe 5 mL | Carl Roth | EP96.1 | |
| dialysis membrane, Molecular Weight Cut-Off (MWCO) 5,000 | Spectrum Medical Industries | Spectra/Por MWCO 5000 dialysis membrane, 133198 | |
| Immobilized Metal ion Affinity Chromatography (IMAC) column 1 mL, Ni-NTA | Macherey Nagel | Protino series, 745410.5 | |
| petri dishes (polystyrene, sterile) | Carl Roth | TA19 | |
| pQE-80L plasmid vector | Qiagen | no longer available | replaced by N-terminus pQE Vector set Cat No./ID: 32915 |
| protein extraction reagent BugBuster | EMB Millipore | 70921-4 | |
| round-bottom polystyrene tubes, 14 mL | Fisher Scientific | Corning Falcon, 14-959-1B | |
| Trp-auxotrophic E. coli strain | ATCC | ATCC 49980 | Bridges BA et al., Chem Biol Interact., 1972, 5(2):77-84; see main text for alternatives |
| Name | Company | Catalog Number | Comments |
| Mass Spectrometry equipment | |||
| mass spectrometer for LC-ESI-TOF-MS | Agilent | Agilent 6530 Accurate-Mass QTOF | coupled with Infinity LC system |
| mass spectrometry data analysis software | Agilent | MassHunter Qualitative Analysis software v. B.06.00 | |
| High-Performance Liquid Chromatography (HPLC) column for LC-ESI-TOF-MS | Sigma-Aldrich | Supelco Discovery BIO Wide Pore C5 HPLC column, 3 µm particle size, 10 cm x 2.1 mm | |
| HPLC autosampler vials 1.5 mL | Sigma-Aldrich | Supelco 854165 | with conical 0.1 mL glass inserts, screw caps and septa |
| Name | Company | Catalog Number | Comments |
| General equipment | |||
| benchtop centrifuge for 1.5 mL Eppendorf tubes | Eppendorf | 5427 R | |
| cooling centrifuge for 50 mL Falcon tubes | Eppendorf | 5810 R | |
| high pressure microfluidizer for bacterial cell disruption | Microfluidics | LM series with “Z” type chamber | |
| peristaltic pump for LC | GE Healthcare | P-1 | |
| Fast Protein Liquid Chromatography (FPLC) system | GE Healthcare | ÄKTA pure 25 L | |
| orbital shaker for bacterial cultivation | Infors HT | Minitron | |
| UV/Vis spectrophotometer | Biochrom | ULTROSPEC 2100 | |
| ultrasonic homogenizer for bacterial cell disruption | Omnilab | Bandelin SONOPULS HD 3200, 5650182 | with MS72 sonifier tip |
| Name | Company | Catalog Number | Comments |
| Fluorescence spectroscopy equipment | |||
| ps-pulsed laser 470 nm | Picoquant GmbH | PDL-470 | |
| time- and wavelength-correlated single photon counting (TWSPC) acquisition software | Picoquant GmbH | SymPhoTime 64 | |
| time- and wavelength-correlated single photon counting (TWSPC) detector | Picoquant GmbH | PML-16C | 16 spectral channels, to be selected by grating settings |
| single photon counting software | Picoquant GmbH | SPCM 9.75 | |
| global fitting software | Picoquant GmbH | SPC2Glo(R) | |
| fluorescence decay data analysis software | Picoquant GmbH | FluoFit program | |
| data analysis software | OriginLab Inc. | Origin 9.2 | |
| neutral density filter set | Schott | NG1 to NG11 | (400 - 650 nm, transmission 50 %, 20%, 10 %, 5 %) |
| 488 nm long-pass emission filter | AHF Analysentechnik | AHF-488 | |
| quartz cuvette | Thorlabs GmbH | CV10Q1400 | 1 cm pathlength |
References
- Shimomura, O., Johnson, F. H., Saiga, Y. Extraction, Purification and Properties of Aequorin, a Bioluminescent Protein from the Luminous Hydromedusan, Aequorea. J Cell Compar Physl. 59 (3), 223-239 (1962).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W. W., Prasher, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Andresen, M., et al. Structure and mechanism of the reversible photoswitch of a fluorescent protein. P Natl Acad Sci USA. 102 (37), 13070-13074 (2005).
- Andresen, M., et al. Structural basis for reversible photoswitching in Dronpa. P Natl Acad Sci USA. 104 (32), 13005-13009 (2007).
- Brakemann, T., et al. A reversibly photoswitchable GFP-like protein with fluorescence excitation decoupled from switching. Nat Biotechnol. 29 (10), 942-947 (2011).
- Kremers, G. -J., Gilbert, S. G., Cranfill, P. J., Davidson, M. W., Piston, D. W. Fluorescent proteins at a glance. J Cell Sci. 124 (Pt 2), 157-160 (2011).
- Shimomura, O. Structure of the chromophore of aequorea 0. shimomura green fluorescent protein. FEBS Lett. 104 (2), 220-222 (1979).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N. G., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Shcherbo, D., et al. Bright far-red fluorescent protein for whole-body imaging. Nat Methods. 4 (9), 741-746 (2007).
- Shcherbakova, D. M., Subach, O. M., Verkhusha, V. V. Red fluorescent proteins: advanced imaging applications and future design. Angew Chem Int Edit. 51 (43), 10724-10738 (2012).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Curr Protein Pept Sc. 9 (4), 338-369 (2008).
- Wang, L., Xie, J., Deniz, A. A., Schultz, P. G. Unnatural amino acid mutagenesis of green fluorescent protein. J Org Chem. 68 (1), 174-176 (2003).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Sharma, N., Furter, R., Kast, P., Tirrell, D. A. Efficient introduction of aryl bromide functionality into proteins in vivo. FEBS Lett. 467 (1), 37-40 (2000).
- Liu, C. C., Schultz, P. G. Adding new chemistries to the genetic code. Annu Rev Biochem. 79, 413-444 (2010).
- Twine, S. M., Murphy, L., Phillips, R. S., Callis, P., Cash, M. T., Szabo, A. G. The Photophysical Properties of 6-Azaindole. J Phys Chem B. 107 (2), 637-645 (2003).
- Lepthien, S., Hoesl, M. G., Merkel, L., Budisa, N. Azatryptophans endow proteins with intrinsic blue fluorescence. P Natl Acad Sci USA. 105 (42), 16095-16100 (2008).
- Budisa, N., et al. Probing the role of tryptophans in Aequorea victoria green fluorescent proteins with an expanded genetic code. Biol Chem. 385 (2), 191-202 (2004).
- Ross, J. B., et al. Spectral enhancement of proteins: biological incorporation and fluorescence characterization of 5-hydroxytryptophan in bacteriophage lambda cI repressor. P Natl Acad Sci USA. 89 (24), 12023-12027 (1992).
- Soumillion, P., Jespers, L., Vervoort, J., Fastrez, J. Biosynthetic incorporation of 7-azatryptophan into the phage lambda lysozyme: Estimation of tryptophan accessibility, effect on enzymatic activity and protein stability. Protein Eng Des Sel. 8 (5), 451-456 (1995).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Bridges, B. A., Mottershead, R. P., Rothwell, M. A., Green, M. H. L. Repair-deficient bacterial strains suitable for mutagenicity screening: tests with the fungicide captain. Chem Biol Interact. 5 (2), 77-84 (1972).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: The Heat Shock Method. J Vis Exp. , (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: Electroporation. J Vis Exp. , (2017).
- Grigorenko, B. L., Krylov, A. I., Nemukhin, A. V. Molecular modeling clarifies the mechanism of chromophore maturation in the green fluorescent protein. J Am Chem Soc. , (2017).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Spectrophotometer. J Vis Exp. , (2017).
- Goedhart, J., et al. Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93%. Nat Commun. 3, 751 (2012).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J Bacteriol. 119 (3), 736-747 (1974).
- Hörnsten, E. G. On culturing Escherichia coli on a mineral salts medium during anaerobic conditions. Bioprocess Eng. 12 (3), 157-162 (1995).
- Davis, B. D. The Isolation of Biochemically Deficient Mutants of Bacteria by Means of Penicillin. P Natl Acad Sci USA. 35 (1), 1-10 (1949).
- Sambrook, J., Russell, D. W. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY, USA. (2001).
- Wang, Y. -S., et al. The de novo engineering of pyrrolysyl-tRNA synthetase for genetic incorporation of L-phenylalanine and its derivatives. Mol Biosyst. 7 (3), 714-717 (2011).
- Bae, J. H., et al. Expansion of the genetic code enables design of a novel "gold" class of green fluorescent proteins. J Mol Biol. 328 (5), 1071-1081 (2003).
- JoVE Science Education Database. Dialysis: Diffusion Based Separation. J Vis Exp. , Cambridge, MA. (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Separating Protein with SDS-PAGE. J Vis Exp. , (2017).
- Petrásek, Z., et al. Excitation energy transfer from phycobiliprotein to chlorophyll d in intact cells of Acaryochloris marina studied by time- and wavelength-resolved fluorescence spectroscopy. Photoch Photobio Sci. 4 (12), 1016-1022 (2005).
- Kolber, Z. S., Barkley, M. D. Comparison of approaches to the instrumental response function in fluorescence decay measurements. Anal Biochem. 152 (1), 6-21 (1986).
- Pelet, S., Previte, M. J. R., Laiho, L. H., So, P. T. C. A fast global fitting algorithm for fluorescence lifetime imaging microscopy based on image segmentation. Biophys J. 87 (4), 2807-2817 (2004).
- Loefroth, J. E. Time-resolved emission spectra, decay-associated spectra, and species-associated spectra. J Phys Chem. 90 (6), 1160-1168 (1986).
- Hartman, M. C. T., Josephson, K., Lin, C. -W., Szostak, J. W. An expanded set of amino acid analogs for the ribosomal translation of unnatural peptides. PLoS One. 2 (10), e972 (2007).
- Budisa, N., et al. Global replacement of tryptophan with aminotryptophans generates non-invasive protein-based optical pH sensors. Angew Chem Int Edit. 41 (21), 4066-4069 (2002).
- Ma, Y., Biava, H., Contestabile, R., Budisa, N., di Salvo, M. L. Coupling bioorthogonal chemistries with artificial metabolism: intracellular biosynthesis of azidohomoalanine and its incorporation into recombinant proteins. Molecules. 19 (1), 1004-1022 (2014).
- Teramoto, H., Kojima, K. Incorporation of Methionine Analogues Into Bombyx mori Silk Fibroin for Click Modifications. Macromol Biosci. 15 (5), 719-727 (2015).
- Deal, R. B., Henikoff, J. G., Henikoff, S. Genome-wide kinetics of nucleosome turnover determined by metabolic labeling of histones. Science. 328 (5982), 1161-1164 (2010).
- Hinz, F. I., Dieterich, D. C., Tirrell, D. A., Schuman, E. M. Non-canonical amino acid labeling in vivo to visualize and affinity purify newly synthesized proteins in larval zebrafish. ACS Chem Neurosci. 3 (1), 40-49 (2012).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat Neurosci. 13 (7), 897-905 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). P Natl Acad Sci USA. 103 (25), 9482-9487 (2006).
- Glenn, W. S., et al. Bioorthogonal Noncanonical Amino Acid Tagging (BONCAT) Enables Time-Resolved Analysis of Protein Synthesis in Native Plant Tissue. Plant Physiol. 173 (3), 1543-1553 (2017).
- Zhou, L., et al. Incorporation of tryptophan analogues into the lantibiotic nisin. Amino Acids. 48 (5), 1309-1318 (2016).
- Acevedo-Rocha, C. G., Budisa, N. Xenomicrobiology: a roadmap for genetic code engineering. Microb Biotechnol. 9 (5), 666-676 (2016).
- Agostini, F., Völler, J. -S., Koksch, B., Acevedo-Rocha, C. G., Kubyshkin, V., Budisa, N. Biocatalysis with Unnatural Amino Acids: Enzymology Meets Xenobiology. Angew Chem Int Edit. 56 (33), 9680-9703 (2017).
- Bacher, J. M., Ellington, A. D. Selection and characterization of Escherichia coli variants capable of growth on an otherwise toxic tryptophan analogue. J Bacteriol. 183 (18), 5414-5425 (2001).
- Wong, J. T. Membership mutation of the genetic code: loss of fitness by tryptophan. Pc Natl Acad Sci USA. 80 (20), 6303-6306 (1983).
- Hoesl, M. G., et al. Chemical Evolution of a Bacterial Proteome. Angew Chem Int Edit. 54 (34), 10030-10034 (2015).
- Italia, J. S., et al. An orthogonalized platform for genetic code expansion in both bacteria and eukaryotes. Nat Chem Biol. 13 (4), 446-450 (2017).
- Völler, J. -S., Thi To, T. M., Biava, H., Koksch, B., Budisa, N. Global substitution of hemeproteins with noncanonical amino acids in Escherichia coli with intact cofactor maturation machinery. Enzyme Microb Tech. 106, 55-59 (2017).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Völler, J. -S., Budisa, N. Coupling genetic code expansion and metabolic engineering for synthetic cells. Curr Opin Biotech. 48, 1-7 (2017).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr Opin Chem Biol. 14 (6), 774-780 (2010).
- Somsen, O. J., van Grondelle, R., van Amerongen, H. Spectral broadening of interacting pigments: polarized absorption by photosynthetic proteins. Biophys J. 71 (4), 1934-1951 (1996).
- Kurschus, F. C., Pal, P. P., Bäumler, P., Jenne, D. E., Wiltschi, B., Budisa, N. Gold fluorescent annexin A5 as a novel apoptosis detection tool. Cytom Part A. 75 (7), 626-633 (2009).
- Lepthien, S., Wiltschi, B., Bolic, B., Budisa, N. In vivo engineering of proteins with nitrogen-containing tryptophan analogs. Appl Microbiol Biot. 73 (4), 740-754 (2006).
- Wachter, R. M., Elsliger, M. -A., Kallio, K., Hanson, G. T., Remington, S. J. Structural basis of spectral shifts in the yellow-emission variants of green fluorescent protein. Structure. 6 (10), 1267-1277 (1998).
- Verkhusha, V. V., Lukyanov, K. A. The molecular properties and applications of Anthozoa fluorescent proteins and chromoproteins. Nat Biotechnol. 22 (3), 289-296 (2004).
- Martynov, V. I., Savitsky, A. P., Martynova, N. Y., Savitsky, P. A., Lukyanov, K. A., Lukyanov, S. A. Alternative cyclization in GFP-like proteins family. The formation and structure of the chromophore of a purple chromoprotein from Anemonia sulcata. J Biol Chem. 276 (24), 21012-21016 (2001).
- Piatkevich, K. D., Malashkevich, V. N., Morozova, K. S., Nemkovich, N. A., Almo, S. C., Verkhusha, V. V. Extended Stokes shift in fluorescent proteins: chromophore-protein interactions in a near-infrared TagRFP675 variant. Sci Rep. 3 (1), 1847 (2013).



