Summary
Toplam hücresel RNA, kısa vadeli RNA sentezi ve çürüme değişikliklerin yanı sıra, RNA işleme kinetiğini incelemek için kötü bir şablon sağlar. Burada, 4-thiouridine ile yeni kopyalanan RNA metabolik etiketlenmesi tarif, bu sınırlılıkların üstesinden gelmek için izin veren yeni transkripsiyonu RNA tiyol-spesifik bir biotinilasyon ve saflaştırma takip etti.
Abstract
Tam transcriptome mikroarrayler ve yeni nesil sıralama gelişimi hücresel gen ifadesinin karmaşıklığı anlayışımızı devrim yarattı. Tutulan moleküler mekanizmaların daha iyi anlaşılması ile birlikte, altta yatan kinetik hassas ölçümler giderek daha önemli hale gelmiştir. Burada, bu güçlü yöntemler şablon örnekleri de çalışma, yani toplam hücresel RNA içsel özellikleri nedeniyle önemli sınırlamalar karşı karşıya. Birçok durumda toplam hücresel RNA değişiklikler yeterli çözünürlüğe sahip temel moleküler olayları ve kinetik temsil etmek için ya çok yavaş ya da çok hızlı bir şekilde ortaya çıkar. Buna ek olarak, RNA sentezi, işlenmesi ve çürüme değişiklikler katkısı kolayca ayırt edilmez.
Biz son zamanlarda bu sınırlamaları aşmak için yüksek çözünürlüklü gen ekspresyon profili geliştirdi. Yaklaşımımız 4-thiouri ile yeni transkripsiyonu RNA metabolik etiketleme dayanmaktadıryemek (böylece de 4SU-etiketleme olarak anılacaktır) tiyol-spesifik bir biotinilasyon ve Streptavidin-kaplı manyetik boncuklar kullanılarak yeni kopyalanan RNA arasında sıkı bir arıtma takip eder. Bu, omurgalılarda, Drosophila ve maya da dahil olmak üzere bir organizma geniş için de geçerlidir. Biz başarılı, transkripsiyon faktörü faaliyetlerinin gerçek zamanlı kinetik çalışma RNA yarı hayatımızın hassas ölçümler sağlamak ve RNA işleme kinetik yeni bakış açıları elde etmek için 4SU-etiketleme uygulanır. Son olarak, sayısal modelleme altta yatan moleküler mekanizmaları entegre, kapsamlı bir analiz oluşturmak için kullanılabilir.
Introduction
Gen ekspresyon profili hücresel süreçleri ve ilgili karmaşık bir etkileşim ağı incelemek için kullanılan önemli bir araçtır. MRNA bolluk ile ilgili çalışmalar genellikle altta yatan moleküler mekanizmaları, temel anlayışlar elde etmek için tercih edilen yöntem olmuştur. Tam transcriptome mikroarrayler 1 ve RNA (RNA-seq) 2-4 daha yakın, yeni nesil sıralama gelişimi bu yaklaşım hızlandırdı. Bu teknolojilerin hücresel gen ifadesinin karmaşıklığı anlayışımızı devrim olsa da, onların şablon örnek doğal özelliklerine, yani toplam hücresel RNA nedeniyle önemli sınırlamalar karşı karşıya. RNA düzeylerinde İlk olarak, kısa vadeli değişiklikler transkripsiyon oranlarındaki değişimler maç, ancak RNA ilgili transkript yarı ömrü üzerinde doğal olarak bağımlı değildir. Bir transkripsiyon faktörü için kısa süreli bir transkript, örneğin kodlama beş kat indüksiyon, toplam RNA kolayca tespit olacak isebir saat içinde, bir metabolik enzim için uzun ömürlü transkript, örneğin kodlama aynı indüksiyon, neredeyse görünmez kalacaktır. Buna ek olarak, hatta bir kapatma (> 1.000 kat yük atma) bir RNA beş saatlik yarılanma ömrü ile ortalama bir genin transkripsiyon oranı tam sadece sadece iki yönlü tarafından azaltmak için, toplam RNA düzeyleri için beş saat sürer . Bu nedenle, toplam RNA analizi düzenleme fonksiyonları 5 ile transkripsiyon faktörleri ve genler için kodlamak birçoğu kısa ömürlü transkript kadar düzenleme tespiti, yanadır. Ayrıca, düzenlemenin gerçek kinetik kaskad gizlenmiş ve birincil sinyal olaylar ikincil ayırt edilemez. Her ikisi de, sırayla, alt biyoinformatik analizler önemli sapmayla neden olabilir. İkinci olarak, toplam RNA düzeylerinde değişiklikler RNA sentezi ya da çürüme değişikliklere mal edilemez. Ikinci ölçümleri transcripti engelleme hücre invaziv yaklaşımlar, örneğin gerektiriraktinomisin D 6, ve zaman içinde devam eden RNA çürüme genişletilmiş izleme kullanarak. 5 memeli hücrelerinde ortalama mRNA yarı ömrü ile - 10 saat 5,7, en genlerin mRNA düzeyleri sadece iki kat transkripsiyonel tutuklama birkaç saat sonra daha az azalmıştır olacaktır. Bunlar, çok küçük farklar temel matematiksel denklem üslü doğası nedeniyle hücresel genlerin çoğu için mRNA'nın yarı ömür kabaca kesin ölçümler ile sonuçlanabilir. Son olarak, süre toplam hücresel RNA RNA-seq genlerimizin yarısını yaklaşık alternatif yapıştırma olayları 8, temel kinetik hem de tam olarak anlaşılamamıştır kalır RNA işleme doku ve bağlama özgü düzenleme rehberlik dinamik mekanizmaları tabi olduğunu ortaya koydu. Buna ek olarak, RNA, özellikle katkı kodlayıcı olmayan RNA'lar için, belirlenecek kalır, diferansiyel gen ekspresyonu için işlem. Toplamda, bu sınırlamalar için önemli engeller temsilaltta yatan moleküler mekanizmaların biyoinformatik kinetik modelleme.
Yakın zamanda bu sorunların 5,7,9 üstesinden gelmek için, olarak adlandırılan yüksek çözünürlüklü gen ekspresyon profili, bir yaklaşım geliştirdi. Bu, 4-thiouridine (4SU-etiketleme), doğal olarak oluşan üridin türev kullanarak yeni transkripsiyonu RNA metabolik etiketleme dayalı ve hücre büyümesi ve gen ekspresyon minimal girişim (bkz. Şekil 1) 5 ile yeni transkripsiyonu transkript doğrudan erişim sağlar 10-12. Hızla alımı, 4SU-trifosfat için fosforilasyon, ve yeni transkripsiyonu RNA içine dahil edilmesi 4SU sonuçlarına ökaryot hücrelerin maruz kalma. Toplam hücresel RNA izole edildikten sonra, 4SU-işaretli RNA fraksiyonu tiol-biotin ve özellikle yeni kopyalanan RNA arasında bir disülfid bağı oluşturma biyotinile olup. 'Toplam hücresel RNA' ardından nicel ('yeni transkripsiyonu') ve etiketsiz ('öncesi existin etiketli ayrılabilirStreptavidin-kaplı manyetik boncuklar kullanılarak yüksek saflıkta G ') RNA dır. Son olarak, etiketli RNA sadece disülfid bağ yarma bir indirgeyici madde (örneğin, ditiyotreitol) eklenmesi ve Tanelerden yeni kopyalanan RNA bırakarak Tanelerden geri kazanılır.
Yeni transkripsiyonu RNA 4SU maruz kalma süre boyunca her genin transkripsiyonel aktivitesini gösteriyor. Dakika ölçeğinde 4SU-etiketleme böylece ökaryotik gen ifadesinin bir anlık görüntü ve aşağı akım biyoinformatik analizleri (promotor analizi) için ideal bir şablon sağlar. Kararlı durum koşulları kabul edilebilir durumda, oranları yeni transkripsiyonu / toplam, yeni transkripsiyonu / etiketsiz ve etiketsiz / RNA hassas RNA yarı ömrü 7,13 için non-invaziv erişim sağlar. Buna ek olarak, 4SU-etiketleme az 5 dakika (5 dakika 4SU-RNA) sonra arıtılmış bu yeni transkripsiyonu RNA dikkat etmek önemlidir 15 ve 60 dakika 4SU-RNA daha gençtir.RNA-seq ile birlikte tek bir deneysel ortamda 4SU-etiketleme ultra-kısa ve giderek daha uzun hem yaparken, RNA işleme kinetik nükleotid çözünürlükte 9 ortaya çıkar. Son olarak, sayısal modelleme ile birlikte yeni transkripsiyonu ve toplam RNA zaman tabii ki analiz RNA sentezi ve çürüme 14 entegre bir analiz sağlar.
Sonuç olarak, bu yaklaşım, ökaryotik hücrelerde RNA sentezi, işleme ve bozulmanın dinamiklerinin doğrudan analizi sağlar. Bu memeliler, böcekler (Drosophila), amfibi (Xenopus), ve maya 5,15,16 dahil olmak üzere tüm büyük model organizmalarda geçerlidir. Bu mikroarray analizi 5,17, RNA-seq 9,13,14 ile doğrudan uyumludur, ve in vivo 12,15 uygulanabilir. Burada, detaylı metodoloji, etiket izole ve kültürlü memeli hücrelerinde yeni transkripsiyonu RNA arındırmak için. Ayrıca, potansiyometre olarakal sorunlar ve tuzaklar tartışılmıştır.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. 4-thiouridine ile Metabolik Etiketleme
Deney düzeneği / program ayrıntılı bir plan yapın, hücre kültürü için 4SU eklemek için ne zaman ve örnekleri hasat zaman örneğin. Her durumda arasında en az 5 dakika boyunca Planı. Bir seferde sadece bir durumun hücre tedavisi. Max tutun. 3 - belirli bir zamanda 5 yemekleri. Sıcaklık ve CO2 düzeylerindeki değişiklikler en aza indirmek için mümkün olduğunca çabuk hücreleri tutun. Bu hücresel proteinlere 4SU-etiketli RNA çapraz neden olabilir 4SU eklendikten sonra parlak ışık hücreleri maruz bırakmayın.
Etiketleme başlangıcı
- Çözülme 4-thiouridine (4SU) sadece steril bir Falcon tüp içine her bir durum için 4SU kullanımı ve pipet gerekli miktarda önce.
- Yemekler kapalı hücre kültür ortamı gerekli miktarda (10 cm tabak başına 5 ml) alın ve 4SU içeren Falcon tüp eklemek ve iyice karıştırın. Çıkarın ve yemekleri kalan orta atın. <li> yemekleri 4SU içeren orta geri uygulayın.
Etiketleme sonu
- Hücrelerinden hücre kültürü ortamı çıkarın. Her plaka için Trizol 5 ml ilave edilir. Çok sayıda zaman noktasında ya da koşullar da dahil olmak üzere karmaşık bir deney için, bu adım en çok iki kişi, orta, Trizol ekleme ve lizat hasat diğer bir kaldırarak yapılır.
- Tam bir hücre parçalama, oda sıcaklığında 5 dakika süreyle inkübe edilir.
- Eklendi Trizol ile dikkatli bir şekilde plaka durulama bir 10 ml pipet kullanın. Bu tam hücre parçalama ve örnek kurtarma yardımcı olur. Cilt veya gözlerle temas alırken Trizol son derece tehlikeli olduğu gibi dikkatli olun! Eldeki fenol yanıklar (örneğin polietilen glikol 300 veya endüstriyel metil alkol (70:30) 400) için panzehir var. Polipropilen tüplere örnekleri aktarın. Standart Falcon tüpler) bu yüksek g kuvvetleri karşı değil lütfen unutmayın. Örnekler I Toplam RNA kadar en az bir ay süreyle -20 ° C'de saklanabilirs hazırlanmıştır.
2. Modifiye Trizol Protokolü kullanarak RNA Hazırlık
- 1 ml kloroform (ml Trizol başına 0.2 ml) ekleyin ve 15 saniye boyunca kuvvetlice çalkalanır. 3 dakika - 2 saat oda sıcaklığında inkübe edin.
- 4 ° C'de 15 dakika 13,000 x g'de santrifüjleyin ° C
- Yeni bir 15 ml polipropilen tüpe sulu üst faz (RNA ihtiva eden) aktarın.
- RNA ekleme ½ Çöktürme tamponu ve izopropanol hem de reaksiyon hacmi (3 ml süpernatant, örneğin, 1.5 ml RNA Çöktürme tamponu ve 1.5 ml izopropanol ekleme).
- İyice karıştırın. 10 dakika oda sıcaklığında inkübe edin.
- 4, 10 dakika boyunca 13.000 x g'de santrifüje ° C Süpernatant atın.
- Kısaca aşağı doğru döndürün (5000 × g 30 saniye) ve 200 ul pipet ile artık izopropanol çıkarın.
- % 75 etanol eşit hacim eklemek ve topak ayrılıncaya kadar tüp çalkalanır. Geride kalan bu kadar çok küçük parçalar halinde o kırılma önlemek yapabilir kaldırmal etanol zor.
- 4, 10 dakika boyunca 13.000 x g'de santrifüje ° C Süpernatant atın.
- Kısaca RNA aşağı Spin ve 200 ul pipet ile kalan etanol çıkarın. 20 ul pipet ile kalan etanol adım ve çıkarın tekrarlayın. Bu iki adım sonra, pelet başka bir kurutma işlemi ifa edilmelidir.
- 100 mg beklenen RNA verim başına H 2 O 100 ul ekleyin ve 5 aşağı yukarı pipetleme ve ile iyice karıştırın - 6 kez RNA çözme yardımcı olmak için.
- Çözülür ve 65 ısıtılarak denatüre RNA ° C'de 10 dakika (çalkalama) ve hemen buz üzerine yerleştirin.
- Üreticinin yönergeleri izleyerek, bir NanoDrop spektrofotometre kullanılarak 260 nm dalga boyunda konsantrasyon RNA ölçün. Bu RNA, en az bir ay boyunca -80 ° C'de muhafaza edilebilir.
3. Yeni Transkripsiyonu RNA tiol özgü biyotinilasyon
- 60 ile başlayın - toplam hücresel RNA 80 mikrogram.
- Reaksiyon etiketleme oluşturmaktadır. Aşağıdaki Pipetsipariş (başına mikrogram RNA):
- 1 ul 10x biyotinilasyon Tampon
- 7 ul RNA (nükleaz içermeyen H 2 O seyreltilmiş 1 mg RNA içeren)
- 2 ul Biotin-HPDP (1 mg / ml DMF)
Her zaman biotin-HPDP son ekleyin ve pipetleme hemen karıştırın. Biyotin çökeltiler durumda, DMF içeriği% 40 arasında bir konsantrasyona kadar arttırılabilir.
- Rotasyonu ile 1.5 saat boyunca oda sıcaklığında inkübe edin.
- Kloroform eşit hacimde ekleyin. Şiddetle karıştırın. Aşamalarını başlar ve kabarcıklar yok başlayana kadar 3 dakika - 2 inkübe edin.
- 4 ° C de 5 dakika için 20,000 x g'de santrifüjleyin Dikkatli bir şekilde yeni bir tüp içine, üst sulu faz transfer.
- Bir kez adımları 3.4 ve 3.5 tekrarlayın. Bu RNA kaybını azaltmak için 2 ml Faz Kilidi Jel Ağır tüplerde bu adımı gerçekleştirmek isteyebilirsiniz.
- RNA ayrılması: eklenti 1/10 5 M NaCI ve hacmi eşit hacimdeSu fazı, izopropanol.
- 4 ° C'de 20 dakika boyunca 20,000 x g'de santrifüjleyin Süpernatant atın.
- Supernatant 10 dakika 4 ° C'de 20,000 azından% 75 etanol, santrifüj eşit bir hacmi x g ekleyin.
- Kısaca Spin ve 200 ul pipet ile artık etanol çıkarın.
- Kısaca Spin ve 20 ul pipet ile artık etanol çıkarın.
- RNA kurumasına izin vermeyin. 50 yeniden askıya - 100 ul H 2 O (1 mcg giriş RNA başına ~ 1 ul). 6 kez - 5 yukarı ve aşağı pipetleme ile iyice karıştırın.
- RNA bozulma dışlamak için electrophoretical analizi ile RNA kalitesini kontrol edin.
4. 4SU-Esas nokta Blot Analizi (İsteğe bağlı)
4SU birleşme kolayca biyotinile edilmiş RNA dot blot analizi ile tespit edilebilir. Bu sorun giderme ve biotinlenmiş DNA oligo kontrole göre 4SU dahil oranlarının tahmin sağlayan isteğe bağlı bir adımdır. Bu tahlil için, biz rAdım 3.2 4SU-etiketli RNA biyotinilasyon için yerine biotin-HPDP bir iyodoasetil-biotin kullanarak ecommend. 4SU-RNA geri dönüşü olmayan bir biyotinilasyon ile sonuçlanır. Bu nedenle, kolon dayalı yöntemler (örneğin, RNeasy) biyotinile edilmiş RNA çok küçük miktarlarda (örneğin, 5 mg) geri kazanılması için kullanılabilir. RNA, bu tahlil için uygun olan biotin-HPDP biotinli olsa da, elde edilen sinyali (Şekil 3) ve zayıf sinyal-gürültü oranı daha az tercih edilir.
- Bölüm 1 ve 2 de tarif edildiği gibi toplam hücresel RNA 4SU-etiketleme ve izolasyonu için protokol takip edin.
- Iyodoasetil-biotin ile biotin-HPDP yerine bölümünde 3 açıklanan ve tamamen aşırı iyodoasetil-biotin artıkları çıkarmak için iki kloroform ekstraksiyon gerçekleştirmek olarak Biotinylate RNA 4SU-etiketli.
- Açıklanan ya da olgu RNA az miktarda ((örneğin RNeasy) bir sütun tabanlı yaklaşım kullanarak olarak izopropanol / etanol yağış biyotinlenmiş RNA Kurtar <10 mg) Kullanılır.
- 10 dakika süre ile sallanan nükleaz içermeyen su içinde Zeta membran inkübe edin.
- Nükleaz içermeyen su dışında zar atın ve iki temiz kağıt havlu ve sıkıca basarak arasında membran yerleştirerek aşırı sıvı çıkarın. Hava ile kurutma 5 dakika boyunca zarın güzel noktalar ile sonuçlanacaktır.
- Her numune için, buz gibi soğuk nokta benek bağlama tamponu (10 mM NaOH, 1 mM EDTA) kullanılarak 200 ng / ul RNA 20 ul hazırlar. Pipetleme Zeta zara bu seyreltme 5 ul (RNA yani 1 ug) ve aynı zamanda takip eden üç 10-kat seyreltme (örneğin, sırasıyla 100, 10 ve 1 ng RNA) uygulanır. Pipet uçları boş bir raf ile pipetleme eşit olarak dağıtılmış boşluk sağlamak için kullanılabilir. Seçenek olarak ise, üreticinin talimatlarına uygun olarak bir nokta lekesi cihazı kullanın.
- Olumlu bir devam olarak 20 ng / ul 20 ug / ml (yani, 100-0,1 ng oligo), değişen konsantrasyonlarda biyotin-etiketli DNA oligo 5 ul uygulayınpipetleme membran rol. Negatif kontrol olarak biotinlenmiş, 4SU-naif örnek kullanın.
- Açık havada kurumaya 5 dakika boyunca membran.
- Sallama ile 40 ml bloke tamponu içinde 30 dakika boyunca zarın inkübe edin.
- 15 dakika (5 ml PBS + 5 ml% 20 SDS + 10 ul streptavidin-yaban turpu peroksidaz) için 1:1,000, streptavidin-yaban turbu peroksidazı 10 mL 'si ile inkübe membran
- 5 dakika boyunca 40 ml PBS +% 10 SDS (20 mi PBS + 20 ml% 20 SDS) içinde iki kez membran yıkayın.
- 40 ml PBS içerisinde iki kez membran yıkayın 5 dakika boyunca +% 1 SDS (38 ml PBS + 2 ml% 20 SDS).
- 40 ml PBS içerisinde iki kez membran yıkayın 5 dakika boyunca +% 0.1 SDS (40 mL PBS + 200 ul% 20 SDS).
- Iki temiz kağıt havlu arasında membran yerleştirerek ve sıkıca bastırarak aşırı sıvı çıkarın.
- Üreticinin talimatlarına göre ECL kullanarak membrana bağlı HRP gözünüzde canlandırın.
- Plastik folyo / torbaya membran yerleştirin, hava kabarcıkları ve karanlıkta 2 dakika süreyle inkübe edin.
- Membran maruz1 Film - 5 dk.
5. Streptavidin kaplı Manyetik Boncuk kullanarak Etiketli ve Etiketsiz RNA ayrılması
- Isı ° C bir su banyosunda 65 (örnek başına 3 ml) bir yıkama tamponu.
- Nükleaz içermeyen H 2 O taze 100 mM ditiyotreitol (DTT) hazırlayın Ultra-ince terazi gözü üzerine yerleştirildi, temiz bir 50 ml Falcon tüpüne DTT toz, 30 mg - 15 şişeden bunu. Nükleaz içermeyen H 2 O gerekli miktarda tartılır ve ekleyin
- Isı ° C 10 denatüre dk ve buz üzerinde hemen yer 65 RNA örnekleri biotinlenmiş.
- Yer manyetik stand içine sütun μMacs. Biz bir anda birden fazla 12 örnek (- 8 örnekleri uygun olan 6) işlemek için değil tavsiye.
- 1 ml oda sıcaklığında tampon yıkama ile ön dengeye Miltenyi sütunlar. Bu yaklaşık 15 dakika sürer.
- Bu arada, 50 streptavidin boncuk 100 ul - biyotinile edilmiş RNA 100 ul. Rotasyonu ile 15 dakika süreyle oda sıcaklığında inkübe edin. < li> sütunları herhangi artık boşaltma başlatılan değilse bu hafifçe bir eldivenli parmak ile sütunun üstüne basarak kolaylaştırılabilir. Bir kez akışını sütunları kolayca tahliye başlamıştır.
- Sütunlara RNA / boncuk uygulayın. Akış-size etiketsiz RNA fraksiyonu (bkz. bölüm 7) kurtarmak istediğiniz sürece atın.
- 0.9 ml 65 ° C yıkama tampon maddesi (65 ° C'de tampon pipetleme 1 ml'lik pipet uçları shrink). Ile üç kez yıkayın
- 0.9 ml oda sıcaklığında yıkama tamponu ile üç kez yıkayın.
- Pipet 700 ul Tampon yeni 2 ml tüpler içine RLT (RNeasy MinElute Temizleme Kiti, Qiagen) ve sütunları altına koyun.
- Sütununa 100 mM DTT, 100 ul ekleyerek RLT tampon içine yeni kopyalanan RNA Zehir.
- 100 mM DTT başka bir 100 ul ekleyerek sonra aynı tüp içine ikinci bir elüsyon turda 3 dk gerçekleştirin.
6. Yeni kopyalanan RNA geri kazanımı
ontent "> üreticinin talimatları izleyerek RNeasy MinElute Temizleme (Qiagen) protokolü ile devam edin. 25 ul nükleaz içermeyen H 2 O ölçün RNA konsantrasyonlarda Zehir bir Nanodrop Spektrofotometre kullanarak. göndermeden önce RNA çözülme ve yeniden dondurmak için ihtiyaç önlemek için bir yüksek verimli test için, hemen yeni transkripsiyonu RNA sonra cDNA hazırlanıyor tavsiye arındırılır. üreticinin talimatları cDNA sentezi için 20 ul cDNA sentezi karışımı yeni transkripsiyonu RNA 2.5 ul kullanın. 1 ile Mah-PCR kontrolleri gerçekleştirin : cDNA karışımı 10 dilüsyonları -80 ° C'de saklayın RNA.7. Etiketsiz, Sınırsız RNA (İsteğe bağlı) geri kazanımı
Durumda ilişkisiz RNA geri gereken, akış yoluyla (sütunlara RNA-streptavidin boncuk çözüm ekledikten sonra) ve sonraki yağış için ilk yıkama toplamak ve birleştirmek. Genellikle thi olarak ilişkisiz RNA sadece 50% çöktürmek için yeterlidirin başlangıç malzemesinin>% 80 içerir.
- Izopropanol eşit hacimde (tuz yıkama tamponu önce, 1 M NaCl içeren olarak ilave edilmesi gerekir).
- 4 ° C'de 20 dakika boyunca 20,000 x g'de santrifüjleyin Süpernatant atın.
- Supernatant 10 dakika 4 ° C'de 20,000 azından% 75 etanol, santrifüj eşit bir hacmi x g ekleyin.
- Kısaca Spin ve 200 ul pipet ile artık etanol çıkarın.
- Kısaca Spin ve 20 ul pipet ile artık etanol çıkarın.
- RNA kurumasına izin vermeyin. 100 ul H 2 O içinde süspanse 6 kez - 5 yukarı ve aşağı pipetleme ile iyice karıştırın. Çalkalama ile 10 dakika boyunca 65 ° C'de inkübe edilir ve buz ile doğrudan transfer.
- RNA bozulma dışlamak için electrophoretical analizi ile RNA kalitesini kontrol edin.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
1. Başlangıç Malzeme ve Beklenen Verim
4SU-maruz kalma 1 saat (saat) ardından yeni transkripsiyonu RNA yaklaşık 1 temsil - toplam hücresel RNA 4%. Artık hücre büyümesi / çoğaltma için hesaba RNA sentezi gibi bu büyüme-tutuklandı hücrelerde daha düşük olacaktır. Toplam RNA 80 mg - 1 saat etiketleme, biz 60 ile tahlil başlayan tavsiye. Biyotinilasyon adımdan sonra görmek zor olan küçük RNA pelet toplam RNA sonuçların az 30 mikrogram ile başlayan ve bu nedenle kolayca kaybolabilir. Giriş RNA düzeylerinin etiketleme çok kısa süreli (- 10 dakika örneğin 5) için olduğu kadar 150 olarak mikrogram yükseltilebilir. RNA etiketleme süresi 1 saat için 5 dakika ~% 60 ~ 80% 9 yeni transkripsiyonu RNA artar kısa ömürlü intronik dizilerinin katkısı kısaltılır zaman. Intronlar önemli ölçüde daha uzun kodlama dizilerinin yanı sıra, 5'-ve 3'-UTRs karşılaştırıldığında olarak, miktarı yeni transkripsiyonuKısa hatta ultra-kısa 4SU-etiketleme sonrasında saf olabilir RNA, doğrusal düşmez. Bu nedenle, biz yapışmaz insan B-hücre hatları 9 4SU-etiketleme 5 dakika sonra toplam RNA>% 0.5 aldı. Ancak, etiketleme 4SU ve biraz daha uzun süreler için daha yüksek konsantrasyonda yapışmayan hücreler içindeki 4SU birleşme oranları elde etmek için gerekli olabilir unutulmamalıdır. Bile düşük 4SU-birleşme oranı izin vermekle birlikte büyük, üridin zengin transkript verimli yakalama ve arıtma, düşük üridin içeriği ile çok kısa transkript (örneğin miRNA'lar) yüksek 4SU konsantrasyonlarda (> 1 mM) kullanırken bile arıtma kaçmak için muhtemeldir. 5 100 nükleotid (nt) - NIH-3T3 fare fibroblast olarak, 200 mcM 4SU maruz kalma 1 saat 50 başına 4SU yaklaşık bir kalıntı ile yeni transkripsiyonu RNA etiketli. Uzunluğunda 1000 nt - Bu transkript> 500 yüksek verimli kurtarma izin vermelidir. Buna uygun olarak, sadece küçük bir transkript büyüklüğü gözlenirmurin fibroblast ve insan B-hücrelerinin 7 hem de 200 uM 4SU kullanarak 1 saat boyunca etiketleme ağırlığı. 200 mcM 4SU 1 saat fare fibroblast, ≥ 200 mcM 4SU için hücrelerin uzun süre maruz hücresel transkript düzeylerinde önemli bir değişiklik neden yoktu da 24 saat (yayınlanmamış veri) içinde ölçülebilir bir büyüme açığı neden yok. Bu nedenle, etiketleme süresi ve 4SU-konsantrasyonu ücret karşılığı ektopik veya toksik etkilerini önlemek için en aza indirilmelidir. Yeni transkripsiyonu RNA etkin kurtarma için gerekli 4SU-konsantrasyonu minimum belirlemek için kolay bir yol 4SU artan konsantrasyonları (- 1.600 mcM örneğin 50) ile 4SU-etiketleme sonrasında yeni transkripsiyonu RNA temizlemektir. Gibi şekillerde gösterildiği 2A ve 2B, primer insan fibroblastları içinde 1 saat boyunca etiketli yeni kopyalanan RNA kurtarma 50 4SU 200 uM büyük ölçüde artmış, ancak daha sonra plato başladı.
2. Nokta4SU Ortaklığın (isteğe bağlı) miktarının leke
Bazı durumlarda toplam RNA 4SU dahil miktarını ölçmek için ilgi çekici olabilir. Bu işlem en iyi konjuge streptavidin kullanılarak biyotinile edilmiş RNA, nokta benek analizi ile yapılır. Kimyasal özelliğinden dolayı iyodoasetil-biyotin yeni kopyalanmış RNA, hemen hemen tüm 4SU kalıntılarının biyotinilasyon sonuçlanan biotin HPDP daha tiol gruplara daha reaktiftir. Bu biyotin-HDPD gibi, iyodoasetil-biyotin suda çözünür değildir ve bu sayede daha verimli olarak biyotin-HPDP için gerçekleştirilen kloroform ekstraksiyonu ile çıkarılır, dikkat etmek önemlidir. Bu nedenle, aynı reaksiyon koşulları ve konsantrasyonlarda biyotin HPDP kullanırken olduğu gibi kullanılabilir. Ancak, iyodoasetil-biotin geri değildir. Bu nedenle sütun tabanlı yaklaşımlarda yeni transkripsiyonu RNA saflaştırma için kullanılamaz. Iyodoasetil-biotin kullanımı 4SU-dahil ölçmek için sağlar, biotin-HPDP tabanlı ölçümleri her iki düşünün4SU-Kuruluş ve biyotinilasyon verimlilik. Aynı örnek için iki biyotinilasyon reaktifler kullanılarak, RNA-Incorporated 4SU arasında biyotinilasyon etkinliğinin ölçümü için izin verir. 4SU-etiketli RNA için biotin-HPDP arasında biyotinilasyon verim yeni kopyalanmış RNA, yaklaşık olarak sadece tek bir 4SU üç kalıntı aslında biotin HPDP (Şekil 3) ile biyotinlenmiştir belirten iyodoasetil-biotin göre yaklaşık üç kat daha az olduğu görülmektedir. Biyotinlenmiş kontrol DNA oligo örnek bir sinyal yoğunlukları karşılaştırarak, biyotinilasyon yoğunluğu ölçülebilir. En memeli hücre hatları için olumlu bir sinyal hala 200 mcM 4SU etiketleme 1 saat sonra biotinlenmiş RNA 10 ng tespit olmalıdır. Zayıf bir arka plan sinyal etiketsiz RNA'nın en yüksek konsantrasyon (1 mcg) için genellikle saptanabilir.
3. Yeni kopyalanan RNA ile saflaştırılması
Yeni transkripsiyonu RNA kurtarma derece Quan olduğutitative. Aynı RNA konsantrasyonu ile başlayan Eğer tüm örnekler için yeni transkripsiyonu RNA aynı miktarda elde etmek için bekleyebilirsiniz. OD 260 ölçümleri etkileyebilir 260 nm (yıkama tamponlar elde edilen deterjan varlığı) - birçok sütun tabanlı deneyleri gibi, RNeasy MinElute kiti kullanılarak yeni transkripsiyonu RNA toplanması 230 ek absorbe edilmesi ile sonuçlanabilir. Her bir santrifüj aşaması için yeni bir 2 ml toplama tüpü kullanılarak, bu daha az bir oranda görülür. Bununla birlikte, herhangi bir makul yüksek OD ölçümleri (diğer örneklere göre> 2 kat daha fazla) OD 260/280 oranı <1.7 olan, özellikle dikkatle düşünülmelidir. Aşağı akım analizleri için tüm örnekler için şablon RNA hacmi aynı miktarda kullanmak genellikle bu nedenle en iyisidir. Etiketli RNA verimi electrophoretical analizi ile RNA bozulma belirtileri için beklenen onay daha düşük olduğu durumlarda. Yeni kopyalanan RNA önemli ölçüde daha büyük bir miktarda içerir tipik rRNA bantları (Şekil 4) çok daha az belirgin olmak üzere büyük, unspliced transkript.
4. Yeni Transkripsiyonu RNA ölçümü
Son olarak, yeni transkripsiyonu RNA 4SU dahil edilmesi oranları doğrudan 330 nm 4SU emilimini maksimum ve OD 330/260 oranı 5,18 dayalı spektrofotometrik analizi ile ölçülebilir. Izopropanol / etanol çökeltme ile - Bu, küçük bir hacme (20 ul 10) konsantre edildi etiketli RNA> 3 ug gerektirir. Nükleaz içermeyen glikojen 30 mg (Fermentas, # R0551) ile küçük RNA pelet eş yağış kaybetmemek için yapılmalıdır. Ek bir tepe yeni transkripsiyonu RNA (Şekil 5) içine 4SU ile birleşmesiyle oranı yansıtan 330 nm görülebilir.
/ Files/ftp_upload/50195/50195fig1highres.jpg "/>
Şekil 1. . Toplam hücresel RNA hazırlanması takip süresi - 4-thiouridine (4SU) metabolik etiketleme prensibi 4SU gerekli (120 dk 5) hücrelere ilave edilir. Tiyol-spesifik bir biotinilasyon ardından, toplam hücresel RNA, streptavidin kaplı manyetik boncuklar kullanılarak 4SU-etiketli, yeni kopyalanan RNA ve etiketsiz, önceden var olan RNA'ya ayrılır. Yeni transkripsiyonu RNA boncuk için yeni transkripsiyonu RNA bağlantı disülfit bağları parçalayarak bir indirgeyici madde kullanılarak boncuk kurtarılır. büyük rakam görmek için buraya tıklayın .
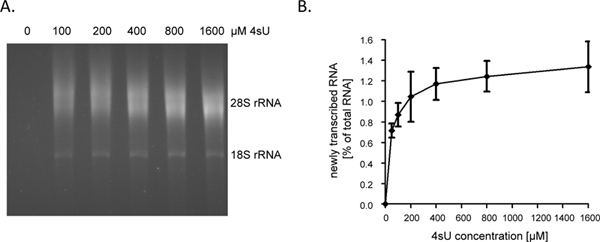
Şekil 2. Aşağıdaki yeni kopyalanan RNA geri kazanımı 4SU artan konsantrasyonları. (a) Primer insan sünnet derisi fibroblast (HFF) 100 ile inkübe edildi, 4SU, 200, 400, 800 veya 1,600 mm. Yeni kopyalanan RNA 50 ug toplam hücresel RNA saflaştırıldı ve electrophoretical analizine tabi tutulmuştur. Beklendiği gibi, geri yeni transkripsiyon RNA, konsantrasyon bağımlı bir artış daha yüksek konsantrasyonlarda plato başladığı gözlenmiştir. (B), yeni kopyalanan RNA saflaştırılmış edilmiştir ImageJ 1.45s yazılımı kullanılarak ölçüldü. 800 mcM 4SU (n = 2) veya 100 - - Yeni transkripsiyonu RNA tutarlar üzerinde dört bağımsız deneyler kombine verileri 50 ya kadar 4SU-etiketleme farklı konsantrasyonlarda ardından iyileşti. 1.600 mcM 4SU (n = 2) gösterilmiştir tıklayınız büyük rakam görmek için .
upload/50195/50195fig3.jpg "alt =" Şekil 3 "fo: içerik-width =" 4.5in "fo: src =" / files/ftp_upload/50195/50195fig3highres.jpg "/>
Şekil 3,. Nokta benek analizi kullanılarak 4SU-etiketli toplam RNA 4SU esas tahmini. Toplam RNA, bir saat süre ile 4SU 200 uM ile inkübe NIH-3T3 fare fibroblastlarının ya da insan sünnet derisi fibroblast (HFF) izole edilmiştir. Resim 4SU negatif kontrol olarak, bir tabak için ilave edildi. HFF için iki kişi inhibe (n = olmayan büyüyen hücreleri) ve büyüyen hücreler (y) dahil edildi. RNA Trizol reaktifi ve biyotin HPDP veya iyodoasetil-biyotin sonradan konjuge kullanılarak izole edilmiştir. Her bir numune konsantrasyonu, bu seyreltme 200 ng / ml ve 5 ul (RNA yani 1 ug), hem de takip eden üç 10-kat seyreltme (yani sırasıyla 100, 10 ve 1 ng RNA) için ayarlandı tüm idi Zeta membran bir parça gördü. Biotin-etiketli DNA oligo 5 ul seyreltme konsantratı pozitif kontroller olarak membran yerleştirilding / ul aşağı 20 ila 20 pg / ml (yani, sırasıyla, 100-0,1 ng) arasında değişen iyonları. Biotin, yoğunluk, bir streptavidin-yaban turpu peroksidaz konjugatı ile problanmıştır.
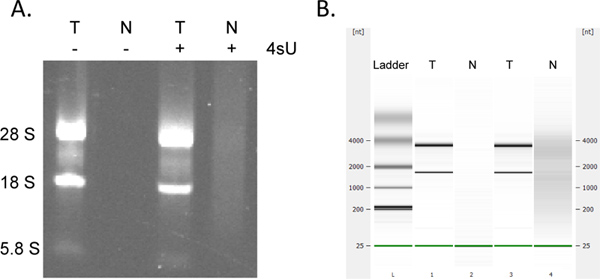
Şekil 4. Yeni transkripsiyonu ve toplam RNA Electrophoretical analizi. Toplam RNA, (T) ve yeni kopyalanan RNA (N) 1 saat agaroz jel elektroforezi ile analiz edilmiştir 500 uM 4SU varlığında ve yokluğunda her ikisi de kültüre sıçangil NIH-3T3 fibroblastlar (A) 'dan hazırlanır ve (aynı sırayla) Agilent BioAnalyser (B) kullanarak. No RNA hücre 4SU tedavi olmadan geri kazanılmıştır. Yeni transkripsiyonu RNA Saf yüksek molekül ağırlıklı mRNA daha fazla miktarda ve toplam önemli ölçüde daha az olgun rRNA içerir28S, 18S ve 5.8S rRNA bantları arasında önemli olarak RNA. büyük rakam görmek için buraya tıklayın .
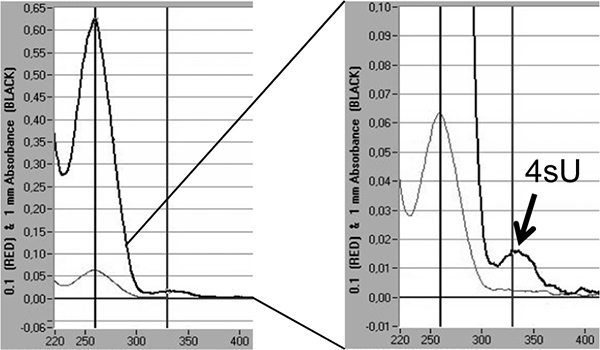
Şekil 5,. Spektrofotometrik analizi ile yeni transkripsiyonu RNA 4SU dahil miktarının. Fare NIH-3T3 fibroblast 4SU 200 mcM 1 saat sonra 2 x 100 mg total RNA arındırılmış Yeni transkripsiyonu RNA. Yeni kopyalanan RNA nükleaz içermeyen glikojen 30 ug ilave edildikten sonra izopropanol / etanol ile çöktürüldü. Bir Nanodrop 1.000 spektrofotometre ile elde edilen yeni transkripsiyonu RNA spektrofotometrik analiz gösterilir. Kalın, koyu gri çizgiler 1 mm sıvı sütun değerleri göstermektedir ise açık gri çizgiler 0.1 mm değerleri göstermektedir. T temsil yok zirvesinde sağında, bir büyütmeO 4SU kalıntısı gösterilmektedir dahil edilmiştir. 4SU 18 katsayısı yok dayanarak 4SU ile birleşmesiyle oranları tahmin edilebilir.
| Etiketleme [dk] Süresi | Önerilen 4SU konsantrasyonu [mcM] |
| 120 | : 100 - 200 |
| 60 | 200-500 |
| 15-30 | 500 - 1000 |
| <10 | 500 - 2000 |
Tablo 1. 4SU konsantrasyonları önerilir.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Yeni transkripsiyonu RNA Metabolik etiketleme ölçüde ilgi biyolojik soruya cevap daha uygun şablonlar sunarak mikroarrayler ve RNA-seq gibi yüksek verimli teknolojilerin gücünü artırır. İşbu Protokol kapsamlı optimizasyon yapıldı. Bu yeni transkripsiyonu RNA> 1000 kat zenginleştirme sağlar ve yüksek tekrarlanabilir sonuçlar sağlar.
Yeni transkripsiyonu RNA sadece 4SU hücrelerin maruz kalma döneminde gerçek zamanlı transkripsiyonel aktivitesini tasvir gibi bir 4SU-etiketleme deney deneysel tasarım büyük önem taşımaktadır. Bir uyaranı takiben transkripsiyon oranlarındaki gerçek değişiklikler zaten sönmeye başladı eşit Toplam RNA düzeylerinde değişiklikler hala tespit olsa bile yeni transkripsiyonu RNA analiz ederken, bu özleyeceğiz. Bu nedenle, temel biyoloji iyi bir anlayış deney sisteminde hem de optimal süre o tanımlamak için önemlidir4SU maruz kalma f zaman. Aşağıda, öneriler ve en önemli adımlar için ortak tuzaklar önlemek için yollar sağlar.
Stok çözümleri ve plastik eşya hazırlanması
Tüm stok çözeltileri, nükleaz içermeyen su ile hazırlanmış olmalıdır. Su indirgeyici ajanlar içerir eğer deiyonize saf su içi kullanırken sorun neden olabilir. Bir durumda, bu tüm etiketli RNA tam kaybı ile sonuçlandı. Bu nedenle, güçlü nükleaz içermeyen NaCl, Tris-Cl, EDTA, sodyum sitrat ve su önceden yapılmış satın öneririz. Her zaman nükleaz içermeyen koşullar emin olun. Dimetilformamid (DMF) gibi bir plastik malzeme çözünür. Biz, biyotin-HPDP stok çözeltisi hazırlamak üzere 50 ml Falcon tüpleri için hisse senedi cam şişe, DMF transfer etmek için 25 ml hücre kültürü plastik pipet kullanılarak esas olarak bütün tahlilinden gelen yeni transkripsiyonu RNA verimi azaltmak için yeterli olduğu bulunmuştur. İlginç olarak, bu olumsuz biotinil etkilemediverim ile (şekilde nokta blot ile test edilmiştir) ancak Tanelerden geri olabilir yeni kopyalanan RNA bir 75 ile>% 90 kaybı ile sonuçlandı. Etiketleme süresi 60 ila 30 dakika ya da daha az düşürülmüştür zaman kaybını en belirgindir. Büyük olasılıkla, DMF plastik pipet ile elüt bir madde kısmen streptavidin boncuk kaplama tahrip. Bu nedenle, DMF ile uyumlu olduğu bilinmemektedir plastik malzemelerin kullanılması elbette kaçınılmalıdır. Aynı nedenlerden dolayı, hücre kazıyıcı hücre kültürü plakaları Trizol numune kazanımı arttırmak için kullanılmamalıdır. Bu DMF veya Trizol ile plastik yıkandı varsayılan maddeler görünüşte ne kloroform ekstraksiyon ne izopropanol / etanol yağış tarafından çıkarıldı bu ilginçtir.
Hücre kültürü
Plakalar üzerinde hücre yoğunluğu önem taşımaktadır. Hücreleri (90 biraz çok birleşik olduğu ortaya çıktı bir deneyde -% 100), interferon, 100 U / ml 'si ile 30 dakika boyunca NIH-3T3 fare fibroblast ile tedavi edilmiş (IFN), α veya γ. Az Konfluent hücrelerde IFN tedavi bile 15 dakika zaten 5 sonuçlandı - irf1 veya socs3 5 gibi genlerin 8 kat indüksiyon için. Hücreleri biraz olmak çok birleşik mikroarray analizi en hızlı irf1 veya socs3 gibi genler indüklenebilir bile IFN-indüklenebilir genlerin herhangi bir indüksiyon göstermedi. Bu nedenle, hücre yoğunluğu önemli bir 4SU etiketleme deneyleri ve hücre kültürü plakaları için bir risk faktörü dikkatlice etiketleme başlamadan önce ele alınmalıdır.
4SU bir photoactivatable ribonükleosit ve 4SU içeren RNA verimli 365 nm ışık kaynağına maruz kaldıktan sonra proteinlere çapraz edilir. 4SU ile tedavi edilen hücreler kaçınılmalıdır karanlık ve parlak ışığa maruz kalma kültüre edilmelidir. Trizol RNA izolasyonu ile hücresel proteinlerin ayrılmasından sonra, bu riski önemli ölçüde azalır.
<p class = "jove_content"> 4SU hücresel DNA dahil değildir. Ancak, halen toplam RNA, hücresel DNA küçük miktarda olacağı unutulmamalıdır. 4SU-etiketleme kullanarak ve q-RT-PCR analizi sitomegalovirüs enfeksiyonu viral gen ekspresyon çalışma ne zaman concatemeric viral genom 19 kaldırmak için protokol bir DNaseI özet adım dahil etmek gerekli bulundu. DNA'nın varlığını duyarlı olmayan alt protokoller kullanılarak, bu muhtemelen gerekli değildir.4SU birleşme oranları ve en uygun 4SU konsantrasyonu
4SU kolayca içi ve dışı hücre düzeyine büyük olasılıkla bir dakikadan 9,16 daha az içinde değişikti ile hücreler tarafından alınır. 4SU alındıktan ve birleşme oranları konsantrasyona bağlıdır. Bu nedenle, 4SU konsantrasyon uygun etiketleme istihdam süresine göre ayarlanabilir. Tablo 1 rel de 4SU konsantrasyonları tavsiye sağlarBizim en iyi kişisel deneyime dayalı etiketleme süresi tirme. Memeli hücrelerinde 4SU etiketleme, 1 saat boyunca, 200 uM 4SU fibroblastlarda yeni kopyalanan RNA içinde 50-100 nükleotid başına yaklaşık bir 4SU tortu ile sonuçlanan bir çok uygulama için yeterli olacaktır.
Son yıllarda çift olarak, fibroblastlar, endotelyal hücreler, epitelyal hücreler, kemik iliği stroma hücreleri, makrofajlar ve T-hücreleri de dahil olmak üzere, insan ve murin kökenli hücre tiplerinin geniş bir yelpazesine 4SU-etiketleme uyguladık. Buna ek olarak, örneğin Drosophila ve Xenopus hücreleri başarılı bir şekilde kullanılmıştır. Bu deneylerin hepsinde, 4SU birleşme farklı hücre tipleri için 4SU konsantrasyonda en az ayarlamalar gerektiren son derece verimli olduğu bulunmuştur. Yeni hücre tipleri için yöntem kurarken, biz 4SU-konsantrasyonları (örneğin 50 ila 1600 mcM kadar) artan etiket hücreleri için tavsiye ve yeni transkripsiyonu saflaştırılmış ilişkiyi analiz olur RUygulanan 4SU-konsantrasyonları NA (Şekil 2A / B). Arıtıldı yeni kopyalanan RNA miktarı bir plato gireceği 4SU konsantrasyon seçilmelidir.
Yüksek birleşmiş, temas inhibe hücreler kullanıldığı durumlarda, biz verimli 4SU dahil olmak için biraz daha yüksek 4SU konsantrasyonları (örneğin 500 yerine 200 mcM) kullanmak tavsiye ederim. Ayrıca, çok kısa yeni transkripsiyonu transkript yakalama (<200 nt) özel ilgi olduğu durumlarda, 4SU konsantrasyonu da artmıştır gerekebilir. Bu ektopik etki veya toksisite önlemek amacıyla uzun süreli etiketleme kez (örneğin> 1 saat) ile kombine edilmemelidir. Son olarak, çok küçük hücre kültür ortamı hacmi kullanılarak 4SU birleşme etkinliğini azaltabilir bulundu. Bu nedenle sırasıyla 5 ml veya 10 cm veya 15 cm çanak başına orta 10 ml kullanmanızı öneririz.
Toplam hücresel RNA hazırlanması
Bu protokolün başarısı için temiz, RNaz-ücretsiz toplam hücresel RNA elde etmek için çok önemlidir. 15 cm tabak başına 5 ml Trizol kullanarak nükleazların ücretsiz temiz RNA üretir. Biz Chomczynski ve arkadaşları tarafından değiştirilmiş Trizol protokolünü kullanmanızı öneririz. 20.. İlk olarak, daha iyi bir yıkama adımları sırasında işlemek daha kolay olan daha sert granüller olarak gelişmiş merkezkaç kuvveti sonuç olarak RNA'nın büyük miktarda (> 100 mg) izole etmek için uygundur. Düzenli 15 ml laboratuvar Falcon tüpler 6.000 'den fazla × g hayatta yok gibi Ancak, bu özel polipropilen borular ve adaptörlerin kullanılması gerekir. İkinci olarak, DNA ve glikoproteinlerin kaldırılması artırır. Organ ya da doku RNA hazırlanması Bu özellikle belirgin hale gelir. Üçüncü olarak, izole edilebilir toplam RNA maksimum miktarını sınırlamak değildir. Biz de bulundu, ancak sütun tabanlı RNA izolasyonu yöntemleri (örneğin RNeasy) uygun kalitede RNA, standart sütun ar sağlamak içine sadece böylece başlangıç malzemesi miktarını sınırlayan toplam RNA 100 mikrogram kadar yakalamak mümkün. Son olarak, kalan etanol kaldırmak için, bir pipet ile iki kez geriye kalan etanol kaldırma RNA kurutulmasıyla artık gerekli değildir. Bu daha sonra tekrar çözünmesi için zor olabilir, RNA, aşırı kurutma riskini ortadan kaldırır. Prensip olarak, 4SU-etiketleme farelerin iv enjeksiyonu ile örneğin, in vivo olarak uygulanabilir. Ancak, RNA saflık yeni transkripsiyonu RNA (yayınlanmamış veri) saflaştırılması önce poliA transkript arıtma gerektiren büyük bir sorun temsil ettiğini kaydetti.
Biyotinilasyon ve bağlanmamış biotin kaldırma
Biotin-HPDP% 100 tiol özgüdür ve tortu, biyotin ve tiyol-etiketli RNA transkripsiyonu yeni moleküller arasında bir disülfid bağı oluşturur. 4SU-işaretli RNA biyotinilasyon verim nokta leke analizi 5 ile belirlenen yaklaşık% 30. Biotin HPDP, suda çözünebilir olduğuverimli bir kloroform ekstraksiyonu ile kaldırılabilir. Tek kloroform ekstraksiyon adım ilişkisiz biotin büyük çoğunluğu kaldırmak için yeterli olsa da düzenli olarak tümüyle kaldırılmasını sağlamak için bu adımı tekrarlayın. Kloroform ekstraksiyon adım 2 ml Faz Kilidi Jel Ağır tüpleri (Eppendorf) sırasında RNA kaybını azaltmak için üreticinin yönergeleri izleyerek kullanılabilir. İlk adım, şablon miktarlar genellikle bu tüpler, doğrudan uyumlu olması için çok yüksek olduğu gibi genellikle sadece ikinci kloroform ekstraksiyon adımı için Faz Kilitli tüpler kullanın. Bağlanmamış biyotin-HPDP çıkarılmasından sonra, RNA, izopropanol / etanol çökeltme ile elde edilir. Bu da, verilen tampon hangi tutunmaya disülfid bağı indirgeme maddeleri içeren ve yeni kopyalanmış RNA, biyotin kaldırmak olarak kullanılmamalıdır biyotinile edilmiş RNA (Qiagen RNeasy örn.) kurtarmak için, ticari sütun tabanlı kitleri dikkat etmek önemlidir .
Yeni saflaştırılmasıly transkripsiyonu RNA
100 ul streptavidin boncuk fazla 100 ul biotinlenmiş RNA eklemeyin. Daha az hacim ekleme tercih edilir. Bununla beraber, RNA'nın aynı hacimde tüm numuneler için ilave edilmelidir. Sadece boncuk 1x TE gerekli hacim ekleyerek streptavidin boncuk eklemek RNA giriş hacmi (örnekler arasında) ayarlayın. Taze nükleaz içermeyen, 100 mM DTT yapmak için kolay bir yol ultra hassas bir terazi gözü üzerine yerleştirildi bir Falcon tüpüne DTT toz yeterli bir miktarı süzün ve sonra, 100 mM DTT oluşturmak için, nükleaz içermeyen H2O gerekli miktarda eklemektir (1 mg DTT başına 64.8 ul su).
4SU-etiketleme geliştirilmesi sırasında çeşitli tedarikçilerden streptavidin boncuk test. Bunların sayısı arka büyük miktarlarda üretilir. Bu nedenle, güçlü şimdiye kadar, biz doku c etiketsiz RNA taşıyan-over ile herhangi bir sorun yaşamamış, Miltenyi Streptavidin boncuklar olarak kullanmanızı öneririzulture türetilmiş RNA numuneleri. Bu şekilde, etiketli RNA az 150 ng özellikle streptavidin boncuk 100 ul ile 150 ug biyotinile edilmiş RNA (su ve 100 ul) den daha da arıtılabilir. Boncuklar ile birlikte dengeleme tamponu ile boncuk dengeleme yapılabilir ve hafif yakalama oranları 13 artırabilir.
Kalite kontrolleri
Biz yüksek verimli analizleri için tabi önce yeni transkripsiyonu RNA q-RT-PCR kontrolleri yapılarak tavsiye ederiz. Bu diferansiyel verilen deneysel ortamda düzenlenir bilinen birkaç referans genlerin miktar içerebilir. 4SU-etiketleme RNA bozulma oranları incelemek için kullanılır durumlarda, kısa süreli transkript (örneğin myc, fos) ve toplam ve yeni transkripsiyonu hem RNA uzun ömürlü bir (örneğin GAPDH) ölçmek için öneriyoruz. Yeni transkripsiyonu / toplam RNA oranı (- ila 10-kat ~ 5) önemli ölçüde daha yüksek olmalıdırkısa ömürlü transkript için. RNA, bir referans gen bölgesinin yarı ömrüne göre, RNA yarı-ömür belirlenebilir. RNA, üç fraksiyonları (toplam RNA, yeni kopyalanan RNA ve etiketlenmemiş önceden var olan RNA) dört ya da daha fazla genin, farklı RNA alt normalleştirilmesi için analiz edilir gibi 7 tarif belirlenebilir lineer regresyon analizi ve kalite kontrol puanları ile gerçekleştirilebilir , 21.
Q-RT-PCR analizi için, 20 ul cDNA sentezi karışımı etiketli RNA 2.5 ul kullanmanızı öneririz. Q-RT-PCR sonuçları en iyi şekilde karşılaştırma için ilk kullanımdan önce 5 ul hacimde cDNA dondurma. Kullanmadan önce çözülme tüpler, H 2 O 45 ul ve q-RT-PCR analizleri için seyreltme konusu 5 ul ekleyin. Bu önemli ölçüde farklı PCR çalışmalar arasında karşılaştırılabilirlik artırır.
Yeni transkripsiyonu RNA örnekleri onlara tabi önce Agilent BioAnalyser kullanarak RNA bozulma belirtileri için kontrol edilmelidiryüksek verimlilik analizi (mikroarrayler veya RNA-sıra). Ancak, ek bant bazen Agilent BioAnalyser tarafından gözlenir not edilmelidir. Bu biyolojik önemi belirsiz kalmaktadır. Yeni transkripsiyonu RNA daha az ribozomal RNA içerdiğinden, bu örnekler bazen Agilent BioAnalyser kalite kontrolleri başarısız. Bu kabul edilebilir kalitede görünür RNA bozulması örnekleri nedeniyle değilse genellikle yüksek verimli analizine tabi olması iyi.
Aşağı akım analizleri ile yeni transkripsiyonu RNA Uyumluluk
Yeni transkripsiyonu RNA RNA çok daha fazla mRNA içerir. Bu 4SU-etiketleme süresi kısalır zaman artırmak yeni transkripsiyonu RNA intronik dizilerinin büyük miktarda kaynaklanmaktadır. Bu başlangıç materyalinin büyük miktarda gerektirir Bu nedenle, düzenli olarak yeni transkripsiyonu RNA örneklerinden rRNA tükenmesi taahhüt yok iken providolmayan rRNA ing daha sonra az (~ iki kat) kazanç okur. Son olarak, yeni nesil sekanslama için cDNA kitaplıkları hazırlama yeni zaman kopyalanan RNA içinde mevcut unspliced, yüksek moleküler ağırlığa sahip transkript daha büyük bir yüzdesi daha fazla parçalanma gerektirebileceği kaydedilmelidir gerekmektedir. Boyutu parçalanma adım Sonuçları nedenle kalite dikkatle kontrol olmalıdır.
RNA yarı canlı ölçümleri için veri normalleştirme
RNA, yarı-ömrü ölçümü için deneysel verileri normalize etmek için standart yaklaşım, RNA, iyi karakterize edilmiş ev tutma geninin yarı ömür ya da orta RNA önceki deneylerde belirlenen bir belirli hücre tipi içinde yarı ömrü tüm verileri normalleştirmek etmektir. Memeli hücrelerinde, ikinci 5-10 saat 6,7 aralığında yer alır. Bu yaklaşım aynı zamanda 4SU tabanlı ölçümler için oldukça güzel çalışıyor iken medyan RNA yarı ömrü ma bilinen veya eğer değilse, normalleşme için başka yollarla gerekliy bile knock-out bir RNA çürüme yolun ile örneğin, çalışma altında cep sisteminde değişiklikler etkilenebilir. 4SU-etiketleme üç RNA kesirler, yani toplam hücresel RNA, yeni transkripsiyonu RNA ve etiketsiz önceden var olan RNA analizine dayalı medyan RNA yarı ömrü tahmin benzersiz bir yol sunuyor. Toplam hücresel RNA, basit doğrusal regresyon modeli birbirine üç RNA fraksiyonlar normalleştirmek ve medyan RNA yarı ömrü 7,16 belirlemek için kullanılabilir ikincisi iki RNA fraksiyon ayrılır gibi. Bir yazılım paketi Bu analizler 22 gerçekleştirmek için çevrimiçi kullanılabilir.
Düşük üridin içerikli transkript verimsiz yakalama yapay olarak düşük yeni transkripsiyonu / total RNA oranları ve uzun süreli RNA yarı ömrü ile sonuçlanan RNA yarı ömrü ölçümü etkileyebilir. Bu sorunun ölçüde uridi karşı RNA yarı ömrü veya günlük (yeni transkripsiyonu / toplam RNA oranları) komplo ile değerlendirilebilirTüm transkript 7,15 ne içeriği. Bu aynı zamanda farklı numune veya koşullar arasında 4SU-birleşme oranlarındaki farklılıkları değerlendirmek için iyi bir kalite kontrol sağlar. Üridin içeriği için önemli bir korelasyon gözlenmiştir durumda bu biyoinformatik aracı 15 tarafından düzeltilebilir. Bununla birlikte, bu yeni kopyalanan RNA olgun transkript katkı kolayca daha büyük ve böylece çok daha üridin zengin öncülerden ayırt edilemez olduğu not edilmelidir. Belirli bir transkript işlenmesi kinetik (ki genellikle değildir) sadece (verimsiz yakalama) düşük üridin içerik düzeltme fena halde RNA yarı ömrü bozabilir bilinmektedir sürece. Bu nedenle, son zamanlarda 9 derece verimsiz olması en insan snoRNAs işlenmesi bulundu. Biz oldukça küçük ve düşük üridin içeriği (70-300 nt) için yeni transkripsiyonu / RNA oranları düzeltilmiş olsaydı snoRNAs, bu son derece kısa snoRNA yarı liv neden olurdu% 100 aşan sayıda yeni transkripsiyonu / toplam RNA oranları ile es (<5 dk). Bu nedenle, genellikle RNA yarı ömrü ölçerken düşük üridin içerik düzeltmek önermiyoruz.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yazarlar hiçbir rakip mali çıkarlarının olmadığını beyan ederim.
Acknowledgments
Biz yazının dikkatli bir okuma için Amie Regan teşekkür etmek istiyorum. Bu çalışma LD ve CCF için DFG hibe FR2938/1-1 için NGFN Artı hibe # 01GS0801, MRC dostluk hibe G1002523 ve NHSBT hibe WP11-05 tarafından desteklenmiştir
Materials
| Name | Company | Catalog Number | Comments |
| 4-thiouridine | Carbosynth | T4509 | Prepare 50 mM stock in sterile H2O, store at -20 °C in aliquots of 50-500 μl, discard unused reagent, do not refreeze. |
| Trizol | Invitrogen | 15596026 (100 ml), 15596018 (200 ml) | WARNING - CORROSIVE and HAZARDOUS TO HEALTH! Ensure immediate access to Phenol antidote (PEG-Methanol); Store at 4 °C. |
| Chloroform | Sigma | 372978 | WARNING - HAZARDOUS TO HEALTH |
| Isopropanol | Sigma | 650447 | |
| Sodium citrate, nuclease-free | Sigma | C8532 | Prepare 1.6 M stock solution using nuclease-free water. |
| 5M nuclease-free NaCl | Sigma | 71386 | Stock solution |
| Nuclease-free H2O | Sigma | W4502 | Make 1 ml aliquots in nuclease-free tubes. |
| RNA precipitation buffer | 1.2 M NaCl, 0.8 M sodium citrate in nuclease free water. Prepare in advance under strictly nuclease-free conditions. Store at room temperature in 50 ml falcon tubes. | ||
| Ethanol | Sigma | 459844 | Use with nuclease-free water to prepare 80% ethanol, store at -20 °C. |
| 1 M nuclease-free Tris Cl, pH 7.5 | Lonza | 51237 | Stock solution |
| 500 mM nuclease-free EDTA, pH 8.0 | Invitrogen | 15575-020 | Stock solution |
| 10x Biotinylation Buffer (BB) | 100 mM Tris pH 7.4, 10 mM EDTA in nuclease-free water, make aliquots of 1 ml. | ||
| Dimethylformamide (DMF) | Sigma | D4551 | |
| EZ-Link biotin-HPDP | Pierce | 21341 | Prepare 1 mg/ml stock solution by dissolving 50 mg biotin-HPDP in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. |
| Phase Lock Gel Heavy tubes 2.0 ml | Eppendorf | 0032 005.152 | Optional for the chloroform extraction step. |
| Zeta membrane | BIORAD | 162-0153 | |
| 10x Dot blot binding buffer | 100 mM NaOH, 10 mM EDTA | ||
| Biotin-oligo | 5'-biotin, 25 nucleotides, any sequence | ||
| Sodium dodecyl sulphate | Fisher | BPE9738 | For 100 ml 20% stock solution, add 20 g SDS to 80 ml PBS pH 7-8 and adjust volume to 100 ml. Keep all high-percentage SDS solutions above 20 °C. Warm the solutions slightly should SDS precipitate. |
| EZ-Link Iodoacetyl-LC-Biotin | Pierce | 21333 | Prepare 1 mg/ml stock solution by dissolving 50 mg iodoacetyl-biotin in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. Generates irreversible, thiol-specific biotinylation. |
| Phosphate buffer saline | Gibco | 10010-015 | |
| Dot blot blocking buffer | Mix 20 ml 20% SDS with 20 ml 1 x PBS pH 7-8 and add EDTA to the final concentration of 1 mM. | ||
| Streptavidin-horseradish peroxidase | Vector Laboratories | SA5004 | Store at -20 °C. Mix 10 ml 20% SDS with 10 ml 1 x PBS. Add 20 μl Streptavidin-HRP before use. |
| ECL reagent | GE Healthcare | RNP2109 | Use following the manufacturer's instructions. |
| Super RX, X-RA Film, 18x24 cm | Fujifilm | 47410 19236 | |
| μMacs Streptavidin Kit | Miltenyi | 130-074-101 | Store the beads at 4 °C. |
| Tween 20 | Sigma | P1379 | |
| Washing buffer | 100 mM Tris pH 7.4, 10 mM EDTA, 1 M NaCl, 0.1% Tween 20 in nuclease-free H2O. | ||
| Dithiothreitol (DTT) | Sigma | 43817 | Prepare as 100 mM DTT in nuclease-free H2O, always prepare fresh before use. |
| RNeasy MinElute Kit | Qiagen | 74204 | Store columns at 4 °C, remaining components of the kit at room temperature. |
| 1.5 ml screw-top polypropylene tubes | Sarstedt | 72.692.005 | Compatible with Dimethylformamide |
| 2.0 ml screw-top polypropylene tubes | Sarstedt | 72.694.005 | Compatible with Dimethylformamide |
| 15 ml tubes | BD Falcon | 352096 | Compatible with Dimethylformamide |
| 50 ml tubes | BD Falcon | 352070 | Compatible with Dimethylformamide |
| All solutions/reagents should be stored at room temperature unless otherwise specified. | |||
| Equipment | |||
| UV/VIS spectrophotometer | Thermo Scientific | NanoDrop 1000 | Or equivalent. Use low volume (1-2 μl) for measurements of low RNA concentrations to avoid excessive sample loss. |
| Polypropylene 15 ml centrifuge tubes | VWR International | 525-0153 | In contrast to standard 15 ml tubes, these tolerate up to 15,000 × g |
| High-speed centrifuge | Beckman Coulter | Avanti J-25 | Or equivalent equipment capable of reaching 13,000×g |
| High-speed rotor | Beckman Coulter | JLA-16250 | Or equivalent equipment capable of reaching 13,000×g |
| Adaptors for 15 ml tubes | Laborgeräte Beranek | 356964 | Or equivalent equipment capable of reaching 13,000×g |
| Refrigerated table-top centrifuge | Eppendorf | 5430 R | Or equivalent. |
| Thermomixer | Eppendorf | Thermomixer compact | Or equivalent. |
| Magnetic stand | Miltenyi Biotec | 130-042-109 | One stand holds 8 μMacs columns. |
| Waterbath | Grant | SUB Aqua 5 | Or equivalent. |
| Ultra-fine scale | A&D | GR-202 | Or equivalent. |
| E-Gel iBase Power System | Invitrogen | G6400UK | For RNA gels; or equivalent. |
| E-Gel EX 1% agarose precast gels | Invitrogen | G4020-01 | For RNA gels; or equivalent. |
References
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Mortazavi, A., Williams, B. A., Mccue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Nagalakshmi, U., Wang, Z., et al. The transcriptional landscape of the yeast genome defined by RNA sequencing. Science. 320, 1344-1349 (2008).
- Wilhelm, B. T., Marguerat, S., et al. Dynamic repertoire of a eukaryotic transcriptome surveyed at single-nucleotide resolution. Nature. 453, 1239-U1239 (2008).
- Dölken, L., Ruzsics, Z., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Yang, E., van Nimwegen, E., et al. Decay rates of human mRNAs: correlation with functional characteristics and sequence attributes. Genome Res. 13, 1863-1872 (2003).
- Friedel, C. C., Dölken, L., Ruzsics, Z., Koszinowski, U. H., Zimmer, R. Conserved principles of mammalian transcriptional regulation revealed by RNA half-life. Nucleic Acids Res. 37, e115 (2009).
- Nilsen, T. W., Graveley, B. R. Expansion of the eukaryotic proteome by alternative splicing. Nature. 463, 457-463 (2010).
- Windhager, L., Bonfert, T., et al. Ultra short and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Res. , (2012).
- Melvin, W. T., Milne, H. B., Slater, A. A., Allen, H. J., Keir, H. M. Incorporation of 6-thioguanosine and 4-thiouridine into RNA. Application to isolation of newly synthesised RNA by affinity chromatography. Eur. J Biochem. 92, 373-379 (1978).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23, 232-237 (2005).
- Kenzelmann, M., Maertens, S., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proc. Natl. Acad. Sci. U.S.A. 104, 6164-6169 (2007).
- Schwanhäusser, B., Busse, D., et al. Global quantification of mammalian gene expression control. Nature. 473, 337-342 (2011).
- Rabani, M., Levin, J. Z., et al. Metabolic labeling of RNA uncovers principles of RNA production and degradation dynamics in mammalian cells. Nat. Biotechnol. , (2011).
- Miller, M. R., Robinson, K. J., Cleary, M. D., Doe, C. Q. TU-tagging: cell type-specific RNA isolation from intact complex tissues. Nat. Methods. 6, 439-441 (2009).
- Miller, C., Schwalb, B., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Mol. Syst. Biol. 7, 458 (2011).
- Weintz, G., Olsen, J. V., et al. The phosphoproteome of toll-like receptor-activated macrophages. Mol. Syst. Biol. 6, 371 (2010).
- Lipsett, M. N. The isolation of 4-thiouridylic acid from the soluble ribonucleic acid of Escherichia coli. Journal of Biological Chemistry. 240, 3975-3978 (1965).
- Marcinowski, L., Liedschreiber, M., et al. Real-time Transcriptional Profiling of Cellular and Viral Gene Expression during Lytic Cytomegalovirus Infection. PLoS Pathog. 8, e1002908 (2012).
- Chomczynski, P., Mackey, K. Short technical reports. Modification of the TRI reagent procedure for isolation of RNA from polysaccharide- and proteoglycan-rich sources. Biotechniques. 19, 942-945 (1995).
- Friedel, C. C., Dölken, L. Metabolic tagging and purification of nascent RNA: implications for transcriptomics. Mol. Biosyst. 5, 1271-1278 (2009).
- Friedel, C. C., Kaufmann, S., Dölken, L., Zimmer, R. HALO - A Java framework for precise transcript half-life determination. Bioinformatics. , (2010).



