Summary
Всего клеточной РНК обеспечивает плохое шаблон для изучения краткосрочных изменений в синтез РНК и распада, а также кинетики процессинга РНК. Здесь мы описываем метаболический маркировки вновь транскрипции РНК с 4-тиоуридина последующим тиол-специфический биотинилирования и очистки вновь транскрипции РНК позволяющих преодолеть эти недостатки.
Abstract
Развитие целого транскриптома микрочипы и следующего поколения секвенирования революцию в нашем понимании сложности клеточной экспрессии гена. Наряду с лучшего понимания молекулярных механизмов, вовлеченных, точные измерения основных кинетики становятся все более важными. Здесь эти мощные методологии сталкиваются с серьезными ограничениями в силу внутренних свойств шаблона образцы, которые они изучают, т.е. общей клеточной РНК. Во многих случаях изменения общей клеточной РНК происходить либо слишком медленно или слишком быстро, чтобы представлять основные молекулярные события и их кинетики с достаточным разрешением. Кроме того, вклад в изменение синтеза РНК, обработки и распада не легко дифференцированы.
Мы недавно разработали высокого разрешения профилирования экспрессии генов для преодоления этих ограничений. Наш подход основан на метаболические маркировки вновь транскрипции РНК с 4-thiouriеды (так называемый также 4SU-тегов), а затем строгий очистки вновь транскрипции РНК с использованием тиол-специфический биотинилирования и покрытые стрептавидином магнитные гранулы. Он применим к широкому кругу организмов, включая позвоночных, дрозофилы и дрожжей. Мы успешно применяется 4SU-тегов для изучения реального времени кинетики активности транскрипционных факторов, обеспечивают точные измерения РНК период полураспада, и получить новые понимания кинетики процессинга РНК. Наконец, компьютерное моделирование может быть использовано для создания комплексного, всестороннего анализа основных молекулярных механизмов.
Introduction
Профилирования экспрессии генов является ключевым инструментом, используемым для изучения клеточных процессов и связанных с ними сложная сеть взаимодействий. Исследования по мРНК изобилие как правило, были методом выбора для получения основной проницательности в основные молекулярные механизмы. Развитие целого транскриптома микрочипов 1 и, совсем недавно, следующего поколения секвенирования РНК (РНК-SEQ) 2-4 подпитывается этот подход. Однако эти технологии пока революцию в нашем понимании сложности клеточной экспрессии гена, они сталкиваются с серьезными ограничениями в силу внутренних свойств их шаблон образца, т.е. общей клеточной РНК. Во-первых, краткосрочные изменения общего уровня РНК не соответствуют изменениям в транскрипции ставки, но по своей природе в зависимости от РНК полураспада соответствующих транскриптов. В то время как пятикратный индукции недолгим стенограммы, например, кодирование для фактора транскрипции, будет легко обнаружить в общей РНКв течение часа, то же индукция долгоживущих стенограммы, например, кодирование для метаболических ферментов, останется практически невидимым. Кроме того, даже полное отключение (> 1000 раз понижающая регуляция) в транскрипции скорость средний ген РНК полураспада пяти часов будут просто принимать пять часов для общего уровня РНК уменьшить лишь два раза . Таким образом, анализ общей РНК способствует обнаружению повышающей регуляции недолго транскриптов, многие из которых кодируют факторы транскрипции и гены регулирующие функции 5. Кроме того, истинная кинетическая каскад регулирования затемняется и первичных сигнальных событий не может быть дифференцирована от вторичной. И, в свою очередь, может привести к существенной смещение в последующих анализов биоинформатики. Во-вторых, изменения в общих уровней РНК не могут быть отнесены к изменениям в синтез РНК или распада. Измерения последние требуют инвазивных подходов клетки, например, блокирование transcriptiпо использованию актиномицином D 6 и расширенный мониторинг текущих распада РНК с течением времени. При средней мРНК период полураспада в клетках млекопитающих, 5 - 10 ч 5,7, уровни мРНК большинство генов только уменьшились на менее чем в два раза после нескольких часов транскрипционный сердца. Эти довольно небольшие различия привести грубую и неточную мРНК измерений периода полураспада для большинства клеточных генов в связи с экспоненциальным характером основного математических уравнений. Наконец, в то время как РНК-сл полной клеточной РНК показал, что приблизительно половина наших генов могут быть альтернативные сплайсинга 8, лежащие в основе кинетики, так и динамических механизмов направляющий ткани и зависит от конкретных условий регулирования процессинга РНК еще мало изучены. Кроме того, вклад процессинг РНК к дифференциальной экспрессии генов, в частности для некодирующих РНК, еще предстоит определить. В целом, эти ограничения представляют собой серьезные препятствия длябиоинформационных кинетических моделей основных молекулярных механизмов.
Недавно мы разработали подход, названный высоким разрешением профилирования экспрессии генов, чтобы преодолеть эти проблемы 5,7,9. Он основан на метаболические маркировки вновь транскрипции РНК с использованием 4-тиоуридина (4SU-тегов), встречающегося в природе уридином производной, и обеспечивает прямой доступ к новой транскрипции стенограммы с минимальным вмешательством в рост клеток и экспрессии генов (см. Рисунок 1) 5, 10-12. Воздействие эукариотической клетки 4SU приводит к ее быстрому поглощению, фосфорилирования 4SU-трифосфат, и включения в новый транскрипции РНК. После выделения общей клеточной РНК, 4SU-меченой РНК фракции тиол-конкретно биотинилированный генерации дисульфидную связь между биотин и вновь транскрипции РНК. "Общую клеточную РНК 'может быть количественно разделить на помечены (' вновь транскрипции ') и немеченого (' предварительно existinг ') РНК с высокой чистотой использованием покрытых стрептавидином магнитных шариков. Наконец, меченой РНК выделяют из шариков путем простого добавления восстанавливающего агента (например дитиотреитола) расщепление дисульфидных связей и выпуск новой транскрипции РНК из шариков.
Недавно транскрипции РНК изображает транскрипционной активности каждого гена в течение срока от 4SU экспозиции. 4SU-тегов в сроки минут таким образом, обеспечивает картинку эукариотической экспрессии генов и идеальный шаблон для вниз по течению биоинформатики анализов (например, промоутер анализ). В случаях, когда стационарных условиях можно предположить, отношения вновь транскрипции / общий, недавно транскрибируется / немеченными и немеченым / общей РНК обеспечивает неинвазивный доступ к точной РНК полураспада 7,13. Кроме того, важно отметить, что недавно транскрипции РНК очищенный после всего лишь 5 мин 4SU-тегов (5 мин 4SU-РНК) моложе 15 и 60 мин 4SU-РНК.При выполнении как ультра-короткие и прогрессивно больше 4SU-тегов в одну экспериментальную установку в сочетании с РНК-SEQ, кинетика процессинг РНК выявлены у нуклеотидных резолюции 9. Наконец, время курса анализы вновь Записал и общей РНК в сочетании с компьютерного моделирования позволяют интегративный анализ синтез РНК и распада 14.
В заключение, этот подход позволяет для прямого анализа динамики синтеза РНК, обработки и деградации в эукариотических клетках. Она применима во всех основных модельных организмов, включая млекопитающих, насекомых (Drosophila), земноводные (Xenopus) и дрожжи 5,15,16. Это непосредственно совместимы с анализ микрочипов 5,17 РНК-далее 9,13,14 и применимо в естественных условиях 12,15. Здесь мы подробно описываем методику на этикетке, изолировать и очистить вновь транскрипции РНК в культивируемых клетках млекопитающих. Кроме того, потенциометрдр. проблем и подводных камней, обсуждаются.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Метаболический маркировки с 4-тиоуридина
Сделать подробный план экспериментальной установки / по расписанию, например, когда, чтобы добавить 4SU в культуре клеток и, когда собрать образцы. План, по крайней мере 5 мин между каждым состоянии. Только лечить клетками одно условие за один раз. Ручка макс. 3 - 5 блюд в данный момент времени. Ручка клетки как можно быстрее, чтобы свести к минимуму изменения температуры и уровень СО2. Избегайте воздействия на клетки яркий свет после 4SU добавляется как это может привести к сшиванию 4SU-меченой РНК с клеточными белками.
Начало маркировки
- Оттепели 4-тиоуридина (4SU) непосредственно перед использованием и пипеткой требуемое количество 4SU для каждого условия в стерильную пробирку Фалькон.
- Отобрать необходимое количество клеточной культуральной среде (5 мл на 10 см чашку) от тарелки и добавить к 4SU содержащие трубки сокола и тщательно перемешать. Снимите и выбросьте оставшийся среды из блюд. <li> Применить 4SU среде, содержащей Вернуться к блюдам.
Конец маркировки
- Удалите среду культуры клеток из клеток. Добавить 5 мл Trizol К каждой пластине. Для сложных экспериментов в том числе различные моменты времени или условий, этот шаг лучше всего сделать два человека, один удаления среды, другой добавления Trizol и уборки лизата.
- Инкубировать в течение 5 мин при комнатной температуре в течение полного лизиса клеток.
- Используйте пипетки на 10 мл для промывки пластины тщательно с дополнительным Trizol. Это помогает полный лизис клеток и восстановления образца. Обращаться с осторожностью, поскольку Trizol крайне опасно при попадании на кожу или в глаза! Есть противоядие фенола ожогов под рукой (например, полиэтиленгликоль 300 или 400 в технический денатурат (70:30)). Передача образцов в полипропиленовые пробирки. Обратите внимание, что стандартные труб Сокол не противостоять этим силам высокие г). Образцы можно хранить при -20 ° С в течение по крайней мере одного месяца до полной РНК яс готовым.
2. Получение РНК с использованием модифицированного протокола Trizol
- Добавить 1 мл хлороформа (0,2 мл на мл Trizol) и энергично встряхивают в течение 15 сек. Инкубируют при комнатной температуре в течение 2 - 3 мин.
- Центрифуга при 13000 х г в течение 15 мин при 4 ° С.
- Передача водный верхнюю фазу (содержащую РНК) в новую 15 мл полипропиленовую пробирку.
- Добавьте ½ реакционного объема и РНК осадки буфер и изопропанола (например, до 3 мл надосадочной жидкости добавляют 1,5 мл РНК осадки буфера и 1,5 мл изопропанола).
- Хорошо перемешать. Инкубируют при комнатной температуре в течение 10 мин.
- Центрифуга при 13000 х г в течение 10 мин при 4 ° С. Удалите супернатант.
- Спином вниз кратко (5000 × г в течение 30 сек) и удаления остаточного изопропанола с 200 мкл пипетки.
- Добавить равный объем 75%-ного этанола и встряхнуть трубку, пока осадок отделяется. Избегайте разбив его на множество мелких частей, как это может сделать удаление остатковл этанола трудно.
- Центрифуга при 13000 х г в течение 10 мин при 4 ° С. Удалите супернатант.
- Спином вниз РНК коротко и удалить остатки этанола с 200 мкл пипетки. Повторите шаг и удалить остатки этанола с 20 мкл пипетки. После этих двух шагов, без дальнейшей сушки гранул должна быть выполнена.
- Добавить 100 мкл H 2 O на 100 мкг РНК ожидается выход и хорошо перемешать с помощью пипетки вверх и вниз 5 - 6 раз, чтобы помочь в растворении РНК.
- Растворить и денатурации РНК при нагревании до 65 ° С в течение 10 мин (шейкер) и сразу же помещают на лед.
- Измерение концентрации РНК при 260 нм с использованием спектрофотометра NanoDrop, следуя инструкциям производителя. Эта РНК можно хранить при -80 ° С в течение по крайней мере одного месяца.
3. Тиол конкретных Биотинилирование Недавно Transcribed РНК
- Начнем с 60 - 80 мкг полной клеточной РНК.
- Составляют маркировки реакции. Пипетка в следующихпоручения (за мкг РНК):
- 1 мкл 10х буфера Биотинилирование
- 7 мкл РНК (содержащего 1 мкг РНК разводят в нуклеазы без H 2 O)
- 2 мкл биотин-HPDP (1 мг / мл ДМФА)
Всегда добавляйте биотин-HPDP последний немедленно и смешайте с помощью пипетки. В случае, биотин осадка, ДМФ содержание может быть увеличено до конечной концентрации 40%.
- Инкубируют при комнатной температуре в течение 1,5 часов с вращением.
- Добавить равный объем хлороформа. Смешайте энергично. Выдержите в течение 2 - 3 минут, пока фаз не начинают отделяться и пузыри начинают исчезать.
- Центрифуга при 20000 х г в течение 5 мин при 4 ° С. Тщательно передачи верхней водной фазы в новую пробирку.
- Повторите шаги 3.4 и 3.5 раза. Вы можете выполнить этот шаг в 2 мл Phase Lock Гель толстостенных труб для уменьшения потери РНК.
- РНК осадков: добавить 1/10 объема 5 М NaCl и равного объемаизопропанола в водной фазе.
- Центрифуга при 20000 х г в течение 20 мин при 4 ° C. Удалите супернатант.
- Добавить равный объем 75%-ного этанола, центрифугируют при 20000 х г в течение 10 мин при 4 ° С, отбросить супернатант.
- Спин коротко и удаления остаточного этанола с 200 мкл пипетки.
- Спин коротко и удаления остаточного этанола с 20 мкл пипетки.
- Не допускать РНК высохнуть. Повторное приостановить его в 50 - 100 мкл H 2 O (~ 1 мкл на 1 мкг входного РНК). Хорошо перемешать с помощью пипетки вверх и вниз 5 - 6 раз.
- Проверьте качество РНК электрофоретического анализа, чтобы исключить деградации РНК.
4. Дот-блот анализ 4SU-включения (необязательно)
4SU регистрации может быть легко определено дот-блот-анализ биотинилированных РНК. Это необязательный шаг, который позволяет устранения неисправностей и оценки 4SU цены по отношению к включению контрольной ДНК биотинилированных олиго. Для этого анализа мы тecommend использованием йодацетильную-биотин вместо биотин-HPDP для биотинилирования 4SU-меченой РНК в пункте 3.2. Это приводит к необратимому биотинилирования 4SU-РНК. Таким образом, на основе столбцов методами (например RNeasy) может быть использован для восстановления значительно меньших количествах биотинилированного РНК (например, 5 мкг). В то время как РНК биотинилированный использованием биотин-HPDP также подходит для этого анализа, результирующий сигнал слабее и отношение сигнал-шум менее благоприятной (фиг. 3).
- Следуйте 4SU протокол для маркировки и выделения общей клеточной РНК, как описано в разделах 1 и 2.
- Biotinylate 4SU-меченой РНК, как описано в разделе 3 заменив биотин-HPDP с йодацетильную-биотин и выполняют две хлороформом, чтобы полностью удалить чрезмерное йодацетильную-биотин остатков.
- Восстановление биотинилированного РНК изопропанол / осаждение этанолом, как описано, или с использованием колонки подход (например RNeasy) в случае небольших количеств РНК (<10 мкг) Используются.
- Выдержите мембраны Zeta в нуклеазы без воды с качания в течение 10 мин.
- Возьмите мембрана из нуклеазы без воды и удаления избытка жидкости путем размещения мембраны в период между двумя чистыми полотенцами бумаги и сильно нажимая. Воздушной сушки мембраны в течение 5 мин приведет к более хорошей точки.
- Для каждого образца, готовят 20 мкл 200 нг / мкл РНК с использованием ледяной дот-блот-связывающем буфере (10 мМ NaOH, 1 мМ ЭДТА). Применение 5 мкл этого раствора (т.е. 1 мкг РНК), а также трех последующих 10-кратного разведения (например, 100, 10 и 1 нг РНК соответственно) на мембрану Zeta с помощью пипетки. Пипетирование через пустой стойке пипеток можно использовать для обеспечения равномерно распределен интервал. Кроме того, использование аппарата дот-блот в соответствии с инструкциями производителя.
- Применение 5 мкл меченного биотином олигонуклеотидов ДНК в концентрациях от 20 нг / мкл до 20 пг / мкл (то есть от 100 до 0,1 нг олиго) в качестве положительного продолжениерол на мембрану с помощью пипетки. Используйте биотинилированными 4SU-наивные образца в качестве отрицательного контроля.
- Воздушной сухой мембраны в течение 5 мин.
- Инкубируйте мембраны в течение 30 мин в 40 мл блокирующего буфера с качалки.
- Инкубируйте мембраны с 10 мл 1:1000 стрептавидин-пероксидаза хрена в течение 15 мин (5 мл PBS + 5 мл 20% SDS + 10 мкл стрептавидин-пероксидаза хрена)
- Мытье мембраны дважды в 40 мл PBS + 10% SDS (20 мл PBS + 20 мл 20% SDS) в течение 5 мин.
- Мытье мембраны дважды в 40 мл PBS + 1% SDS (38 мл PBS + 2 мл 20% SDS) в течение 5 мин.
- Мытье мембраны дважды в 40 мл PBS + 0,1% SDS (40 мл PBS + 200 мкл 20% SDS) в течение 5 мин.
- Удалить излишки жидкости путем размещения мембраны в период между двумя чистыми бумажными полотенцами и нажав на них твердо.
- Визуализируйте мембраносвязанные HRP использованием ECL соответствии с инструкциями производителя.
- Поместите мембраны в пленку / мешок, удаления пузырьков воздуха и инкубировать в течение 2 мин в темноте.
- Expose мембраныфильм в течение 1 - 5 мин.
5. Разделение мечеными и немечеными РНК с использованием Стрептавидин-магнитных шариков, покрытых
- Тепло промывочным буфером (3 мл на образец) до 65 ° С на водяной бане.
- Подготовьте свежий 100 мМ дитиотреитола (DTT) в нуклеазы без H 2 O. Сделайте так, отстаивают 15 - 30 мг DTT порошок в чистый 50 мл Сокол трубки размещены на ультра-тонкого масштаба. Взвесьте и добавьте необходимое количество нуклеазы без H 2 O.
- Тепло биотинилированный образцы РНК до 65 ° С в течение 10 минут для денатурации и сразу же помещают на лед.
- Место μMacs столбцов в магнитную подставку. Мы рекомендуем не обрабатывать более 12 образцов одновременно (6 - 8 проб оптимальны).
- Предварительно равновесие Miltenyi колонн с 1 мл комнатной температуры промывочного буфера. Это займет около 15 мин.
- Между тем, добавляют 100 мкл шариков стрептавидином до 50 - 100 мкл биотинилированного РНК. Инкубируют при комнатной температуре в течение 15 мин с вращением. < литий> Если любой из столбцов не инициировал слива уже это может способствовать легком нажатии на верхнюю часть колонны с палец в перчатке. Как только поток начал столбцов легко слить.
- Применить РНК / шарики к колоннам. Откажитесь от проточных если вы не хотите, чтобы восстановить немеченой фракций РНК (см. раздел 7).
- Вымойте три раза с 0,9 мл 65 ° C стиральными буфер (1 мл пипеток уменьшаться, когда пипетки буферов при 65 ° C).
- Мытье три раза с 0,9 мл комнатной температуры промывочного буфера.
- Внесите 700 мкл буфера RLT (RNeasy Kit Очистка MinElute, Qiagen) в новые 2 мл пробирок и поместите их под колоннами.
- Элюировать вновь транскрипции РНК в RLT буфер добавлением 100 мкл 100 мМ DTT в столбцах.
- Выполните второй элюирования раунд 3 минут позже в ту же трубу, добавив еще 100 мкл 100 мМ DTT.
6. Восстановление Недавно Transcribed РНК
ontent "> Продолжить RNeasy Очистка MinElute (Qiagen) протокол в соответствии с инструкциями производителя. Elute в 25 мкл нуклеазы без H 2 O. измерения концентрации РНК с использованием спектрофотометра Nanodrop. Чтобы избежать необходимости оттаивать и повторно заморозить РНК перед отправкой его высокой пропускной способности анализа, рекомендуется подготовке кДНК сразу же после недавно транскрипции РНК очищали. Использование 2,5 мкл вновь транскрипции РНК в 20 мкл кДНК, смесь синтез для синтеза кДНК в соответствии с инструкциями производителя. Выполнить QRT-PCR управления, используя один : 10 разведений кДНК смесь магазин РНК при -80 ° С..7. Восстановление без маркировки, несвязанный РНК (необязательно)
В случае несвязанного РНК должна быть восстановлена, собирать и комбинировать проточные (после добавления РНК-стрептавидин бисер решение столбцы) и первой стирки для последующего осаждения. Обычно достаточно, чтобы осадить только 50% от несвязанного РНК, как эту темусек будет содержать> 80% от исходного материала.
- Добавить равным объемом изопропанола (соль не должен быть добавлен в качестве промывочного буфера уже содержит 1 М NaCl).
- Центрифуга при 20000 х г в течение 20 мин при 4 ° C. Удалите супернатант.
- Добавить равный объем 75%-ного этанола, центрифугируют при 20000 х г в течение 10 мин при 4 ° С, отбросить супернатант.
- Спин коротко и удаления остаточного этанола с 200 мкл пипетки.
- Спин коротко и удаления остаточного этанола с 20 мкл пипетки.
- Не допускать РНК высохнуть. Ресуспендируйте это в 100 мкл H 2 O. Хорошо перемешать с помощью пипетки вверх и вниз 5 - 6 раз. Инкубируют при 65 ° С в течение 10 мин при встряхивании и передавать непосредственно в лед.
- Проверьте качество РНК электрофоретического анализа, чтобы исключить деградации РНК.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
1. Исходного материала и ожидаемых выходов
После 1 часа (ч) 4SU экспозиции вновь транскрипции РНК составляет около 1 - 4% от общей клеточной РНК. Это будет ниже роста арестован клетки, поскольку они больше не синтезировать РНК на счет для роста клеток / репликации. Когда маркировка в течение 1 ч, рекомендуется началом анализа с 60 - 80 мкг общей РНК. Начиная с менее чем 30 мкг общей РНК результаты в небольших гранул РНК, которые трудно увидеть после биотинилированием шаг и, следовательно, может быть легко потерян. Входные уровни РНК может быть увеличен до целых 150 мкг на очень короткие длительности маркировки (например, 5 - 10 мин). Когда продолжительность РНК маркировки укорачивается от 1 часа до 5 мин вклад короткоживущих последовательности интронов во вновь транскрипции РНК увеличивается от ~ 60% и ~ 80% 9. Как интроны существенно дольше по сравнению с кодирующими последовательностями, а также 5'-и 3'-НТО, количество вновь транскрибируетсяРНК, которая может быть очищена после короткого или даже ультра-короткие 4SU-тегов, не падает линейно. Таким образом, мы получили> 0,5% от общего числа РНК после 5 мин 4SU-тегов в неадгезивные человеческих В-клеточных линий 9. Следует, однако, отметить, что более высокая концентрация 4SU и немного более длительные периоды маркировки могут быть необходимы для достижения аналогичных 4SU цены включение в прилипшие клетки. В то время как даже низкая 4SU-включение Курс позволит эффективного захвата и очистки больших, богатых уридином стенограммы, очень короткая стенограмма с низким содержанием уридином (например, микроРНК), вероятно, чтобы избежать очистки даже при использовании высоких концентраций 4SU (> 1 мм). В NIH-3T3 мышиных фибробластов, 1 час 200 мкМ 4SU воздействия помечены вновь транскрипции РНК с приблизительно одним 4SU остатка на 50 - 100 нуклеотидов (нт) 5. Это должно позволить высокоэффективного восстановления транскриптов> 500 - 1000 NT в длину. Соответственно, мы только наблюдали незначительные размеры стенограммысмещения при маркировке в течение 1 часа с использованием 200 мкМ 4SU как в мышиных фибробластов и человеческих В-клеток 7. В то время как 1 час 200 4SU мкм не приводит к существенным изменениям в клеточном уровнях транскриптов в мышиных фибробластов, длительного воздействия на клетки ≥ 200 мкмоль 4SU действительно приводит к измеримым рост дефицита в течение 24 ч (неопубликованные данные). Поэтому и продолжительность маркировки и занятого 4SU-концентрация должна быть сведена к минимуму, чтобы избежать внематочной или токсических эффектов. Самый простой способ определить минимальные 4SU-концентрация, необходимая для эффективного восстановления вновь транскрипции РНК состоит в очищении вновь транскрипции РНК после 4SU маркировки с увеличением концентрации 4SU (например, 50 - 1600 мкм). Как показано на фигурах 2А и 2В, восстановление недавно транскрипции РНК помечены в течение 1 часа в первичных человеческих фибробластов значительно увеличилось от 50 до 200 мкм 4SU но потом начал плато.
2. ТочкаТех Количественная 4SU регистрации (опционально)
В некоторых случаях это может представлять интерес для измерения количества 4SU включение в общей РНК. Это лучше всего сделать, дот-блот анализ на биотинилированному РНК с использованием сопряженных стрептавидином. Благодаря своей химической природе йодацетильную-биотин более реактивный тиоловых групп, чем биотин-HPDP в результате чего биотинилирования 4SU практически все остатки в новой транскрипции РНК. Важно отметить, что, как биотин-HDPD, йодацетильную-биотин не растворимы в воде и, таким образом, эффективно удалены хлороформом как выполняется для биотин-HPDP. Таким образом, идентичных условиях реакции и концентрации может быть использован как при использовании биотин-HPDP. Тем не менее, йодацетильную-биотин не является обратимым. Она может, следовательно, не будет использоваться для очистки вновь транскрипции РНК в колонке подходов. Хотя использование йодацетильную-биотин позволяет количественно 4SU-регистрации, биотин-HPDP измерениям учитывать как4SU-регистрации и биотинилированием эффективности. Применение двух реагентов биотинилирования в том же образце дает возможность измерения биотинилирования эффективность РНК-объединенный 4SU. Биотинилирование эффективность биотин-HPDP для 4SU-меченой РНК, кажется, примерно в три раза меньше, чем у йодацетильную-биотин, указывающее, что только один из трех 4SU остатков в новой транскрипции РНК фактически биотинилированный по биотин-HPDP (фиг. 3). Путем сравнения интенсивности образец сигнала с биотинилированным контрольной ДНК олигонуклеотидов, биотинилирования плотность может быть измерена. Для большинства клеточных линий млекопитающих положительный сигнал все еще должен быть обнаружен в 10 нг биотинилированному РНК после 1 часа 200 мкМ 4SU маркировки. Слабый сигнал фона, как правило, обнаруживаемых на самой высокой концентрацией (1 мкг) немеченого РНК.
3. Очистка Недавно Transcribed РНК
Восстановление вновь транскрипции РНК очень Цюаньколичественные. Если Вы начали с той же концентрацией РНК вы можете рассчитывать на получение таких же количествах вновь транскрипции РНК для всех образцов. Как и многие колонке основе анализов, сбора вновь транскрипции РНК с использованием набора RNeasy MinElute может привести к дополнительному поглощению при 230 - 260 нм (наличие моющие средства, полученные из стиральной буферов), которые могут мешать OD 260 измерений. Это видно в меньшей степени при использовании свежего 2 мл пробирку для каждой стадии центрифугирования. Тем не менее, любой OD необоснованно высоких измерений (> 2 раз больше, чем другие образцы) следует рассматривать с осторожностью, особенно, если OD 260/280 соотношения <1,7. Для вниз по течению анализов, таким образом, часто лучше всего использовать такое же количество объем РНК-матрицы для всех образцов. В случаях, когда выходы меченой РНК ниже, чем ожидалось, проверка на наличие признаков деградации РНК с помощью электрофоретического анализа. Недавно транскрибируется РНК содержит значительно большее количество большой, несплайсированные стенограммы с типичной полосы рРНК будучи гораздо менее заметными (рис. 4).
4. Количественная Недавно Transcribed РНК
Наконец, включение цены 4SU во вновь транскрипции РНК может быть непосредственно количественно спектрофотометрически по поглощению 4SU максимум при 330 нм и OD 330/260 соотношение 5,18. Это требует> 3 мкг меченой РНК сосредоточены в небольшом количестве (10 - 20 мкл) при изопропанол / осаждением этанолом. Чтобы избежать потери малых РНК гранул соосаждения с 30 мкг нуклеазы без гликогена (Fermentas, # R0551) должны быть выполнены. Дополнительный пик виден при 330 нм, отражающие включение скорость 4SU во вновь транскрипции РНК (рис. 5).
/ Files/ftp_upload/50195/50195fig1highres.jpg "/>
Рисунок 1. . Принцип метаболический маркировки 4-тиоуридин (4SU) 4SU добавляют к клеткам для требуемого (5 - 120 мин) время последующей подготовки общей клеточной РНК. После тиол-специфический биотинилирования общей клеточной РНК разделяется на 4SU меченные, недавно транскрипции РНК и немеченого, уже существующие РНК с использованием покрытых стрептавидином магнитных шариков. Недавно транскрибируется РНК выделяют из бисера с использованием восстановителя, который расщепляет дисульфидные связи, которые связывают вновь транскрипции РНК с шариками. Нажмите здесь, чтобы увеличить рисунок .
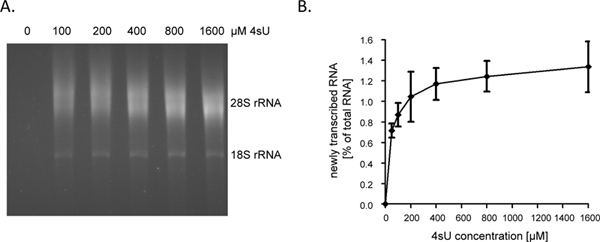
Рисунок 2. Восстановление вновь транскрипции РНК после возрастающих концентраций 4SU. (А) Первичный фибробласты крайней плоти человека (HFF) инкубировали с 100, 200, 400, 800 или 1600 мкМ 4SU. Недавно транскрипции РНК очищали от 50 мкг полной клеточной РНК и подвергали электрофоретического анализа. Как и ожидалось, зависимое от концентрации увеличение восстановлены вновь транскрипции РНК наблюдалось который начал плато при более высоких концентрациях. (B) Средства очищенной вновь транскрипции РНК оценивали количественно с помощью программного обеспечения ImageJ 1.45s. Комбинированные данные четырех независимых экспериментах на суммы, вновь транскрипции РНК восстановился после различных концентраций 4SU маркировки либо в пределах от 50 - 800 мкм 4SU (N = 2) или 100 -. 4SU 1600 мкМ (n = 2) показаны Нажмите здесь , чтобы увеличить рисунок .
upload/50195/50195fig3.jpg "Alt =" Рисунок 3 "FO: Content-ширина =" 4,5-дюймовой "FO: SRC =" / files/ftp_upload/50195/50195fig3highres.jpg "/>
Рисунок 3. Оценка 4SU включение в 4SU меченных общей РНК с использованием дот-блот-анализа. Тотальная РНК была выделена из NIH-3T3 фибробластов мыши или фибробласты крайней плоти человека (HFF) инкубировали с 200 мкМ 4SU течение одного часа. Нет 4SU не был добавлен в одно блюдо в качестве отрицательного контроля. Для HFF ингибирует оба контакта (N = межвегетационных клетки) и растущие клетки (у) были включены. РНК выделяли с помощью Trizol реагента и впоследствии конъюгированного с биотином HPDP или йодацетильную-биотин. Концентрация каждого образца доводили до 200 нг / мкл и 5 мкл этого раствора (т.е. 1 мкг РНК), а также трех последующих 10-кратного разведения (т.е. 100, 10 и 1 нг РНК, соответственно), были наносили на кусок мембраны Зета. 5 мкл разведения меченого биотином ДНК олиго были размещены на мембраны в качестве положительного контроля в Concentratионов в диапазоне от 20 нг / мкл до 20 пг / мкл (то есть от 100 до 0,1 нг соответственно). Биотин плотность зондировали использованием стрептавидин-пероксидазы хрена.
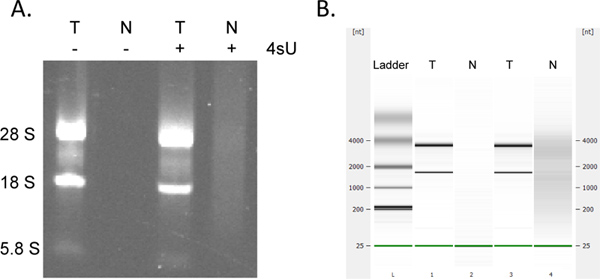
Рисунок 4. Электрофоретического анализа вновь транскрипции и тотальную РНК. Общую РНК (Т) и вновь транскрипции РНК (N), полученных из мышиных NIH-3T3 фибробласты культивировали как в присутствии и в отсутствие 500 мкМ 4SU в течение 1 часа анализировали с помощью электрофореза в агарозном геле (A) и (в том же порядке), используя Bioanalyser Agilent (B). Нет РНК был извлечен без 4SU обработка клеток. Очищенная РНК транскрибируется вновь содержит большее количество мРНК высоким молекулярным весом и значительно меньше, чем зрелых рРНК общегоРНК в качестве заметных между 28S, 18S, 5.8S рРНК и групп. Нажмите здесь, чтобы увеличить рисунок .
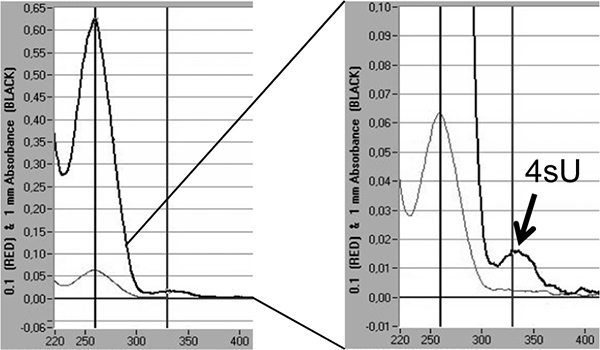
Рисунок 5. Количественная оценка 4SU включения во вновь транскрипции РНК спектрофотометрически. Недавно транскрипции РНК очищали от 2 х 100 мкг общей РНК после 1 часа 200 мкМ 4SU в мышиных NIH-3T3 фибробластов. Недавно транскрипции РНК осаждали с помощью изопропилового спирта / этанола после добавления 30 мкг нуклеазы без гликогена. Спектрофотометрический анализ вновь транскрипции РНК, полученной по Nanodrop 1000 спектрофотометр показано. Светлые линии серым цветом, измерения на 0,1 мм, а более толстая, темно серые линии представляют измерения в 1 мм столба жидкости. На правом, увеличением пика вымирания представляющих тон включил 4SU-остатков показана. На основе вымирания коэффициентом 4SU 18 включение цены 4SU может быть оценена.
| Продолжительность маркировки [мин] | Рекомендуемая концентрация 4SU [мкм] |
| 120 | 100 - 200 |
| 60 | 200 - 500 |
| 15 - 30 | 500 - 1000 |
| <10 | 500 - 2000 |
Таблица 1. Рекомендуем 4SU концентрациях.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Метаболический маркировки вновь транскрипции РНК существенно повышает мощность высокопроизводительных технологий, как микрочипы и РНК-SEQ, предоставляя более подходящие шаблоны для решения биологических вопросов, представляющих интерес. Настоящий Протокол проводился капитальный оптимизации. Это позволяет> 1000-кратное обогащение вновь транскрипции РНК и обеспечивает высокую воспроизводимость результатов.
Экспериментальный дизайн 4SU-тегов эксперимент имеет решающее значение, как недавно транскрипции РНК будет изображать в реальном времени активность транскрипции только во время воздействия на клетки 4SU. Если фактические изменения в транскрипции цены после стимул уже утихли, эти будут пропущены при анализе вновь транскрипции РНК даже если изменения общего уровня РНК может все еще быть обнаружены. Таким образом, хорошее понимание основных биологии важно определить экспериментальную установку, а также оптимальные сроки выводаF 4SU время для экспозиции. Ниже мы приводим рекомендации и способы для избежания типичных ошибок для наиболее важных шагов.
Приготовление растворов и пластиковая посуда
Все растворы должны быть приготовлены с использованием нуклеазы без воды. Использование в доме очищенной деионизированной воды может привести к проблемам, если в воде содержится восстановителей. В одном случае это привело к полной потере всех меченой РНК. Поэтому мы настоятельно рекомендуем покупать предварительно сделаны нуклеазы без NaCl, Трис-Cl, EDTA, цитрат натрия и воды. Убедитесь, нуклеазы без условия в любое время. Диметилформамид (ДМФА) растворяет некоторые пластмассы. Мы обнаружили, что использование 25 мл пипетки клетки культуры пластиковых передать DMF от стеклянной бутылки Фондовой 50 мл Сокол труб подготовить биотин-HPDP маточный раствор недостаточно для существенного сокращения выходы вновь транскрипции РНК из цельной. Интересно, что это отрицательно не влияет на биотинилAtion эффективность (как проверено дот-блот), но в результате 75 до> 90% потери вновь транскрипции РНК, которые могут быть извлечены из шариков. Потеря была наиболее выраженной, когда длительность маркировки была уменьшена с 60 до 30 мин или меньше. Скорее всего, вещество элюировали из пластиковой пипетки в ДМФА частично разрушены покрытия стрептавидином шариками. Таким образом, использование пластиковых материалов, как известно, не совместимы с ДМФА следует избегать всеми средствами. По тем же причинам, сотовые скребки не должны быть использованы для повышения восстановление Trizol образцы пластин клеточной культуры. Интересно отметить, что предполагаемый веществ элюированы из пластмасс ДМФ или Trizol не было видимо ни удалить, ни хлороформом изопропанол / осаждением этанолом.
Клеточные культуры
Плотность клеток на пластинах имеет решающее значение. В одном эксперименте, где клетки оказались немного слишком сливающийся (90 -100%), мы рассмотрели NIH-3T3 фибробластов мыши в течение 30 мин с 100 ЕД / мл интерферона (IFN) α или γ. В менее сливной клетки даже 15 минут лечения IFN уже привели к 5 - 8-кратную индукцию генов, как irf1 или SOCS3 5. С клетки быть немного слишком сливающийся анализа микрочипов не показали каких-либо индукции IFN генов, индуцируемых даже для самых быстро генов, индуцируемых как irf1 или SOCS3. Таким образом, плотность клеток является решающим фактором для 4SU маркировки экспериментов и все Клеточные культуры должны быть тщательно изучены перед началом маркировки.
4SU является фотоактивируемый рибонуклеозидом и 4SU содержащих РНК эффективно сшитый к белкам после контакта с источником света 365 нм. 4SU обработанные клетки должны быть выращены в темноте, и воздействие яркого света следует избегать. После удаления клеточных белков путем выделения РНК Trizol этот риск значительно уменьшается.
<р = класса "jove_content"> 4SU не включена в клеточную ДНК. Следует, однако, отметить, что общей РНК-прежнему будет содержать небольшие количества клеточной ДНК. При использовании 4SU-тегов и Q-RT-PCR анализа для изучения экспрессии вирусных генов в цитомегаловирусной инфекции мы сочли необходимым включить шаг DNaseI дайджеста в протокол, чтобы удалить concatemeric вирусных геномов 19. Это, вероятно, нет необходимости при использовании по потоку протоколов, которые не чувствительны к наличию ДНК.4SU цены регистрации и оптимальной концентрации 4SU
4SU подхватывается, клетками с внутри-и внеклеточного уровня, скорее всего, уравновешивающая в течение менее чем за минуту 9,16. Поглощение и включения цены 4SU являются зависимые от концентрации. Поэтому 4SU концентрация может быть удобно регулируется в зависимости от занятого продолжительность маркировки. Таблица 1 дает советы по 4SU концентрации в отнAtion продолжительности маркировке основаны на нашем личном опыте. В течение 1 часа из 4SU маркировки в клетках млекопитающих, 200 мкм 4SU будет достаточно для большинства применений в результате чего около одного 4SU остатка на 50 до 100 нуклеотидов во вновь транскрипции РНК в фибробласты.
В последние пару лет, мы применили 4SU-тегов для широкого спектра типов клеток человека и мыши происхождения, включая фибробласты, эндотелиальные клетки, эпителиальные клетки, клетки костного мозга стромы, макрофагов и Т-клеток. Кроме того, клетки из Drosophila и Xenopus были успешно использованы. Во всех этих экспериментах 4SU включение было установлено, что высокоэффективные требует минимальной корректировки 4SU концентрации для различных типов клеток. При создании нового метода типов клеток, мы рекомендовали бы этикетку клетки с увеличением 4SU-концентрации (например от 50 до 1600 мкм) и анализировать отношение очищенной вновь транскрибируемых RNA приложенному 4SU-концентраций (см. Фиг.2А / B). 4SU концентрации, при которой количество очищенной вновь транскрипции РНК входит плата должна быть выбрана.
В случаях, когда высоко сливающиеся, контакт ингибирует клетки используются, мы рекомендовали бы использовать немного выше 4SU концентрации (например, 500 вместо 200 мкм) для обеспечения эффективного 4SU регистрации. Кроме того, в случаях, когда захват очень короткий вновь транскрибируемых транскриптов (<200 NT) представляет особый интерес, 4SU концентрация может также должны быть увеличены. Это не должно быть объединено с длительным раз маркировка (например,> 1 час), чтобы избежать внематочной эффектов или токсичности. Наконец, мы обнаружили, что использование слишком малого объема среды для культивирования клеток может уменьшить эффективность 4SU регистрации. Поэтому мы рекомендуем использовать 5 мл или 10 мл среды на 10 см или 15 см блюдо, соответственно.
Приготовление общей клеточной РНК
Для успеха этого протокола крайне важно для получения чистой, свободной от РНКазы общей клеточной РНК. Использование Trizol 5 мл на 15 см блюдо производит чистую РНК бесплатно нуклеаз. Мы рекомендуем использовать модифицированный протокол Trizol Chomczynski соавт. 20. Во-первых, она лучше подходит для изоляции больших количеств РНК (> 100 мкг) в качестве расширенной центробежный сила приводит к прочной гранулы, которые более просты в обращении в течение этапов промывки. Однако, это требует использования специальных полипропиленовых пробирках и адаптеров, как регулярный лабораторный 15 мл Сокол труб не выживают более 6000 × г. Во-вторых, это улучшает удаление ДНК и гликопротеинов. Это становится особенно очевидным при подготовке РНК из органов или тканей. В-третьих, это не ограничивает максимальное количество общей РНК, которые могут быть изолированы. Хотя мы также обнаружили, на основе столбцов Выделение РНК методами (например RNeasy), чтобы обеспечить РНК соответствующего качества, стандартные колонки ARэлектронной только возможность захвата до 100 мкг общей РНК, тем самым ограничивая количество исходного материала. Наконец, путем удаления оставшегося этанола в два раза с помощью пипетки, сушка РНК для удаления остаточного этанола больше не требуется. Это устраняет риск чрезмерного высушивания РНК, которая может быть трудно растворить снова после этого. В принципе, 4SU-тегов применим в естественных условиях, например, путем внутривенной инъекции мышам. Тем не менее, мы отметили, что РНК представляет чистоту основной проблемой, требующей очистки стенограммы пола до очистки вновь транскрипции РНК (неопубликованные данные).
Биотинилирование и удалением несвязанного биотина
Биотин-HPDP на 100% тиол-специфических и образует дисульфидную связь между остатком биотина и тиол-меченой РНК вновь транскрипции молекул. Биотинилирование эффективность 4SU-меченой РНК составляет около 30%, как определено дот-блот-анализ 5. Как биотин HPDP не растворимые в водеон может быть эффективно удален экстракцией хлороформом. При одном этапе экстракции хлороформом достаточно для удаления большинства несвязанного биотина мы регулярно Повторите этот шаг, чтобы обеспечить полное удаление. Чтобы снизить потери РНК во время стадии экстракции хлороформом 2 мл фазовой автоподстройки гель тяжелых труб (Eppendorf) могут быть использованы в соответствии с инструкциями производителя. Обычно мы используем трубы фазовой автоподстройки только на второй стадии экстракции хлороформом в качестве шаблона объемов первый шаг зачастую слишком высокой, чтобы быть совместим с этими трубами. После удаления несвязанного биотина HPDP, РНК выделяют изопропанол / осаждением этанолом. Важно отметить, что коммерческий колонке на основе наборов для восстановления биотинилированного РНК (например, RNeasy от QIAGEN) не должны быть использованы, поскольку они содержат восстановители в буферах при условии, что расщеплять дисульфидные связи и удалить биотин от вновь транскрипции РНК .
Очистка новыхLY транскрипции РНК
Не добавляйте более 100 мкл биотинилированному РНК до 100 мкл стрептавидин бисер. Добавление меньший объем, является предпочтительным. Однако в том же объеме РНК должны быть добавлены для всех образцов. Регулировка громкости РНК вход (между образцами), которые вы добавляете к стрептавидин бисер, просто добавляя необходимый объем 1x TE к шарикам. Простой способ сделать свежий нуклеазы без 100 мМ DTT является сцеживать достаточное количество порошка в DTT трубки сокола помещены на сверхчувствительных масштабе, а затем добавить необходимое количество нуклеазы без H 2 O для получения 100 мМ DTT (64,8 мкл воды на 1 мг DTT).
В ходе разработки 4SU-тегов мы тестировали стрептавидин бусы из различных поставщиков. Некоторые из них генерируется большое количество фоне. Поэтому мы настоятельно рекомендуем использовать Miltenyi Стрептавидин бисера как, до сих пор, мы никогда не испытывали проблем с переходящих немеченных РНК из тканей сulture полученных образцов РНК. Таким образом, всего 150 нг меченой РНК может быть специально очищенный от 150 мкг биотинилированного РНК (в 100 мкл воды) с использованием 100 мкл стрептавидина шариков. Равновесие из бисера с уравновешивающим буфером поставляется с бисером может быть выполнена и может немного повышения скорости захвата 13.
Контроль качества
Рекомендуется выполнить Q-RT-PCR управления на вновь транскрипции РНК перед подачей для высокопроизводительного анализа. Это может включать количественное нескольких генов ссылкой известно, дифференциально регулируется в данной экспериментальной установки. В случаях, когда 4SU пометки используются для изучения РНК цены распада, мы рекомендовали бы количественно недолгим стенограмму (например, Мус, ФОС) и долгоживущие один (например, GAPDH) как в общем и недавно транскрипции РНК. Отношение вновь транскрипции / тотальную РНК должна быть существенно выше (~ 5 - 10 раз)для недолгой транскриптов. На основе РНК полураспада ссылкой ген, РНК полураспада может быть определена. Если все три фракции РНК (тотальной РНК, недавно транскрипции РНК и немеченого ранее существовавшего РНК), анализируют на четыре или более генов, нормализация различные подмножества РНК может быть выполнена линейного регрессионного анализа и оценки качества управления может быть определено, как описано семь 21.
Для Q-ОТ-ПЦР анализа, мы рекомендуем использовать 2,5 мкл меченой РНК в 20 мкл смеси кДНК синтеза. Для оптимального сравнения Q-RT-PCR результаты заморозить кДНК в аликвоты 5 мкл до первого использования. Оттепель труб непосредственно перед употреблением, добавьте 45 мкл H 2 O и с учетом 5 мкл разведения в Q-RT-ПЦР. Это значительно повышает сопоставимость различных работает ПЦР.
Недавно транскрипции РНК образцы должны быть проверены на признаки деградации РНК с использованием Bioanalyser Agilent до применения к нимвысокопроизводительного анализа (микрочипы или РНК-SEQ). Следует, однако, отметить, что дополнительные полосы иногда наблюдались Bioanalyser Agilent. Биологическое значение этого остается неясным. Как недавно транскрибируется РНК содержит значительно меньше рибосомной РНК, эти образцы иногда провалить Agilent Bioanalyser контроля качества. Если это не связано с видимой деградации образцов РНК приемлемого качества, как правило, прекрасно, чтобы быть подвергнут высокопроизводительного анализа.
Совместимость вновь транскрипции РНК с вниз по течению анализы
Недавно транскрибируется РНК содержится значительно больше, чем общая мРНК РНК. Это в основном за счет большего количества последовательностей интронов во вновь транскрипции РНК, которая увеличивается, когда продолжительность 4SU-тегов сокращается. Таким образом, мы не регулярно проводить истощение рРНК от вновь транскрипции образцов РНК, так как это требует больших количеств исходного материала в то время как ProvidIng довольно мало (~ в два раза) выигрыш в не-рРНК читает. Наконец, остается отметить, что больший процент несплайсированные с высоким молекулярным весом транскриптов, присутствующих в новой транскрипции РНК может потребовать дополнительных фрагментации при подготовке библиотек кДНК для следующего поколения секвенирования. Результаты этапа фрагментации Размер Поэтому следует тщательно контролировать качество.
Данные нормализации для РНК половины проживанию измерений
Стандартный подход к нормализации экспериментальные данные для РНК полураспада измерений нормализовать все данные в РНК полураспада хорошо охарактеризованы уборки генов или РНК средний период полураспада в данном типе клеток определяли в предыдущих экспериментах. В клетках млекопитающих, причем последний находится в диапазоне от 5 до 10 ч 6,7. Хотя этот подход также работает достаточно хорошо для 4SU на основе измерений, другие средства для нормализации необходимы, если среднее РНК период полураспада не известно, или, если это маY даже пострадать от изменения в клеточном изучаемой системы, например, путем выбивания пути распада РНК. 4SU-тегов предлагает уникальный способ оценки РНК средний период полураспада на основе анализа всех трех фракций РНК, т.е. общей клеточной РНК, недавно транскрипции РНК и немеченого ранее существовавшего РНК. В общей клеточной РНК разделяется на два последних фракций РНК простую линейную регрессионную модель может быть использована для нормализации три фракции РНК друг к другу и определения среднего РНК полураспада 7,16. Пакет программного обеспечения доступны в Интернете для выполнения этих анализов 22.
Неэффективное захвата стенограммы с низким содержанием уридином может повлиять РНК полураспада измерений в результате искусственно заниженным вновь транскрипции / общее соотношение РНК и РНК длительного периода полураспада. Масштабы этой проблемы может быть оценена путем построения РНК полураспада или журнала (вновь транскрипции / общее соотношение РНК) против uridiNE содержание всех стенограмм 7,15. Это также обеспечивает хороший контроль качества для оценки различий в 4SU-включение курсов между различными образцами или условий. В случаях, когда существенной корреляции с содержанием уридин наблюдается это может быть скорректирована на биоинформационных средства 15. Тем не менее, следует отметить, что вклад зрелых транскриптов в недавно транскрипции РНК не может быть легко отличить от гораздо больше и, следовательно, гораздо более уридин-богатые прекурсоров. Если обработка кинетики данной транскрипт не известны (что они обычно не) просто поправкой на низкое содержание уридина (неэффективное захвата) может грубо искажающие РНК полураспада. Таким образом, мы недавно обнаружили обработки большинства человеческих snoRNAs, весьма неэффективны 9. Если бы мы исправили вновь транскрипции / общей РНК отношения для низкого содержания уридином на довольно небольшой (70 - 300 NT) snoRNAs, это привело бы к очень коротким snoRNA половиной-LIVES (<5 мин) с многочисленными вновь транскрипции / общее соотношение РНК превышает 100%. Таким образом, мы вообще не рекомендуем поправкой на низкое содержание уридином при измерении РНК полураспада.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Авторы заявляют, что у них нет конкурирующих финансовых интересов.
Acknowledgments
Мы хотели бы поблагодарить Amie Риган за внимательное чтение рукописи. Эта работа была поддержана грантом NGFN Plus # 01GS0801, MRC G1002523 стипендии, гранты и NHSBT WP11-05 LD и гранта DFG FR2938/1-1 к CCF
Materials
| Name | Company | Catalog Number | Comments |
| 4-thiouridine | Carbosynth | T4509 | Prepare 50 mM stock in sterile H2O, store at -20 °C in aliquots of 50-500 μl, discard unused reagent, do not refreeze. |
| Trizol | Invitrogen | 15596026 (100 ml), 15596018 (200 ml) | WARNING - CORROSIVE and HAZARDOUS TO HEALTH! Ensure immediate access to Phenol antidote (PEG-Methanol); Store at 4 °C. |
| Chloroform | Sigma | 372978 | WARNING - HAZARDOUS TO HEALTH |
| Isopropanol | Sigma | 650447 | |
| Sodium citrate, nuclease-free | Sigma | C8532 | Prepare 1.6 M stock solution using nuclease-free water. |
| 5M nuclease-free NaCl | Sigma | 71386 | Stock solution |
| Nuclease-free H2O | Sigma | W4502 | Make 1 ml aliquots in nuclease-free tubes. |
| RNA precipitation buffer | 1.2 M NaCl, 0.8 M sodium citrate in nuclease free water. Prepare in advance under strictly nuclease-free conditions. Store at room temperature in 50 ml falcon tubes. | ||
| Ethanol | Sigma | 459844 | Use with nuclease-free water to prepare 80% ethanol, store at -20 °C. |
| 1 M nuclease-free Tris Cl, pH 7.5 | Lonza | 51237 | Stock solution |
| 500 mM nuclease-free EDTA, pH 8.0 | Invitrogen | 15575-020 | Stock solution |
| 10x Biotinylation Buffer (BB) | 100 mM Tris pH 7.4, 10 mM EDTA in nuclease-free water, make aliquots of 1 ml. | ||
| Dimethylformamide (DMF) | Sigma | D4551 | |
| EZ-Link biotin-HPDP | Pierce | 21341 | Prepare 1 mg/ml stock solution by dissolving 50 mg biotin-HPDP in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. |
| Phase Lock Gel Heavy tubes 2.0 ml | Eppendorf | 0032 005.152 | Optional for the chloroform extraction step. |
| Zeta membrane | BIORAD | 162-0153 | |
| 10x Dot blot binding buffer | 100 mM NaOH, 10 mM EDTA | ||
| Biotin-oligo | 5'-biotin, 25 nucleotides, any sequence | ||
| Sodium dodecyl sulphate | Fisher | BPE9738 | For 100 ml 20% stock solution, add 20 g SDS to 80 ml PBS pH 7-8 and adjust volume to 100 ml. Keep all high-percentage SDS solutions above 20 °C. Warm the solutions slightly should SDS precipitate. |
| EZ-Link Iodoacetyl-LC-Biotin | Pierce | 21333 | Prepare 1 mg/ml stock solution by dissolving 50 mg iodoacetyl-biotin in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. Generates irreversible, thiol-specific biotinylation. |
| Phosphate buffer saline | Gibco | 10010-015 | |
| Dot blot blocking buffer | Mix 20 ml 20% SDS with 20 ml 1 x PBS pH 7-8 and add EDTA to the final concentration of 1 mM. | ||
| Streptavidin-horseradish peroxidase | Vector Laboratories | SA5004 | Store at -20 °C. Mix 10 ml 20% SDS with 10 ml 1 x PBS. Add 20 μl Streptavidin-HRP before use. |
| ECL reagent | GE Healthcare | RNP2109 | Use following the manufacturer's instructions. |
| Super RX, X-RA Film, 18x24 cm | Fujifilm | 47410 19236 | |
| μMacs Streptavidin Kit | Miltenyi | 130-074-101 | Store the beads at 4 °C. |
| Tween 20 | Sigma | P1379 | |
| Washing buffer | 100 mM Tris pH 7.4, 10 mM EDTA, 1 M NaCl, 0.1% Tween 20 in nuclease-free H2O. | ||
| Dithiothreitol (DTT) | Sigma | 43817 | Prepare as 100 mM DTT in nuclease-free H2O, always prepare fresh before use. |
| RNeasy MinElute Kit | Qiagen | 74204 | Store columns at 4 °C, remaining components of the kit at room temperature. |
| 1.5 ml screw-top polypropylene tubes | Sarstedt | 72.692.005 | Compatible with Dimethylformamide |
| 2.0 ml screw-top polypropylene tubes | Sarstedt | 72.694.005 | Compatible with Dimethylformamide |
| 15 ml tubes | BD Falcon | 352096 | Compatible with Dimethylformamide |
| 50 ml tubes | BD Falcon | 352070 | Compatible with Dimethylformamide |
| All solutions/reagents should be stored at room temperature unless otherwise specified. | |||
| Equipment | |||
| UV/VIS spectrophotometer | Thermo Scientific | NanoDrop 1000 | Or equivalent. Use low volume (1-2 μl) for measurements of low RNA concentrations to avoid excessive sample loss. |
| Polypropylene 15 ml centrifuge tubes | VWR International | 525-0153 | In contrast to standard 15 ml tubes, these tolerate up to 15,000 × g |
| High-speed centrifuge | Beckman Coulter | Avanti J-25 | Or equivalent equipment capable of reaching 13,000×g |
| High-speed rotor | Beckman Coulter | JLA-16250 | Or equivalent equipment capable of reaching 13,000×g |
| Adaptors for 15 ml tubes | Laborgeräte Beranek | 356964 | Or equivalent equipment capable of reaching 13,000×g |
| Refrigerated table-top centrifuge | Eppendorf | 5430 R | Or equivalent. |
| Thermomixer | Eppendorf | Thermomixer compact | Or equivalent. |
| Magnetic stand | Miltenyi Biotec | 130-042-109 | One stand holds 8 μMacs columns. |
| Waterbath | Grant | SUB Aqua 5 | Or equivalent. |
| Ultra-fine scale | A&D | GR-202 | Or equivalent. |
| E-Gel iBase Power System | Invitrogen | G6400UK | For RNA gels; or equivalent. |
| E-Gel EX 1% agarose precast gels | Invitrogen | G4020-01 | For RNA gels; or equivalent. |
References
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Mortazavi, A., Williams, B. A., Mccue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Nagalakshmi, U., Wang, Z., et al. The transcriptional landscape of the yeast genome defined by RNA sequencing. Science. 320, 1344-1349 (2008).
- Wilhelm, B. T., Marguerat, S., et al. Dynamic repertoire of a eukaryotic transcriptome surveyed at single-nucleotide resolution. Nature. 453, 1239-U1239 (2008).
- Dölken, L., Ruzsics, Z., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Yang, E., van Nimwegen, E., et al. Decay rates of human mRNAs: correlation with functional characteristics and sequence attributes. Genome Res. 13, 1863-1872 (2003).
- Friedel, C. C., Dölken, L., Ruzsics, Z., Koszinowski, U. H., Zimmer, R. Conserved principles of mammalian transcriptional regulation revealed by RNA half-life. Nucleic Acids Res. 37, e115 (2009).
- Nilsen, T. W., Graveley, B. R. Expansion of the eukaryotic proteome by alternative splicing. Nature. 463, 457-463 (2010).
- Windhager, L., Bonfert, T., et al. Ultra short and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Res. , (2012).
- Melvin, W. T., Milne, H. B., Slater, A. A., Allen, H. J., Keir, H. M. Incorporation of 6-thioguanosine and 4-thiouridine into RNA. Application to isolation of newly synthesised RNA by affinity chromatography. Eur. J Biochem. 92, 373-379 (1978).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23, 232-237 (2005).
- Kenzelmann, M., Maertens, S., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proc. Natl. Acad. Sci. U.S.A. 104, 6164-6169 (2007).
- Schwanhäusser, B., Busse, D., et al. Global quantification of mammalian gene expression control. Nature. 473, 337-342 (2011).
- Rabani, M., Levin, J. Z., et al. Metabolic labeling of RNA uncovers principles of RNA production and degradation dynamics in mammalian cells. Nat. Biotechnol. , (2011).
- Miller, M. R., Robinson, K. J., Cleary, M. D., Doe, C. Q. TU-tagging: cell type-specific RNA isolation from intact complex tissues. Nat. Methods. 6, 439-441 (2009).
- Miller, C., Schwalb, B., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Mol. Syst. Biol. 7, 458 (2011).
- Weintz, G., Olsen, J. V., et al. The phosphoproteome of toll-like receptor-activated macrophages. Mol. Syst. Biol. 6, 371 (2010).
- Lipsett, M. N. The isolation of 4-thiouridylic acid from the soluble ribonucleic acid of Escherichia coli. Journal of Biological Chemistry. 240, 3975-3978 (1965).
- Marcinowski, L., Liedschreiber, M., et al. Real-time Transcriptional Profiling of Cellular and Viral Gene Expression during Lytic Cytomegalovirus Infection. PLoS Pathog. 8, e1002908 (2012).
- Chomczynski, P., Mackey, K. Short technical reports. Modification of the TRI reagent procedure for isolation of RNA from polysaccharide- and proteoglycan-rich sources. Biotechniques. 19, 942-945 (1995).
- Friedel, C. C., Dölken, L. Metabolic tagging and purification of nascent RNA: implications for transcriptomics. Mol. Biosyst. 5, 1271-1278 (2009).
- Friedel, C. C., Kaufmann, S., Dölken, L., Zimmer, R. HALO - A Java framework for precise transcript half-life determination. Bioinformatics. , (2010).



