Summary
סה"כ הסלולרי RNA מספק תבנית עניים לחקר שינויים קצרי טווח בסינתזה וריקבון RNA, כמו גם את הקינטיקה של רנ"א עיבוד. כאן, אנו מתארים תיוג חילוף חומרים של RNA החדש שעבד עם 4-thiouridine אחרי biotinylation תיאול ספציפי ולטיהור של RNA החדש שעבד ומאפשר להתגבר על המגבלות האלה.
Abstract
הפיתוח של microarrays השלם transcriptome והדור הבא של רצף יש מהפכה ההבנה של המורכבות של ביטוי גני הסלולר שלנו. יחד עם הבנה טובה יותר של המנגנונים המולקולריים המעורבים, מידות מדויקות של קינטיקה הבסיסית הפכו להיות חשובה יותר ויותר. כאן מתודולוגיות רבי עוצמה אלה, עומדות בפני מגבלות עיקריות בשל מאפיינים פנימיים של דגימות תבניתו הם לומדים, RNA הסלולרי כלל כלומר. במקרים רבים שינויים ברנ"א הסלולרי כלל להתרחש גם לאט מדי או מהר מדי כדי לייצג את האירועים המולקולריים שבבסיס וקינטיקה שלהם עם רזולוציה מספיקה. בנוסף, התרומה של שינויים בסינתזת רנ"א, עיבוד, וריקבון אינה מובחנות בקלות.
לאחרונה פיתחנו פרופיל גנטי ביטוי ברזולוציה גבוה כדי להתגבר על המגבלות האלה. הגישה שלנו מבוססת על תיוג חילוף חומרים של RNA החדש שעבד עם 4-thiouriלסעוד (ובכך גם המכונה 4sU תיוג) ואחריו טיהור קפדני של RNA עבד מחדש באמצעות biotinylation תיאול ספציפי וחרוזים מגנטיים streptavidin מצופים. זה ישים למגוון רחב של יצורים בעלי חוליות, לרבות תסיסנית, ושמרים. אנחנו מיושמים בהצלחה 4sU תיוג ללמוד קינטיקה בזמן אמת של פעילות גורם שעתוק, לספק מדידות מדויקות של RNA מחצית חיים, ולקבל תובנות לתוך רומן קינטיקה של רנ"א עיבוד. לבסוף, יכולים להיות מועסקים מודלים חישוביים ליצירת ניתוח משולב, מקיף של המנגנונים המולקולריים שבבסיס.
Introduction
פרופיל גנטי ביטוי הוא כלי מרכזי המשמש ללמוד תהליכים תאיים וברשת יחסי הגומלין המורכבת המשויכת. שפע מחקרים על mRNA היו בדרך כלל בשיטה של בחירה כדי להשיג תובנות בסיסיות לתוך המנגנונים המולקולריים שבבסיס. הפיתוח של microarrays השלם transcriptome 1, ולאחרונה רצף של הדור הבא, של רנ"א (RNA-אילך) 2-4 מונע את הגישה הזו. בעוד טכנולוגיות אלה חוללו מהפכה ההבנה של המורכבות של ביטוי גני הסלולר שלנו שהם עומדים בפני מגבלות עיקריות בשל מאפיינים פנימיים של תבנית המדגם שלהם, כלומר סך הסלולרי RNA. שינויים ראשונים, לטווח קצר ברמות רנ"א הכל אינם תואמים שינויים בשערי שעתוק, אבל מטבעם תלויים ברנ"א מחצית החיים של התמלילים, בהתאמה. בעוד אינדוקציה פי חמישה מתמליל, קידוד לדוגמה קצר לגורם שעתוק, תהיה בקלות להבחין ברנ"א הכלבתוך שעה, אותו הגיוס של תמליל, למשל קידוד חיים ארוכים לאנזים המטבולי, יישאר כמעט בלתי נראה. בנוסף, גם שלם כיבוי (> למטה רגולציה של פי 1,000) בשיעור השעתוק של גן ממוצע עם RNA מחצית חיים של חמש שעות תהיה פשוט לקחת חמש שעות לרמות רנ"א הכל לירידה של רק כפולה . לכן, ניתוח של RNA הכולל מעדיף את זיהוי של עד הוויסות של תמלילים קצרים ימים, שרבים מהם לקודד לגורמי שעתוק וגנים עם פונקציות רגולטוריות 5. בנוסף, המפל קינטית הנכון של רגולציה הוא מוסתר ולא יכול להיות מובחן אירועי איתות ראשוניים משני. שניהם, בתורו, עלולים לגרום להטיה משמעותית בניתוחים ביואינפורמטיקה במורד הזרם. שנית, לא ניתן לייחס שינויים ברמות RNA הכולל לשינויים בסינתזה או ריקבון רנ"א. מדידות של זה האחרון דורשות גישות פולשניות סלולריים, כגון חסימת transcriptiעל שימוש actinomycin 6 ד ', וניטור מורחב של ריקבון מתמשך RNA לאורך זמן. עם ה-mRNA ממוצע מחצית חיים בתאי יונקים של 5 - 10 שעות 5,7, רמות ה-mRNA של רוב הגנים תהיה ירדו רק על ידי פחות מכפול לאחר כמה שעות של מעצר תעתיק. הבדלים קטנים ולא אלו לגרום למדידות לא מדויקות בעליל של mRNA מחצית חיים עבור רוב הגנים סלולריים בשל אופי מעריכי של משוואות המתמטיות המשמשות כבסיס. לבסוף, בעוד RNA-ואילך בסך הכל הסלולרי RNA גילה כי כמחצית מהגנים שלנו כפופה לאירועי שחבור חלופיים 8, קינטיקה הבסיסית, כמו גם את המנגנונים הדינמיים מנחים רגולציה רקמות והקשר ספציפי של RNA עיבוד יישאר הבין היטב. בנוסף, התרומה של רנ"א עיבוד לביטוי ההפרש גן, במיוחד עבור RNAs ללא קידוד, עדיין לא נקבע. בסך הכל, על המגבלות האלה מייצגות מכשולים עיקריים לדוגמנות הקינטית bioinformatic של המנגנונים המולקולריים שבבסיס.
לאחרונה פתחנו גישה, פרופיל גנטי ביטוי ברזולוציה גבוה המכונה, כדי להתגבר על בעיות אלה 5,7,9. היא מבוססת על תיוג חילוף חומרים של RNA החדש שעבד באמצעות 4-thiouridine (4sU תיוג), נגזרת uridine מתרחש באופן טבעי, ומספקת גישה ישירה לתמלילים חדשים שעבדו עם התערבות מינימאלית בצמיחת תאים וביטוי גנים (ראה איור 1) 5, 10-12. חשיפה של תאים אוקריוטים לתוצאות 4sU בספיגתו המהירה, זירחון 4sU-אדנוזין, והכללתו RNA החדש שעבד. בעקבות בידוד בסך הכל הסלולרי רנ"א, רנ"א שבריר 4sU שכותרתו הוא biotinylated תיאול-במיוחד ליצירת קשר דיסולפיד בין ביוטין ואת הרנ"א חדש שעבד. לאחר מכן ניתן להפריד 'סה"כ סלולרי RNA' כמותית לכותרתו ("זה עתה עיבד ') וללא תווית (' מראש existinרנ"א) "G עם טוהר גבוה באמצעות חרוזים מגנטיים streptavidin מצופים. לבסוף, שכותרתו RNA הוא התאושש מן החרוזים פשוט על ידי הוספת סוכן הפחתה (למשל dithiothreitol) ביקוע אג"ח דיסולפיד ומשחרר את הרנ"א חדש שעבד מהחרוזים.
RNA עיבד חדש מתאר את פעילות תעתיק של כל גן במהלך פרק הזמן של חשיפת 4sU. 4sU תיוג בלוח הזמנים של דקות ובכך מספק תמונת תמונת מצב של ביטוי אוקריוטים גן ותבנית אידיאלית לניתוחי bioinformatic מורד זרם (לדוגמה: ניתוח אמרגן). במקרים בהם ניתן להניח תנאי מצב יציב, את היחס של זה עתה עיבד / סך הכל, שזה עתה עיבד / ללא תווית וללא תווית / RNA הכולל לספק גישה לא פולשנית לרנ"א המדויק מחצית חיים 7,13. בנוסף, חשוב לציין כי RNA החדש שעבד מטוהר לאחר קטן כמו 5 דקות של 4sU תיוג (5 דקות 4sU-RNA) הוא צעיר מ -15 ו -60 דקות 4sU-RNA.בעת ביצוע שני אולטרה קצר וארוך יותר בהדרגה 4sU תיוג בהגדרה ניסיונית אחת בשילוב עם RNA-ואילך, קינטיקה של רנ"א עיבוד מתגלה ברזולוציה של נוקלאוטיד 9. לבסוף, ניתוחים בזמן קורס של RNA חדש שעבד ומוחלט בשילוב עם מודלים חישוביים לאפשר ניתוח אינטגרטיבי של סינתזה וריקבון 14-RNA.
לסיכום, גישה זו מאפשרת ניתוח הישיר של הדינמיקה של סינתזת RNA, עיבוד, והשפלה בתאים אוקריוטים. זה ישים בכל אורגניזמים המודל הגדולים, כולל יונקים, חרקים (דרוזופילה), דו חיים (Xenopus), ושמרים 5,15,16. זה תואם באופן ישיר עם microarray ניתוח 5,17, RNA-9,13,14 ואילך, והוא ישים in vivo 12,15. הנה, אנחנו פירוט המתודולוגיה לתייג, לבודד, ולטהר את RNA החדש שעבד בתאי יונקים בתרבית. בנוסף, potentiבעיות ואת החסרונות al הם דנו.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. תיוג מטבולית עם 4-thiouridine
הפוך את תכנית מפורטת של ההתקנה / לוח הזמנים של הניסוי, למשל מתי להוסיף 4sU לתרבית תאים ומתי לקצור את הדגימות. תכנית במשך לפחות 5 דקות בין כל מצב. טיפול בתאים של תנאי אחד בלבד בכל פעם. להתמודד עם מקסימום. 3 - 5 מנות בכל זמן נתון. להתמודד עם תאים במהירות אפשרית כדי למזער את השינויים בטמפרטורה וברמות CO 2. הימנע מחשיפת התאים לאור בהיר לאחר 4sU הוא הוסיף שדבר עלול לגרום לcrosslinking של רנ"א 4sU שהכותרת לחלבונים תאיים.
התחל של תיוג
- הפשירו 4-thiouridine (4sU) ממש לפני שימוש וטפטף כמות הנדרשת של 4sU עבור כל תנאי לתוך צינור פלקון סטרילי.
- קח את הכמות הנדרשת של תרבית תאים בינוניות (5 מ"ל לכל צלחת 10 ס"מ) את המנות ולהוסיף לצינור פלקון המכיל 4sU ומערבבים היטב. להסיר ולסלק את המדיום שנותר מאת הכלים. <li> החל אחורי בינונית מכיל 4sU את הכלים.
הסוף של תיוג
- סור בינוני תרבות תא מהתאים. הוסף 5 מ"ל של Trizol לכל צלחת. לניסויים מורכבים, כולל נקודות זמן מרובות או תנאים, צעד זה נעשה הכי טוב על ידי שני אנשים, אחד הסרת בינונית, אחר הוספת Trizol וקציר lysate.
- דגירה במשך 5 דקות בטמפרטורת חדר למשך תמוגה תא שלמה.
- השתמש פיפטה 10 מ"ל כדי לשטוף את הצלחת בזהירות עם Trizol הוסיף. זה מסייע תמוגה תא שלמה ומדגם התאוששות. להתמודד עם טיפול כTrizol הוא מסוכן ביותר כאשר מקבלים במגע עם עור או עיניים! יש תרופה לכוויות פנול בהישג היד (למשל פוליאתילן גליקול 300 או 400 בספירט תעשייתי (70:30)). העבר את הדגימות לצינור פלסטיק. שים לב כי צינורות פלקון סטנדרטיים אינם להתנגד כוחות g גבוהים אלה). דוגמאות ניתן לאחסן ב -20 ° C לחודש אחד לפחות עד רנ"א הכל אנישל מוכן.
2. RNA הכנה באמצעות שינוי Trizol פרוטוקול
- הוסף 1 מיליליטר כלורופורם (0.2 מ"ל למ"ל Trizol) ולנער במרץ במשך 15 שניות. דגירה בטמפרטורת חדר למשך 2 - 3 דקות.
- צנטריפוגה ב 13,000 × גרם במשך 15 דקות ב 4 ° C.
- העבר את השלב עליון מימית (המכיל RNA) לצינור חדש 15 מיליליטר פוליפרופילן.
- הוסף ½ נפח התגובה של רנ"א ממטרים חיץ וisopropanol (למשל כדי 3 מ"ל supernatant חיץ להוסיף 1.5 מיליליטר RNA ממטרים ו1.5 מ"ל isopropanol).
- מערבבים היטב. דגירה בטמפרטורת חדר למשך 10 דקות.
- צנטריפוגה ב 13,000 × גרם במשך 10 דקות ב 4 ° C. בטל supernatant.
- ספין למטה בקצרה (5,000 × גרם במשך 30 שניות) ולהסיר isopropanol שייר עם 200 פיפטה μl.
- הוסף נפח שווה של 75% אתנול ולנער את הצינור עד לניתוק גלולה. ההסרה הימנע לשבור אותו לחתיכות קטנות רבות מכיוון שהדבר עלול לגרום לresiduaאתנול L קשה.
- צנטריפוגה ב 13,000 × גרם במשך 10 דקות ב 4 ° C. בטל supernatant.
- ספין למטה בקצרה רנ"א ולהסיר אתנול נותר עם טפטפת μl 200. חזור על השלב ולהסיר אתנול נותר עם טפטפת μl 20. לאחר שני שלבים אלה, אין ייבוש נוסף של גלולה צריכה להתבצע.
- הוסף 100 μl של H 2 O לתשואה צפוי RNA 100 מיקרוגרם ומערבבים היטב על ידי pipetting למעלה ולמטה 5 - 6 פעמים כדי לסייע בהמסת רנ"א.
- ממיסים ולפגל RNA על ידי חימום עד 65 מעלות צלזיוס למשך 10 דקות (ייקר) ומקום באופן מיידי על קרח.
- למדוד ריכוז RNA ב 260 ננומטר באמצעות ספקטרופוטומטר NanoDrop, בעקבות הוראות יצרן. ניתן לאחסן את זה RNA ב -80 מעלות צלזיוס למשך חודש אחד לפחות.
3. Biotinylation תיאול ספציפי של RNA חדש בתעתיק
- התחל עם 60-80 מיקרוגרם של רנ"א הסלולרי בסך הכל.
- התיוג מהווה תגובה. פיפטה בפעולות הבאותסדר (RNA מיקרוגרם לכל):
- מאגר biotinylation 10x μl 1
- 7 RNA μl (המכיל 1 מיקרוגרם דילול RNA בnuclease ללא H 2 O)
- 2 ביוטין-HPDP μl (1 מ"ג / מיליליטר DMF)
תמיד להוסיף שעבר ביוטין, HPDP ולערבב באופן מיידי על ידי pipetting. במקרה ביוטין משקעים, תוכן DMF יכול להיות מוגבר לריכוז סופי של 40%.
- דגירה בטמפרטורת חדר למשך 1.5 שעות עם סיבוב.
- הוסף נפח שווה של כלורופורם. מערבבים במרץ. דגירה של 2 - 3 דקות, עד השלבים מתחילים להפריד ובועות מתחילות להיעלם.
- צנטריפוגה ב 20,000 × גרם במשך 5 דקות ב 4 ° C. להעביר את השלב המימית העליון לתוך צינור חדש בזהירות.
- חזור על שלבים 3.4 ו -3.5 ועונה אחת. ייתכן שתרצה לבצע שלב זה ב2 צינורות ג'ל נעל שלב מ"ל כבדים כדי לצמצם את האובדן של רנ"א.
- RNA משקעים: תוספת 1/10 נפח 5 M NaCl ונפח שווה שלisopropanol לשלב מים.
- צנטריפוגה ב 20,000 × גרם במשך 20 דקות ב 4 ° C. בטל supernatant.
- הוסף נפח שווה של 75% אתנול, צנטריפוגות ב 20,000 × גרם במשך 10 דקות ב 4 ° C, להשליך supernatant.
- ספין בקצרה ולהסיר אתנול שיורי עם 200 פיפטה μl.
- ספין בקצרה ולהסיר אתנול שיורי עם 20 פיפטה μl.
- אל תאפשר לרנ"א לייבוש. Re-להשעות אותו ב 50 - H 2 O μl 100 (~ 1 μl לכל 1 מיקרוגרם קלט RNA). מערבבים היטב על ידי pipetting למעלה ולמטה 5 - 6 פעמים.
- לבדוק את איכות הרנ"א על ידי ניתוח electrophoretical להוציא השפלה RNA.
4. ניתוח דוט כתם של 4sU-התאגדות (אופציונלי)
התאגדות 4sU ניתן לקבוע בקלות על ידי ניתוח כתם נקודה של רנ"א biotinylated. זהו צעד אופציונלי המאפשר פתרון בעיות והערכה של שיעורי התאגדות 4sU ביחס לשליטת אוליגו DNA biotinylated. עבור assay זה אנו Recommend באמצעות iodoacetyl-ביוטין במקום ביוטין-HPDP לbiotinylation של רנ"א 4sU שהכותרת בשלב 3.2. התוצאה היא בלתי הפיך של biotinylation 4sU-RNA. לכן, שיטות המבוססות על העמודה (RNeasy למשל) יכולות לשמש להתאוששות של כמויות הרבה יותר קטנות של RNA (למשל biotinylated 5 מיקרוגרם). אמנם RNA biotinylated באמצעות ביוטין-HPDP המתאים גם לassay זה, האות המתקבל הוא חלש יותר ויחס אות לרעש פחות נוח (איור 3).
- לעקוב אחר הפרוטוקול ל4sU תיוג ובידוד של רנ"א הסלולרי כלל, כמתואר בסעיפי 1 ו -2.
- Biotinylate 4sU שכותרתו RNA כאמור בסעיף 3 החלפת ביוטין-HPDP עם iodoacetyl-ביוטין ולבצע שתי עקירות כלורופורם כדי להסיר שאריות iodoacetyl-ביוטין מוגזמים לחלוטין.
- לשחזר RNA biotinylated על ידי המשקעים isopropanol / אתנול כמתואר או באמצעות גישה המבוססת על העמודה (למשל RNeasy) במקרה כמויות קטנות של RNA (<10 מיקרוגרם) נמצאים בשימוש.
- דגירה הממברנה זיטה במי nuclease עם הנדנדה למשך 10 דקות.
- קח את הקרום מחוץ למים nuclease ללא נוזלים עודפים ולהסיר על ידי הנחת בקרום בין שתי מגבות נייר נקיות ולחיצה בחוזקה. Air-ייבוש הקרום למשך 5 דקות יגרום נקודות יותר נחמדות.
- עבור כל דגימה, להכין 20 μl של 200 רנ"א ng / μl באמצעות חיץ קרח קר נקודת כתם מחייב (10 מ"מ NaOH, 1 mM EDTA). החל 5 μl דילול זה (כלומר 1 מיקרוגרם של רנ"א), כמו גם שלושה פי 10 דילולים הבאים (כלומר 100, 10, ו 1 ng-RNA, בהתאמה) לקרום זיטה על ידי pipetting. Pipetting דרך מדף ריק של טיפים פיפטה יכול להיות מועסק על מנת לספק מרווח מופץ באופן שווה. לחלופין, השתמש במנגנון כתם נקודה על פי הוראות היצרן.
- החל 5 μl של ביוטין שכותרתו DNA אוליגו בריכוזים הנעים בין 20 ng / μl עד 20 pg / μl (כלומר 100-0.1 ng אוליגו) כהמשך חיובירול לממברנה על ידי pipetting. השתמש מדגם biotinylated, 4sU-נאיבי כביקורת שלילית.
- אוויר יבש הקרום למשך 5 דקות.
- דגירה הממברנה למשך 30 דקות בחיץ 40 מיליליטר חסימה עם נדנדה.
- דגירה הממברנה עם 10 מ"ל של 1:1,000 peroxidase streptavidin-חזרת במשך 15 דקות (5 מ"ל + 20% + 10 SDS peroxidase streptavidin-חזרת μl 5 מ"ל PBS)
- לשטוף קרום פעמיים ב40 מ"ל PBS + 10% SDS (20 מ"ל PBS + 20 מ"ל 20% SDS) למשך 5 דקות.
- לשטוף קרום פעמיים ב40 מ"ל PBS + 1% SDS (38 מ"ל PBS + 2 מ"ל 20% SDS) למשך 5 דקות.
- לשטוף קרום פעמיים ב40 מ"ל PBS + 0.1% SDS (40 מ"ל PBS + μl 200 20% SDS) למשך 5 דקות.
- הסר עודף של נוזל על ידי הצבת בקרום בין שתי מגבות נייר נקיות ולחיצה עליהם בתקיפות.
- דמיינו HRP קרום הנכנס באמצעות ECL לפי הוראות יצרן.
- מניחים את הקרום בנייר כסף / שקית ניילון, להסיר בועות אוויר ודגירה במשך 2 דקות בחושך.
- לחשוף את הקרום לסרט 1 - 5 דקות.
5. הפרדה של רנ"א תווית וללא תווית באמצעות חרוזים מגנטיים streptavidin מצופים
- חום כביסה חיץ (3 מ"ל לדגימה) עד 65 מעלות צלזיוס באמבט מים.
- הכן טרי 100 מ"מ dithiothreitol (DTT) בnuclease ללא H 2 O. לעשות זאת על ידי decanting 15 - 30 מ"ג של אבקת DTT לתוך צינור פלקון 50 מ"ל נקי הונח על קנה המידה Ultra-בסדר. לשקול ולהוסיף כמות הנדרשת של nuclease ללא H 2 O.
- חום biotinylated דגימות RNA ל -65 מעלות צלזיוס במשך 10 דקות כדי לפגל ומקום באופן מיידי על קרח.
- מקום μMacs עמודות לדוכן המגנטי. אנו ממליצים שלא לעבד יותר מ -12 דגימות בכל פעם (6 - 8 דגימות הן אופטימליים).
- עמודות Miltenyi טרום לאזן עם טמפרטורת חדר 1 מיליליטר הכביסה חיץ. זה ייקח בערך 15 דקות.
- בינתיים, להוסיף 100 μl של חרוזים streptavidin ל50 - 100 μl של רנ"א biotinylated. דגירה בטמפרטורת חדר למשך 15 דקות עם רוטציה. < li> אם כל אחת מהעמודות לא יזם ניקוז על ידי החברה ניתן להקל על ידי לחיצה קלה על החלק העליון של העמודה עם אצבע עטוית כפפה. ברגע שהזרימה החלה לנקז את העמודות בקלות.
- להחיל את הרנ"א / חרוזים לעמודות. מחק את הזרימה דרך אלא אם כן אתה רוצה לשחזר את שבר RNA ללא תווית (ראה סעיף 7).
- לשטוף שלוש פעמים עם 0.9 מ"ל של 65 מעלות צלזיוס חיץ כביסה (1 טיפים פיפטה מ"ל להתכווץ כאשר pipetting חוצצים על 65 מעלות צלזיוס).
- לשטוף שלוש פעמים עם 0.9 מיליליטר טמפרטורת חדר הכביסה חיץ.
- RLT 700 פיפטה μl חוצץ (ערכת ניקוי MinElute RNeasy, Qiagen) לתוך צינורות חדשים 2 מ"ל ומניח אותם מתחת לעמודים.
- Elute את הרנ"א חדש שעבד לתוך מאגר RLT על ידי הוספת 100 μl של DTT מ"מ 100 לעמודות.
- בצע elution 3 דקות סיבוב שנייה מאוחר יותר לתוך הצינור אותו על ידי הוספת 100 μl נוסף של DTT מ"מ 100.
6. שחזור של רנ"א חדש תעתיק
ontent "> המשך עם ניקוי MinElute RNeasy (Qiagen) הפרוטוקול בעקבות הוראות היצרן. Elute ב25 2 ריכוזי μl nuclease ללא H O. מדוד RNA באמצעות ספקטרופוטומטר Nanodrop. כדי למנוע את הצורך להפשיר ולהקפיא מחדש RNA לפני הגשה זה assay תפוקה גבוהה, אנו ממליצים הכנת cDNA מייד לאחר RNA החדש שעבד הוא מטוהרים. השתמש 2.5 μl של RNA החדש שעבד ב20 תמהיל סינתזת cDNA μl לסינתזת cDNA בעקבות הוראות היצרן. ביצוע בקרות qRT-PCR באמצעות 1 : 10 דילולים של תמהיל cDNA החנות RNA ב -80 ° C..7. שחזור של ללא תווית, לא כרוך רנ"א (אופציונלי)
במקרה מאוגד צריך RNA להיות התאושש; לאסוף ולשלב את הזרימה דרך (לאחר הוספת פתרון RNA-streptavidin חרוזים לעמודות) וההדחה ראשונה לממטרים שלאחר מכן. בדרך כלל זה מספיק כדי לזרז רק 50% מהמאוגדים RNA כthiים יכיל> 80% מהחומר המוצא.
- הוסף נפח שווה של isopropanol (מלח לא צריך להוסיף כחיץ כביסה כבר מכיל 1 M NaCl).
- צנטריפוגה ב 20,000 × גרם במשך 20 דקות ב 4 ° C. בטל supernatant.
- הוסף נפח שווה של 75% אתנול, צנטריפוגות ב 20,000 × גרם במשך 10 דקות ב 4 ° C, להשליך supernatant.
- ספין בקצרה ולהסיר אתנול שיורי עם 200 פיפטה μl.
- ספין בקצרה ולהסיר אתנול שיורי עם 20 פיפטה μl.
- אל תאפשר לרנ"א לייבוש. Resuspend זה בH μl 100 2 א ' מערבבים היטב על ידי pipetting למעלה ולמטה 5 - 6 פעמים. לדגור על 65 מעלות צלזיוס למשך 10 דקות עם רעד ולהעביר ישירות לקרח.
- לבדוק את איכות הרנ"א על ידי ניתוח electrophoretical להוציא השפלה RNA.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
1. חומר מוצא ותשואות צפויות
בעקבות 1 שעה (HR) של 4sU-RNA החדש שעבד חשיפה מייצג כ 4 - 1% מכלל RNA הסלולרי. זה יהיה נמוך יותר ב- נעצר גדילת תאים כפי שהם כבר לא לסנתז RNA לחשבון לצמיחת תאים / שכפול. כאשר תיוג עבור שעה 1, אנו ממליצים להתחיל את assay עם 60-80 מיקרוגרם של רנ"א הכל. החל עם פחות מ -30 מיקרוגרם של תוצאות כלל RNA בכדורי RNA קטנים שקשה לראות אחרי צעד biotinylation ובכך עלולים ללכת לאיבוד בקלות. רמות RNA קלט ניתן להעלות ל ככל 150 מיקרוגרם לתקופות קצרות מאוד של תיוג (לדוגמה: 5 - 10 דקות). כאשר משך הזמן של רנ"א תיוג מתקצר משעת 1 עד 5 דק 'התרומה של רצפים קצרי חי intronic בתמללו חדש עליות RNA מ ~ 60% עד 80% ~ 9. כאינטרונים מושווים באופן משמעותי יותר לקידוד רצפים כמו גם 5'-וUTRs 3'-, כמות עיבד חדשהרנ"א, אשר יכול להיות מטוהר בעקבות קצר או 4sU תיוג אולטרה קצר אפילו, לא לרדת באופן ליניארי. ככזה, השגנו> 0.5% מרנ"א הכל לאחר 5 דקות של 4sU תיוג בקווי B-תאים אנושיים שאינם חסיד 9. צריך, עם זאת, לציין כי ריכוז גבוה יותר של משכי 4sU ומעט יותר של תיוג עשוי להידרש כדי להשיג שיעורי התאגדות 4sU דומים בתאים חסיד. אמנם גם שיעור 4sU-התאגדות נמוכה יאפשר לכידה יעילה וטיהור של תמלילים גדולים, uridine עשירים, תמליל קצר מאוד עם תוכן uridine נמוך (miRNAs למשל) עשויה לברוח טיהור אפילו בעת שימוש בריכוזים גבוהים (4sU> 1 מ"מ). ב fibroblasts העכברי-NIH 3T3, שעה 1 חשיפת 200 מיקרומטר 4sU שכותרתו RNA החדש שעבד עם על אחד 4sU שאריות ל100 - 50 נוקלאוטידים (NT) 5. זה אמור לאפשר התאוששות יעילה ביותר של תמלילים> 500 - 1000 NT באורך. בהתאם לכך, אנו נצפו גודל תמליל קל בלבדהטיה כאשר תיוג עבור שעה 1 באמצעות 200 4sU מיקרומטר בשני fibroblasts Murine וB-תאים אנושיים 7. תוך שעה 1 של 200 מיקרומטר 4sU לא הביאה לכל שינוי משמעותי ברמות תעתיקים סלולריות בfibroblasts Murine, חשיפה ממושכת של תאים ל≥ 4sU מיקרומטר 200 עושה מביא לגרעון צמיחה למדידה בתוך 24 שעות (נתונים שלא פורסמו). לכן, גם את משך הזמן של תיוג ו4sU-ריכוז המועסק צריך להיות ממוזער כדי למנוע תופעות מחוץ לרחם או רעילות. דרך קלה כדי לקבוע את הריכוז מינימאלי 4sU-הנדרש להתאוששות יעילה של RNA החדש שעבד היא לטהר את RNA החדש שעבד הבאים 4sU תיוג עם ריכוז גדל והולך של 4sU (למשל 50-1600 מיקרומטר). כפי שניתן לראות באיורים 2 א ו -2, התאוששות של RNA החדש שכותרתו עיבד במשך שעה 1 בfibroblasts אדם הראשוני גדלו באופן משמעותי בין 50 ל 200 מיקרומטר 4sU אבל אז התחילו לרמה.
2. נקודהכתם כימות של התאגדות 4sU (אופציונלי)
במקרים מסוימים זה יכול להיות עניין כדי למדוד את כמות התאגדות 4sU ברנ"א הכל. זה נעשה הכי טוב על ידי ניתוח כתם נקודה על רנ"א biotinylated באמצעות המצומד streptavidin. בשל האופי הכימי שלה iodoacetyl-ביוטין הוא תגובתי יותר תיאול קבוצות מאשר ביוטין, וכתוצאה מכך HPDP biotinylation של כמעט כל שאריות 4sU ב-RNA החדש שעבד. חשוב לציין כי, כמו ביוטין, HDPD, iodoacetyl-ביוטין אינו מסיס במים ולכן היא הוסר ביעילות על ידי מיצוי כלורופורם כפי שבוצע עבור ביוטין-HPDP. לכן, יכולים להיות מועסקים תנאי תגובה זהים וריכוזים כבעת שימוש ביוטין, HPDP. עם זאת, iodoacetyl-ביוטין אינו הפיך. זה יכול אפוא לא ישמש לטיהור של RNA החדש שעבד בגישות המבוססות על עמודות. תוך השימוש בiodoacetyl-ביוטין מאפשר לכמת 4sU-התאגדות, מבוססות מדידות ביוטין-HPDP לשקול גם4sU-התאגדות ויעילות biotinylation. העסקת שני ריאגנטים biotinylation לאותו המדגם מאפשרת למדידת יעילות biotinylation של RNA-Incorporated 4sU. נראה biotinylation יעילות של ביוטין-HPDP ל4sU שכותרתו RNA להיות כ פי שלוש פחות מזה של iodoacetyl-ביוטין מצביע על כך שרק אחד מכל שלושה על 4sU שאריות ב-RNA החדש שעבד למעשה biotinylated ידי ביוטין-HPDP (איור 3). על ידי השוואת עוצמות אות לדוגמה עם biotinylated השליטה DNA אוליגו, ניתן למדוד צפיפויות biotinylation. עבור רוב שורות תאי יונקים איתות חיובית עדיין צריכה להתגלות ב10 ננוגרם של רנ"א biotinylated לאחר שעה 1 של 200 תיוג 4sU מיקרומטר. אות רקע חלשה היא בדרך כלל זיהוי לריכוז הגבוה ביותר (1 מיקרוגרם) של RNA ללא תווית.
3. טיהור של רנ"א פרסום חדש בתעתיק
שחזור של RNA החדש שעבד הוא קוואן ביותרtitative. אם התחיל עם אותו ריכוז RNA אתה יכול לצפות להשיג את אותן כמויות של RNA החדש שעבד עבור כל הדגימות. כמו מבחני מבוססי עמודה רבים, אוסף חדש של RNA עיבד באמצעות ערכת MinElute RNeasy עלול לגרום לספיגה נוספת ב230-260 ננומטר (נוכחות של חומרי ניקוי הנגזרים ממאגרי הכביסה) שעלול להפריע למדידות OD 260. הדבר באו לידי הביטוי במידה פחותה יותר בעת שימוש בצינור טרי 2 מיליליטר אוסף לכל שלב צנטריפוגה. עם זאת, כל מדידות OD באופן בלתי סביר גבוהות (> פי 2 יותר מאשר דוגמאות אחרות) צריכים להיחשב בזהירות, במיוחד אם 260/280 יחס OD הם <1.7. עבור ניתוחים למטה זרם זה ובכך לעתים קרובות הטוב ביותר לשימוש באותה הכמות של תבנית נפח RNA לכל הדגימות. במקרים שבו התשואות של רנ"א שכותרתו הן נמוכות מצפוי לסימון סימנים של השפלה RNA על ידי ניתוח electrophoretical. RNA עבד חדש מכיל כמויות גדולות יותר באופן משמעותי של תמלילים גדולים, unspliced עם להקות rRNA הטיפוסיות להיות הרבה פחות בולט (איור 4).
4. כימות של RNA חדש בתעתיק
לבסוף, שיעורי התאגדות של 4sU ב-RNA החדש שעבד ניתן לכמת באופן ישיר על ידי ניתוח spectrophotometric מבוסס על ספיגת מקסימלית 4sU ב 330 ננומטר וOD 330/260 היחס 5,18. זה דורש> 3 מיקרוגרם של רנ"א שכותרתו מרוכז בנפח קטן (10 - 20 μl) על ידי isopropanol / משקעים אתנול. כדי להימנע מאיבוד RNA גלולה הקטנה שיתוף עם הממטרים 30 מיקרוגרם של גליקוגן nuclease חינם (Fermentas, # R0551) צריכה להתבצע. שיא נוסף הוא גלוי ב 330 ננומטר המשקף את שיעור שילוב של 4sU לרנ"א עיבד מחדש (איור 5).
/ Files/ftp_upload/50195/50195fig1highres.jpg "/>
איור 1. . העיקרון של תיוג מטבולית עם 4-thiouridine (4sU) 4sU מתווסף לתאים לנדרש (5 - 120 דקות) ואחרי זמן הכנה של רנ"א הסלולרי בסך הכל. בעקבות biotinylation תיאול ספציפי, סך הסלולרי RNA הוא מופרד ל4sU שכותרתו RNA, שזה עתה עיבד, ו-RNA ללא תווית, קיים בשימוש בחרוזים מגנטיים streptavidin מצופים. RNA עיבד לאחרונה הוא התאושש מאת החרוזים באמצעות סוכן צמצום אשר cleaves אגרות חוב דיסולפיד המקשרים את הרנ"א חדש שעבד לחרוזים. לחץ כאן לצפייה בדמות גדולה.
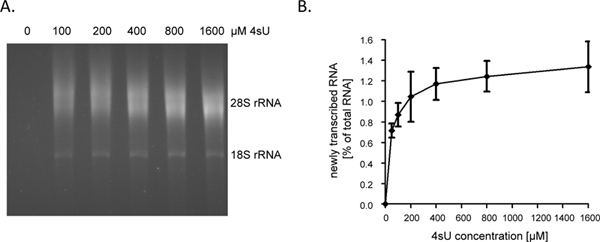
איור 2. שחזור של RNA החדש שעבד לאחר ריכוז גדל והולך של 4sU. () fibroblasts עורלה האנושי הראשוני (HFF) הודגרו עם 100, 200, 400, 800 או 1,600 מיקרומטר של 4sU. RNA החדש שעבד היה מטוהר מ50 מיקרוגרם הכולל סלולרי RNA ונתונים לניתוח electrophoretical. כצפוי, עלייה תלויה בריכוז התאושש RNA עבד לאחרונה נצפתה שהתחילה מישור בריכוזים גבוהים יותר. (ב) סכומים של מטוהרים RNA חדשים שעבד היו לכמת באמצעות תוכנת 1.45s ImageJ. הנתונים משולבים של ארבעה ניסויים בלתי תלויים על הסכומים של RNA החדש שעבד התאוששו בעקבות ריכוזים שונים של 4sU תיוג הנעים בין שני 50-800 מיקרומטר 4sU (N = 2) או 100 -. 1,600 מיקרומטר 4sU (N = 2) מוצג לחץ כאן כדי להציג דמות גדולה.
upload/50195/50195fig3.jpg "alt =" איור 3 "עבור: תוכן-width =" 4.5in "עבור: src =" / files/ftp_upload/50195/50195fig3highres.jpg "/>
איור 3. הערכת התאגדות 4sU ברנ"א הכל 4sU שכותרתו באמצעות ניתוח כתם נקודה. סך RNA הייתה מבודדת מfibroblasts העכברי-NIH 3T3 או fibroblasts עורלת אדם (HFF) מודגרות עם 200 מיקרומטר 4sU במשך שעה אחת. אין 4sU התווסף למנה אחת כביקורת שלילית. לשניהם קשר HFF עכבות (n = תאים שאינם צומחים) וגידול בתאים (y) נכללו במחקר. RNA היה מבודד באמצעות מגיב Trizol ולאחר מכן מוצמד לביוטין-HPDP או iodoacetyl-ביוטין. ריכוז של כל דגימה הותאם לμl 200 ng / μl ו5 של דילול זה (כלומר 1 מיקרוגרם של רנ"א), וכן שלושה פי 10 דילולים הבאים (כלומר 100, 10, ו 1 ng-RNA, בהתאמה), היו כל הבחין על פיסת קרום זיטה. 5 דילולים μl של ביוטין שכותרתו DNA אוליגו הונחו על הממברנה כפקדים חיוביים בconcentratיונים החל ng / μl למטה 20-20 pg / μl (כלומר 100-0.1 ng, בהתאמה). צפיפות ביוטין הייתה נחקר באמצעות המצומד peroxidase streptavidin-חזרת.
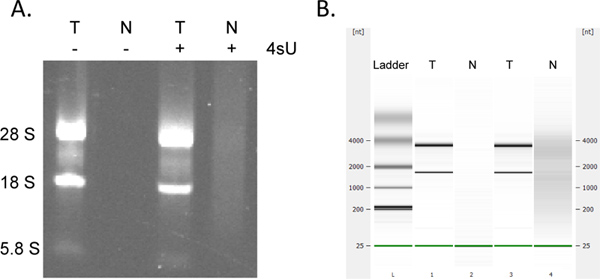
איור 4. ניתוח Electrophoretical של RNA חדש שעבד ומוחלט. סה"כ רנ"א (T) ו-RNA החדש שעבד (N) שהוכן מfibroblasts 3T3-NIH העכברי בתרבית הן בנוכחות והיעדר 500 מיקרומטר 4sU עבור שעה 1 נותחה על ידי ג'ל אלקטרופורזה agarose () ו( באותו סדר) באמצעות Bioanalyser Agilent (ב '). אין RNA היה התאושש ללא טיפול 4sU של תאים. מטוהרים RNA עיבד חדש מכיל כמויות גדולות יותר של mRNAs משקל המולקולרית גבוהה וrRNAs משמעותי פחות בוגר מסךRNA כבולט בין 28S, 18S, ולהקות rRNA 5.8S. לחץ כאן לצפייה בדמות גדולה.
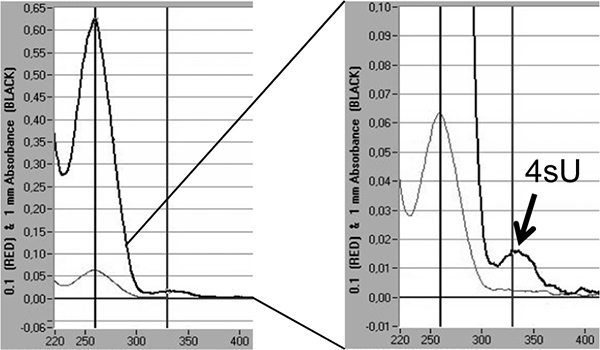
איור 5. כימות התאגדות 4sU בRNA חדש עיבד ידי ניתוח spectrophotometric. RNA חדש עיבד מטוהר מ2 100 מיקרוגרם רנ"א הכל x לאחר שעה 1 של 200 מיקרומטר 4sU בfibroblasts 3T3-NIH Murine. RNA החדש שעבד היה זירז עם isopropanol אתנול / לאחר הוספת 30 מיקרוגרם של גליקוגן nuclease חינם. ניתוח spectrophotometric של RNA החדש שעבד מתקבל על ידי 1000 ספקטרופוטומטר Nanodrop מוצג. הקווים האפורים הבהירים מייצגים מדידות על 0.1 מ"מ ואילו קווים האפורים העבים יותר, כהים מייצגים מדידות בעמודת נוזל 1 מ"מ. בצד ימין, הגדלה של שיא הכחדה מייצג tהוא שילבו 4sU-שאריות מוצגת. בהתבסס על הכחדת שיתוף היעילה של 4sU 18 שיעורי ההתאגדות של 4sU ניתן לאמוד.
| משך תיוג [דקות] | ריכוז 4sU מומלץ [מיקרומטר] |
| 120 | 100 - 200 |
| 60 | 200-500 |
| 15 - 30 | 500 - 1000 |
| <10 | 500 - 2000 |
טבלת מס '1. מומלץ ריכוזי 4sU.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
תיוג חילוף חומרים של RNA החדש שעבד באופן משמעותי מגביר את העצמה של טכנולוגיות תפוקה גבוהה כמו microarrays וRNA-Seq על ידי מתן תבניות מתאימות יותר להתייחס לשאלה הביולוגית של עניין. הפרוטוקול הנוכחי עבר אופטימיזציה נרחבת. היא מאפשרת העשרה> 1,000 פי חדש של RNA עיבד ומספקת תוצאות לשחזור מאוד.
עיצוב ניסיוני של ניסוי 4sU תיוג הוא בעלת חשיבות מכרעת כRNA חדש שעבד יהיה לתאר את פעילות תעתיק בזמן אמת רק בזמן חשיפה של תאים ל4sU. אם את השינויים בפועל בשיעורי שעתוק לאחר גירוי כבר שככו, אלה יחסרו בעת ניתוח RNA החדש שעבד אף שהשינויים ברמות רנ"א הכל עדיין עשויים להתגלות. לכן, הבנה טובה של הביולוגיה הבסיסית חשוב להגדיר התקנה הניסיונית כמו גם אופטימלי תקופות Oזמן F לחשיפת 4sU. להלן, אנו מספקים המלצות ודרכים להימנעות ממכשולים נפוצים לשלבים החשובים ביותר.
הכנה של פתרונות מניות וכלי פלסטיק
כל פתרונות המניות חייבים להיות מוכנים להשתמש במי nuclease חינם. שימוש בתוך בית מים מטוהר deionized עלול לגרום לבעיות אם המים מכיל צמצום הסוכנים. במקרה אחד, זה גרם לאובדן של כל RNA שכותרתו המלא. לכן, אנו ממליצים בחום לקנות מראש עשה nuclease ללא NaCl, טריס-Cl, EDTA, ציטראט ומים נתרן. להבטיח תנאי nuclease חינם בכל העת. Dimethylformamide (DMF) מתמוסס כמה חומרים פלסטיים. מצאנו כי שימוש ב25 pipettes פלסטיק תרבית תאי מ"ל להעביר DMF מבקבוק זכוכית המניות שלה ל50 צינורות מ"ל פלקון להכין פתרון מניות ביוטין-HPDP היה די כדי להפחית באופן משמעותי תשואות של RNA החדש שעבד מ כל assay. מעניין, זה לא להשפיע לרעה על biotinylיעילות ation (כפי שנבדק על ידי נקודת כתם) אבל הביא 75 ל> אובדן 90% של RNA החדש שעבד שיכול להיות התאושש את החרוזים. ההפסד היה בולט ביותר כאשר משך התיוג הופחת 60-30 דקות או פחות. סביר להניח, חומר eluted מפלסטיק pipettes ידי DMF חלקית הרס את הציפוי של חרוזי streptavidin. לכן, השימוש בחומרים פלסטיים אינו ידועים כתואם עם DMF יש להימנע בכל האמצעים. מאותן סיבות, לא אמורים לשמש מגרדים סלולריים כדי לשפר את ההתאוששות של דגימות Trizol מצלחות תרבית תאים. זה מעניין לציין כי חומרים המשוערת מזוקקים מהפלסטיק על ידי DMF או Trizol כנראה לא הוסרו על ידי מיצוי כלורופורם ולא isopropanol / משקעים אתנול.
תרבית תאים
צפיפות תאים על הצלחות היא בעלת חשיבות מכרעת. בניסוי אחד שבו התאים שנראה מעט מדי מחוברות (90 -100%), התייחסנו fibroblasts העכברי-NIH 3T3 למשך 30 דקות עם 100 U / מ"ל של אינטרפרון (IFN) או α γ. בתאים פחות מחוברות אפילו 15 דקות של טיפול IFN כבר הביאו 5 - לאינדוקציה של פי 8 של גנים כמו irf1 או socs3 5. עם תאים להיות מעט ניתוח microarray מחוברות מדי לא הראה שום אינדוקציה של גני IFN-מושרים לאף בקצב המהיר ביותר עין מתנהל כמו גני irf1 או socs3. לכן, צפיפות תאים היא גורם מכריע עבור ניסויי 4sU תיוג ואת כל צלחות תרבית התאים יש לבחון היטב לפני שמתחיל את התיוג.
4sU הוא ribonucleoside photoactivatable ו-RNA המכיל 4sU הוא crosslinked יעילות לחלבונים לאחר חשיפה למקור אור 365 ננומטר. התאים שטופלו ב-4sU צריכים להיות מתורבתים בחושך והחשיפה לאור בהיר יש להימנע. לאחר ההסרה של חלבונים תאיים על ידי בידוד RNA Trizol סיכון זה מצטמצם באופן משמעותי.
<כיתת p = "jove_content"> 4sU לא שולבה ה-DNA התאי. צריך, עם זאת, לציין כי רנ"א הכל עדיין מכיל כמויות קטנות של ה-DNA התאי. בעת שימוש ב4sU תיוג וניתוח Q-RT-PCR ללמוד ביטוי גנים בזיהום נגיפי ציטומגלווירוס מצאנו לנכון לכלול צעד DNaseI לעכל בפרוטוקול כדי להסיר את הגנום הנגיפי concatemeric 19. זה כנראה לא הכרחי בעת שימוש בפרוטוקולים במורד הזרם שאינם רגישים לנוכחותו של ה-DNA.שיעורי התאגדות 4sU וריכוז 4sU אופטימלי
4sU הוא נלקח בקלות על ידי תאים עם רמות התוך תאיים וחוץ כנראה equilibrating בתוך פחות מ 9,16 דקות. שיעורי ספיגה והטמעה של 4sU הם ריכוז תלויים. לכן, ריכוז 4sU יכול להיות מותאם בנוחות על פי המשך העסיק של תיוג. טבלת 1 מספקת ייעוץ בריכוזי 4sU בrelation כדי משך סימון המבוסס על הניסיון האישי הטוב ביותר שלנו. במשך שעה 1 של תיוג 4sU בתאי יונקים, 200 מיקרומטר 4sU יהיה מספיק עבור מרבית היישומים וכתוצאה מכך על אחת שאריות 4sU ל50 עד 100 נוקלאוטידים ברנ"א חדש שעבד בfibroblasts.
בשנים האחרונה, יש לנו להחיל 4sU תיוג למגוון רחב של סוגי תאים ממקור אנושי ועכברי כולל fibroblasts, בתאי אנדותל, תאי אפיתל, תאי stroma מח עצם, מקרופאגים וT-תאים. בנוסף לכך, תאי דרוזופילה וXenopus שמשו בהצלחה. בכל הניסויים הללו, התאגדות 4sU נמצאה להיות יעיל ביותר הדורש התאמות מינימאליות בריכוז 4sU לתאים מסוגים השונים. בעת הגדרת השיטה לסוגים חדשים של תאים, היינו ממליץ לתאי תווית עם הגדלת 4sU-ריכוזים (למשל החל 50-1600 מיקרומטר) ולנתח את הקשר של מטוהרים חדשים שעבד RNA ל4sU-ריכוזים יושמו (ראה איור 2 א / ב '). 4sU-הריכוז שבו הסכום של מטוהרים RNA עיבד לאחרונה נכנס מישור צריך להיבחר.
במקרים שבם נעשו שימוש, עכבות תאי קשר מחוברות ביותר, אנו ממליצים להשתמש בריכוזי 4sU מעט גבוהים יותר (למשל 500 במקום 200 מיקרומטר), כדי להבטיח שילוב 4sU יעיל. בנוסף, במקרים בהם לכידה של תמלילים חדשים שעבדו קצרים מאוד (<200 NT) היא עניין מיוחד, ריכוז 4sU עשוי גם צריך להיות מוגבר. זה לא צריך להיות בשילוב עם תיוג זמנים ממושכים (למשל HR> 1) על מנת להימנע מהשפעות מחוץ לרחם או רעילות. לבסוף, מצאנו כי שימוש בנפח קטן מדי של תקשורת בתרבות סלולרי עלולה להפחית את יעילות שילוב 4sU. לפיכך, אנו ממליצים על שימוש 5 מ"ל או 10 מ"ל של מדיום לכל 10 ס"מ או 15 ס"מ צלחת, בהתאמה.
אופן ההכנה בסך הכל הסלולרי RNA
להצלחתו של פרוטוקול זה הוא חיוני כדי להשיג רנ"א הכל נקי נטול RNase סלולרי. שימוש מ"ל Trizol 5 לכל מנה 15 ס"מ מייצר RNA נקי ללא nucleases. אנו ממליצים להשתמש בפרוטוקול Trizol שונה על ידי Chomczynski et al. 20. ראשית, היא מתאימה יותר לבודד כמויות גדולות של RNA (> 100 מיקרוגרם), כמו התוצאות המשופרות בכח הצנטריפוגלי כדורים מוצקים יותר אשר קלים יותר לטפל בשלבי הכביסה. עם זאת, זה דורש שימוש בצינור פלסטיק מיוחד ומתאמים כמו 15 הצינורות הרגילים מ"ל המעבדה פלקון לא שורדים יותר מ 6,000 × גרם. שנית, זה משפר את ההסרה של ה-DNA וגליקופרוטאינים. זה הופך להיות ברור במיוחד בעת הכנת RNA מן האיברים או רקמות. שלישית, זה אינו מגביל את הסכום המקסימאלי של RNA הכולל שיכול להיות מבודד. למרות שאנחנו גם מצאנו שיטות בידוד RNA המבוססים על העמודה (RNeasy למשל) כדי לספק RNA באיכות מתאימה, טורים סטנדרטיים arדואר רק מסוגל ללכוד עד 100 מיקרוגרם של רנ"א הכל וכך להגביל את הכמות של חומר מוצא. לבסוף, על ידי הסרת אתנול נותר פעמיים עם פיפטה, ייבוש של רנ"א כדי להסיר אתנול השיורי אינם נדרש עוד. זה מבטל את הסיכון של ייבוש על RNA, אשר עלול להיות קשה כדי לפזר שוב לאחר מכן. באופן עקרוני, 4sU התיוג הוא ישים in vivo, למשל על ידי הזרקת IV של עכברים. עם זאת, אנו מציינים כי טוהר RNA מייצג בעיה רצינית הדורשת טיהור של תמלילים פולים לפני הטיהור של RNA החדש שעבד (נתונים שלא פורסמו).
Biotinylation והסרה של ביוטין מאוגד
ביוטין-HPDP הוא 100% תיאול ספציפיים ויוצר קשר דיסולפיד בין שאריות ביוטין ומולקולות RNA תיאול שכותרתו חדשה עיבד. Biotinylation יעילות של רנ"א 4sU שכותרתו היא כ -30%, כפי שנקבע על ידי ניתוח כתם נקודה 5. כמו ביוטין, HPDP אינו מסיסים במיםניתן להסירו ביעילות על ידי מיצוי כלורופורם. בעוד שלב חילוץ כלורופורם אחת מספיק כדי להסיר את הרוב המכריע של ביוטין מאוגד אנו חוזרים באופן קבוע על שלב זה, כדי להבטיח הסרה מלאה. כדי לצמצם את אובדן RNA בשלב חילוץ כלורופורם 2 צינורות מ"ל שלב נעילת ג'ל כבדים (אפנדורף) עשוי לשמש בעקבות הוראות היצרן. בדרך כלל אנו משתמשים בצינורות נעילת השלב רק לשלב חילוץ כלורופורם השני ככרכי התבנית של הצעד הראשון הם לעתים קרובות גבוהים מדי כדי להיות תואם באופן ישיר עם הצינורות האלה. לאחר פינוי מאוגד ביוטין, HPDP, RNA הוא התאושש על ידי isopropanol / משקעים אתנול. חשוב לשים לב כי ערכות המבוססות על העמודה מסחריות להתאושש RNA biotinylated (למשל RNeasy מQIAGEN) לא אמור לשמש כהם מכילים צמצום סוכנים במאגרים שסופקו, אשר ידבק בונד דיסולפיד ולהסיר ביוטין מRNA חדש שעבד .
טיהור החדשהly RNA עיבד
אל תוסיף יותר מ -100 RNA biotinylated μl 100 חרוזים streptavidin μl. הוספת נפח פחות עדיף. עם זאת, את אותו הנפח של רנ"א יש להוסיף לכל הדגימות. התאם את עוצמת קלט רנ"א (בין דגימות) שבו אתה מוסיף את חרוזי streptavidin פשוט על ידי הוספת הנפח הנדרש 1x TE את החרוזים. דרך קלה לעשות 100 nuclease ללא טרי מ"מ DTT היא למזוג כמות מספקת של אבקת DTT לתוך צינור פלקון דגש על קנה מידה אולטרה רגישה ולאחר מכן להוסיף את הכמות הנדרשת של nuclease ללא H 2 O לייצר DTT מ"מ 100 (64.8 מים μl לכל 1 מ"ג DTT).
במהלך הפיתוח של 4sU תיוג בדקנו חרוזים streptavidin מהספקים שונים. מספרם נוצר כמויות גדולות של רקע. לכן, אנו ממליצים בחום להשתמש בחרוזי streptavidin Miltenyi כ, עד כה, מעולם לא חוו שום בעיות עם לשאת מעל ללא תווית של רנ"א מהרקמה גדגימות RNA-ulture נגזרו. בדרך זו, קטנה כמו 150 ננוגרם של רנ"א שכותרתו יכול להיות מטוהר במיוחד מ150 מיקרוגרם RNA biotinylated (ב100 μl מים) באמצעות 100 μl של חרוזים streptavidin. איזון של החרוזים עם חיץ האיזון מסופק עם החרוזים עשוי להתבצע ועשוי לשפר את השיעורים ללכוד מעט 13.
בקרת איכות
אנו ממליצים ביצוע בקרות Q-RT-PCR על RNA החדש שעבד לפני העמדתו לניתוח תפוקה גבוהה. זה עשוי לכלול כימות של מספר גנים הידועים להתייחסות להיות מוסדר באופן דיפרנציאלי בהגדרה הניסיונית נתון. במקרים בהם 4sU התיוג מועסק ללמוד שיעורי ריקבון RNA, אנו ממליצים לכמת תמליל קצר מועד (למשל myc, FOS) וחיים ארוכים אחד (למשל GAPDH) ב-RNA הן כולל ועתה עיבד. היחס של RNA החדש שעבד / הכולל צריך להיות גבוה יותר באופן משמעותי (~ 5 - עד 10 פי)לתעתיקים קצרי החיים. המבוסס על RNA מחצית החיים של גן התייחסות, ניתן לקבוע RNA מחצית חיים. אם כל שלושת שברי RNA (RNA סך הכל, RNA החדש שעבד וRNA קיים מראש ללא תווית) מנותחים לארבעה או יותר גנים, נורמליזציה של תת RNA השונים יכול להתבצע על ידי ניתוח רגרסיה לינארית ועשרות בקרת איכות יכולה להיקבע כמתואר 7 , 21.
לניתוח Q-RT-PCR, אנו ממליצים להשתמש 2.5 μl של RNA שבכותרתו 20 תמהיל סינתזת cDNA μl. לשם השוואת תוצאות אופטימלית של Q-RT-PCR להקפיא cDNA בaliquots של 5 μl לפני השימוש ראשון. צינורות להפשיר רק לפני שימוש, מוסיפים 45 μl של H 2 O ו -5 μl הנושא של דילולים לניתוחי Q-RT-PCR. זה משפר השוואתיות בין ריצות PCR שונות באופן משמעותי.
דגימות RNA חדשים שעבדו יש לבדוק סימנים של השפלה RNA באמצעות Bioanalyser Agilent לפני העמדת להםניתוח תפוקה גבוהה (microarrays או RNA-seq). צריך, עם זאת, לציין כי להקות נוספות לעתים נצפו על ידי Bioanalyser Agilent. המשמעות הביולוגית של זה עדיין לא ברור. כRNA עיבד חדש מכיל RNA באופן משמעותי פחות ריבוזומלי, דגימות אלה נכשלות בקרות איכות Bioanalyser Agilent מדי פעם. אם זה לא עקב דגימות השפלה RNA גלויות של איכות מקובלת הם בדרך כלל בסדר להיות חשוף לניתוח תפוקה גבוהה.
תאימות של RNA החדש שעבד עם ניתוחים למטה זרם
RNA עבד חדש מכיל באופן משמעותי יותר מאשר ה-mRNA רנ"א הכל. זה נובע בעיקר לכמויות הגדולות יותר של רצפים ברנ"א intronic חדשים עיבד המגבירים כאשר משך 4sU התיוג מתקצר. לכן, אנחנו לא מתחייבים דלדול של rRNAs מדגימות RNA החדש שעבדו באופן קבוע כמו זה דורש כמויות גדולות יותר של חומר מוצא תוך providרווח (~ כפול) קטן ולא ing שבאינם קורא rRNA. לבסוף, הוא נשאר יש לציין, כי אחוז גבוה יותר של תמלילי unspliced, משקל מולקולרי גבוהים הנוכחיים ב-RNA שזה עתה עיבד עשוי לדרוש פיצול נוסף בעת הכנת ספריות cDNA עבור הדור הבא של רצף. תוצאות של צעד פיצול הגודל ולכן צריכה להיות באיכות מבוקרת בקפידה.
נורמליזציה נתונים למדידות חצי חיות RNA
הגישה הסטנדרטית ללנרמל את נתוני ניסוי למדידת זמן מחצית החיים RNA היא לנרמל את כל הנתונים לרנ"א מחצית החיים של גן בית שמירה-מאופיין היטב או RNA חציון זמן מחצית החיים בסוג תא נתון שנקבע בניסויים קודמים. בתאי יונקים, האחרון טמון בטווח של 5 עד 10 שעות 6,7. בעוד גישה זו גם עובדת די יפה למדידות מבוססות 4sU, אמצעים אחרים לנורמליזציה נדרשים אם RNA חציון זמן מחצית החיים אינו ידוע או אם זה מאy אפילו להיות מושפע משינויים במערכת התאית תחת מחקר, למשל על ידי עקום החוצה ממסלול דעיכת רנ"א. 4sU התיוג מציע דרך ייחודית של אומדן RNA חציון זמן מחצית החיים המבוססים על הניתוח של כל שלושת שברי RNA, כלומר סך הסלולרי RNA, RNA החדש שעבד, ו-RNA קיים ללא תווית. ככולל סלולרי RNA הוא מופרד לשני שברי RNA האחרונים יכול להיות מועסק מודל רגרסיה לינארית פשוט לנרמל את שלושה שברי RNA זה לזה ולקבוע את RNA חציון זמן מחצית החיים 7,16. חבילת תוכנה זמינה באינטרנט כדי לבצע ניתוחים אלה 22.
לכידה יעילה של תמלילים עם תוכן uridine נמוך עלולה להשפיע על מדידות מחצית החיים RNA וכתוצאה מכך באופן מלאכותי יחסי RNA חדש שעבדו / כולל נמוכים וRNA מחצית חיים ממושכים. היקף הבעיה ניתן להעריך על ידי קשירת קשר RNA מחצית חיים או יומן (שזה עתה עיבד / רנ"א הכל יחסים) נגד uridiתוכן ne של כל התעתיקים 7,15. זה גם מספק בקרת איכות טובה כדי להעריך את ההבדלים בשיעורי 4sU-התאגדות בין דגימות או תנאים שונים. במקרים בהם קשר משמעותי לתוכן uridine הוא ציין זה יכול להיות מתוקן על ידי bioinformatic פירושו 15. עם זאת, יש לציין כי התרומה של תמלילים בוגרים בRNA החדש שעבד לא יכולה להיות מובחנת בקלות מהרבה יותר גדול ולכן הרבה יותר מבשרים uridine עשירים. אלא אם כן את הקינטיקה העיבוד של תמליל נתון ידועות (שהם בדרך כלל לא) פשוט תיקון לתוכן uridine נמוך (לכידה יעילה) עשוי לעוות גס RNA מחצית חיים. ככזה, לאחרונה מצאו עיבוד של רוב snoRNAs אדם להיות יעיל מאוד 9. אם היינו תיקן את היחסים חדשים שעבדו / סה"כ RNA לתוכן uridine הנמוך של קטן למדי (70-300 NT) snoRNAs, זה היה גורם snoRNA הקצר מאוד חצי ליבes (<5 דקות) עם יחס RNA החדש שעבד / סה"כ רב העולים על 100%. לכן, בדרך כלל אנחנו לא ממליצים על תיקון לתוכן uridine נמוך כאשר מודדים RNA מחצית חיים.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
החוקרים מצהירים כי אין להם אינטרסים כלכליים מתחרים.
Acknowledgments
ברצוננו להודות לאיימי רגן לקריאה מדוקדקת של כתב היד. עבודה זו נתמכה על ידי מענק NGFN פלוס # 01GS0801, MRC מלגת מענק G1002523 וNHSBT מענק WP11-05 לLD וDFG מענק FR2938/1-1 לCCF
Materials
| Name | Company | Catalog Number | Comments |
| 4-thiouridine | Carbosynth | T4509 | Prepare 50 mM stock in sterile H2O, store at -20 °C in aliquots of 50-500 μl, discard unused reagent, do not refreeze. |
| Trizol | Invitrogen | 15596026 (100 ml), 15596018 (200 ml) | WARNING - CORROSIVE and HAZARDOUS TO HEALTH! Ensure immediate access to Phenol antidote (PEG-Methanol); Store at 4 °C. |
| Chloroform | Sigma | 372978 | WARNING - HAZARDOUS TO HEALTH |
| Isopropanol | Sigma | 650447 | |
| Sodium citrate, nuclease-free | Sigma | C8532 | Prepare 1.6 M stock solution using nuclease-free water. |
| 5M nuclease-free NaCl | Sigma | 71386 | Stock solution |
| Nuclease-free H2O | Sigma | W4502 | Make 1 ml aliquots in nuclease-free tubes. |
| RNA precipitation buffer | 1.2 M NaCl, 0.8 M sodium citrate in nuclease free water. Prepare in advance under strictly nuclease-free conditions. Store at room temperature in 50 ml falcon tubes. | ||
| Ethanol | Sigma | 459844 | Use with nuclease-free water to prepare 80% ethanol, store at -20 °C. |
| 1 M nuclease-free Tris Cl, pH 7.5 | Lonza | 51237 | Stock solution |
| 500 mM nuclease-free EDTA, pH 8.0 | Invitrogen | 15575-020 | Stock solution |
| 10x Biotinylation Buffer (BB) | 100 mM Tris pH 7.4, 10 mM EDTA in nuclease-free water, make aliquots of 1 ml. | ||
| Dimethylformamide (DMF) | Sigma | D4551 | |
| EZ-Link biotin-HPDP | Pierce | 21341 | Prepare 1 mg/ml stock solution by dissolving 50 mg biotin-HPDP in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. |
| Phase Lock Gel Heavy tubes 2.0 ml | Eppendorf | 0032 005.152 | Optional for the chloroform extraction step. |
| Zeta membrane | BIORAD | 162-0153 | |
| 10x Dot blot binding buffer | 100 mM NaOH, 10 mM EDTA | ||
| Biotin-oligo | 5'-biotin, 25 nucleotides, any sequence | ||
| Sodium dodecyl sulphate | Fisher | BPE9738 | For 100 ml 20% stock solution, add 20 g SDS to 80 ml PBS pH 7-8 and adjust volume to 100 ml. Keep all high-percentage SDS solutions above 20 °C. Warm the solutions slightly should SDS precipitate. |
| EZ-Link Iodoacetyl-LC-Biotin | Pierce | 21333 | Prepare 1 mg/ml stock solution by dissolving 50 mg iodoacetyl-biotin in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. Generates irreversible, thiol-specific biotinylation. |
| Phosphate buffer saline | Gibco | 10010-015 | |
| Dot blot blocking buffer | Mix 20 ml 20% SDS with 20 ml 1 x PBS pH 7-8 and add EDTA to the final concentration of 1 mM. | ||
| Streptavidin-horseradish peroxidase | Vector Laboratories | SA5004 | Store at -20 °C. Mix 10 ml 20% SDS with 10 ml 1 x PBS. Add 20 μl Streptavidin-HRP before use. |
| ECL reagent | GE Healthcare | RNP2109 | Use following the manufacturer's instructions. |
| Super RX, X-RA Film, 18x24 cm | Fujifilm | 47410 19236 | |
| μMacs Streptavidin Kit | Miltenyi | 130-074-101 | Store the beads at 4 °C. |
| Tween 20 | Sigma | P1379 | |
| Washing buffer | 100 mM Tris pH 7.4, 10 mM EDTA, 1 M NaCl, 0.1% Tween 20 in nuclease-free H2O. | ||
| Dithiothreitol (DTT) | Sigma | 43817 | Prepare as 100 mM DTT in nuclease-free H2O, always prepare fresh before use. |
| RNeasy MinElute Kit | Qiagen | 74204 | Store columns at 4 °C, remaining components of the kit at room temperature. |
| 1.5 ml screw-top polypropylene tubes | Sarstedt | 72.692.005 | Compatible with Dimethylformamide |
| 2.0 ml screw-top polypropylene tubes | Sarstedt | 72.694.005 | Compatible with Dimethylformamide |
| 15 ml tubes | BD Falcon | 352096 | Compatible with Dimethylformamide |
| 50 ml tubes | BD Falcon | 352070 | Compatible with Dimethylformamide |
| All solutions/reagents should be stored at room temperature unless otherwise specified. | |||
| Equipment | |||
| UV/VIS spectrophotometer | Thermo Scientific | NanoDrop 1000 | Or equivalent. Use low volume (1-2 μl) for measurements of low RNA concentrations to avoid excessive sample loss. |
| Polypropylene 15 ml centrifuge tubes | VWR International | 525-0153 | In contrast to standard 15 ml tubes, these tolerate up to 15,000 × g |
| High-speed centrifuge | Beckman Coulter | Avanti J-25 | Or equivalent equipment capable of reaching 13,000×g |
| High-speed rotor | Beckman Coulter | JLA-16250 | Or equivalent equipment capable of reaching 13,000×g |
| Adaptors for 15 ml tubes | Laborgeräte Beranek | 356964 | Or equivalent equipment capable of reaching 13,000×g |
| Refrigerated table-top centrifuge | Eppendorf | 5430 R | Or equivalent. |
| Thermomixer | Eppendorf | Thermomixer compact | Or equivalent. |
| Magnetic stand | Miltenyi Biotec | 130-042-109 | One stand holds 8 μMacs columns. |
| Waterbath | Grant | SUB Aqua 5 | Or equivalent. |
| Ultra-fine scale | A&D | GR-202 | Or equivalent. |
| E-Gel iBase Power System | Invitrogen | G6400UK | For RNA gels; or equivalent. |
| E-Gel EX 1% agarose precast gels | Invitrogen | G4020-01 | For RNA gels; or equivalent. |
References
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Mortazavi, A., Williams, B. A., Mccue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Nagalakshmi, U., Wang, Z., et al. The transcriptional landscape of the yeast genome defined by RNA sequencing. Science. 320, 1344-1349 (2008).
- Wilhelm, B. T., Marguerat, S., et al. Dynamic repertoire of a eukaryotic transcriptome surveyed at single-nucleotide resolution. Nature. 453, 1239-U1239 (2008).
- Dölken, L., Ruzsics, Z., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Yang, E., van Nimwegen, E., et al. Decay rates of human mRNAs: correlation with functional characteristics and sequence attributes. Genome Res. 13, 1863-1872 (2003).
- Friedel, C. C., Dölken, L., Ruzsics, Z., Koszinowski, U. H., Zimmer, R. Conserved principles of mammalian transcriptional regulation revealed by RNA half-life. Nucleic Acids Res. 37, e115 (2009).
- Nilsen, T. W., Graveley, B. R. Expansion of the eukaryotic proteome by alternative splicing. Nature. 463, 457-463 (2010).
- Windhager, L., Bonfert, T., et al. Ultra short and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Res. , (2012).
- Melvin, W. T., Milne, H. B., Slater, A. A., Allen, H. J., Keir, H. M. Incorporation of 6-thioguanosine and 4-thiouridine into RNA. Application to isolation of newly synthesised RNA by affinity chromatography. Eur. J Biochem. 92, 373-379 (1978).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23, 232-237 (2005).
- Kenzelmann, M., Maertens, S., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proc. Natl. Acad. Sci. U.S.A. 104, 6164-6169 (2007).
- Schwanhäusser, B., Busse, D., et al. Global quantification of mammalian gene expression control. Nature. 473, 337-342 (2011).
- Rabani, M., Levin, J. Z., et al. Metabolic labeling of RNA uncovers principles of RNA production and degradation dynamics in mammalian cells. Nat. Biotechnol. , (2011).
- Miller, M. R., Robinson, K. J., Cleary, M. D., Doe, C. Q. TU-tagging: cell type-specific RNA isolation from intact complex tissues. Nat. Methods. 6, 439-441 (2009).
- Miller, C., Schwalb, B., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Mol. Syst. Biol. 7, 458 (2011).
- Weintz, G., Olsen, J. V., et al. The phosphoproteome of toll-like receptor-activated macrophages. Mol. Syst. Biol. 6, 371 (2010).
- Lipsett, M. N. The isolation of 4-thiouridylic acid from the soluble ribonucleic acid of Escherichia coli. Journal of Biological Chemistry. 240, 3975-3978 (1965).
- Marcinowski, L., Liedschreiber, M., et al. Real-time Transcriptional Profiling of Cellular and Viral Gene Expression during Lytic Cytomegalovirus Infection. PLoS Pathog. 8, e1002908 (2012).
- Chomczynski, P., Mackey, K. Short technical reports. Modification of the TRI reagent procedure for isolation of RNA from polysaccharide- and proteoglycan-rich sources. Biotechniques. 19, 942-945 (1995).
- Friedel, C. C., Dölken, L. Metabolic tagging and purification of nascent RNA: implications for transcriptomics. Mol. Biosyst. 5, 1271-1278 (2009).
- Friedel, C. C., Kaufmann, S., Dölken, L., Zimmer, R. HALO - A Java framework for precise transcript half-life determination. Bioinformatics. , (2010).



