Summary
Totalt cellulært RNA giver en dårlig skabelon for at studere kortvarige ændringer i RNA-syntese og forfald samt kinetik RNA forarbejdning. Her beskriver vi metabolisk mærkning af nyligt transkriberede RNA med 4-thiouridine efterfulgt af thiol-specifikke biotinylering og oprensning af nyligt transkriberede RNA gør det muligt at overvinde disse begrænsninger.
Abstract
Udviklingen af hel-transkriptomet microarrays og næste generation sekventering har revolutioneret vores forståelse af kompleksiteten af cellulære genekspression. Sammen med en bedre forståelse af de involverede molekylære mekanismer, har præcise målinger af de underliggende kinetik blevet stadig vigtigere. Her disse kraftfulde metoder står over for store begrænsninger på grund iboende egenskaber af skabelonen prøver de studerer, dvs totale cellulære RNA. I mange tilfælde ændringer i totalt cellulært RNA enten forekomme for langsomt eller for hurtigt at repræsentere de underliggende molekylære begivenheder og deres kinetik med tilstrækkelig opløsning. Desuden er bidraget fra ændringer i RNA-syntese, forarbejdning og forfald ikke let differentierede.
Vi har for nylig udviklet høj opløsning genekspressionsprofilering at overvinde disse begrænsninger. Vores tilgang er baseret på metabolisk mærkning af nyligt transskriberede RNA med 4-thiourispise (således også kaldet 4SU-tagging) efterfulgt af stringent oprensning af nyligt transskriberet RNA under anvendelse thiol-specifikke biotinylering og streptavidin-overtrukne magnetiske perler. Det gælder for en bred vifte af organismer, herunder hvirveldyr, Drosophila og gær. Vi har med held anvendt 4SU-tagging at studere real-time kinetik for transkriptionsfaktor aktiviteter, give præcise målinger af RNA halveringstider, og opnå nye indsigter i kinetikken af RNA-behandling. Endelig kan datamodellering være ansat til at generere en integreret, omfattende analyse af de underliggende molekylære mekanismer.
Introduction
Genekspressionsprofilering er et vigtigt værktøj, der anvendes til at studere cellulære processer og den tilhørende komplekse samspil netværk. Undersøgelser af mRNA overflod har typisk været den foretrukne metode til at opnå grundlæggende indsigt i de underliggende molekylære mekanismer. Udviklingen af hel-transkriptomet microarrays 1 og, for nylig, næste generation sekventering af RNA (RNA-seq) 2-4 næret denne tilgang. Mens disse teknologier har revolutioneret vores forståelse af kompleksiteten af cellulære genekspression, de står over for store begrænsninger på grund iboende egenskaber deres template stikprøven, dvs total cellulær RNA. Først, kortvarige ændringer i den samlede RNA-niveauer svarer ikke ændringer i transskription satser, men i sagens natur er afhængig af RNA halveringstid de respektive udskrifter. Mens en femdobbelt induktion af en kortvarig transkript, fx koder for en transskriptionsfaktor, vil være let påviselig i total RNAinden for en time, det samme induktion af en langlivet transkript, fx koder for et metabolisk enzym forbliver næsten usynlig. Hertil kommer en selv fuldstændig lukning (> 1.000-fold ned-regulering) i transkription på en gennemsnitlig gen med en RNA halveringstid på fem timer vil simpelthen tage fem timer for dens samlede RNA-niveauer kun at falde med dobbelt . Derfor analyse af total RNA favoriserer påvisning af opregulering af kortlivede udskrifter, hvoraf mange koder for transkriptionsfaktorer og gener med reguleringsopgaver 5. Desuden er den sande kinetiske kaskade af regulering tilsløret og primære signaleringsbegivenheder kan ikke skelnes fra sekundær. Både til gengæld kan resultere i betydelig skævhed i downstream bioinformationsafdeling analyser. For det andet, kan ændringer i den samlede RNA-niveauer ikke tilskrives ændringer i RNA-syntese eller forfald. Målinger af de sidstnævnte kræver celle invasive metoder, f.eks blokerer transcriptiom brug af actinomycin D 6, og udvidet overvågning af igangværende RNA henfald over tid. Med en gennemsnitlig mRNA halveringstid i pattedyrceller af 5 - 10 hr 5,7, vil mRNA-niveauer i de fleste gener kun er faldet med mindre end to efter flere timers transkriptionel anholdelse. Disse ret små forskelle resultere i groft upræcise målinger af mRNA halveringstider for hovedparten af cellulære gener på grund af den eksponentielle karakter af de underliggende matematiske ligninger. Endelig mens RNA-seq af totalt cellulært RNA viste, at cirka halvdelen af vores gener er underlagt alternative splejsningsbegivenheder 8, de underliggende kinetik samt de dynamiske mekanismer styrende væv og kontekst-specifikke regulering af RNA behandling forbliver dårligt forstået. Desuden forarbejdning bidrag af RNA til forskellen i genekspression, især for ikke-kodende RNA, skal bestemmes tilbage. Tilsammen disse begrænsninger udgør væsentlige hindringer forbioinformatik kinetisk modellering af de underliggende molekylære mekanismer.
Vi har for nylig udviklet en strategi, kaldet høj opløsning genekspression profilering, for at overvinde disse problemer 5,7,9. Den er baseret på metabolisk mærkning af nyligt transskriberede RNA ved hjælp af 4-thiouridine (4SU-tagging), et naturligt forekommende uridin derivat og giver direkte adgang til nyligt transskriberede udskrifter med minimal indblanding i cellevækst og genekspression (se figur 1) 5, 10-12. Eksponering af eukaryote celler til 4SU resultater i sin hurtige optagelse, phosphorylering til 4SU-triphosphat, og inkorporering i nyligt transkriberede RNA. Efter isolering af totalt cellulært RNA, er den 4SU-mærkede RNA-fraktion thiol-specifikt biotinyleret generere en disulfidbinding mellem biotin og de nyligt transkriberede RNA. "Totalt cellulært RNA 'kan derefter kvantitativt opdeles i mærkede (" nyligt transskriberet) og umærket (' pre-existing ') RNA med høj renhed ved hjælp af streptavidin-coatede magnetiske perler. Endelig er mærkede RNA udvindes fra perlerne ved blot at tilføje et reduktionsmiddel (f.eks dithiothreitol) spaltning af disulfidbinding og frigivelse af de nyligt transkriberede RNA fra perlerne.
Nyligt transskriberet RNA skildrer transkriptionsaktivitet hvert gen under tidsramme på 4SU eksponering. 4SU-tagging i tidshorisont minutter giver således et øjebliksbillede af eukaryot genekspression og en ideel skabelon for down-stream bioinformatiske analyser (f.eks promotor analyse). I tilfælde, hvor steady-state kan antages, forholdene mellem nyligt transkriberet / total, nyligt transskriberet / umærkede og umærkede / total RNA giver non-invasiv adgang til præcise RNA halveringstider 7,13. Desuden er det vigtigt at bemærke, at nyligt transskriberede RNA oprenset efter så lidt som 5 min af 4SU-tagging (5 min 4SU-RNA) er yngre end 15 og 60 min 4SU-RNA.Når du udfører både ultrakorte og gradvist længere 4SU-tagging i et enkelt eksperimenterende indstilling kombineret med RNA-seq, er kinetik RNA behandling afsløret ved nukleotid opløsning 9.. Endelig tid-retters analyser af nyligt transskriberede og total RNA kombineret med datamodellering tillade en integrativ analyse af RNA-syntese og forfald 14..
Afslutningsvis giver denne tilgang til direkte analyse af dynamikken i RNA-syntese, forarbejdning og nedbrydning i eukaryote celler. Det gælder i alle større model organismer, herunder pattedyr, insekter (Drosophila), padder (Xenopus) og gær 5,15,16. Det er direkte kompatibel med microarray analyse 5,17, RNA-seq 9,13,14, og gælder in vivo 12,15. Her har vi detaljeret den metode til at mærke, isolere og rense nyligt transskriberede RNA i dyrkede pattedyrceller. Desuden potential problemer og faldgruber diskuteres.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1.. Metabolisk mærkning med 4-thiouridine
Lav en detaljeret plan af forsøgsopstillingen / tidsplan, fx ved at tilføje 4SU til cellekultur, og når at høste prøverne. Plan for mindst 5 minutter mellem hver tilstand. Kun behandle celler i en tilstand på et tidspunkt. Håndtag max. 3-5 retter på et givet tidspunkt. Håndter celler så hurtigt som muligt for at minimere ændringer i temperatur og CO 2 niveauer. Undgå at udsætte cellerne for lys efter 4SU tilføjes, da dette kan resultere i tværbinding af 4SU-mærkede RNA til cellulære proteiner.
Start på mærkning
- Thaw 4-thiouridine (4SU) lige inden brug og pipette nødvendige mængde 4SU for hver betingelse i en steril Falcon rør.
- Tag den nødvendige mængde cellekulturmedium (5 ml pr 10 cm skål) off retter og tilføje til 4SU-holdige Falcon rør og bland grundigt. Fjern og kassér den resterende medium fra skålene. <li> Anvend 4SU-holdigt medium tilbage til retterne.
Slutningen af mærkning
- Fjern cellekulturmedium fra celler. Tilsæt 5 ml Trizol til hver plade. For komplekse eksperimenter, herunder forskellige tidspunkter eller betingelser, dette trin gøres bedst af to personer, den ene fjerne mediet, det andet tilføjer Trizol og høst af lysatet.
- Inkuber i 5 minutter ved stuetemperatur til fuldstændig cellelysis.
- Brug en 10 ml pipette at skylle pladen forsigtigt med den ekstra Trizol. Dette hjælper fuldstændig cellelyse og prøve opsving. Håndteres med forsigtighed, da Trizol er ekstremt farligt, når det kommer i kontakt med hud eller øjne! Har modgift mod phenol forbrændinger ved hånden (f.eks Polyethylenglycol 300 eller 400 i industrielle denatureret sprit (70:30)). Overføre prøver til polypropylenrør. Bemærk venligst, at standard Falcon rør ikke modstå disse høje g styrker). Prøver kan opbevares ved -20 ° C i mindst en måned, indtil total RNA is forberedt.
2.. RNA Forberedelse Brug Modified Trizol Protocol
- Tilsæt 1 ml chloroform (0,2 ml pr ml Trizol) og omrystes kraftigt i 15 sek. Inkuber ved stuetemperatur til 2 - 3 min.
- Centrifugeres ved 13.000 x g i 15 minutter ved 4 ° C.
- Overfør vandig øvre fase (indeholdende RNA) til en ny 15 ml polypropylenrør.
- Tilsæt ½ reaktionen volumen både RNA præcipitationsbuffer og isopropanol (f.eks til 3 ml supernatant tilsættes 1,5 ml RNA præcipitationsbuffer og 1,5 ml isopropanol).
- Bland godt. Inkuber ved stuetemperatur i 10 min.
- Centrifugeres ved 13.000 x g i 10 minutter ved 4 ° C. Supernatanten kasseres.
- Centrifuger kortvarigt (5.000 × g i 30 sek), og fjerne resterende isopropanol med 200 ul pipette.
- Tilføj et tilsvarende volumen 75% ethanol og ryste røret, indtil pelleten løsnes. Undgå at bryde det op i mange små stykker, da dette kan gøre fjernelse af residual ethanol vanskelig.
- Centrifugeres ved 13.000 x g i 10 minutter ved 4 ° C. Supernatanten kasseres.
- Spin ned RNA kortvarigt og fjerne resterende ethanol med en 200 gl pipette. Gentag trin og fjerne resterende ethanol med en 20 ul pipette. Efter disse to trin, bør yderligere tørring af pelleten skal udføres.
- Tilføj 100 ul H2O 100 ug forventede RNA udbytte og bland godt ved pipettering op og ned 5 - 6 gange for at hjælpe med at opløse RNA.
- Opløses og denaturere RNA ved opvarmning til 65 ° C i 10 min (shaker) og straks placeres på is.
- Måle RNA koncentration ved 260 nm under anvendelse af en NanoDrop spektrofotometer efter producentens anvisninger. Dette RNA kan opbevares ved -80 ° C i mindst en måned.
3.. Thiol specifik Biotinylering af Newly transskriberet RNA
- Start med 60-80 ug totalt cellulært RNA.
- Udgør mærkning reaktion. Afpipetteres i det følgenderækkefølge (per ug RNA):
- 1 pi 10x Biotinylering Buffer
- 7 pi RNA (indeholdende 1 ug RNA fortyndet i nuklease-fri H2O)
- 2 pi biotin-HPDP (1 mg / ml DMF)
Altid tilføje biotin-HPDP sidste og bland straks ved pipettering. I tilfælde af biotin udfælder, kan DMF indhold øges til en endelig koncentration på 40%.
- Inkuber ved stuetemperatur i 1,5 time med rotation.
- Tilføj et lige så stort volumen chloroform. Bland kraftigt. Inkuber i 2 - 3 minutter, indtil faserne begynder at adskille og bobler begynder at forsvinde.
- Centrifugeres ved 20.000 x g i 5 minutter ved 4 ° C. Omhyggeligt overføre den øvre vandige fase ind i et nyt rør.
- Gentag trin 3.4 og 3.5 en gang. Du ønsker måske at udføre dette trin i 2 ml faselåsning Gel Heavy rør for at mindske tabet af RNA.
- RNA nedbør: add 1/10 volumen 5 M NaCl og et tilsvarende volumenisopropanol til vandfasen.
- Centrifugeres ved 20.000 x g i 20 minutter ved 4 ° C. Supernatanten kasseres.
- Tilføj et tilsvarende volumen 75% ethanol, centrifugeres ved 20.000 x g i 10 minutter ved 4 ° C, kasseres supernatanten.
- Spin kortvarigt og fjerne resterende ethanol med 200 ul pipette.
- Spin kortvarigt og fjerne resterende ethanol med 20 ul pipette.
- Lad ikke RNA at tørre. Resuspender den i 50-100 pi H2O (~ 1 ml per 1 ug indgang RNA). Bland godt ved pipettering op og ned 5 - 6 gange.
- Check RNA kvalitet ved electrophoretical analyse at udelukke RNA nedbrydning.
4.. Dot blot analyse af 4SU-inkorporering (valgfri)
4SU inkorporering kan let bestemmes ved dot blot-analyse af biotinylerede RNA. Dette er et valgfrit trin, der giver fejlfinding og estimering af 4SU iblandes i forhold til et biotinyleret DNA oligo kontrol. Til dette assay vi recommend hjælp iodacetyl-biotin i stedet for biotin-HPDP til biotinylering af 4SU-mærkede RNA i trin 3.2. Dette resulterer i en irreversibel biotinylering af 4SU-RNA. Derfor, kolonne-baserede metoder (f.eks RNeasy) kan anvendes til genindvinding af meget mindre mængder af biotinyleret RNA (fx 5 ug). Mens RNA biotinyleret anvendelse af biotin-HPDP er også velegnet til dette assay, det resulterende signal er svagere og signal-støjforhold mindre gunstig (figur 3).
- Følg protokollen for 4SU-mærkning og isolation af den samlede cellulære RNA, som beskrevet i punkt 1 og 2.
- Biotinylere 4SU-mærket RNA, som beskrevet i afsnit 3 erstatter biotin-HPDP med iodacetyl-biotin og udføre to chloroformekstraktioner til helt at fjerne store iodacetyl-biotin rester.
- Recover biotinylerede RNA ved isopropanol / ethanoludfældning som beskrevet eller ved hjælp af en søjle-tilgang (f.eks RNeasy) i tilfælde små mængder af RNA (<10 ug) Anvendes.
- Inkuber Zeta membranen i nuklease-frit vand med vuggende i 10 min.
- Tag membranen ud af nuklease-frit vand og fjerne overdrevne væsker ved at placere membranen mellem to rene papirservietter og fast tryk. Lufttørring membranen i 5 min, vil resultere i pænere prikker.
- For hver prøve, forberede 20 pi 200 ng / pi RNA ved anvendelse iskold dot blot bindingsbuffer (10 mM NaOH, 1 mM EDTA). Påfør 5 ul af denne fortynding (dvs. 1 ug RNA) samt tre efterfølgende 10-folds fortyndinger (dvs. 100, 10 og 1 ng RNA, henholdsvis) til Zeta membranen ved pipettering. Pipettering gennem en tom rack pipettespidser kan anvendes til at tilvejebringe jævnt fordelt afstand. Alternativt med dot blot apparat ifølge producentens anvisninger.
- Påfør 5 ul af biotin-mærkede DNA-oligo i koncentrationer i området fra 20 ng / pi til 20 pg / pi (dvs. 100 til 0,1 ng oligo) som en positiv control til membranen ved pipettering. Brug en biotinyleret, 4SU-naive prøve som negativ kontrol.
- Air-tørre membranen i 5 min.
- Inkuber membranen i 30 minutter i 40 ml blokerende buffer med vuggende.
- Inkuber membranen med 10 ml 1:1000 streptavidin-peberrodsperoxidase i 15 minutter (5 ml PBS + 5 ml 20% SDS + 10 pi streptavidin-peberrodsperoxidase)
- Vask membranen to gange i 40 ml PBS + 10% SDS (20 ml PBS + 20 ml 20% SDS) i 5 minutter.
- Vask membranen to gange i 40 ml PBS + 1% SDS (38 ml PBS + 2 ml 20% SDS) i 5 minutter.
- Vask membranen to gange i 40 ml PBS + 0,1% SDS (40 ml PBS + 200 pi 20% SDS) i 5 minutter.
- Fjern overdreven væske ved at placere membranen mellem to rene papirservietter og trykke på dem fast.
- Visualisere membran-bundne HRP anvendelse af ECL ifølge producentens anvisninger.
- Anbring membranen i plastfolie / pose, fjerne luftbobler og inkuberes i 2 min i mørke.
- Expose membranfilm til 1 - 5 min.
5.. Adskillelse af mærkede og umærkede RNA Brug Streptavidin-coatede Magnetic Beads
- Heat vaskebuffer (3 ml per prøve) til 65 ° C i et vandbad.
- Forbered friske 100 mM dithiothreitol (DTT) i nuklease-fri H 2 O. Gøre det ved dekantering 15-30 mg DTT pulver i en ren 50 ml Falcon-rør placeret på ultrafine skala. Afvejes og tilsættes nødvendige mængde nuklease-fri H 2 O.
- Heat biotinyleret RNA-prøver til 65 ° C i 10 minutter for at denaturere og straks sted på is.
- Place μMacs kolonner i den magnetiske stand. Vi anbefaler ikke at behandle mere end 12 prøver ad gangen (6 - 8 prøver er optimale).
- Pre-Ækvilibrer Miltenyi søjler med 1 ml stuetemperatur vaskepuffer. Det vil tage omkring 15 min.
- I mellemtiden tilsættes 100 ul streptavidinkugler til 50-100 ul biotinylerede RNA. Inkuber ved stuetemperatur i 15 minutter med rotation. < li> Hvis nogen af kolonnerne ikke har indledt tømning nu dette kan fremmes ved at trykke forsigtigt på toppen af søjlen med en behandsket finger. Når strømmen er startet kolonnerne dræne let.
- Anvend RNA / perler til søjlerne. Kassér gennemstrømning, medmindre du ønsker at gendanne det umærkede RNA fraktion (se afsnit 7).
- Der vaskes tre gange med 0,9 ml 65 ° C vaskebuffer (1 ml pipettespidser krymper ved pipettering buffere ved 65 ° C).
- Der vaskes tre gange med 0,9 ml stuetemperatur vaskepuffer.
- Pipette 700 gl Buffer RLT (RNeasy MinElute Cleanup Kit, Qiagen) i nye 2 ml rør og placere dem under søjlerne.
- Elueres nyligt transkriberede RNA ind i RLT buffer ved tilsætning af 100 pi 100 mM DTT til søjlerne.
- Udfør en anden eluering runde 3 min senere i det samme rør ved at tilføje en anden 100 ul 100 mM DTT.
6.. Inddrivelse af Newly transskriberet RNA
NDHOLDET "> Fortsæt med RNeasy MinElute Cleanup (Qiagen) protokol efter fabrikantens anvisninger. Elute i 25 ul nuklease-fri H 2 O. Measure RNA koncentrationer ved hjælp af en NanoDrop spektrofotometer. at undgå behovet for at tø og re-fryse RNA før du indsender det til en high-throughput assay, anbefaler vi forbereder cDNA umiddelbart efter nyligt transkriberede RNA oprenses. Brug 2,5 ul af de nyligt transkriberede RNA i 20 ul cDNA-syntese mix for cDNA-syntese efter fabrikantens anvisninger. Udfør QRT-PCR-kontroller med 1 : 10 fortyndinger af cDNA mix Store RNA ved -80 ° C..7.. Inddrivelse af Umærket, Ubundet RNA (valgfri)
I tilfælde af ubundet RNA skal inddrives, indsamle og kombinere gennemstrømning (efter tilsætning af RNA-streptavidinkugler løsning på kolonner) og den første vask til efterfølgende udfældning. Normalt er det tilstrækkeligt at udfælde kun 50% af ubundet RNA som This vil indeholde> 80% af udgangsmaterialet.
- Tilføj en tilsvarende mængde isopropanol (ingen salt skal tilføjes som vaskebuffer allerede indeholder 1 M NaCl).
- Centrifugeres ved 20.000 x g i 20 minutter ved 4 ° C. Supernatanten kasseres.
- Tilføj et tilsvarende volumen 75% ethanol, centrifugeres ved 20.000 x g i 10 minutter ved 4 ° C, kasseres supernatanten.
- Spin kortvarigt og fjerne resterende ethanol med 200 ul pipette.
- Spin kortvarigt og fjerne resterende ethanol med 20 ul pipette.
- Lad ikke RNA at tørre. Opblande det i 100 pi H2O Bland godt ved pipettering op og ned 5 - 6 gange. Inkuber ved 65 ° C i 10 min under omrystning og overføres direkte til is.
- Check RNA kvalitet ved electrophoretical analyse at udelukke RNA nedbrydning.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
1.. Udgangsmateriale, og Forventede Udbytter
Efter 1 time (hr) i 4SU eksposition nyligt transkriberet RNA repræsenterer ca 1-4% af den samlede cellulære RNA. Dette vil være lavere i vækststandsede celler som de ikke længere syntetisere RNA højde for cellevækst / replikation. Ved mærkning i 1 time, anbefaler vi analysen startes med 60-80 ug af total RNA. Startende med mindre end 30 ug total RNA resulterer i små RNA pellets, der er svært at se efter biotinylation trin og kan således let tabt. Input RNA niveauer kan øges til så meget som 150 ug til meget korte varigheder af mærkning (f.eks 5 - 10 min). Når varigheden af RNA mærkning afkortes fra 1 time til 5 min bidrag kortlivede intronsekvenser i nyligt transkriberet RNA stiger fra -60% til -80% 9. Som introner væsentligt længere sammenlignet med kodende sekvenser samt 5'-og 3'-UTR'er, mængden af nyligt transkriberetRNA, som kan renses efter kort eller endda ultrakorte 4SU-tagging, ikke falder lineært. Som sådan opnåede vi> 0,5% af total RNA efter 5 min af 4SU-tagging i ikke-klæbende humane B-cellelinjer 9. Det skal dog bemærkes, at højere koncentration af 4SU og lidt længere varighed af mærkning kan være påkrævet for at opnå tilsvarende 4SU iblandes i vedhæftende celler. Mens selv et lavt 4SU-iblandingen vil give en effektiv opsamling og rensning af store, Uridin-rige udskrifter, meget kort udskrift med lavt uridin indhold (f.eks miRNA) er tilbøjelige til at undslippe rensning selv når du bruger høje 4SU koncentrationer (> 1 mm). I NIH-3T3 musefibroblaster, mærket 1 hr 200 uM 4SU eksponering nyligt transkriberede RNA med omkring en 4SU rest pr 50-100 nukleotider (nt) 5. Dette skulle give særdeles effektiv inddrivelse af udskrifter> 500 - 1000 nt i længden. Derfor har vi kun observeret en mindre udskrift størrelseskævhed når mærkningen i 1 time med 200 uM 4SU i både murine fibroblaster og humane B-celler 7. Mens 1 time på 200 pM 4SU ikke resulterede i nogen væsentlige ændringer i cellulære udskrifter niveauer i murine fibroblaster, langvarig eksponering af celler til ≥ 200 uM 4SU medfører en målbar vækst underskud inden 24 timer (upublicerede data). Derfor bør både varigheden af mærkning og den beskæftigede 4SU koncentration minimeres for at undgå ektopisk eller toksiske virkninger. En nem måde at bestemme den minimale 4SU-koncentration, der kræves for effektiv inddrivelse af nyligt transskriberede RNA er at rense nyligt transskriberede RNA efter 4SU-mærkning med stigende koncentrationer af 4SU (f.eks 50-1600 uM). Som vist i figur 2A og 2B, genvinding af nyligt transskriberede RNA mærket i 1 time i primære humane fibroblaster steg betydeligt fra 50 til 200 pM 4SU men derefter begyndte at plateau.
2.. PunktumSkamplet Kvantificering af 4SU Indbygning (valgfrit)
I nogle tilfælde kan det være af interesse at måle mængden af 4SU inkorporering i totale RNA. Dette gøres bedst ved dot blot analyse på de biotinylerede RNA ved hjælp af en streptavidin konjugat. På grund af sin kemiske natur iodacetyl-biotin er mere reaktiv over for thiol-grupper end biotin-HPDP resulterer i biotinylering af næsten alle 4SU rester i nyligt transkriberet RNA. Det er vigtigt at bemærke, at ligesom biotin-HDPD, iodacetyl-biotin er ikke vandopløselige og således effektivt fjernes ved ekstraktion med chloroform som udført for biotin-HPDP. Derfor kan identiske reaktionsbetingelser og koncentrationer anvendes som når du bruger biotin-HPDP. Imidlertid iodacetyl-biotin er ikke reversibel. Det kan således ikke bruges til rensning af nyligt transskriberede RNA i kolonne tilgange. Mens brugen af iodacetyl-biotin gør det muligt at kvantificere 4SU-inkorporering, biotin-HPDP målinger overveje både4SU-stiftelse og biotinylation effektivitet. Aktiveringen af to biotinyleringsbetingelserne reagenser til den samme prøve giver mulighed for måling af biotinylering effektivitet af RNA-inkorporerede 4SU. Biotinylering effektivitet af biotin-HPDP til 4SU-mærkede RNA synes at være cirka tre gange mindre end for iodacetyl-biotin indikerer, at kun omkring en ud af tre 4SU rester i nyligt transkriberet RNA faktisk biotinyleres af biotin-HPDP (figur 3). Ved at sammenligne prøven signalintensiteter med den biotinylerede kontrol-DNA oligo kan biotinyleringsbetingelserne tætheder måles. For de fleste mammaliacellelinier et positivt signal, bør stadig være påviselig i 10 ng af biotinylerede RNA efter 1 time på 200 pM 4SU mærkning. En svag baggrund signal er normalt påvises for den højeste koncentration (1 ug) af umærkede RNA.
3.. Oprensning af Nyligt transskriberet RNA
Inddrivelse af nyligt transskriberede RNA er meget mængdertive. Hvis du startede med det samme RNA-koncentrationen, du kan forvente at opnå de samme mængder af nyligt transskriberede RNA for alle prøver. Ligesom mange kolonne-baserede assays, kan samling af nyligt transkriberede RNA ved anvendelse RNeasy MinElute kit resultere i yderligere absorption ved 230-260 nm (tilstedeværelse af detergenter afledt fra vask buffere), som kan interferere med OD260 målinger. Dette ses i mindre omfang, når den anvender et nyt 2 ml opsamlingsrør for hver centrifugation skridt. Ikke desto mindre bør eventuelle urimeligt høje OD målinger (> 2 gange større end andre prøver) overvejes med omhu, især hvis OD 260/280 nøgletal er <1.7. For down-stream-analyser er det således ofte bedst at bruge den samme mængde template RNA volumen for alle prøver. I tilfælde, hvor udbyttet af mærket RNA er lavere end forventet kontrollere for tegn på RNA nedbrydning af electrophoretical analyse. Nyligt transskriberet RNA indeholder væsentligt større mængder af store, ikke-splejsede transkripter med de typiske rRNA bands er langt mindre fremtrædende (figur 4).
4.. Kvantificering af Newly transskriberet RNA
Endelig kan iblandes af 4SU i nyligt transskriberede RNA direkte kvantificeres ved spektrofotometrisk analyse baseret på absorptionen maksimalt 4SU ved 330 nm og OD 330/260 forholdet 5,18. Dette kræver> 3 ug af mærkede RNA koncentreret i et lille volumen (10 - 20 ul) af isopropanol / ethanol udfældning. For at undgå at miste den lille RNA pellet udfældes sammen med 30 ug nukleasefrit glykogen (Fermentas, # R0551) bør udføres. En yderligere top er synlig ved 330 nm afspejler iblandingen af 4SU i nyligt transkriberet RNA (figur 5).
/ Files/ftp_upload/50195/50195fig1highres.jpg "/>
Figur 1. . Princippet om metabolisk mærkning med 4-thiouridine (4SU) 4SU tilsættes til celler for den krævede (5 - 120 min) tid efterfulgt af udarbejdelse af det samlede cellulære RNA. Efter thiol-specifik biotinylering er totalt cellulært RNA separeret i 4SU-mærkede, nyligt transkriberede RNA og umærkede, præ-eksisterende RNA anvendelse af streptavidin-coatede magnetiske perler. Nyligt transskriberet RNA udvindes fra perlerne ved hjælp af et reduktionsmiddel, som spalter disulfidbindingerne, der forbinder de nyligt transskriberede RNA til perlerne. Klik her for at se større figur .
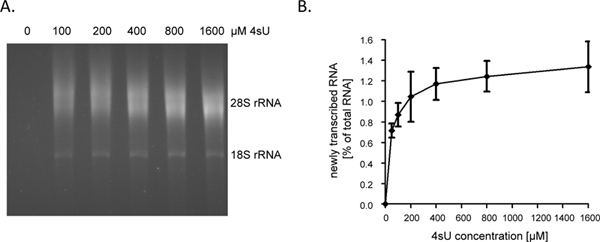
Figur 2. Inddrivelse af nyligt transskriberede RNA efter stigende koncentrationer af 4SU. (A) Primære humane forhudsfibroblaster (HFF), blev inkuberet med 100, 200, 400, 800 eller 1.600 uM 4SU. Nyligt transkriberede RNA blev oprenset fra 50 ug totalt cellulært RNA og underkastet electrophoretical analyse. Som forventet var en koncentrationsafhængig stigning i genvundne nyligt transskriberet RNA observeret som begyndte at plateau ved højere koncentrationer. (B) Mængden af renset nyligt transskriberede RNA blev kvantificeret ved hjælp af ImageJ 1.45s software. Kombinerede data fra fire uafhængige eksperimenter på beløbene for nyligt transskriberede RNA inddrives som følge af forskellige koncentrationer af 4SU mærkning spænder fra enten 50-800 uM 4SU (n = 2) eller 100 -. 1.600 uM 4SU (n = 2) er vist Klik her for at se større figur .
upload/50195/50195fig3.jpg "alt =" Figur 3 "fo: content-width =" 4.5in "fo: src =" / files/ftp_upload/50195/50195fig3highres.jpg "/>
Figur 3. Estimering af 4SU inkorporering i 4SU mærkede total RNA ved hjælp af dot blot analyse. Totalt RNA blev isoleret fra NIH-3T3 musefibroblaster eller humane forhudsfibroblaster (HFF) inkuberet med 200 pM 4SU i en time. Ingen 4SU blev tilføjet til en skål som negativ kontrol. For HFF både kontakt hæmmede (n = ikke-voksende celler) og voksende celler (y) blev inkluderet. RNA blev isoleret under anvendelse af Trizol-reagens og efterfølgende konjugeret til biotin-HPDP eller iodacetyl-biotin. Koncentrationen af hver prøve blev justeret til 200 ng / pi og 5 pi af denne fortynding (dvs. 1 ug RNA), samt tre efterfølgende 10-folds fortyndinger (dvs. 100, 10 og 1 ng RNA, henholdsvis), blev alle spottet på et stykke Zeta membran. 5 gl fortyndinger af biotin-mærkede DNA oligo blev placeret på membranen som positive kontroller i koncentreioner fra 20 ng / ul ned til 20 pg / pi (dvs. 100 til 0,1 ng, henholdsvis). Biotin densitet blev probet under anvendelse af en streptavidin-peberrodsperoxidase-konjugat.
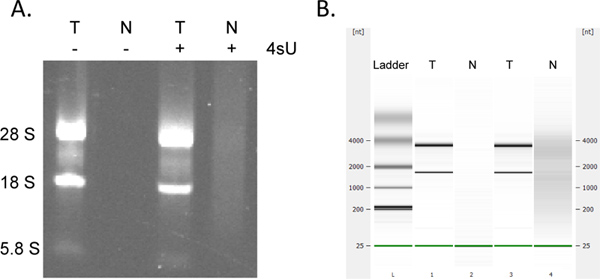
Figur 4.. Electrophoretical analyse af nyligt transkriberet og totalt RNA. Totalt RNA (T) og nyligt transkriberet RNA (N) fremstillet ud fra murine NIH-3T3 fibroblaster dyrket både i nærvær og fravær af 500 uM 4SU i 1 time blev analyseret ved agarosegelelektroforese (A) og (i samme rækkefølge) ved hjælp af Agilent Bioanalyser (B). Ingen RNA blev udvundet uden 4SU behandling af celler. Renset nyligt transskriberet RNA indeholder større mængder af højmolekylære mRNA og betydeligt mindre modne rRNA'er end totalRNA bemærkelsesværdig mellem 28S, 18S og 5.8S rRNA bands. Klik her for at se større figur .
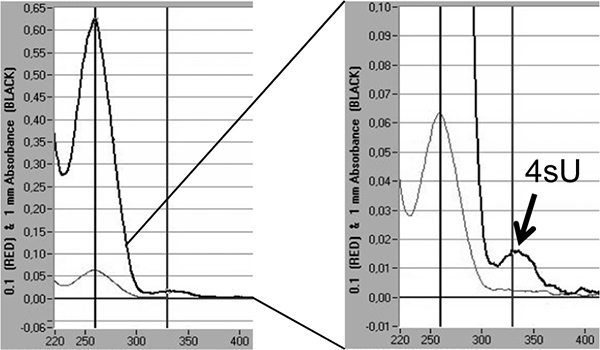
Figur 5. Kvantificering af 4SU inkorporering i nyligt transkriberede RNA ved spektrofotometrisk analyse. Newly transskriberede RNA oprenset fra 2 x 100 ug totalt RNA efter 1 time på 200 uM 4SU i murine NIH-3T3 fibroblaster. Nyligt transkriberede RNA blev udfældet med isopropanol / ethanol efter tilsætning 30 ug nuklease-fri glykogen. Spektrofotometrisk analyse af nyligt transkriberede RNA opnået ved en NanoDrop 1000 spektrofotometer vises. De lysegrå linier repræsenterer målinger ved 0,1 mm, mens de tykkere, mørkegrå linier repræsenterer målinger ved 1 mm væskesøjle. Til højre forstørrelse en af toppen af udryddelse repræsenterer tHan indarbejdet 4SU-rester vises. Baseret på udslettelse co-effektive af 4SU 18 inkorporeringen satser 4SU kan estimeres.
| Varigheden af mærkning [min] | Anbefalet 4SU koncentration [pM] |
| 120 | 100-200 |
| 60 | 200-500 |
| 15-30 | 500 - 1000 |
| <10 | 500 - 2000 |
Tabel 1. Anbefalet 4SU koncentrationer.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Metabolisk mærkning af nyligt transskriberede RNA væsentlig styrkelse af den magt high-throughput teknologier som microarrays og RNA-seq ved at stille mere velegnede skabeloner til at tage fat på biologiske spørgsmål af interesse. Den nuværende protokol gennemgik en omfattende optimering. Det tillader> 1000 fold berigelse af nyligt transkriberede RNA og giver meget reproducerbare resultater.
Det eksperimentelle design af et 4SU-tagging forsøget er af afgørende betydning, da nyligt transskriberede RNA vil skildre realtid transkriptionsaktivitet kun i den tid af eksponering af celler til 4SU. Hvis de faktiske ændringer i transskription satser efter en stimulus allerede har lagt sig, vil disse blive savnet, når man analyserer nyligt transskriberede RNA selvom ændringerne i totale RNA-niveauer kan stadig være søgbar. Derfor er en god forståelse af de underliggende biologi er vigtigt at definere forsøgsopstillingen samt den optimale perioder of tid til 4SU eksponering. Nedenfor giver vi anbefalinger og måder til at undgå almindelige faldgruber for de mest afgørende skridt.
Udarbejdelse af stamopløsninger og plast ware
Alle stamopløsninger skal fremstilles ved nuklease-frit vand. Ved hjælp af internt renset deioniseret vand kan resultere i problemer, hvis vandet indeholder reduktionsmidler. I et tilfælde resulterede dette i et fuldstændigt tab af alle mærkede RNA. Derfor anbefaler vi at købe pre-made nukleasefrit NaCl, Tris-Cl, EDTA, natriumcitrat og vand. Sikre nuklease-frie betingelser på alle tidspunkter. Dimethylformamid (DMF) opløser nogle plastmaterialer. Vi fandt, at anvendelse af 25 ml cellekultur plastpipetter at overføre DMF fra sit lager glasflaske til 50 ml Falcon-rør til fremstilling af biotin-HPDP stamopløsning var tilstrækkelig til væsentligt at reducere udbyttet af nyligt transkriberede RNA fra hele prøveperioden. Interessant, dette ikke negativ indvirkning på biotinylation effektivitet (som testet ved dot blot) men resulterede i en 75 til> 90% tab af nyligt transskriberede RNA, der kunne inddrives fra perlerne. Tabet var mest udtalt, når varigheden af mærkning blev reduceres fra 60 til 30 min eller mindre. Mest sandsynligt, en substans elueret fra plastpipetter af DMF delvist ødelagt belægningen på streptavidinkugler. Derfor bør anvendelsen af plastmaterialer ikke er kendt for at være forenelig med DMF undgås med alle midler. Af samme grunde bør celle skrabere ikke bruges til at fremme genanvendelsen af Trizol prøver fra cellekultur plader. Det er interessant at bemærke, at de formodede stoffer, der elueres fra plast med DMF eller Trizol tilsyneladende hverken blev fjernet ved ekstraktion med chloroform eller isopropanol / ethanol udfældning.
Cellekultur
Celletæthed på pladerne er af afgørende betydning. I et eksperiment, hvor cellerne viste sig at være lidt for sammenflydende (90 -100%), behandlede vi NIH-3T3 musefibroblaster i 30 minutter med 100 U / ml interferon (IFN) α eller γ. På mindre sammenflydende celler endda 15 min af IFN behandling allerede resulteret i en 5 - til 8-fold induktion af gener som irf1 eller socs3 5.. Med celler bliver lidt for sammenflydende microarray analyse viste ingen induktion af IFN-inducerbare gener for selv de mest hurtigt inducerbare gener som irf1 eller socs3. Derfor celledensiteten er en afgørende faktor for 4SU-mærkning eksperimenter og alle cellekultur plader bør undersøges nøje, inden du starter mærkning.
4SU er en fotoaktiverbar ribonukleosid og 4SU-holdige RNA effektivt krydsbundet til proteiner efter udsættelse for 365 nm lyskilde. 4SU-behandlede celler bør dyrkes i mørke og udsættelse for lys bør undgås. Efter fjernelse af cellulære proteiner ved Trizol RNA-isolering denne risiko reduceres væsentligt.
<p class = "jove_content"> 4SU er ikke indarbejdet i cellulært DNA. Det skal dog bemærkes, at total RNA stadig vil indeholde små mængder af cellulært DNA. Når du bruger 4SU-tagging og q-RT-PCR-analyse til at studere viral genekspression i cytomegalovirusinfektion fandt vi det nødvendigt at medtage en DNaseI digest skridt i protokollen til at fjerne de concatemeric virusgenomer 19.. Dette er sandsynligvis ikke nødvendig ved brug downstream protokoller, som ikke er følsom over for tilstedeværelsen af DNA.4SU iblandes og optimal 4SU koncentration
4SU let taget op af celler med intra-og extra-cellulær niveauer sandsynligvis equilibrating inden for mindre end et minut 9,16. Optagelses-og indarbejdelse satser 4SU er koncentrationsafhængig. Derfor kan 4SU koncentration bekvemt justeres efter den anvendte varighed af mærkning. Tabel 1 giver råd om 4SU koncentrationer i relation til varigheden af mærkning baseret på vores bedste personlig erfaring. I 1 time af 4SU mærkning i pattedyrceller, vil 200 pM 4SU være tilstrækkelig til de fleste anvendelser resulterer i omkring en 4SU rest pr 50 til 100 nukleotider i nyligt transkriberede RNA i fibroblaster.
I de sidste par år har vi anvendt 4SU-tagging til en bred vifte af celletyper af human og murin oprindelse, herunder fibroblaster, endotelceller, epitelceller, knoglemarvsstroma celler, makrofager og T-celler. Desuden blev celler fra Drosophila og Xenopus held brugt. I alle disse eksperimenter blev 4SU inkorporering sig at være yderst effektiv kræver kun minimale justeringer i 4SU koncentration for de forskellige celletyper. Ved opsætning af metoden til nye celletyper, vil vi anbefale at mærke celler med stigende 4SU-koncentrationer (fx spænder 50-1600 uM) og analysere forholdet af renset nyligt transskriberede RNA til de anvendte 4SU-koncentrationer (se figur 2A / B). Den 4SU-koncentration, ved hvilken mængden af renset nyligt transskriberet RNA ind i en plateau bør vælges.
I tilfælde, hvor meget sammenflydende, kontakt hæmmede celler anvendes, vil vi anbefale at bruge lidt højere 4SU koncentrationer (f.eks 500 i stedet for 200 uM) for at sikre en effektiv 4SU inkorporering. Derudover er der i tilfælde, hvor erobringen af meget korte nyligt transskriberede udskrifter (<200 nt) er af særlig interesse, kan 4SU koncentration skal også øges. Dette bør ikke kombineres med forlængede mærkning gange (f.eks> 1 time) for at undgå ektopiske effekter eller toksicitet. Endelig fandt vi at bruge for lille et volumen celledyrkningsmedier kan reducere 4SU inkorporering effektivitet. Vi anbefaler derfor at anvende 5 ml eller 10 ml medium pr 10 cm eller 15 cm parabol, hhv.
Fremstilling af totalt cellulært RNA
For succes i denne protokol er det afgørende at få rene, RNase-fri samlede cellulære RNA. Brug 5 ml Trizol per 15 cm parabol producerer rene RNA fri for nucleaser. Vi anbefaler at bruge den modificerede Trizol protokollen af Chomczynski et al. 20.. Først er det bedre egnet til at isolere store mængder af RNA (> 100 ug) som de forbedrede centrifugalkraften resulterer i fastere pellets, som er lettere at håndtere under vasketrinene. Men det kræver anvendelse af særlige polypropylen rør og adaptere, da de regelmæssige 15 ml laboratorium Falcon rør ikke overlever mere end 6.000 × g. Andet, det forbedrer fjernelse af DNA og glycoproteiner. Dette bliver især tydeligt, når de udarbejder RNA fra organer og væv. For det tredje betyder det ikke begrænse den maksimale samlede mængde af RNA, som kan isoleres. Selvom vi fandt også kolonne-baserede RNA isolation metoder (f.eks RNeasy) for at give RNA af passende kvalitet, standard kolonner are kun i stand til at fange op til 100 ug af total RNA og derved begrænser mængden af udgangsmateriale. Endelig er ved at fjerne den resterende ethanol to gange med en pipette, tørring af RNA til fjernelse af resterende ethanol ikke længere er nødvendig. Dette eliminerer risikoen for over-tørring RNA, som kan være vanskeligt at opløse igen bagefter. I princippet er 4SU-tagging anvendelse in vivo, fx ved intravenøs injektion af mus. Vi konstaterede imidlertid, at RNA renhed udgør et stort problem, der kræver rensning af polyA transkripter før oprensning af nyligt transkriberet RNA (upublicerede data).
Biotinylering og fjernelse af ubundet biotin
Biotin-HPDP er 100% thiol-specifik og danner en disulfidbinding mellem biotin rester og thiol-mærkede RNA nyligt transskriberede molekyler. Biotinylering effektivitet 4SU-mærkede RNA er omkring 30% som bestemt ved dot blot-analyse 5.. Såsom biotin-HPDP ikke er vandopløseligtdet kan fjernes effektivt ved ekstraktion med chloroform. Mens en enkelt chloroformekstraktion trin er tilstrækkelig til at fjerne størstedelen af ubundet biotin vi jævnligt gentage dette trin for at sikre fuldstændig fjernelse. For at reducere RNA tab i chloroformekstraktion trin 2 ml faselåsning Gel Heavy rør (Eppendorf) kan anvendes efter fabrikantens anvisninger. Normalt bruger vi faselåsning rørene kun for det andet chloroformekstraktion trin som skabelon mængder af det første trin er ofte alt for høj til at være direkte kompatible med disse rør. Efter fjernelse af ubundet biotin-HPDP, RNA udvindes ved isopropanol / ethanol udfældning. Det er vigtigt at bemærke, at kommercielle kolonne-baserede kits til at inddrive det biotinylerede RNA (f.eks RNeasy fra QIAGEN) bør ikke anvendes, da de indeholder reduktionsmiddel i de leverede buffere, som spalter disulfidbindingen og fjern biotin fra det nyligt transkriberede RNA .
Oprensning af nyely transskriberede RNA
Må ikke tilføje mere end 100 ul biotinylerede RNA til 100 gl streptavidinkugler. Tilføjelse mindre volumen foretrækkes. Dog bør den samme mængde RNA blive tilføjet til alle prøver. Juster RNA input volumen (mellem prøverne), som du føjer til de streptavidinkugler ved blot at tilføje den nødvendige mængde 1x TE til perlerne. En nem måde at lave frisk nukleasefrit 100 mM DTT er at dekantere en tilstrækkelig mængde af DTT pulver i en falk rør, der anbringes på en ultra-følsomme skala og derefter tilsættes den nødvendige mængde nuklease-fri H2O at generere 100 mM DTT (64.8 pi vand per 1 mg DTT).
Under udviklingen af 4SU-tagging testede vi streptavidinkugler fra forskellige leverandører. En række af dem genererede store mængder baggrund. Derfor anbefaler vi kraftigt at bruge Miltenyi streptavidinkugler som hidtil, har vi aldrig oplevet nogen problemer med fremførsel af umærkede RNA fra væv cULTUR-afledte RNA-prøver. På denne måde, kan så lidt som 150 ng af mærkede RNA specifikt oprenset fra 150 ug biotinyleret RNA (i 100 ul vand) under anvendelse af 100 ul streptavidinkugler. Ligevægtsindstilling af perlerne med ækvilibreringspuffer leveres med perlerne kan udføres, og kan let forbedre opsamlingsprocent 13..
Kvalitetskontrol
Vi anbefaler at udføre Q-RT-PCR-kontroller på nyligt transskriberede RNA før udsætte det for high-throughput analyser. Dette kan omfatte kvantificering af flere referencepunkter gener vides at være differentielt reguleret i givet eksperimentelle indstilling. I tilfælde, hvor 4SU-tagging er ansat til at studere RNA henfaldshastigheder, vil vi anbefale at kvantificere en kortvarig udskrift (f.eks myc, fos) og en langlivet én (f.eks GAPDH) i både alt og nyligt transkriberet RNA. Forholdet af nyligt transkriberet / totalt RNA bør være betydeligt højere (~ 5 - til 10-fold)for kortlivede udskrifter. Baseret på RNA-halveringstiden for en reference-gen, kan RNA halveringstider bestemmes. Hvis alle tre RNA fraktioner (total RNA, nyligt transkriberede RNA og umærket præeksisterende RNA) analyseres for fire eller flere gener, normalisering af de forskellige RNA-delmængder kan udføres ved lineær regressionsanalyse og kvalitetskontrol scoringer kan bestemmes som beskrevet 7 , 21..
Til Q-RT-PCR-analyse anbefaler vi at bruge 2,5 pi mærkede RNA i 20 ul cDNA syntese mix. For optimal sammenligning af q-RT-PCR resultater fastfryse cDNA i portioner af 5 pi før første brug. Tø rør lige før brug, tilsættes 45 ul H 2 O og emne 5 ul af fortyndingerne til Q-RT-PCR-analyser. Dette forbedrer sammenligneligheden mellem forskellige PCR kørsler.
Nyligt transskriberede RNA-prøver skal kontrolleres for tegn på RNA nedbrydning ved hjælp af Agilent Bioanalyser før udsætte dem forhigh-throughput analyse (microarrays eller RNA-seq). Det skal dog bemærkes, at yderligere bands undertiden observeret af Agilent Bioanalyser. Den biologiske betydning af dette er uklart. Som nyligt transskriberet RNA indeholder betydeligt mindre ribosomale RNA, disse prøver lejlighedsvis mislykkes Agilent Bioanalyser kvalitetskontrol. Hvis dette ikke skyldes synlige RNA nedbrydning prøver af acceptabel kvalitet er normalt fint at blive udsat for high-throughput analyse.
Kompatibilitet af nyligt transskriberede RNA med down-stream analyser
Nyligt transskriberet RNA indeholder væsentligt mere mRNA end total RNA. Dette skyldes primært de større mængder af intronsekvenser i nyligt transskriberede RNA, som stiger, når varigheden af 4SU tagging forkortes. Derfor mener vi ikke regelmæssigt foretage udtømning af rRNAs fra nyligt transskriberede RNA-prøver, da dette kræver større mængder af udgangsmateriale, mens providing temmelig lidt (~ dobbelt) gevinst i ikke-rRNA læser. Endelig skal det bemærkes, at den største procentdel af splejsede højmolekylære transkripter til stede i nyligt transkriberede RNA kan kræve yderligere fragmentering ved udarbejdelsen cDNA-biblioteker for næste generation sekventering. Resultaterne af størrelsen fragmentering skridt bør derfor være kvalitet kontrolleres omhyggeligt.
Data normalisering til RNA halv levende målinger
Standarden tilgang til at normalisere forsøgsdata for RNA halveringstiden målinger er at normalisere alle data til RNA halveringstid et godt karakteriseret hus-føring genet eller median RNA halveringstid i en given celletype bestemt i tidligere forsøg. I pattedyrceller, ligger sidstnævnte i området fra 5 til 10 timer 6,7. Mens denne tilgang virker også ganske pænt for 4SU-baserede målinger, er andre midler til normalisering nødvendig, hvis median RNA halveringstiden ikke er kendt, eller hvis det may selv blive påvirket af ændringer i det cellulære system under undersøgelse, fx ved knock-out af et RNA henfald vej. 4SU-tagging byder på en unik måde at estimere den mediane RNA halveringstid baseret på en analyse af alle tre RNA fraktioner, nemlig samlet cellulært RNA, nyligt transskriberede RNA og umærkede allerede eksisterende RNA. Som total cellulært RNA adskilles i de to sidstnævnte RNA fraktioner en simpel lineær regressionsmodel kan anvendes til at normalisere de tre RNA fraktionerne til hinanden og bestemme median RNA halveringstiden 7,16. En software pakke er tilgængelig online for at udføre disse analyser 22..
Ineffektiv indfangning af udskrifter med lavt uridin indhold kan påvirke RNA halveringstid målinger resulterer i kunstigt lave nyligt transskriberede / total RNA nøgletal og forlængede RNA halveringstider. Omfanget af dette problem kan vurderes ved at plotte RNA halveringstider eller log (nyligt transkriberet / total RNA-forhold) mod uridine indholdet af alle udskrifter 7,15. Dette giver også en god kvalitetskontrol for at vurdere forskellene i 4SU-iblandes mellem forskellige prøver eller betingelser. I tilfælde, hvor en betydelig korrelation til uridin indhold observeret det kan korrigeres for ved bioinformatik betyder 15.. Dog skal det bemærkes, at bidraget fra modne transkripter i nyligt transkriberet RNA ikke let kan skelnes fra den meget større og dermed langt mere uridin-rige forstadier. Medmindre behandlingen kinetik en given transkript er kendte (som de normalt ikke) bare korrigere for lav uridin indhold (ineffektiv capture) kan forvrænger RNA halveringstider. Som sådan har vi for nylig fundet behandling af de fleste menneskelige snoRNAs at være meget ineffektiv 9.. Hvis vi havde korrigeret de nyligt transskriberede / total RNA-forhold til den lave uridin indholdet af temmelig små (70-300 nt) snoRNAs, ville det have resulteret i en ekstremt kort snoRNA halv-lives (<5 min) med talrige nyligt transskriberede / totalt RNA forhold over 100%. Derfor er vi generelt ikke anbefale korrigere for lavt uridin indhold, når måling RNA halveringstider.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne erklærer, at de ikke har nogen konkurrerende finansielle interesser.
Acknowledgments
Vi vil gerne takke Amie Regan for omhyggelig læsning af manuskriptet. Dette arbejde blev støttet af NGFN Plus tilskud # 01GS0801, MRC stipendium G1002523 og NHSBT tilskud WP11-05 til LD og DFG tilskud FR2938/1-1 til CCF
Materials
| Name | Company | Catalog Number | Comments |
| 4-thiouridine | Carbosynth | T4509 | Prepare 50 mM stock in sterile H2O, store at -20 °C in aliquots of 50-500 μl, discard unused reagent, do not refreeze. |
| Trizol | Invitrogen | 15596026 (100 ml), 15596018 (200 ml) | WARNING - CORROSIVE and HAZARDOUS TO HEALTH! Ensure immediate access to Phenol antidote (PEG-Methanol); Store at 4 °C. |
| Chloroform | Sigma | 372978 | WARNING - HAZARDOUS TO HEALTH |
| Isopropanol | Sigma | 650447 | |
| Sodium citrate, nuclease-free | Sigma | C8532 | Prepare 1.6 M stock solution using nuclease-free water. |
| 5M nuclease-free NaCl | Sigma | 71386 | Stock solution |
| Nuclease-free H2O | Sigma | W4502 | Make 1 ml aliquots in nuclease-free tubes. |
| RNA precipitation buffer | 1.2 M NaCl, 0.8 M sodium citrate in nuclease free water. Prepare in advance under strictly nuclease-free conditions. Store at room temperature in 50 ml falcon tubes. | ||
| Ethanol | Sigma | 459844 | Use with nuclease-free water to prepare 80% ethanol, store at -20 °C. |
| 1 M nuclease-free Tris Cl, pH 7.5 | Lonza | 51237 | Stock solution |
| 500 mM nuclease-free EDTA, pH 8.0 | Invitrogen | 15575-020 | Stock solution |
| 10x Biotinylation Buffer (BB) | 100 mM Tris pH 7.4, 10 mM EDTA in nuclease-free water, make aliquots of 1 ml. | ||
| Dimethylformamide (DMF) | Sigma | D4551 | |
| EZ-Link biotin-HPDP | Pierce | 21341 | Prepare 1 mg/ml stock solution by dissolving 50 mg biotin-HPDP in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. |
| Phase Lock Gel Heavy tubes 2.0 ml | Eppendorf | 0032 005.152 | Optional for the chloroform extraction step. |
| Zeta membrane | BIORAD | 162-0153 | |
| 10x Dot blot binding buffer | 100 mM NaOH, 10 mM EDTA | ||
| Biotin-oligo | 5'-biotin, 25 nucleotides, any sequence | ||
| Sodium dodecyl sulphate | Fisher | BPE9738 | For 100 ml 20% stock solution, add 20 g SDS to 80 ml PBS pH 7-8 and adjust volume to 100 ml. Keep all high-percentage SDS solutions above 20 °C. Warm the solutions slightly should SDS precipitate. |
| EZ-Link Iodoacetyl-LC-Biotin | Pierce | 21333 | Prepare 1 mg/ml stock solution by dissolving 50 mg iodoacetyl-biotin in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. Generates irreversible, thiol-specific biotinylation. |
| Phosphate buffer saline | Gibco | 10010-015 | |
| Dot blot blocking buffer | Mix 20 ml 20% SDS with 20 ml 1 x PBS pH 7-8 and add EDTA to the final concentration of 1 mM. | ||
| Streptavidin-horseradish peroxidase | Vector Laboratories | SA5004 | Store at -20 °C. Mix 10 ml 20% SDS with 10 ml 1 x PBS. Add 20 μl Streptavidin-HRP before use. |
| ECL reagent | GE Healthcare | RNP2109 | Use following the manufacturer's instructions. |
| Super RX, X-RA Film, 18x24 cm | Fujifilm | 47410 19236 | |
| μMacs Streptavidin Kit | Miltenyi | 130-074-101 | Store the beads at 4 °C. |
| Tween 20 | Sigma | P1379 | |
| Washing buffer | 100 mM Tris pH 7.4, 10 mM EDTA, 1 M NaCl, 0.1% Tween 20 in nuclease-free H2O. | ||
| Dithiothreitol (DTT) | Sigma | 43817 | Prepare as 100 mM DTT in nuclease-free H2O, always prepare fresh before use. |
| RNeasy MinElute Kit | Qiagen | 74204 | Store columns at 4 °C, remaining components of the kit at room temperature. |
| 1.5 ml screw-top polypropylene tubes | Sarstedt | 72.692.005 | Compatible with Dimethylformamide |
| 2.0 ml screw-top polypropylene tubes | Sarstedt | 72.694.005 | Compatible with Dimethylformamide |
| 15 ml tubes | BD Falcon | 352096 | Compatible with Dimethylformamide |
| 50 ml tubes | BD Falcon | 352070 | Compatible with Dimethylformamide |
| All solutions/reagents should be stored at room temperature unless otherwise specified. | |||
| Equipment | |||
| UV/VIS spectrophotometer | Thermo Scientific | NanoDrop 1000 | Or equivalent. Use low volume (1-2 μl) for measurements of low RNA concentrations to avoid excessive sample loss. |
| Polypropylene 15 ml centrifuge tubes | VWR International | 525-0153 | In contrast to standard 15 ml tubes, these tolerate up to 15,000 × g |
| High-speed centrifuge | Beckman Coulter | Avanti J-25 | Or equivalent equipment capable of reaching 13,000×g |
| High-speed rotor | Beckman Coulter | JLA-16250 | Or equivalent equipment capable of reaching 13,000×g |
| Adaptors for 15 ml tubes | Laborgeräte Beranek | 356964 | Or equivalent equipment capable of reaching 13,000×g |
| Refrigerated table-top centrifuge | Eppendorf | 5430 R | Or equivalent. |
| Thermomixer | Eppendorf | Thermomixer compact | Or equivalent. |
| Magnetic stand | Miltenyi Biotec | 130-042-109 | One stand holds 8 μMacs columns. |
| Waterbath | Grant | SUB Aqua 5 | Or equivalent. |
| Ultra-fine scale | A&D | GR-202 | Or equivalent. |
| E-Gel iBase Power System | Invitrogen | G6400UK | For RNA gels; or equivalent. |
| E-Gel EX 1% agarose precast gels | Invitrogen | G4020-01 | For RNA gels; or equivalent. |
References
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Mortazavi, A., Williams, B. A., Mccue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Nagalakshmi, U., Wang, Z., et al. The transcriptional landscape of the yeast genome defined by RNA sequencing. Science. 320, 1344-1349 (2008).
- Wilhelm, B. T., Marguerat, S., et al. Dynamic repertoire of a eukaryotic transcriptome surveyed at single-nucleotide resolution. Nature. 453, 1239-U1239 (2008).
- Dölken, L., Ruzsics, Z., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Yang, E., van Nimwegen, E., et al. Decay rates of human mRNAs: correlation with functional characteristics and sequence attributes. Genome Res. 13, 1863-1872 (2003).
- Friedel, C. C., Dölken, L., Ruzsics, Z., Koszinowski, U. H., Zimmer, R. Conserved principles of mammalian transcriptional regulation revealed by RNA half-life. Nucleic Acids Res. 37, e115 (2009).
- Nilsen, T. W., Graveley, B. R. Expansion of the eukaryotic proteome by alternative splicing. Nature. 463, 457-463 (2010).
- Windhager, L., Bonfert, T., et al. Ultra short and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Res. , (2012).
- Melvin, W. T., Milne, H. B., Slater, A. A., Allen, H. J., Keir, H. M. Incorporation of 6-thioguanosine and 4-thiouridine into RNA. Application to isolation of newly synthesised RNA by affinity chromatography. Eur. J Biochem. 92, 373-379 (1978).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23, 232-237 (2005).
- Kenzelmann, M., Maertens, S., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proc. Natl. Acad. Sci. U.S.A. 104, 6164-6169 (2007).
- Schwanhäusser, B., Busse, D., et al. Global quantification of mammalian gene expression control. Nature. 473, 337-342 (2011).
- Rabani, M., Levin, J. Z., et al. Metabolic labeling of RNA uncovers principles of RNA production and degradation dynamics in mammalian cells. Nat. Biotechnol. , (2011).
- Miller, M. R., Robinson, K. J., Cleary, M. D., Doe, C. Q. TU-tagging: cell type-specific RNA isolation from intact complex tissues. Nat. Methods. 6, 439-441 (2009).
- Miller, C., Schwalb, B., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Mol. Syst. Biol. 7, 458 (2011).
- Weintz, G., Olsen, J. V., et al. The phosphoproteome of toll-like receptor-activated macrophages. Mol. Syst. Biol. 6, 371 (2010).
- Lipsett, M. N. The isolation of 4-thiouridylic acid from the soluble ribonucleic acid of Escherichia coli. Journal of Biological Chemistry. 240, 3975-3978 (1965).
- Marcinowski, L., Liedschreiber, M., et al. Real-time Transcriptional Profiling of Cellular and Viral Gene Expression during Lytic Cytomegalovirus Infection. PLoS Pathog. 8, e1002908 (2012).
- Chomczynski, P., Mackey, K. Short technical reports. Modification of the TRI reagent procedure for isolation of RNA from polysaccharide- and proteoglycan-rich sources. Biotechniques. 19, 942-945 (1995).
- Friedel, C. C., Dölken, L. Metabolic tagging and purification of nascent RNA: implications for transcriptomics. Mol. Biosyst. 5, 1271-1278 (2009).
- Friedel, C. C., Kaufmann, S., Dölken, L., Zimmer, R. HALO - A Java framework for precise transcript half-life determination. Bioinformatics. , (2010).



