Summary
Totaal cellulair RNA verschaft een slechte template voor het bestuderen korte termijn veranderingen in RNA synthese en verval en de kinetiek van RNA processing. We beschrijven hier metabolisch merken van nieuw getranscribeerde RNA met 4-thiouridine gevolgd door thiol-specifieke biotinylering en zuivering van nieuw getranscribeerde RNA waardoor deze beperkingen te overwinnen.
Abstract
De ontwikkeling van de hele-transcriptome microarrays en next-generation sequencing heeft een revolutie ons begrip van de complexiteit van de cellulaire genexpressie. Naast een beter begrip van de moleculaire mechanismen betrokken zijn nauwkeurige metingen van de onderliggende kinetiek steeds belangrijker geworden. Hier, deze krachtige methodieken staan voor grote beperkingen als gevolg van intrinsieke eigenschappen van de sjabloon monsters ze studeren, dwz totaal cellulair RNA. In veel gevallen optreden veranderingen in totaal cellulair RNA ofwel te langzaam of te snel om de onderliggende moleculaire gebeurtenissen en hun kinetiek vertegenwoordigen voldoende resolutie. Daarnaast wordt de bijdrage van veranderingen in RNA synthese, verwerking en verval niet gemakkelijk onderscheiden.
We hebben onlangs ontwikkelde hoge-resolutie genexpressieprofilering om deze beperkingen te overwinnen. Er wordt gebruik gemaakt van metabolisch merken van nieuw getranscribeerde RNA met 4-thiouridine (dus ook wel 4SU-tagging) gevolgd door strenge zuivering van nieuw RNA getranscribeerd met thiol-specifieke biotinylering en streptavidine beklede magnetische korrels. Het is toepasbaar op een breed scala aan organismen, waaronder gewervelden, Drosophila en gist. Wij hebben met succes 4SU-tagging toegepast om te studeren real-time kinetiek van transcriptiefactor activiteiten, nauwkeurige metingen van RNA halfwaardetijden, en het verkrijgen van nieuwe inzichten in de kinetiek van RNA processing. Tenslotte kunnen computermodellen worden gebruikt om een geïntegreerde, alomvattende analyse van de onderliggende moleculaire mechanismen te genereren.
Introduction
Genexpressie is een belangrijk instrument dat wordt gebruikt om cellulaire processen en de bijbehorende complexe interactie netwerk bestuderen. Studies over mRNA overvloed hebben meestal al de methode van keuze voor fundamentele inzichten te krijgen in de onderliggende moleculaire mechanismen. De ontwikkeling van de hele-transcriptome microarrays 1 en, meer recentelijk, next-generation sequencing van RNA (RNA-seq) 2-4 aangewakkerd deze aanpak. Hoewel deze technologieën ons begrip van de complexiteit van de cellulaire genexpressie hebben een revolutie, waarmee zij worden geconfronteerd grote beperkingen als gevolg van de intrinsieke eigenschappen van hun template monster, dwz totaal cellulair RNA. Eerste, korte-termijn veranderingen in totaal RNA niet overeenkomen veranderingen in transcriptiesnelheden, maar inherent afhankelijk RNA halfwaardetijd van de respectievelijke transcripten. Terwijl een vijfvoudige inductie van een kortstondige transcript, bijv. coderend voor een transcriptiefactor, zal gemakkelijk detecteerbaar in totaal RNAbinnen een uur, dezelfde inductie van een langlevende transcript, bijv. codeert voor een metabolisch enzym, zal vrijwel onzichtbaar blijven. Bovendien, zelfs een complete shut-down (> 1.000-voudig down-regulatie) in de transcriptie snelheid van een gemiddelde gen met een RNA halfwaardetijd van vijf uur zal nemen gewoon vijf uur om zijn totaal RNA niveaus dalen met slechts tweeledig . Daarom analyse van totaal RNA bevordert de detectie van opwaartse regulatie van kortlevende transcripten, waarvan vele coderen voor transcriptiefactoren en genen met regulerende functie 5. Daarnaast wordt de ware kinetische cascade van regulering verduisterd en primaire signalering evenementen kunnen niet worden onderscheiden van de secundaire. Beide zijn beurt kan leiden tot aanzienlijke vertekening in daaropvolgende bioinformatica analyses. Ten tweede, kunnen veranderingen in totaal RNA niveaus niet worden toegeschreven aan veranderingen in RNA synthese of verval. Metingen van de laatste eisen cel invasieve benaderingen, zoals het blokkeren van Transcriptiover het gebruik van actinomycine D 6, en uitgebreide monitoring van lopende RNA verval in de tijd. Met een gemiddelde mRNA halfwaardetijd in zoogdiercellen van 5-10 uur 5,7, wordt mRNA van de meeste genen slechts gedaald met minder dan twee keer na verscheidene uren van transcriptionele arrest. Deze vrij kleine verschillen leiden tot grove onnauwkeurige metingen van mRNA halfwaardetijden van de meeste cellulaire genen als gevolg van de exponentiële aard van de onderliggende wiskundige vergelijkingen. Tenslotte, terwijl de RNA-seq van totaal cellulair RNA bleek dat ongeveer de helft van onze genen kunnen alternatieve splicing gebeurtenissen 8, de onderliggende kinetiek en de dynamische mechanismen leidende weefsel-en context-specifieke regulatie van RNA processing blijven slecht begrepen. Bovendien is de bijdrage van RNA verwerking differentiële genexpressie, in het bijzonder voor niet-coderende RNA's, nog worden vastgesteld. Al met al, deze beperkingen vormen de belangrijkste obstakels voorbioinformatic kinetische modellering van de onderliggende moleculaire mechanismen.
Onlangs hebben we een aanpak ontwikkeld, genaamd hoge resolutie genexpressieprofilering, om deze problemen te overwinnen 5,7,9. Het is gebaseerd op metabolisch merken van nieuw getranscribeerde RNA onder toepassing van 4-thiouridine (4SU-tagging), een natuurlijk voorkomende derivaat uridine en geeft direct toegang tot nieuw getranscribeerde transcripten met minimale tussenkomst in celgroei en genexpressie (zie figuur 1) 5, 10-12. Blootstelling van eukaryote cellen om 4SU resultaten in zijn snelle opname, fosforylering aan 4SU-trifosfaat, en incorporatie in nieuw getranscribeerde RNA. Na isolatie van totaal cellulair RNA, de 4SU-gelabelde RNA fractie thiol-specifiek gebiotinyleerd genereren van een disulfidebinding tussen de biotine en nieuw getranscribeerde RNA. 'Totaal cellulair RNA' kan dan kwantitatief worden gescheiden in het label ('nieuw getranscribeerd') en niet-gemerkte ('pre-existing ') RNA met hoge zuiverheid met streptavidine beklede magnetische korrels. Tenslotte wordt gelabeld RNA gewonnen uit de parels door het toevoegen van een reductiemiddel (bijvoorbeeld dithiothreitol) splitsen van het disulfide binding en het vrijgeven van de nieuw getranscribeerde RNA van de bolletjes.
Nieuw getranscribeerd RNA toont de transcriptionele activiteit van elk gen in het tijdsbestek van 4SU blootstelling. 4SU-tagging in de tijdschaal van minuten verschaft dus een momentopname van eukaryote genexpressie en een ideale sjabloon voor downstream-bioinformatica analyses (bv promotor analyse). In gevallen waarin steady-state omstandigheden kan worden aangenomen, de verhoudingen van nieuw getranscribeerd / totaal, opnieuw opgeschreven / ongelabelde en ongelabelde / totaal RNA bieden niet-invasieve en toegang tot nauwkeurige RNA halfwaardetijden 7,13. Bovendien is het belangrijk op te merken dat nieuw getranscribeerde RNA gezuiverd na slechts 5 min 4SU-tagging (5 min 4SU-RNA) jonger dan 15 en 60 min 4SU-RNA.Bij het uitvoeren van zowel de ultra-korte en progressief langere 4SU-tagging in een experimentele setting gecombineerd met RNA-seq, zijn de kinetiek van RNA processing onthuld op nucleotide resolutie 9. Tenslotte tijdsverloop analyses van nieuw getranscribeerd en totaal RNA gecombineerd met computermodellen laten een geïntegreerde analyse van RNA synthese en verval 14.
Kortom, deze benadering maakt de directe analyse van de dynamica van RNA-synthese, verwerking en afbraak in eukaryote cellen. Het is in alle belangrijke modelorganismen zoals zoogdieren, insecten (Drosophila), amfibieën (Xenopus), en gist 5,15,16 toepassing. Het is direct compatibel met microarray analyse 5,17, RNA-seq 9,13,14, en is in vivo 12,15 toepassing. Hier hebben we detail de methodologie te labelen, te isoleren en te zuiveren nieuw getranscribeerd RNA in gekweekte zoogdiercellen. Bovendien, potentiometeral problemen en valkuilen worden besproken.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Metabole Labeling met 4-thiouridine
Maak een gedetailleerd plan van de experimentele opstelling / schema, bijv. bij de 4SU toevoegen aan celkweek en wanneer de monsters te oogsten. Plan ten minste 5 minuten tussen elke conditie. Slechts behandelen cellen van een voorwaarde tegelijk. Handvat max. 3 - 5 gerechten op een gegeven moment. Behandel cellen zo snel mogelijk veranderingen in temperatuur en CO2 te minimaliseren. Vermijd blootstelling van de cellen aan fel licht na 4SU wordt toegevoegd, omdat dit kan leiden tot verknoping van 4SU-gelabelde RNA aan cellulaire eiwitten.
Start van etikettering
- Dooi 4-thiouridine (4SU) vlak voor gebruik en pipet benodigde hoeveelheid 4SU voor elke aandoening in een steriele Falcon buis.
- Neem de benodigde hoeveelheid celcultuurmedium (5 ml per 10 cm schaal) van de borden en voeg aan 4SU bevattende Falcon buis en meng. Verwijder de resterende medium uit de gerechten. <li> Breng 4SU-bevattend medium terug naar de gerechten.
Einde van de etikettering
- Verwijder celkweekmedium van cellen. Voeg 5 ml Trizol aan elke plaat. Voor complexe experimenten, inclusief verschillende tijdstippen of voorwaarden, wordt deze stap best gedaan door twee mensen, een verwijdering van het medium, de andere het toevoegen Trizol en het oogsten van de lysaat.
- Incubeer gedurende 5 minuten bij kamertemperatuur volledige cellysis.
- Gebruik een pipet 10 ml om de plaat af te spoelen met de toegevoegde Trizol. Dit helpt volledige cellysis en sample herstel. Voorzichtig behandelen als Trizol is zeer gevaarlijk bij contact met de huid of ogen! Hebben tegengif voor fenol brandwonden bij de hand (bv. polyethyleen glycol 300 of 400 in industriële spiritus (70:30)). Overdracht monsters polypropyleenbuizen. Houd er rekening mee dat standaard Falcon buizen niet deze hoge g krachten te weerstaan). Monsters kunnen bij -20 ° C gedurende ten minste een maand bewaard tot totale RNA is bereid.
2. RNA Voorbereiding behulp Gewijzigd Trizol Protocol
- Voeg 1 ml chloroform (0,2 ml per ml Trizol) en schud krachtig gedurende 15 sec. Incubeer bij kamertemperatuur gedurende 2-3 minuten.
- Centrifugeer bij 13.000 xg gedurende 15 min bij 4 ° C.
- Breng waterige bovenste fase (die het RNA) een nieuwe 15 ml polypropyleen buis.
- Voeg ½ het reactievolume van beide RNA neerslag buffer en isopropanol (bijv. 3 ml supernatant voeg 1,5 ml RNA neerslag buffer en 1,5 ml isopropanol).
- Meng goed. Incubeer bij kamertemperatuur gedurende 10 minuten.
- Centrifugeer bij 13.000 xg gedurende 10 min bij 4 ° C. Gooi supernatant.
- Kort spin down (5000 g gedurende 30 seconden) en verwijder resterende isopropanol met 200 ul pipet.
- Voeg een gelijk volume van 75% ethanol en schud buis totdat de pellet loskomt. Vermijd breken in vele kleine stukjes als deze kunnen maken verwijdering van residuenl ethanol moeilijk.
- Centrifugeer bij 13.000 xg gedurende 10 min bij 4 ° C. Gooi supernatant.
- Spin down RNA kort en verwijder resterende ethanol met een 200 ul pipet. Herhaal stap en verwijder de resterende ethanol met een 20 ul pipet. Na deze twee stappen is nadere drogen van de pellet wordt uitgevoerd.
- Voeg 100 ui H2O per 100 ug verwachte RNA opbrengst en meng goed door en neer te pipetteren 5-6 keer om te helpen bij het oplossen van het RNA.
- Oplossen en denatureren RNA door verwarming tot 65 ° C gedurende 10 min (shaker) normaal en plaats op ijs.
- Meet RNA concentratie bij 260 nm met behulp van een NanoDrop spectrofotometer, volgens de instructies van de fabrikant. Dit RNA kan bij -80 ° C gedurende ten minste een maand worden bewaard.
3. Thiol specifieke Biotinylatie van Newly Transcribed RNA
- Begin met 60 - 80 ug totaal cellulair RNA.
- Vormen etikettering reactie. Pipetteer in de volgendeOm (per ug RNA):
- 1 pi 10x Buffer Biotinylering
- 7 pl RNA (met 1 ug RNA verdund in nuclease-vrij H2O)
- 2 ui biotine-HPDP (1 mg / ml DMF)
Voeg altijd de biotine-HPDP laatste en meng onmiddellijk door pipetteren. In geval precipiteert het biotine, kan DMF inhoud worden verhoogd tot een eindconcentratie van 40%.
- Incubeer bij kamertemperatuur gedurende 1,5 uur met rotatie.
- Voeg een gelijk volume chloroform. Meng krachtig. Incubeer gedurende 2-3 minuten totdat de fasen te scheiden en beginnen bellen beginnen te verdwijnen.
- Centrifugeer bij 20.000 x g gedurende 5 min bij 4 ° C. Breng de bovenste waterige fase naar een nieuwe buis zorgvuldig.
- Herhaal de stappen 3.4 en 3.5 keer. Misschien wilt u deze stap uit te voeren in 2 ml Phase Lock Gel Zware buizen om het verlies van RNA te verminderen.
- RNA neerslag: voeg 1/10 van het volume 5 M NaCl en een gelijk volumeisopropanol aan de waterfase.
- Centrifugeer bij 20.000 xg gedurende 20 min bij 4 ° C. Gooi supernatant.
- Voeg een gelijk volume van 75% ethanol, centrifugeer bij 20.000 xg gedurende 10 min bij 4 ° C, gooi supernatant.
- Spin kort en verwijder de resterende ethanol met 200 ul pipet.
- Spin kort en verwijder de resterende ethanol met 20 ul pipet.
- Sta niet toe dat RNA om te drogen. Resuspendeer in 50 - 100 ul H 2 O (~ 1 ui per 1 pg ingang RNA). Meng goed door en neer te pipetteren 5-6 keer.
- Controleer RNA kwaliteit door elektroforetische analyse om RNA degradatie te sluiten.
4. Dot Blot Analyse van 4SU-opname (optioneel)
4SU opname kan gemakkelijk worden bepaald door dot blot analyse van gebiotinyleerd RNA. Dit is een optionele stap die trouble shooting en de schatting van 4SU bijmengingspercentages ten opzichte van een gebiotinyleerd DNA-oligo-controle mogelijk maakt. Voor deze test we recommend met joodacetyl-biotine in plaats van biotine-HPDP voor biotinylering van 4SU-gelabelde RNA in stap 3.2. Dit leidt tot een onomkeerbare biotinylering van 4SU-RNA. Daarom kolom-gebaseerde methoden (bijv. RNeasy) kan worden gebruikt voor het herstel van veel kleinere hoeveelheden gebiotinyleerd RNA (bijvoorbeeld 5 ug). Terwijl RNA gebiotinyleerd met biotine-HPDP is ook geschikt voor deze test, het resulterende signaal zwakker en de signaal-ruis-verhouding ongunstiger (figuur 3).
- Volg het protocol voor 4SU-etikettering en de isolatie van totaal cellulair RNA, zoals beschreven in de paragrafen 1 en 2.
- Biotinyleren 4SU-gelabelde RNA zoals beschreven in paragraaf 3 vervangen biotine-HPDP met joodacetyl-biotine en voeren twee chloroformextracties aan overmatige joodacetyl-biotine resten volledig te verwijderen.
- Recover gebiotinyleerd RNA door isopropanol / ethanol precipitatie zoals beschreven of met behulp van een kolom-gebaseerde benadering (bijv. RNeasy) bij kleine hoeveelheden RNA (<10 ug) Gebruikt.
- Incubeer de Zeta membraan in nuclease-vrij water met rockende gedurende 10 minuten.
- Neem het membraan uit de nuclease-vrij water en verwijder overmatig vocht door het plaatsen membraan tussen twee schone papieren handdoeken en stevig te drukken. Lucht-drogen van het membraan gedurende 5 min leidt tot mooier dots.
- Voor elk monster bereid 20 pi 200 ng / ul RNA met ijskoud dot blot bindingsbuffer (10 mM NaOH, 1 mM EDTA). Breng 5 ul van deze verdunning (dus 1 ug RNA) en drie verdere 10-voudige verdunningen (bijvoorbeeld 100, 10 en 1 ng RNA respectievelijk) de Zeta membraan door pipetteren. Pipetteren door een leeg rek van pipet tips kunnen worden om gelijkmatig verdeeld afstand bieden. Alternatief, een dot blot apparaat volgens de instructies van de fabrikant.
- Breng 5 ul van de biotine-gelabelde DNA oligo bij concentraties variërend van 20 ng / ul tot 20 pg / pl (ie 100-0,1 ng oligo) als positieve control het membraan door pipetteren. Gebruik een gebiotinyleerd, 4SU-naïeve sample als negatieve controle.
- Lucht-drogen van het membraan gedurende 5 minuten.
- Incubeer het membraan gedurende 30 minuten in 40 ml blokkeringsbuffer met schommelen.
- Incubeer het membraan met 10 ml 1:1000 streptavidine-mierikswortel peroxidase gedurende 15 min. (5 ml PBS + 5 ml 20% SDS + 10 ul streptavidine-mierikswortel peroxidase)
- Was tweemaal membraan in 40 ml PBS + 10% SDS (20 ml PBS + 20 ml 20% SDS) gedurende 5 minuten.
- Was tweemaal membraan in 40 ml PBS + 1% SDS (38 ml PBS + 2 ml 20% SDS) gedurende 5 minuten.
- Was tweemaal membraan in 40 ml PBS + 0,1% SDS (40 ml PBS + 200 ul 20% SDS) gedurende 5 minuten.
- Verwijder overmatig vocht door het plaatsen membraan tussen twee schone papieren handdoeken en drukken op hen stevig.
- Visualiseer membraangebonden HRP met ECL per instructies van de fabrikant.
- Plaats het membraan in plastic folie / zak, verwijdert luchtbellen en incubeer gedurende 2 min in het donker.
- Expose membraan naarfilm voor 1-5 minuten.
5. Scheiding van gelabelde of niet gelabelde RNA Met streptavidine beklede magnetische korrels
- Heat wasbuffer (3 ml per monster) tot 65 C in een waterbad.
- Bereid verse 100 mM dithiothreitol (DTT) in nuclease-free H 2 O. Doen dat door decanteren 15-30 mg DTT poeder in een schone 50 ml Falcon buis geplaatst op de ultrafijne schaal. Weeg en voeg vereiste hoeveelheid nuclease-vrij H2O
- Heat gebiotinyleerd RNA monsters tot 65 ° C gedurende 10 min denatureren en onmiddellijk plaats op ijs.
- Plaats μMacs kolommen in de magnetische stand. Wij adviseren om niet meer dan 12 monsters tegelijk (6 - 8 monsters zijn optimaal) te verwerken.
- Pre-evenwicht Miltenyi zuilen met 1 ml kamertemperatuur wasbuffer. Dit zal ongeveer 15 minuten duren.
- Ondertussen, voeg 100 ul van streptavidine kralen 50-100 pl gebiotinyleerd RNA. Incubeer bij kamertemperatuur gedurende 15 minuten met rotatie. < li> Als een van de kolommen heeft ingeleid aftappen inmiddels kan dit worden vergemakkelijkt door voorzichtig drukken op de top van de kolom met een gehandschoende vinger. Zodra de stroom is begonnen met de kolommen goed leeglopen.
- Breng de RNA / beads aan de kolommen. Gooi de doorstroom tenzij je wilt het niet-gemerkte RNA-fractie (zie paragraaf 7) herstellen.
- Was driemaal met 0,9 ml 65 ° C wasbuffer (1 ml pipet tips krimpen bij het pipetteren buffers bij 65 ° C).
- Spoel driemaal met 0,9 ml kamertemperatuur wasbuffer.
- Pipetteer 700 ul Buffer RLT (RNeasy MinElute Cleanup Kit, Qiagen) in nieuwe 2 ml buisjes en leg ze onder de kolommen.
- Elueer het nieuw getranscribeerde RNA in de buffer RLT met toevoeging van 100 ul 100 mM DTT aan de kolommen.
- Voer een tweede elutie ronde 3 min later in hetzelfde buisje toevoegen van een 100 pi 100 mM DTT.
6. Terugwinning van opnieuw opgeschreven RNA
nhoud "> Ga verder met de RNeasy MinElute Cleanup (Qiagen) protocol volgens de instructies van de fabrikant. Elute in 25 ul nuclease-free H 2 O. Meet RNA-concentraties met een Nanodrop spectrofotometer. Om de behoefte te ontdooien en opnieuw invriezen RNA voorkomen voordat het indienen aan een high-throughput assay aanbevolen bereiden cDNA direct na de nieuw getranscribeerde RNA gezuiverd. gebruik 2.5 ul van de nieuw getranscribeerde RNA in 20 pi cDNA synthese mix voor cDNA-synthese volgens de instructies van de fabrikant. Voer qRT-PCR controles met 1 : 10 verdunningen van cDNA mengsel WINKEL RNA bij -80 ° C..7. Herstel van Ongelabelde, geconsolideerd RNA (optioneel)
Indien het ongebonden RNA moet worden hersteld, verzamel en combineren de doorstroming (na toevoeging van het RNA-streptavidineparels oplossing voor de kolommen) en de eerste wasbeurt voor daaropvolgende precipitatie. Gewoonlijk volstaat het om slechts 50% van het ongebonden RNA precipiteren als this zal bevatten> 80% van het uitgangsmateriaal.
- Voeg een gelijk volume isopropanol (geen zout toegevoegd moet worden als de wasbuffer bevat reeds 1 M NaCl).
- Centrifugeer bij 20.000 xg gedurende 20 min bij 4 ° C. Gooi supernatant.
- Voeg een gelijk volume van 75% ethanol, centrifugeer bij 20.000 xg gedurende 10 min bij 4 ° C, gooi supernatant.
- Spin kort en verwijder de resterende ethanol met 200 ul pipet.
- Spin kort en verwijder de resterende ethanol met 20 ul pipet.
- Sta niet toe dat RNA om te drogen. Resuspendeer in 100 pi H2O Meng goed door en neer te pipetteren 5-6 keer. Incubeer bij 65 ° C gedurende 10 min onder schudden en rechtstreeks over ijs.
- Controleer RNA kwaliteit door elektroforetische analyse om RNA degradatie te sluiten.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
1. Uitgangsmateriaal en verwachte rendementen
Na 1 uur (hr) van 4SU-exposure nieuw getranscribeerde RNA vertegenwoordigt ongeveer 1-4% van de totale cellulaire RNA. Dit zal lager zijn in de groei-gearresteerd cellen als ze niet langer RNA te synthetiseren om rekening te houden voor de celgroei / replicatie. Bij etikettering voor 1 uur, raden wij het starten van de test met 60 - 80 ug totaal RNA. Beginnend met minder dan 30 ug totaal RNA resulteert in kleine RNA pellets die moeilijk te zien na de biotinylering stap en kan dus gemakkelijk verloren. Input RNA niveaus kan worden verhoogd tot wel 150 pg van zeer korte duur van labeling (bijvoorbeeld 5-10 min). Wanneer de duur van RNA labeling wordt verkort van 1 uur tot 5 min de bijdrage van kortlevende intronsequenties in opnieuw opgeschreven RNA stijgt van ~ 60% tot 80% ~ 9. Aangezien introns in hoofdzaak langer is dan coderende sequenties en 5'-en 3'-UTR's, de hoeveelheid opnieuw opgeschrevenRNA, dat kan worden gezuiverd na korte of ultrakorte 4SU-tagging, niet lineair vallen. Als zodanig, verkregen we> 0,5% van de totale RNA na 5 min 4SU-tagging in niet-adherente humane B-cellijnen 9. Er zij echter op gewezen dat hogere concentratie 4SU en iets langere duur van etikettering nodig vergelijkbare 4SU bijmengingspercentages in hechtende cellen bereiken. Terwijl zelfs een lage 4SU-bijmengingsgehalte zal toestaan efficiënte afvang en zuivering van grote, uridine-rijke transcripten, zeer korte transcript met lage uridine content (miRNA) zijn waarschijnlijk zuivering te ontsnappen, zelfs bij gebruik van hoge 4SU concentraties (> 1 mM). In NIH-3T3 muis fibroblasten, 1 uur van 200 uM 4SU blootstelling bestempeld nieuw getranscribeerde RNA met ongeveer een 4SU residu per 50 - 100 nucleotiden (nt) 5. 1000 nt in lengte - dit moet zeer efficiënte invordering van transcripten> 500 toestaan. Daarom hebben we alleen waargenomen een kleine transcriptiegroottevertekening bij het benoemen gedurende 1 uur met 200 uM 4SU in zowel murine fibroblasten en humane B-cellen 7. Terwijl 1 hr 200 uM 4SU resulteerde niet in enige significante verandering in cellulaire transcripten niveaus in muizen fibroblasten, langdurige blootstelling van cellen aan ≥ 200 uM 4SU resulteert wel in een meetbare groeistoornis binnen 24 uur (ongepubliceerde gegevens). Daarom moeten zowel de duur van de etikettering en de werkenden 4SU-concentratie worden geminimaliseerd om ectopische of toxische effecten te vermijden. Een gemakkelijke manier om de minimale 4SU-concentratie die nodig is voor een efficiënte invordering van nieuw getranscribeerde RNA te bepalen is om nieuw getranscribeerd RNA na 4SU-labeling zuiveren met toenemende concentraties van 4SU (bv 50-1600 uM). Zoals getoond in figuren 2A en 2B, terugwinning van nieuw getranscribeerde RNA gemerkt gedurende 1 uur in primaire menselijke fibroblasten aanzienlijk toegenomen 50-200 uM 4SU maar daarna begon te plateau.
2. StipDep Kwantificering van 4SU Oprichting (optioneel)
In sommige gevallen kan het van belang zijn om de hoeveelheid 4SU incorporatie meten in totaal RNA. Dit kan het beste gebeuren door dot blot analyse van de gebiotinyleerde RNA met behulp van een streptavidineconjugaat. Vanwege de chemische aard joodacetyl-biotine reactiever te thiolgroepen dan biotine-HPDP waardoor de biotinylering van vrijwel alle 4SU residuen in nieuw getranscribeerde RNA. Het is belangrijk op te merken dat, zoals biotine-HDPD, joodacetyl-biotine is niet oplosbaar in water en wordt dus efficiënt verwijderd door chloroform extractie zoals uitgevoerd voor biotine-HPDP. Daarom kan dezelfde reactieomstandigheden en concentraties worden toegepast wanneer gebruik biotine-HPDP. Echter, joodacetyl-biotine is niet omkeerbaar. Er kan dus niet worden gebruikt voor de zuivering van nieuw getranscribeerde RNA in kolom benaderingen. Terwijl het gebruik van joodacetyl-biotine maakt om 4SU-opname kwantificeren, biotine-HPDP gebaseerde metingen zich zowel4SU-integratie en biotinylatie efficiëntie. Gebruikmakend van de twee biotinylering reagentia hetzelfde monster maakt de meting van de biotinylering efficiëntie van RNA-opgenomen 4SU. Biotinylering efficiëntie van biotine-HPDP voor 4SU-gelabelde RNA lijkt ongeveer een factor drie kleiner dan joodacetyl-biotine aangeeft dat slechts een op drie 4SU residuen in nieuw getranscribeerde RNA werkelijk door biotine-HPDP (figuur 3) wordt gebiotinyleerd. Door het monster signaal intensiteiten te vergelijken met de gebiotinyleerde controle DNA oligo, kan biotinylatie dichtheden worden gemeten. Voor de meeste zoogdierlijke cellijnen een positief signaal nog detecteerbaar moeten zijn in 10 ng gebiotinyleerd RNA na 1 uur van 200 uM 4SU labeling. Een zwak achtergrondsignaal gewoonlijk detecteerbaar de hoogste concentratie (1 ug) niet-gemerkt RNA.
3. Zuivering van opnieuw opgeschreven RNA
Herstel van nieuw getranscribeerde RNA is zeer quantitatieve. Als je bent begonnen met hetzelfde RNA-concentratie kunt u verwachten om dezelfde hoeveelheden nieuw getranscribeerde RNA te verkrijgen voor alle monsters. Net als veel andere kolom-gebaseerde testen, kunnen verzameling van nieuw getranscribeerde RNA met behulp van de RNeasy MinElute kit resulteren in extra absorptie bij 230-260 nm (aanwezigheid van detergenten afgeleid van de wasbuffers) die kunnen interfereren met de OD 260 metingen. Dit wordt gezien in mindere mate wanneer een nieuwe 2 ml verzamelbuis per centrifugatie stap. Toch moet elke onredelijk hoge OD metingen (> 2-voudig hoger dan andere monsters) voorzichtig worden bekeken, vooral als OD 260/280 verhoudingen <1.7. Voor stroomafwaartse analyses dus vaak het beste om dezelfde hoeveelheid van matrijs RNA volume gebruiken voor alle monsters. Indien de opbrengsten van gelabelde RNA lager dan verwachte controle op tekenen van RNA afbraak door elektroforetische analyse. Opnieuw opgeschreven RNA bevat aanzienlijk grotere hoeveelheden grote, unspliced transcripten met de typische rRNA bands die minder prominent (figuur 4).
4. Kwantificering van Nieuw Transcribed RNA
Ten slotte kan bijmengingspercentages van 4SU in nieuw getranscribeerde RNA rechtstreeks worden gekwantificeerd door spectrofotometrische analyse op basis van de absorptie maximum van 4SU bij 330 nm en de OD 330/260 verhouding 5,18. Dit vereist> 3 ug van gelabelde RNA geconcentreerd in een klein volume (10-20 pi) van isopropanol / ethanol precipitatie. Om te voorkomen dat de kleine RNA pellet co-precipitatie met 30 ug nuclease-free glycogeen (Fermentas, # R0551) moet worden uitgevoerd. Een extra piek is zichtbaar bij 330 nm als gevolg van de bijmengingsgehalte van 4SU in nieuw uit getranscribeerd RNA (figuur 5).
/ Files/ftp_upload/50195/50195fig1highres.jpg "/>
Figuur 1. . Principe van metabole labeling met 4-thiouridine (4SU) 4SU wordt toegevoegd aan cellen voor de vereiste (5-120 min) tijd gevolgd door de voorbereiding van totaal cellulair RNA. Na thiol-specifieke biotinylering wordt totaal cellulair RNA gescheiden in 4SU-label nieuw getranscribeerde RNA en ongelabelde, bestaande RNA met streptavidine beklede magnetische korrels. Opnieuw opgeschreven RNA wordt uit de korrels met een reducerend middel dat disulfidebindingen dat de nieuw getranscribeerde RNA koppelen aan de kralen splitst. Klik hier om een grotere afbeelding te bekijken .
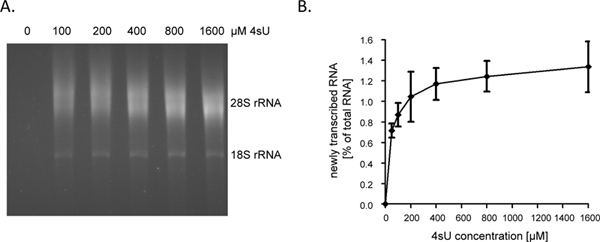
Figuur 2. Herstel van nieuw getranscribeerde RNA na toenemende concentraties 4SU. (A) primaire humane voorhuid fibroblasten (HFF) werden geïncubeerd met 100, 200, 400, 800 of 1600 uM 4SU. Nieuw getranscribeerde RNA werd gezuiverd uit 50 ug totaal cellulair RNA en onderworpen aan elektroforetische analyse. Zoals verwacht, werd een concentratie-afhankelijke stijging van de herstelde pas getranscribeerd RNA waargenomen die begon te plateau bij hogere concentraties. (B) Bedragen van gezuiverd nieuw getranscribeerde RNA werd gekwantificeerd met behulp van de ImageJ 1.45s software. Gecombineerde gegevens van vier onafhankelijke experimenten op de bedragen van de nieuw getranscribeerde RNA teruggevorderd in verschillende concentraties van 4SU-etikettering variërend van ofwel 50-800 uM 4SU (n = 2) of 100 -. 1600 uM 4SU (n = 2) worden getoond Klik hier om een grotere afbeelding te bekijken .
upload/50195/50195fig3.jpg "alt =" Figuur 3 "fo: content-width =" 4.5in "fo: src =" / files/ftp_upload/50195/50195fig3highres.jpg "/>
Figuur 3. Schatting van 4SU incorporatie in 4SU-gelabeld totaal RNA met dot blot analyse. Totaal RNA werd geïsoleerd uit NIH-3T3 murine fibroblasten of humane voorhuid fibroblasten (HFF) geïncubeerd met 200 uM 4SU een uur. Geen 4SU werd aan een gerecht als negatieve controle toegevoegd. Voor HFF zowel contact geremd (n = niet-groeiende cellen) en de groeiende cellen (y) werden opgenomen. RNA werd geïsoleerd met behulp van Trizol reagens en vervolgens geconjugeerd aan biotine-HPDP of joodacetyl-biotine. Concentratie van elk monster werd ingesteld op 200 ng / pl en 5 pl van deze verdunning (dus 1 ug RNA), en drie verdere 10-voudige verdunningen (bijvoorbeeld 100, 10 en 1 ng RNA, respectievelijk), allemaal gespot op een stuk van Zeta membraan. 5 pl verdunningen van biotine-gelabelde DNA oligo werden op het membraan geplaatst positieve controles op CONCENTRATionen variërend van 20 ng / ul tot 20 pg / pl (ie 100-0,1 ng respectievelijk). Biotine dichtheid werd gesondeerd met een streptavidine horseradish peroxidase conjugaat.
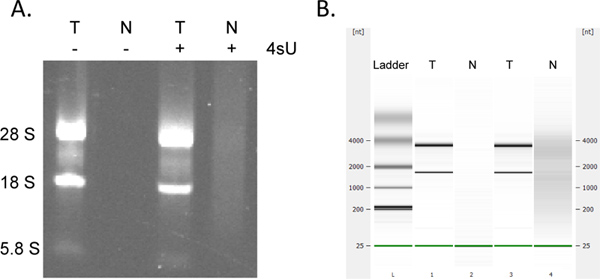
Figuur 4. Elektroforetische analyse van nieuw getranscribeerde en totaal RNA. Totaal RNA (T) en nieuw getranscribeerde RNA (N) bereid uit muizen NIH-3T3 fibroblasten zowel met als zonder 500 uM 4SU gedurende 1 uur werd geanalyseerd door agarose gel elektroforese (A) en (in dezelfde volgorde) met de Agilent bioanalyser (B). Geen RNA werd teruggevonden zonder 4SU behandeling van de cellen. Gezuiverd opnieuw opgeschreven RNA bevat grotere hoeveelheden hoogmoleculaire mRNAs en aanzienlijk minder dan de totale volwassen rRNAsRNA als opmerkelijk tussen de 28S, 18S, en 5.8S rRNA bands. Klik hier om een grotere afbeelding te bekijken .
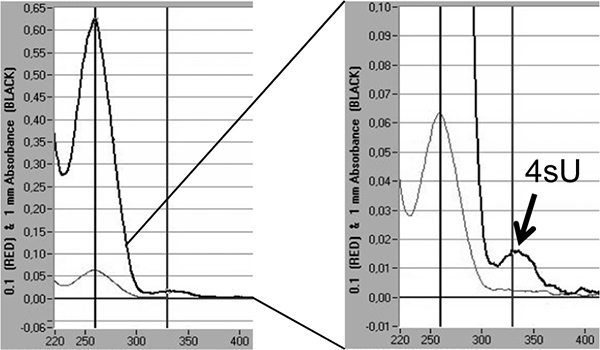
Figuur 5. Kwantificering van 4SU incorporatie in nieuw getranscribeerde RNA door spectrofotometrische analyse. Newly getranscribeerd RNA gezuiverd uit 2 x 100 ug totaal RNA na 1 uur van 200 uM 4SU in muizen NIH-3T3 fibroblasten. Nieuw getranscribeerde RNA werd geprecipiteerd met isopropanol / ethanol na het toevoegen van 30 ug nuclease-vrij glycogeen. Spectrofotometrische analyse van nieuw getranscribeerde RNA verkregen door een Nanodrop 1000 spectrofotometer wordt getoond. De lichtgrijze lijnen geven de metingen bij 0,1 mm, terwijl de dikkere, donkere grijze lijnen vertegenwoordigen metingen bij 1 mm kolom vloeistof. Aan de rechterkant, een vergroting van de piek van uitsterven vertegenwoordigen tHij verwerkt 4SU-resten wordt getoond. Op basis van de extinctie-coëfficiënt van 4SU 18 de incorporatie tarieven van 4SU kan worden geschat.
| Duur van de etikettering [min] | Aanbevolen 4SU concentratie [uM] |
| 120 | 100-200 |
| 60 | 200 - 500 |
| 15-30 | 500 - 1000 |
| <10 | 500 - 2000 |
Tabel 1. Aanbevolen 4SU concentraties.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Metabole etikettering van nieuw getranscribeerde RNA aanzienlijk versterkt de kracht van high-throughput technologieën zoals microarrays en RNA-seq door meer geschikte templates om de biologische vraag van belang aan te pakken. Het huidige protocol werd na optimalisatie. Het maakt> 1000-voudige verrijking van nieuw getranscribeerde RNA en biedt zeer reproduceerbare resultaten.
Het experimentele ontwerp van een 4SU-tagging experiment is van cruciaal belang als nieuw getranscribeerde RNA zal de real-time transcriptieactiviteit verbeelden enkel op het moment van de blootstelling van cellen aan 4SU. Als de werkelijke veranderingen in transcriptiesnelheden na een stimulus reeds verdwenen, zullen deze missen bij het analyseren nieuw getranscribeerde RNA terwijl de veranderingen in totale RNA nog steeds detecteerbaar. Daarom is een goed begrip van de onderliggende biologie belangrijk voor de experimentele opstelling en de optimale perioden o definiërenf tijd voor 4SU blootstelling. Hieronder geven we aanbevelingen en manieren voor het vermijden van valkuilen voor de meest cruciale stappen.
Voorbereiding van de stamoplossingen en plastic ware
Alle voorraad-oplossingen moeten worden bereid met nuclease-vrij water. Met behulp van in-house gezuiverd gedemineraliseerd water kan leiden tot problemen als het water bevat reducerende middelen. In een geval resulteerde in het volledig verlies van gelabeld RNA. Daarom raden wij het kopen van pre-gemaakt nuclease-vrij NaCl, Tris-Cl, EDTA, natriumcitraat en water. Zorgen nuclease-vrije omstandigheden te allen tijde. Dimethylformamide (DMF) opgelost sommige kunststoffen. We vonden dat het gebruik van 25 ml celkweek plastic pipetten om DMF te brengen van zijn voorraad glazen fles naar 50 ml Falcon buizen naar de biotine-HPDP stockoplossing bereid was voldoende om de opbrengsten van de nieuw getranscribeerd RNA uit de hele test aanzienlijk verminderen. Interessant is dat deze geen negatieve invloed hebben op de biotinylatie efficiëntie (zoals getest door dot blot), maar resulteerde in een 75 tot> 90% verlies van de nieuw getranscribeerde RNA die kunnen worden verhaald op de kralen. Het verlies was het meest uitgesproken wanneer de duur van de etikettering, daalde van 60 tot 30 minuten of minder. Waarschijnlijk een stof geëlueerd uit de plastic pipetten door de DMF gedeeltelijk de bekleding van de streptavidineparels vernietigd. Daarom moet het gebruik van kunststoffen niet bekend compatibel met DMF te worden vermeden worden. Om dezelfde redenen moet cel schrapers niet worden gebruikt voor het herstel van Trizol monsters van celcultuur platen te verbeteren. Het is interessant op te merken dat de vermeende stoffen uitgewassen uit de kunststof door de DMF of Trizol blijkbaar niet werden verwijderd door chloroform extractie noch isopropanol / ethanol precipitatie.
Celcultuur
Celdichtheid op de platen is van cruciaal belang. In een experiment waarbij de cellen bleek iets te confluent (90 -100%), behandeld we NIH-3T3 murine fibroblasten gedurende 30 min met 100 U / ml interferon (IFN) α en γ. In minder confluente cellen nog 15 min van IFN reeds resulteerde in een 5 - tot 8-voudige inductie van genen zoals IRF1 of SOCS3 5. Met cellen die lichtjes te samenvloeiing microarray analyse eventuele inductie van IFN-induceerbare genen niet tonen voor zelfs de meest snel induceerbare genen zoals IRF1 of SOCS3. Daarom celdichtheid is een cruciale factor voor 4SU-labeling experimenten en alle celkweek platen moeten zorgvuldig voordat u het etiket worden onderzocht.
4SU is een fotoactiveerbare ribonucleoside en 4SU bevattende RNA wordt efficiënt verknoopt aan eiwitten na blootstelling aan 365 nm lichtbron. 4SU behandelde cellen worden gekweekt in het donker en blootstelling aan licht te vermijden. Na verwijdering van cellulaire eiwitten door Trizol RNA isolatie dit risico aanzienlijk wordt verminderd.
<p class = "jove_content"> 4SU is niet opgenomen in cellulair DNA. Er zij echter op gewezen dat de totale RNA nog kleine hoeveelheden cellulair DNA bevat. Bij gebruik 4SU-tagging en q-RT-PCR-analyse van virale genexpressie te bestuderen in cytomegalovirusinfectie vonden we het nodig om een DNaseI digest stap in het protocol bij de concatemere virale genomen te verwijderen 19 omvatten. Dit is waarschijnlijk niet nodig bij gebruik downstream protocollen die niet gevoelig zijn voor de aanwezigheid van DNA.4SU bijmengingspercentages en optimale 4SU concentratie
4SU wordt gemakkelijk opgenomen door cellen met intra-en extra-cellulair niveau het meest waarschijnlijk tot evenwicht binnen minder dan een minuut 9,16. Opname en incorporatie tarieven van 4SU zijn concentratie-afhankelijk. Daarom kan 4SU concentratie gemakkelijk worden aangepast aan de dienst duur van de etikettering. Tabel 1 geeft advies over 4SU concentraties in relatie om de duur van de etikettering op basis van onze beste persoonlijke ervaring. 1 uur van 4SU labeling in zoogdiercellen, wordt 200 pM 4SU voldoende voor de meeste toepassingen resulteert in ongeveer een 4SU residu per 50 tot 100 nucleotiden nieuw getranscribeerde RNA in fibroblasten zijn.
In de laatste paar jaren hebben we 4SU-tagging toegepast op een brede waaier van celtypes van mensen en muizen oorsprong waaronder fibroblasten, endotheelcellen, epitheelcellen, beenmerg stroma cellen, macrofagen en T-cellen. Verder werden cellen van Drosophila en Xenopus succes gebruikt. In al deze experimenten werd 4SU opname gevonden zeer efficiënt om met minimale aanpassingen 4SU concentratie voor de verschillende celtypes. Bij het opzetten van de methode voor het nieuwe celtypen, zouden we aanraden om label cellen met toenemende 4SU-concentraties (bijv. variërend 50-1600 uM) en analyseert de relatie van gezuiverde nieuw getranscribeerd RNA de toegepaste 4SU-concentraties (zie figuur 2A / B). De 4SU-concentratie waarbij de hoeveelheid gezuiverde opnieuw opgeschreven RNA gaat een plateau worden gekozen.
In gevallen waarin zeer samenvloeiing, remde contact cellen worden gebruikt, raden we aan iets hogere 4SU concentraties (bv 500 ipv 200 uM) gebruiken om efficiënt 4SU incorporatie garanderen. Bovendien, in gevallen waarin de vangst van zeer korte onlangs uitgeschreven transcripten (<200 nt) is van bijzonder belang, de 4SU concentratie kan ook moeten worden verhoogd. Dit moet niet worden gecombineerd met verlengde labeling tijd (bijv.> 1 uur) om ectopische effecten of toxiciteit te vermijden. Tenslotte vonden we dat het gebruik van een te klein volume van media voor celkweek kan 4SU incorporatie efficiëntie verminderen. Wij adviseren daarom 5 ml of 10 ml medium per 10 cm of 15 cm schotel, respectievelijk.
Bereiding van totaal cellulair RNA
Voor het welslagen van dit protocol is het cruciaal om schoon, RNase-free totaal cellulair RNA te verkrijgen. Met behulp van 5 ml Trizol per 15 cm schotel produceert schone RNA vrij van nucleases. Wij raden het gebruik van de gemodificeerde Trizol protocol door Chomczynski et al.. 20. Ten eerste is het beter geschikt om grote hoeveelheden RNA (> 100 mg) te isoleren als verbeterde centrifugale kracht resulteert in een steviger pellets die makkelijker te hanteren tijdens de wasstappen. Dit vereist echter het gebruik van speciale polypropyleen buizen en adapters als de reguliere 15 ml laboratorium Falcon buizen overleven niet meer dan 6000 × g. Ten tweede bevordert de verwijdering van DNA en glycoproteïnen. Dit wordt met name duidelijk bij het opstellen van RNA van organen of weefsels. Ten derde is het niet de maximale hoeveelheid totaal RNA dat geïsoleerd kan worden beperken. Hoewel we vonden ook de column-gebaseerde RNA isolatie methoden (bv. RNeasy) om RNA van geschikte kwaliteit, standaard kolommen ar biedene alleen in staat om opname tot 100 ug totaal RNA waardoor de hoeveelheid uitgangsmateriaal beperken. Tenslotte, door het verwijderen van de resterende ethanol tweemaal met een pipet, het drogen van het RNA om resterende ethanol te verwijderen is niet meer nodig. Dit elimineert het risico van over-drogen RNA, die moeilijk om achteraf weer oplossen mogelijk. In principe 4SU-tagging is in vivo toepassing, bijv. door iv injectie van muizen. Maar we vastgesteld dat RNA zuiverheid vormt een groot probleem dat de zuivering van polyA transcripten vóór de zuivering van nieuw getranscribeerde RNA (ongepubliceerde gegevens).
Biotinylatie en verwijdering van ongebonden biotine
Biotine-HPDP is 100% thiol-specifiek en vormt een disulfidebinding tussen de biotine en het residu thiol-gemerkte RNA-moleculen getranscribeerd nieuw. Biotinylering efficiëntie van 4SU-gelabelde RNA is ongeveer 30% zoals bepaald door dot blot analyse 5. Als biotine-HPDP is niet oplosbaar in waterefficiënte wijze kunnen worden verwijderd door extractie met chloroform. Een enkele chloroform extractiestap voldoende is om de meeste niet-gebonden biotine verwijderen regelmatige Herhaal deze stap om volledige verwijdering te verzekeren. Om RNA te beperken tijdens de chloroform extractiestap 2 ml Gel Heavy Phase Lock buizen (Eppendorf) worden gebruikt volgens instructies van de fabrikant. Meestal gebruiken we de Phase Lock buizen alleen de tweede chloroform extractiestap als de matrijs volumes van de eerste stap dikwijls te groot rechtstreeks compatibel met deze buizen zijn. Na verwijdering van ongebonden biotine-HPDP wordt RNA teruggewonnen door isopropanol / ethanol precipitatie. Het is belangrijk op te merken dat commerciële kolom gebaseerde kits de gebiotinyleerde RNA (bijv. RNeasy van QIAGEN) terug mag niet worden gebruikt Bevat reductiemiddelen in de buffers voorzien die klieven disulfidebinding en verwijder het biotine uit de nieuw getranscribeerde RNA .
Zuivering van nieuwely getranscribeerd RNA
Niet meer dan 100 ul gebiotinyleerde RNA tot 100 ul streptavidineparels voegen. Minder volume toevoegen voorkeur. Toch moet hetzelfde volume van RNA worden toegevoegd voor alle monsters. Pas het RNA-ingang volume (tussen monsters) die u toevoegt aan de streptavidineparels door simpelweg het vereiste volume 1x TE toe te voegen aan de kralen. Een gemakkelijke manier om verse nuclease-free 100 mM DTT maken is om een voldoende hoeveelheid DTT poeder in een valk buis geplaatst op een ultra-gevoelige schaal decanteren en voeg de benodigde hoeveelheid nuclease-free H 2 O tot 100 mM DTT genereren (64,8 pi water per 1 mg DTT).
Tijdens de ontwikkeling van 4SU-tagging we getest streptavidineparels van verschillende leveranciers. Een aantal van hen gegenereerde grote hoeveelheden achtergrond. Daarom raden wij het gebruik van de Miltenyi Streptavidine kralen als, tot nu toe, hebben we nooit problemen ondervonden met de overdracht van niet-gemerkt RNA uit weefsel cULTUUR afkomstige RNA monsters. Op deze wijze kan slechts 150 ng gelabelde RNA specifiek gezuiverd uit 150 ug gebiotinyleerd RNA (in 100 pl water) met 100 ui streptavidine beads. Equilibration van de kralen met de equilibratiebuffer met de kralen geleverd kan worden uitgevoerd en kunnen enigszins verbeteren capture tarieven 13.
Kwaliteitscontroles
Wij raden het uitvoeren van q-RT-PCR controle op pas getranscribeerd RNA voordat u deze blootstelt voor high-throughput analyse. Dit kan kwantificering van verscheidene verwijzing genen waarvan bekend differentieel gereguleerd in de gegeven experimentele setting. In gevallen waarin 4SU-tagging wordt gebruikt om RNA vervalsnelheden bestuderen, zouden we aanraden om een kortstondige transcript (bijvoorbeeld myc, fos) en een lange levensduur een (bv. GAPDH) in zowel de totale en nieuw getranscribeerde RNA kwantificeren. De verhouding van nieuw getranscribeerde / totaal RNA zou aanzienlijk hoger zijn (~ 5 - tot 10-voudig)voor de kortstondige transcripten. Op basis van de RNA halfwaardetijd van een referentie-gen kan RNA halfwaardetijden vastgesteld. Als alle drie RNA fracties (totaal RNA, nieuw getranscribeerde RNA en ongelabelde bestaande RNA) geanalyseerd voor vier of meer genen, normalisatie van de verschillende subsets RNA kan door lineaire regressieanalyse en kwaliteitscontrole scores kunnen worden bepaald zoals beschreven 7 , 21.
Voor q-RT-PCR-analyse, raden wij u 2,5 pl gelabelde RNA in 20 pi cDNA-synthese mix. Voor een optimale vergelijking van q-RT-PCR resultaten bevriezen het cDNA in hoeveelheden van 5 pi vóór het eerste gebruik. Dooi buisjes vlak voor gebruik, voeg 45 ul van H 2 O en onder 5 ul van de verdunningen aan q-RT-PCR analyses. Dit verhoogt aanzienlijk de vergelijkbaarheid tussen verschillende PCR-runs.
Nieuw getranscribeerde RNA monsters moet worden gecontroleerd op tekenen van RNA degradatie met behulp van de Agilent bioanalyser alvorens hen te onderwerpen aanhigh-throughput analyse (microarrays of RNA-seq). Hierbij moet echter worden opgemerkt dat aanvullende bands soms worden waargenomen door de Agilent bioanalyser. De biologische betekenis van deze onduidelijk. Zoals onlangs getranscribeerd RNA bevat aanzienlijk minder ribosomaal RNA, deze monsters af mislukken van de Agilent bioanalyser kwaliteitscontroles. Indien dit niet het gevolg toegankelijk RNA afbraak voorbeelden van aanvaardbare kwaliteit zijn gewoonlijk geschikt worden onderworpen aan hoge-doorvoer analyse.
Compatibiliteit van nieuw getranscribeerde RNA met down-stream analyses
Nieuw getranscribeerd RNA bevat aanzienlijk meer mRNA dan totaal RNA. Dit is voornamelijk te wijten aan de grotere hoeveelheden intronsequenties in nieuw getranscribeerd RNA die toenemen wanneer de duur van 4SU-tagging wordt verkort. Daarom hebben we niet periodiek de uitputting van rRNA's van nieuw getranscribeerde RNA monsters als dit vereist grotere hoeveelheden uitgangsmateriaal blijven terwijl deing nogal klein (~ tweevoudige) toename in niet-rRNA leest. Tenslotte moet nog worden opgemerkt dat het grootste percentage van ongesplitste hoog molecuulgewicht transcripten aanwezig in nieuw getranscribeerde RNA bijkomende versnippering vereisen bij de voorbereiding cDNA bibliotheken voor de volgende generatie sequencing. Resultaten van de grootte fragmentatie stap moet daarom kwaliteit zorgvuldig gecontroleerd.
Gegevensnormalisatie voor RNA half-live-metingen
De standaardbenadering van de experimentele gegevens voor RNA halfwaardetijd meting aanpassen om alle gegevens te normaliseren aan het RNA halfwaardetijd van een goed gekarakteriseerde huishoudgen gen of de mediaan RNA halfwaardetijd in een gegeven celtype bepaald eerdere experimenten. In zoogdiercellen, deze in het bereik van 5 tot 10 uur 6,7. Deze aanpak werkt ook heel goed voor 4SU gebaseerde metingen, zijn andere middelen voor normalisatie vereist als het mediane RNA halfwaardetijd niet bekend of het may ook worden beïnvloed door veranderingen in het cellulaire systeem onder studie, bijvoorbeeld door de knock-out van een RNA verval pad. 4SU-tagging biedt een unieke manier van het schatten van de mediane RNA halfwaardetijd gebaseerd op de analyse van alle drie RNA-fracties, dwz totaal cellulair RNA, nieuw getranscribeerd RNA, en ongelabelde reeds bestaande RNA. Zoals totaal cellulair RNA wordt gescheiden in de laatste twee fracties RNA een eenvoudig lineair regressiemodel worden gebruikt om de drie RNA fracties normaliseren elkaar en bepalen de RNA mediane halveringstijd 7,16. Een softwarepakket is online beschikbaar om deze analyses uit te voeren 22.
Inefficiënte opname van transcripten met lage uridine inhoud kan beïnvloeden RNA halfwaardetijd metingen resulteert in een kunstmatig lage pas getranscribeerd / totaal RNA ratio's en langdurige RNA halfwaardetijden. De omvang van dit probleem kan worden beoordeeld door het plotten van RNA halveringstijden of log (nieuw getranscribeerd / totaal RNA's) tegen de uridine inhoud van alle transcripten 7,15. Dit biedt ook een goede kwaliteitscontrole aan verschillen in 4SU-bijmengingspercentages tussen verschillende monsters of voorwaarden te beoordelen. Wanneer een sterke correlatie aan uridine inhoud wordt waargenomen kan dit worden gecorrigeerd door bio middelen 15. Er moet worden opgemerkt dat de bijdrage van rijpe transcripten in nieuw getranscribeerde RNA niet gemakkelijk kan worden onderscheiden van de veel grotere en daardoor veel meer uridine-rijk precursors. Tenzij de verwerking kinetiek van een bepaalde transcript bekend zijn (wat ze meestal niet) eenvoudig te corrigeren voor laag uridine inhoud (inefficiënte opname) kan schromelijk RNA halfwaardetijden verstoren. Als zodanig hebben wij onlangs vonden verwerking van de meeste menselijke snoRNAs tot zeer inefficiënt 9. Als we de pas getranscribeerd / totaal RNA's voor de lage uridine inhoud van de vrij kleine (70-300 nt) had gecorrigeerd snoRNAs, zou dit hebben geresulteerd in extreem korte snoRNA half-lives (<5 min) met talrijke nieuwe getranscribeerd / totaal RNA ratio van meer dan 100%. Daarom hebben we in het algemeen niet aan te raden te corrigeren voor laag uridine inhoud bij het meten van RNA halfwaardetijden.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
De auteurs verklaren dat zij geen concurrerende financiële belangen.
Acknowledgments
Wij willen Amie Regan bedanken voor zorgvuldige lezing van het manuscript. Dit werk werd ondersteund door NGFN Plus subsidie # 01GS0801, MRC fellowship subsidie G1002523 en NHSBT subsidie WP11-05 op LD en DFG subsidie FR2938/1-1 naar CCF
Materials
| Name | Company | Catalog Number | Comments |
| 4-thiouridine | Carbosynth | T4509 | Prepare 50 mM stock in sterile H2O, store at -20 °C in aliquots of 50-500 μl, discard unused reagent, do not refreeze. |
| Trizol | Invitrogen | 15596026 (100 ml), 15596018 (200 ml) | WARNING - CORROSIVE and HAZARDOUS TO HEALTH! Ensure immediate access to Phenol antidote (PEG-Methanol); Store at 4 °C. |
| Chloroform | Sigma | 372978 | WARNING - HAZARDOUS TO HEALTH |
| Isopropanol | Sigma | 650447 | |
| Sodium citrate, nuclease-free | Sigma | C8532 | Prepare 1.6 M stock solution using nuclease-free water. |
| 5M nuclease-free NaCl | Sigma | 71386 | Stock solution |
| Nuclease-free H2O | Sigma | W4502 | Make 1 ml aliquots in nuclease-free tubes. |
| RNA precipitation buffer | 1.2 M NaCl, 0.8 M sodium citrate in nuclease free water. Prepare in advance under strictly nuclease-free conditions. Store at room temperature in 50 ml falcon tubes. | ||
| Ethanol | Sigma | 459844 | Use with nuclease-free water to prepare 80% ethanol, store at -20 °C. |
| 1 M nuclease-free Tris Cl, pH 7.5 | Lonza | 51237 | Stock solution |
| 500 mM nuclease-free EDTA, pH 8.0 | Invitrogen | 15575-020 | Stock solution |
| 10x Biotinylation Buffer (BB) | 100 mM Tris pH 7.4, 10 mM EDTA in nuclease-free water, make aliquots of 1 ml. | ||
| Dimethylformamide (DMF) | Sigma | D4551 | |
| EZ-Link biotin-HPDP | Pierce | 21341 | Prepare 1 mg/ml stock solution by dissolving 50 mg biotin-HPDP in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. |
| Phase Lock Gel Heavy tubes 2.0 ml | Eppendorf | 0032 005.152 | Optional for the chloroform extraction step. |
| Zeta membrane | BIORAD | 162-0153 | |
| 10x Dot blot binding buffer | 100 mM NaOH, 10 mM EDTA | ||
| Biotin-oligo | 5'-biotin, 25 nucleotides, any sequence | ||
| Sodium dodecyl sulphate | Fisher | BPE9738 | For 100 ml 20% stock solution, add 20 g SDS to 80 ml PBS pH 7-8 and adjust volume to 100 ml. Keep all high-percentage SDS solutions above 20 °C. Warm the solutions slightly should SDS precipitate. |
| EZ-Link Iodoacetyl-LC-Biotin | Pierce | 21333 | Prepare 1 mg/ml stock solution by dissolving 50 mg iodoacetyl-biotin in 50 ml DMF. Gentle warming enhances solubilisation. Store at 4 °C in aliquots of 1 ml. Generates irreversible, thiol-specific biotinylation. |
| Phosphate buffer saline | Gibco | 10010-015 | |
| Dot blot blocking buffer | Mix 20 ml 20% SDS with 20 ml 1 x PBS pH 7-8 and add EDTA to the final concentration of 1 mM. | ||
| Streptavidin-horseradish peroxidase | Vector Laboratories | SA5004 | Store at -20 °C. Mix 10 ml 20% SDS with 10 ml 1 x PBS. Add 20 μl Streptavidin-HRP before use. |
| ECL reagent | GE Healthcare | RNP2109 | Use following the manufacturer's instructions. |
| Super RX, X-RA Film, 18x24 cm | Fujifilm | 47410 19236 | |
| μMacs Streptavidin Kit | Miltenyi | 130-074-101 | Store the beads at 4 °C. |
| Tween 20 | Sigma | P1379 | |
| Washing buffer | 100 mM Tris pH 7.4, 10 mM EDTA, 1 M NaCl, 0.1% Tween 20 in nuclease-free H2O. | ||
| Dithiothreitol (DTT) | Sigma | 43817 | Prepare as 100 mM DTT in nuclease-free H2O, always prepare fresh before use. |
| RNeasy MinElute Kit | Qiagen | 74204 | Store columns at 4 °C, remaining components of the kit at room temperature. |
| 1.5 ml screw-top polypropylene tubes | Sarstedt | 72.692.005 | Compatible with Dimethylformamide |
| 2.0 ml screw-top polypropylene tubes | Sarstedt | 72.694.005 | Compatible with Dimethylformamide |
| 15 ml tubes | BD Falcon | 352096 | Compatible with Dimethylformamide |
| 50 ml tubes | BD Falcon | 352070 | Compatible with Dimethylformamide |
| All solutions/reagents should be stored at room temperature unless otherwise specified. | |||
| Equipment | |||
| UV/VIS spectrophotometer | Thermo Scientific | NanoDrop 1000 | Or equivalent. Use low volume (1-2 μl) for measurements of low RNA concentrations to avoid excessive sample loss. |
| Polypropylene 15 ml centrifuge tubes | VWR International | 525-0153 | In contrast to standard 15 ml tubes, these tolerate up to 15,000 × g |
| High-speed centrifuge | Beckman Coulter | Avanti J-25 | Or equivalent equipment capable of reaching 13,000×g |
| High-speed rotor | Beckman Coulter | JLA-16250 | Or equivalent equipment capable of reaching 13,000×g |
| Adaptors for 15 ml tubes | Laborgeräte Beranek | 356964 | Or equivalent equipment capable of reaching 13,000×g |
| Refrigerated table-top centrifuge | Eppendorf | 5430 R | Or equivalent. |
| Thermomixer | Eppendorf | Thermomixer compact | Or equivalent. |
| Magnetic stand | Miltenyi Biotec | 130-042-109 | One stand holds 8 μMacs columns. |
| Waterbath | Grant | SUB Aqua 5 | Or equivalent. |
| Ultra-fine scale | A&D | GR-202 | Or equivalent. |
| E-Gel iBase Power System | Invitrogen | G6400UK | For RNA gels; or equivalent. |
| E-Gel EX 1% agarose precast gels | Invitrogen | G4020-01 | For RNA gels; or equivalent. |
References
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Mortazavi, A., Williams, B. A., Mccue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Nagalakshmi, U., Wang, Z., et al. The transcriptional landscape of the yeast genome defined by RNA sequencing. Science. 320, 1344-1349 (2008).
- Wilhelm, B. T., Marguerat, S., et al. Dynamic repertoire of a eukaryotic transcriptome surveyed at single-nucleotide resolution. Nature. 453, 1239-U1239 (2008).
- Dölken, L., Ruzsics, Z., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Yang, E., van Nimwegen, E., et al. Decay rates of human mRNAs: correlation with functional characteristics and sequence attributes. Genome Res. 13, 1863-1872 (2003).
- Friedel, C. C., Dölken, L., Ruzsics, Z., Koszinowski, U. H., Zimmer, R. Conserved principles of mammalian transcriptional regulation revealed by RNA half-life. Nucleic Acids Res. 37, e115 (2009).
- Nilsen, T. W., Graveley, B. R. Expansion of the eukaryotic proteome by alternative splicing. Nature. 463, 457-463 (2010).
- Windhager, L., Bonfert, T., et al. Ultra short and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Res. , (2012).
- Melvin, W. T., Milne, H. B., Slater, A. A., Allen, H. J., Keir, H. M. Incorporation of 6-thioguanosine and 4-thiouridine into RNA. Application to isolation of newly synthesised RNA by affinity chromatography. Eur. J Biochem. 92, 373-379 (1978).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23, 232-237 (2005).
- Kenzelmann, M., Maertens, S., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proc. Natl. Acad. Sci. U.S.A. 104, 6164-6169 (2007).
- Schwanhäusser, B., Busse, D., et al. Global quantification of mammalian gene expression control. Nature. 473, 337-342 (2011).
- Rabani, M., Levin, J. Z., et al. Metabolic labeling of RNA uncovers principles of RNA production and degradation dynamics in mammalian cells. Nat. Biotechnol. , (2011).
- Miller, M. R., Robinson, K. J., Cleary, M. D., Doe, C. Q. TU-tagging: cell type-specific RNA isolation from intact complex tissues. Nat. Methods. 6, 439-441 (2009).
- Miller, C., Schwalb, B., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Mol. Syst. Biol. 7, 458 (2011).
- Weintz, G., Olsen, J. V., et al. The phosphoproteome of toll-like receptor-activated macrophages. Mol. Syst. Biol. 6, 371 (2010).
- Lipsett, M. N. The isolation of 4-thiouridylic acid from the soluble ribonucleic acid of Escherichia coli. Journal of Biological Chemistry. 240, 3975-3978 (1965).
- Marcinowski, L., Liedschreiber, M., et al. Real-time Transcriptional Profiling of Cellular and Viral Gene Expression during Lytic Cytomegalovirus Infection. PLoS Pathog. 8, e1002908 (2012).
- Chomczynski, P., Mackey, K. Short technical reports. Modification of the TRI reagent procedure for isolation of RNA from polysaccharide- and proteoglycan-rich sources. Biotechniques. 19, 942-945 (1995).
- Friedel, C. C., Dölken, L. Metabolic tagging and purification of nascent RNA: implications for transcriptomics. Mol. Biosyst. 5, 1271-1278 (2009).
- Friedel, C. C., Kaufmann, S., Dölken, L., Zimmer, R. HALO - A Java framework for precise transcript half-life determination. Bioinformatics. , (2010).



