Introduction
Antistoffer, der kan folde og fungerer i det intracellulære miljø er lovende redskaber til både forskning og terapeutiske anvendelser. De har evnen til at modulere protein-aktivitet ved binding til et målprotein i celler for at forhindre protein-protein-interaktioner, forstyrre protein-nukleinsyre-interaktioner, eller forhindre substrat adgang til enzymer 1-5.
Selvom antistoffer har stort potentiale for intracellulære applikationer, ingeniør dem for korrekt foldning og opløselighed i det intracellulære miljø under opretholdelse af evnen til at binde til et målantigen er udfordrende. Den reducerende cytoplasmatiske miljø forhindrer dannelsen af disulfidbindinger normalt kræves til den stabile foldning af fuld-længde antistoffer og antistoffragmenter, herunder enkelt-kæde variable fragment (scFv) antistoffer 6,7. En række rettet evolution tilgange er blevet anvendt til at konstruere antistoffer med hIgH tilhørsforhold til target antigener 8-10. Disse tilgange almindeligt brug fagdisplay, gær overflade display, eller bakteriel overflade display at screene store biblioteker af antistoffer 11-13. Disse metoder er kraftfulde og effektive til identificering af antistoffer, der binder til mål, men de afhænger af sekretoriske sti for at transportere proteiner, der vises 14-16. Den sekretoriske pathway translokerer ufoldede proteiner fra den reducerende cytoplasma ind i det endoplasmatiske reticulum lumen i gær eller i periplasmaet i bakterier. Proteinerne så fold under oxiderende betingelser og vises på celleoverfladen eller pakket i fagpartikler til screening for bindingsaffinitet 17,18. Som følge heraf vil antistoffer isoleret ved hjælp af disse teknikker ikke nødvendigvis fold godt i cytoplasmaet, og intracellulær opløselighed skal ofte manipuleret særskilt, hvis antistofferne vil blive anvendt i intracellulære applikationer.
At forbedreeffektiviteten af tekniske antistoffer, der er godt foldet i cytoplasmaet, vi tidligere rapporteret succes MAD-TRAP (membran-forankret display til Tat-baseret genkendelse af associerende proteiner), en fremgangsmåde til screening af et scFv-antistof-bibliotek under anvendelse af Escherichia coli inder- membran display 19. Bakteriel indre membran display afhængig af twin-arginin translokation (Tat) pathway til transport viste antistoffer, i modsætning til andre almindeligt display, som bruger den sekretoriske pathway. Tat-banen indeholder en kvalitetskontrol mekanisme, der kun tillader opløselige, korrekt foldede proteiner, som skal transporteres fra E. coli cytoplasma, på tværs af indre membran, og i periplasmaet 20,21. Overudtrykt Tat substrater (dvs.., Proteiner målrettet mod Tat-banen med en N-terminal fusion til Tat signalpeptidet ssTorA), der er godt foldet i cytoplasmaet danne en lang levetid translokation mellemprodukt med N-terminalen in cytoplasmaet og C-terminalen i periplasmaet 19. Dette tillader visning af korrekt foldede Tat substrater, herunder antistoffragmenter, om det periplasmiske side af E. coli indre membran. Efter fjernelse af ydre membran ved enzymatisk spaltning til dannelse sfæroplaster er antistoffer udsat for det ekstracellulære rum (figur 1). Dette tillader Tat substrater vises på den indre membran, der skal screenes for binding til et bestemt mål. Vigtigt er det, at udnytte Tat-banen for celle-overflade display sikrer, at kun de antistoffer i biblioteket, som er godt foldet i cytoplasmaet, vil blive afhørt for binding, tillader samtidig manipulation af bindingsaffinitet og intracellulær foldning. I denne protokol, beskriver vi, hvordan at vise et scFv-bibliotek på E. coli indre membran, panorere biblioteket mod en mål-antigen, og udføre en sekundær skærm til at identificere de mest lovende bestanddele af biblioteket. Mens vi fokuserer protokollen om scFv'er, kunne metoden anvendes på engineering enhver protein, hvis applikation kræver binding og intracellulær foldning.
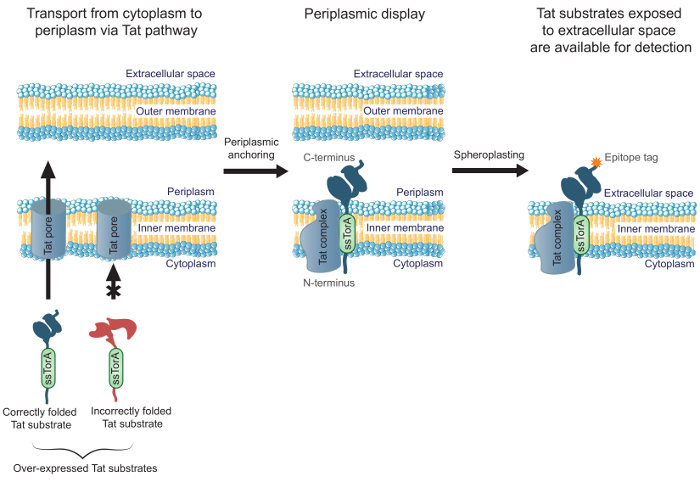
Figur 1. Tat indre membran display. I E. coli, scFv-antistoffer, der udtrykkes som en fusion til den ssTorA signalsekvensen og korrekt foldede i cytoplasmaet transporteres over den indre membran. En translokation mellemformer, hvor scFv'erne forankres i den indre membran med N-terminalen i cytoplasmaet og C-terminalen i periplasmaet. E. coli ydre membran fordøjes enzymatisk for at danne sfæroplaster og derved udsætte den forankrede antistoffer til det ekstracellulære rum og gøre dem tilgængelige for påvisning ved anvendelse af et antistof, som binder til C-terminalt smeltet epitopmærke på det viste antistof.belastning / 54.583 / 54583fig1large.jpg "target =" _ blank "> Klik her for at se en større version af dette tal.
Protocol
1. Klargør scFv-bibliotek som en fusion til ssTorA signalsekvensen
- Opnå en deoxyribonukleinsyre (DNA) bibliotek indeholdende varianter af et scFv-gen.
BEMÆRK: Biblioteket kan også konstrueres ved hjælp af enhver egnet tilstand til at generere mangfoldighed over hele scFv genet eller målrettede domæner 22 (f.eks den tredje komplementaritetsbestemmende regioner, CDR3'er.). - Indsæt DNA-biblioteket ind i pIMD plasmidet (figur 2) ved anvendelse af standard molekylær kloningsmetoder 23.
BEMÆRK: Dette plasmid udtrykker scFv'er som en genetisk fusion til ssTorA signalsekvensen (N-terminalt for scFv) og FLAG-epitopmærke (C-terminalt for scFv). Udformningen af plasmid til indre membran display er tidligere 19 beskrevet. Den pIMD plasmid er tilgængelig fra forfatterne.

Figur 2. Inner-membran display plasmid (pIMD) kort (trin 1.2 gennem 1.3). Dette plasmid indeholder en lac-promotor, ColE1 replikationsstartområde, og et chloramphenicol-resistensgen. Den indsatte scFv-genet er fusioneret til ssTorA signalsekvensen at målrette scFv til Tat-banen og et FLAG-epitopmærke, med alle tre i den samme læseramme. Restriktionsenzymsites er indikeret. For et bibliotek indsat mellem Xbal- og Notl- restriktionsenzymsteder, størrelsen af plasmidet er 2219 bp plus størrelsen af scFv. Klik her for et større version af denne figur.
- Omdan plasmid DNA indeholdende biblioteket i MC4100 E. coli-celler 23. Restituere sig og dyrke denne bakterielle form af biblioteket. Der centrifugeres ved 4.000 x g i 15 minutter ved stuetemperatur for at opsamle cellerne. Fjern supernatanten og resuspender opsamlede celleri 25% glycerol i Luria-Bertani (LB) medium. Opbevar portioner ved -80 ° C indtil brug, eller fortsætte til trin 2.
BEMÆRK: Protokollen er blevet verificeret med MC4100 celler, men andre E. coli stammer forventes også at være forenelig med protokollen. Elektroporering er den foretrukne fremgangsmåde til transformation, på grund af sin høje transformationseffektivitet. Biblioteket bør typisk bestå af mindst 10 9 scFv-varianter på dette stadium, og hver alikvot bør indeholde nok celler, således at biblioteket er dækket 100 gange.
2. Udtryk biblioteket og Forbered Sfæroplaster
- Tø en alikvot af den bakterielle bibliotek (fra trin 1.3) ved stuetemperatur, og tilsæt portionen til en kolbe indeholdende 100 ml LB medium med 20 ug / ml chloramphenicol (Cm). Grow i 3 timer ved 37 ° C og 225 rpm i en inkuberet rysteapparat.
- Efter 3 timer, kolben fra 37 ° C inkuberet shaker fjerne. Muliggøre ekspression af scFv-biblioteket tilFortsæt O / N for 15 til 22 timer ved 20 ° C og 225 rpm i en inkuberet rysteapparat.
BEMÆRK: Der kræves ingen inducer, når du bruger pIMD plasmidet, som promotoren er utæt. Bemærk at MC4100 celler ikke overudtrykke Lac repressor (og LacI ikke findes på plasmidet).

Figur 3. E. coli-celler og sfæroplaster. (A) E. coli-celler er cylindriske i form. (B) Efter spheroplasting hjælp EDTA og lysozym, den ydre membran af E. coli-celler brister, og de resulterende sfæroplaster er sfæriske i form. Differential interferens kontrast (DIC) mikroskopi billeder blev opnået under anvendelse af en 100X objektiv på et inverteret mikroskop. Zoom cslikke her for at se en større version af dette tal.
- Forbered bibliotekets sfæroplaster.
BEMÆRK: Sfæroplaster dannes ved at bryde den ydre membran af E. coli og er sfærisk form (figur 3).- Forbered de nødvendige buffere.
BEMÆRK: Alle buffere skal være sterile.- Forberede 1 × phosphatbufret saltvand (PBS; pH 7,4) ved opløsning af 8 g NaCl, 0,2 g KCI, 1,44 g Na 2 HPO 4, og 0,24 g KH 2 PO 4 i destilleret H2O til et slutvolumen på 1000 ml. Hold på is.
- Forbered PBS med 0,1% (w / v) bovint albumin serum (BSA) ved at opløse 0,2 g BSA i 200 ml 1 x PBS. Hold på is.
- Forbered fraktionering puffer (FB) ved at blande 7,5 ml sterilfiltreret 1 M saccharose, 1 ml 1 M Tris-buffer (pH 8,0), og 1,5 ml destilleret H2O Hold på is.
- Forberede 1 mM ethylendiamintetraeddikesyre (EDTA) ved tilsætning 30 pi of 0,5 M EDTA til 14,97 ml destilleret H2O
- Forbered 0,5 M MgCl2 ved at opløse 4,76 g MgCI2 i 100 ml destilleret H2O Hold på is.
- Kolben fjernes fra rysteren, og måle den optiske densitet (OD) ved 600 nm ved anvendelse af et spektrofotometer til bestemmelse af celletætheden. Beregn nødvendigt volumen af induceret kultur, således at hver prøve til spheroplasting har 1 × 10 10 celler.
BEMÆRK: tilnærmelse af en OD 600 på 1 indikerer en koncentration på 10 9 celler / ml for E. coli kan anvendes 24. - Centrifuger den beregnede mængde af induceret kultur i et 1,5 ml mikrocentrifugerør ved 12.000 x g ved stuetemperatur i 5 min. Fremstilles mindst to prøver i tilfælde af, at et problem opstår i forberedelsen.
- Fjern supernatanten fra de centrifugerede kulturer og resuspender hver cellepellet i 100 pi iskold FB. Centrifuge ved 12.000 ×g ved stuetemperatur i 1 min, og derefter fjerne supernatanten ved pipettering. Resuspender hver pellet i 350 pi iskold FB suppleret med 3,5 pi 10 mg / ml lysozym.
- Langsomt vortexes hvert rør under tilsætning dråbevis 700 pi 1 mM EDTA, og derefter inkuberes rørene ved stuetemperatur i 20 minutter, medens langsomt roterende på et rør rotator at blande prøverne. Fjern rørene fra rotator, tilsættes 50 pi iskold 0,5 M MgCl2 til hvert rør, og inkuber dem på is i 10 min. Centrifuger rørene ved 11.000 x g ved 4 ° C i 10 minutter.
- Isoler sfæroplast pellet.
- Brug en mikropipette med en 1 ml tip til langsomt at trække en del af pillen. Hold røret i en vinkel med åbningen direkte over en ny 1,5 ml rør, langsomt løfte pipettespidsen ud af supernatanten og skub pillen ind i det nye rør.
- Hvis en betydelig mængde supernatant overføres til det nye rør, fjerne den ved pipettering. Hvis pelleten er ikke fast enough at overføre, re-centrifuger ved 11.000 x g i 2 min og forsøg pellet isolation igen.
- Resuspender spheroplast pellet i hvert rør i 1 ml iskold 1 × PBS. Skift mellem pipettering og langsomt vortex på en vortex holder indtil pillen er fuldt resuspenderes. Ikke holde prøver ud af isen i mere end 2 minutter ad gangen, og vende tilbage til is i mindst 5 minutter før fjernelse fra isen igen. Hold sfæroplasterne ved 4 ° C (i op til 2 dage) indtil anvendelse til panning i trin 4.
- Forbered de nødvendige buffere.
3. Immobilisere målantigenet på magnetiske perler
- Biotinylere målantigenet in vivo under rekombinant produktion i E. coli-celler. Alternativt kan du bruge kemisk konjugation 25 eller køb mål-antigen, der allerede er biotinyleret, og fortsætte til trin 3.2.
- Tilføj 816 g bicin til 50 ml vand til fremstilling 10 × bicin-buffer. Fortynd buffer til 1 × i destilleret H2O og opvarm til 50 ° C. Tilføj 14,7 mg biotin til 12 ml af den opvarmede 1 × bicin-puffer til at gøre et biotin løsning, der er 5 mM biotin i 10 mM bicin-buffer. Opbevar ved -20 ° C indtil brug.
- Udtrykke og biotinylere målproteinet hjælp af pAK400cb-BCCP plasmid 26, som muliggør produktion af målantigenet som en fusion til biotin carboxyl carrier protein (BCCP).
BEMÆRK: E. coli-celler nativt biotinylere BCCP, eliminerer behovet for at oprense og kemisk biotinylering målproteinet før immobilisering på streptavidin-coatede kugler. De indfødte E. coli biotinligase BirA er tilstrækkelig til biotinylering af fusionsproteinet.- Grow E. coli indeholdende biotinylering plasmidet (med målantigenet indsat som en fusion til den N-terminale ende af BCCP) O / N i 15 til 18 timer i 5 ml LB medium suppleret med 20 ug / ml Cm ved 37 ° C under omrystning ved 225 rpm.
- Mål OD ved 600 nm ved anvendelse af et spektrofotometer og beregne volumenet af kulturen nødvendige (V add) til subkultur ved en start OD på 0,05 i 25 ml frisk LB-medium med 20 ug / ml Cm anvendelse af ligningen: V add = (0,05 × 25 ml) / (OD600 - 0,05), hvor OD600 er den optiske densitet af O / N-kulturen og V add er rumfanget af O / N-kulturen til at føje til den friske LB. Subkultur og vokse til en OD på 0,5 til 0,8 i en inkuberet rysteapparat ved 37 ° C og 225 rpm.
- Tilføj isopropyl β-D-1-thiogalactopyranosid til en slutkoncentration på 100 uM og biotin til en endelig koncentration på 5 uM. Inducere ekspression i et inkuberet rysteapparat i 15 til 22 timer ved 20 ° C og 225 rpm.
- Harvest bakterier ved centrifugering ved 4.000 x g ved 4 ° C i 10 min. Fjern supernatanten. Opbevar pellet ved -20 ° C indtil den er klar til brug.
- Tilføje1 ml af en cellelyse detergent pr 0,2 g cellepellet. Opblande ved pipettering og forsigtigt rotere i 20 min for at lysere cellerne. Efter lyse centrifuge ved 16.000 x g og 4 ° C i 20 min. Pipettere det opløselige lysat (supernatant) i en ny 1,5 ml rør.
- Brug en 3 kDa molekylvægtsafskæring kolonne for at fjerne det ubundne biotin. Pipettere lysatet ind i søjlen, og der centrifugeres ved 20 ° C ifølge producentens anvisninger. Vask med 1 × PBS indtil biotin i lysatet er blevet fortyndet 100 gange, og volumenet af den vaskede lysat er lig med det oprindelige volumen af lysat. Overfør lysatet til et nyt rør.
- Immobilisere det biotinylerede målantigen på streptavidin-coatede magnetiske perler.
- Forberede 1 × PBS og 1 × PBS med 0,1% (vægt / volumen) BSA som beskrevet i trin 2.3.1.
- Forbered de magnetiske perler.
BEMÆRK: Dette kræver anvendelse af en magnetisk separation rack.- Resuspender streptavIDIN-coatede magnetiske perler i deres originale glas. Enten vortex i mindst 30 sek eller rotere i 5 min.
- Overfør 7-10 × 10 9 perler til et 1,5 ml rør.
BEMÆRK: Volumenet kræves vil afhænge af perlekoncentration leveret af producenten. - Glasset anbringes indeholdende perler på magneten rack i 2 minutter for at indsamle perlerne på siden af røret. Med røret stadig på magneten, forsigtigt fjerne supernatanten ved pipettering uden at forstyrre perlerne.
- At vaske, fjerne røret fra magneten, og resuspender perlerne i 1 ml 1 x PBS ved pipettering uden at generere bobler. Retur røret til magneten i 2 minutter for at opsamle perlerne, og fjern forsigtigt supernatanten ved pipettering. Gentag processen to gange mere til i alt tre vaske. Sikre, at ingen er væske i røret efter den sidste vask.
- Tilføj lysatet indeholdende biotinyleret antigen til den magnetiske beads.
- Fjern røret fra magneten og resuspender perlerne i 1 ml lysat (fra trin 3.1.5). Inkuber ved stuetemperatur i 30 minutter under forsigtig rotation.
- Glasset anbringes på magneten i 3 min for at indsamle de antigen-coatede perler. Vask de coatede perler fem gange med 1 × PBS med 0,1% BSA i den samme måde som beskrevet i trin 3.2.2.3 til 3.2.2.4. Efter den sidste vask resuspenderes perlerne i 1 × PBS med 0,1% BSA op til samme volumen anvendes i trin 3.2.2.2.
- Hvis det immobiliserede målantigenet er stabilt ved 4 ° C, opbevares de overtrukne perler ved 4 ° C indtil det skal bruges til panning. Ellers skal du fortsætte til Trin 4.
4. Skærm scFv Bibliotek ved panorering mod målantigenet (figur 4)

Figur 4. panorering (Trin 4). Antigen-coatede magnetiske perler are inkuberet med sfæroplaster, der udtrykker antistofbibliotek varianter. Plasmid-DNA fra perle-bundne sfæroplaster genvindes og anvendes til at generere et underbibliotek, som er screenet under anvendelse af ELISA-baserede sekundær screening. Tilsvarende protokoltrin er noteret. Klik her for at se en større version af dette tal.
- Inkuber de coatede perler med sfæroplaster.
- Brug en sfæroplast til vulst forhold på ca. 5: 1. Tilsæt 4 × 10 9 sfæroplaster og 8 × 10 8 perler til et sterilt 15 ml rør.
BEMÆRK: Antag, at ingen celler blev tabt under spheroplasting processen, så koncentrationen er stadig 1 × 10 10 sfæroplaster / ml. - Tilsæt 1 × PBS med 0,1% BSA for at bringe det totale volumen til 4 ml. Alikvot i fire 1,5 ml rør med 1 ml hver. Inkuber reaktionerne ved 4 ° C i 5 timer under forsigtig rotation.
- Brug en sfæroplast til vulst forhold på ca. 5: 1. Tilsæt 4 × 10 9 sfæroplaster og 8 × 10 8 perler til et sterilt 15 ml rør.
- prepare vulsten-bundne sfæroplaster for polymerasekædereaktion (PCR).
- Placer panorering reaktionsrør på magneten i 3 min. Fjern supernatanten ved pipettering, og vaske perle-bundne sfæroplaster fire gange med iskold 1 × PBS med 0,1% BSA i den samme måde som beskrevet i trin 3.2.2.3 til 3.2.2.4. Resuspendere perle-bundne sfæroplaster i hvert rør i 25 pi destilleret H2O Opbevar perlerne ved -20 ° C eller gå til trin 4.3.
- Udføre hel-plasmidet PCR på perle-bundne sfæroplaster at opformere plasmider indeholdende generne for perle-bundne scFv'er.
- Opnå primere med følgende sekvenser: 5'CCAACTCTTTTTCCGAAGGTAACTG3 '(fremadrettet primer) og 5'TAGCTCTTGATCCGGCAAACAAA3' (reverse primer).
BEMÆRK: Disse vil binde end-to-end på modsatte strenge af pIMD plasmidet (figur 2) og er designet til at anneale til et fælles træk ved pIMD, så amplifikation sker, uanset denscFv variant sekvens. - Phosphorylere primerne.
BEMÆRK: Uden fosforylering, religering ikke vil forekomme. Primere kan også bestilles med 5'-fosforylering, frem for at bruge denne phosphorylering metode i denne protokol.- I en 0,5 ml rør, der er nedsat en phosphoryleringsreaktion for den fremadrettede PCR-primer som beskrevet i tabel 1. Gentag denne proces for den reverse primer.
- Inkuber reaktionerne ved 37 ° C i 1 time. Derefter inkubere dem ved 65 ° C i 20 minutter for at deaktivere T4 polynukleotidkinase (PNK). Opbevar phosphorylerede primere ved -20 ° C.
- Udføre PCR.
- I en PCR-rør, forberede PCR-reaktionen som beskrevet i tabel 2.
BEMÆRK: Flere reaktioner kan være forberedt til højere udbytte. De ubenyttede perle-bundne sfæroplaster kan opbevares ved -20 ° C. - Varm PCR reaktioner ved 98 ° C i 15 min i en thermal cycler at sikre fuld lysis af sfæroplasterne. Fjern rørene fra den termiske cyklusapparat, og der tilsættes 0,5 pi af en high fidelity polymerase til hver. Retur rør til gennemførelse af termiske cykler og køre ved hjælp af programmet beskrevet i tabel 3.
- I en PCR-produkter efter behov. Opbevar ved -20 ° C eller gå til trin 4.4.
- I en PCR-rør, forberede PCR-reaktionen som beskrevet i tabel 2.
- Opnå primere med følgende sekvenser: 5'CCAACTCTTTTTCCGAAGGTAACTG3 '(fremadrettet primer) og 5'TAGCTCTTGATCCGGCAAACAAA3' (reverse primer).
Tabel 1. PNK phosphorylering reaktion (trin 4.3.2.1).
| Reagens | Volumen (pi) |
| Destilleret H2O | 15 |
| 10x T4 DNA ligase reaktionsbuffer | 2 |
| 100 pM primer | 2 |
| T4-polynukleotidkinase (PNK) | 1 |
| Reagens | Volumen (pi) |
| Destilleret H2O | 28.5 |
| 5x High-fidelity polymerase buffer | 10 |
| 10 uM Phosphoryleret fremadrettet primer | 2.5 |
| 10 uM Phosphoryleret reverse primer | 2.5 |
| 40 mM dNTP-blanding (10 mM hver dNTP) | 1 |
| Bead-bundne sfæroplaster | 5 |
Tabel 3. PCR program (trin 4.3.3.2).
| Trin | Temperatur (° C) | Tid (min: sek) | Antal cykler |
| initial denaturering | 98 | 00:30 | 1 |
| denaturere | 98 | 00:10 | 35 |
| annealing | 69 | 00:30 | |
| Udvidelse | 72 | 00:30 per kb | |
| Final udvidelse | 72 | 06:00 | 1 |
| Holde | 12 | Uendelig | 1 |
- Re-cirkularisere hele-plasmidet PCR-produkter, og bruge det ligerede produkt til at transformere MC4100 E. coli-celler.
- Oprens PCR-produktet ved at køre PCR-reaktionen på en agarosegel 23, farvning DNA'et i gelen 23, og under anvendelse af engel oprydning kit til at rense lineariserede plasmid ved at følge instruktionerne, der leveres af producenten. Måle koncentrationen under anvendelse af et spektrofotometer ved 260 nm. Opbevar oprenset fragment ved -20 ° C indtil brug, eller fortsætte til trin 4.4.2.
- Re-cirkularisere plasmid fra PCR-produktet.
- At forhindre intermolekylær ligering af PCR-produktet, udføre ligeringsreaktionen med en lav koncentration 27 af 1 ng / pi PCR-produkt. Beregn rumfanget nødvendig til fremstilling af en 800 pi ligeringsreaktion ved denne koncentration.
- Forbered ligeringsreaktionen på is. I et rør, tilsæt volumen, beregnet i trin 4.4.2.1 af PCR-produktet, 80 pi 10 × DNA ligasepuffer og destilleret H2O op til 800 pi. Tilsættes 4 pi T4 DNA-ligase, og straks placere rørene ved 16 ° C i et vandbad eller termisk cykliseringsapparat. Der inkuberes ved 16 ° CO / N for 14 til 18 timer. Opbevar de udfyldte ligeringsreaktioner ved -20 ° Cindtil de skal bruges, eller fortsætte til trin 4.4.3.
- Placer ligeringsreaktionen på en varmeblok ved 65 ° C i 15 min for at varme inaktivere DNA-ligase. Brug derefter en mikrodialyse membran eller DNA oprydning kit til de-salt det ligerede DNA. Opbevar ved 20 ° C eller fortsætte til trin 4.4.4.
- Brug hele varmeinaktiveret, de-saltet ligeringsprodukt at transformere MC4100 E. coli-celler 23. Forbered glycerolstamopløsninger, som beskrevet i trin 1.3, i de celler, der indeholder det resulterende panoreret underbibliotek og opbevar prøver ved -80 ° C.
- Gentag trin 4 i sin helhed ved hjælp af en portion fra trin 4.4.4 til at gøre en anden panorering på underbibliotek.
BEMÆRK: En anden panning hjælper berige for bibliotekets bestanddele, som binder godt til målantigenet 19.
5. Udfør en sekundær skærm Brug af en enzymmaerket Assay Metode til identificere lovende kloner til yderligere karakterisering (figur 5) </ P>

Figur 5. ELISA-baserede sekundær screening (trin 5). (A) Bibliotek varianter fra underbibliotek beriget under panning inokuleres i individuelle brønde i en dyrkningsplade for vækst og ekspression. (B) en ELISA-plade coates med mål-antigen. (C) Biblioteket screenes ved hjælp af ELISA-baserede sekundære screening beskrevet i protokollen. Ved analyse af data fra den sekundære skærm, er varianter af interesse udvalgt og karakteriseret yderligere. Tilsvarende protokoltrin er noteret. Klik her for at se en større version af dette tal.
- Tø ét rør af panoreres underbibliotek (fra trin 4.4.4) og plade på LB-agarplader. Plate flere fortyndingerved koncentrationer lave nok til at sikre individuelle kolonier (f.eks 10 2 - til 10 6 fold fortyndinger). Pladerne inkuberes i 15 til 18 timer ved 37 ° C. pladerne opbevares ved 4 ° C eller gå til trin 5.2.
- Kultur og inducerer kolonier fra panorerede underbibliotek. Udfør alle trin under sterile forhold. Brug en multikanalpipette for trin, der involverer plader med 96 brønde.
- Tilsæt 200 pi LB med 20 ug / ml Cm i hver brønd i en rundbundet 96-brønds dyrkningsplade.
- Pick en individuel koloni fra agarpladen med en pipettespids, placere spidsen i den første brønd af 96-brønds plade, og forsigtigt omrøres til podning. Brug en ny spids til hver brønd. Inokulere en koloni i hver brønd. Som en kontrol, omfatte mindst én sterilitet kontrol godt med nogen koloni podet.
- Gentag trin 5.2.1 og 5.2.2 at pode flere plader med 96 brønde.
- Placer plader med 96 brønde på en mikroplade ryster ved 310 rpm. Der inkuberes ved 376 C i 20 til 24 timer for at udtrykke scFv'erne.
- For hver kultur plade fremstillet i trin 5.2, frakke en 96-brønd ELISA-plade med målantigenet.
- Fortynd oprenset målantigen til en passende koncentration (f.eks, 1 ug / ml til 4 ug / ml) i 1 × PBS for at gøre overtræksopløsningen. Foretag 5 ml coatingopløsning for hver 96-brønds plade.
BEMÆRK: Den passende koncentration afhænger af det specifikke antigen, der anvendes, og kan være nødvendigt at justere. - Der tilsættes 50 ul af coatingopløsningen til hver brønd i en 96-brønds høj-bindende klar polystyren ELISA plade. Bank let på pladen på benchtop overflade for at sikre, at hele overfladen af hver brønd coatet. Gentag for hver plade. Pladerne inkuberes ved 4 ° CO / N.
- Fortynd oprenset målantigen til en passende koncentration (f.eks, 1 ug / ml til 4 ug / ml) i 1 × PBS for at gøre overtræksopløsningen. Foretag 5 ml coatingopløsning for hver 96-brønds plade.
- Replikere kolonier fra de 96-brønds dyrkningsplader på agarplader.
- Placer en steril polystyren replikator i brøndene på en dyrkningsplade at indsamle en lille mængde liquid. hæve forsigtigt Replicator og overførsel til en 15 cm LB agarplade sådan, at alle tips rører pladen. Når væsken er overført, løft Replicator lige op. Gentag for hver kultur plade.
- Mærk agarplade med den korrekte orientering, således at resultaterne fra den sekundære skærm i 96-brønds plade kan matches med den korrekte replikeret koloni på pladen, hvis der ønskes yderligere karakterisering. Vokse ved 37 ° C i 15 til 18 timer, og derefter opbevares ved 4 ° C indtil brug.
- Udfør ELISA sekundære screening.
- Forbered blokerende opløsning ved at gøre 2% (vægt / volumen) tørmælk i 1 × PBS. Tøm coatingopløsningen fra ELISA-pladerne. Tilsættes 100 pi af blokeringsopløsningen til hver brønd. Inkuber ved stuetemperatur i mindst 2 timer eller blokere O / N ved 4 ° C.
- Forbered vaskepufferen ved tilsætning polysorbat 20 til en slutkoncentration på 0,05% i 1 × PBS. Gør 250 ml pr ELISA-plade.
- Tilsæt 20 gl af enkoncentreret cellelyse detergent til hver brønd i rundbundede dyrkningsplade og inkuberes kulturen plade på en mikropladeryster ved stuetemperatur i 15 til 20 min. Begynd lysis samtidig, at blokering af ELISA-pladerne er afsluttet, så der kan udføres lysis og vasketrinet 5.5.4 samtidigt.
- Tøm blokeringsopløsning fra ELISA-pladerne. Vask de blokerede ELISA plader fire gange med 200 pi vaskebuffer per brønd per vask. Tøm vaskepufferen fra brøndene.
- Transfer 50 pi fra hver brønd i cellelysis pladen til den tilsvarende brønd i ELISA-pladen ved hjælp af en ny spids til hver brønd. Inkubér ELISA-pladen ved stuetemperatur i 1 til 2 timer.
- Forbered antistof løsning til at detektere bundne scFv'er.
- Brug en peberrodsperoxidase (HRP) -konjugeret primære antistof der binder til FLAG-epitopen fusioneret til biblioteket scFv'er.
- Fortynd antistoffet til passende fortynding til brug i et ELISA (se leverandør & #39; s anbefalinger) i 2% (vægt / volumen) tørmælk i 0,05% polysorbat 20 i 1 × PBS. Forbered 5 ml for hver plade.
- Vask ELISA-pladerne fire gange med vaskebuffer som beskrevet i trin 5.5.4.
- Der tilsættes 50 ul af antistofopløsningen til hver brønd i ELISA-pladen. Der inkuberes i 1 til 2 timer ved stuetemperatur.
- Forbered HRP substrat ved opløsning o-phenylendiamin (OPD) -tabletter i destilleret H2O pr producentens protokol samtidig undgå lys. Forbered 20 ml pr ELISA-plade.
- Forbered 3 MH 2 SO 4 ved fortynding koncentreret H 2 SO 4 med destilleret H2O som nødvendigt. Forbered 5 ml pr ELISA-plade.
Forsigtig: H 2 SO 4 er en stærk syre. Sørg for at bære egnede personlige værnemidler. - Vask ELISA-pladerne fire gange med vaskebuffer, som beskrevet i trin 5.5.4.
- Inkuberes ELISA-plader med HRP substrate.
- Tilsæt 200 pi af HRP substrat til hver brønd. For at minimere lyseksponering, tilføje substratet til en ELISA-plade ad gangen, og wrap med aluminiumfolie før man går videre til den næste plade. Pladerne inkuberes i 30 til 60 minutter ved stuetemperatur i mørke.
- Efter de første 30 min, kontrollere pladerne i mørkfarvning af substratet, og der inkuberes længere, hvis nødvendigt for at visualisere udvikle farve.
- Der tilsættes 50 pi 3 MH 2 SO 4 til hver brønd for at standse reaktionen. Brug af en anden spids for hver brønd, bland opløsningen i brøndene ved forsigtigt at pipettere op og ned uden skumdannelse. For konsistens og for at forhindre mætning, tilsæt H 2 SO 4 hurtigt og omhyggeligt til alle ELISA-pladerne inden blanding løsningen for hver plade.
- Absorbansen af opløsningen i brøndene i hver plade ved 492 nm under anvendelse af en pladelæser.
- Analyser absorbansdata at identificere scFv- varianter at exhibdet lovende bindende signaler og karakterisere disse lovende scFv'er. Vælg scFv'er, der udviser absorbans-signaler højere end baggrundssignalet og højere end den gennemsnitlige signal på hver plade.
BEMÆRK: Absorbansen niveau vil afhænge af egenskaberne af antigenet og anti-FLAG-antistof anvendte, sammen med styrken af scFv-varianter, der blev isoleret i screeningen.
Representative Results
Den intracellulære protein foldemekanisme kvalitetskontrol af Tat-banen i E. coli begrænser transport over den indre cellemembran til proteiner, der er godt foldet i reducerende cytoplasmatiske miljø. Ved overekspression en fusion af en scFv til ssTorA signalsekvensen (signalsekvensen fra Tora protein, som er naturligt transporteres af Tat-banen 20), translokation gået i stå, hvilket resulterer i visning af scFv'er på den indre membran 19. Efter enzymatisk sprængning af den ydre membran, er de viste antistoffer til rådighed til screening for antigenbindende aktivitet. Evnen til at drage fordel af den Tat-banen for scFv display blev vist af Karlsson et al. 19 (figur 6). ScFv-antistoffer scFv13 og scFv13.R4 blev fusioneret til enten den native ssTorA sekvens eller en modificeret ssTorA der mangler arginin-argininrest pair anerkendt afTat-banen. scFv13.R4 blev manipuleret af Martineau et al. fra scFv13 gennem fire runder af målrettet evolution og er kendt at folde godt i cytoplasmaet 9. Dette scFv blev vist på den indre membran, men kun når det udtrykkes som en fusion til den native ssTorA signalsekvensen (figur 6). Modsat er scFv13 ikke godt foldet cytoplasmatisk 9, så det ikke vises godt på den indre membran, uanset signalsekvensen, hvortil det er fusioneret. Desuden, hvis de scFv'er blev udtrykt i celler, der manglede den TatC protein, en vital komponent af Tat maskiner 20,28, display blev ikke observeret, viser den vigtige forbindelse mellem indre membran display og Tat-banen. Disse resultater viser, at kun proteiner, der indeholder Tat signalpeptidet, og som er korrekt foldet i cytoplasmaet er vist på den indre membran, hvilket tillader transport gennem Tat-banen til at fungere som en skærm til intracellulær folding.

Figur 6. Påvisning af viste scFv'er på den indre membran. Flowcytometri-analyse blev udført for at detektere visningen af dårligt foldet scFv13 og godt foldede scFv13.R4 på den indre membran. scFv'er blev fusioneret til nativt ssTorA eller ssTorA (KK), hvor Arg-Arg pair i ssTorA sekvensen blev modificeret til Lys-Lys. Den C-terminale FLAG epitopmærker på scFv'er blev detekteret med et fluoresceinisothiocyanat (FITC) -konjugeret anti-FLAG-antistof. Celler uden den TatC-proteinet (ΔtatC) og ssTorA-scFv13 uden FLAG tag blev testet som kontroller. M angiver medianen fluorescensværdi. Gengivet fra henvisning 19 med tilladelse. Klik her for at se en større version af dette tal.
19. For at demonstrere dette, en fejlbehæftet PCR bibliotek baseret på scFv13, som har et lavt niveau af bindingsaffinitet for β-galactosidase, blev panned mod målantigenet β-galactosidase via display og panorering fremgangsmåde beskrevet i protokollen. scFv 1-4 blev isoleret efter en runde af mutagenese og panorering og udviste højere bindingsaffinitet til p-galactosidase end scFv13 (figur 7A) og en højere grad af cytoplasmatisk opløselighed (figur 7B).
Et nyt bibliotek, baseret på scFv 1-4, blev fremstillet ved anvendelse fejlbehæftet PCR, og panorering af denne anden generation bibliotek modβ-galactosidase blev udført ved anvendelse af en modifikation af den beskrevne protokol. Den panning mod β-galactosidase for anden runde af evolutionen blev udført i nærvær af oprenset, opløselig scFv 14 som en konkurrent for at forbedre sandsynligheden for at isolere kloner med højere affinitet end scFv 1-4. Efter denne anden runde af mutagenese og panorering, blev scFv 2-1 og scFv 2-3 isoleret under anvendelse af ELISA-baserede sekundær screening. Disse scFv'er ikke kun udviste højere bindingsaffinitet for β-galactosidase end scFv13, men også udviste bedre binding end den første runde klon scFv 1-4. scFv 2-1 udviste β-galactosidase binding sammenlignes med scFv13.R4 (figur 7A). scFv 2-3 viser også en yderligere stigning i cytoplasmatisk opløselighed sammenlignet med scFv 14, fremhæver den samtidige engineering af opløselighed og antigen-binding. Da affinitet og opløselig ekspression af scFv'er screenes for samtidigt, er det muligt, at en udvalgt scFv har Modbejder opløselighed, men høj binding eller omvendt. F.eks scFv 2-1 har lavere opløseligt ekspression end scFv 2-3, men den udviser højere bindingsaffinitet til p-galactosidase.

Figur 7. Target-binding og cytoplasmatisk ekspression af scFv-varianter isoleret under anvendelse indre membran display. (A) scFv'er blev udtrykt i cytoplasmaet i E. coli-celler (f.eks., uden ssTorA signalsekvensen) med en hexahistidin (6 × -His) tag og oprenset under anvendelse af nikkel-nitrilotrieddikesyre spin-søjler. Bindingen af de oprensede scFv'er til p-galactosidase blev målt med en ELISA. Oprensede scFv'er blev påsat β-galactosidase-overtrukne ELISA-plader, og de bundne scFv'er blev detekteret med et anti-6 × His antistof. Dataene er et gennemsnit af seks replikater, og fejlen bar viser standardfejl på middelværdien.(B) De opløselige og uopløselige fraktioner af cellelysater fra celler, der udtrykker scFv cytoplasmatisk blev analyseret ved en Western blot probet med et anti-6 × His antistof. Total proteinkoncentration blev anvendt til at normalisere lastning af prøverne. Genoptrykt (A) og tilpasset (B) fra henvisning 19 med tilladelse. Klik her for at se en større version af dette tal.
Discussion
Engineering antistoffer til cytoplasmatisk aktivitet er en vanskelig opgave på grund af den reducerende miljø i cytoplasmaet, hvilket vanskeliggør dannelsen af stabiliserende disulfidbindinger 6,7. Dette forårsager de fleste antistoffer, der skal cytoplasmatisk inaktive, medmindre de er konstrueret for stabilitet og opløselighed i cytoplasmaet, ud over at være udviklet til bindingsaffinitet. De eksisterende metoder til fagvisning, bakteriel overflade display, og gær overfladefremvisning metoder bruger alle den sekretoriske pathway 14-16 til udstilling af manipulerede antistoffer, men disse fremgangsmåder har ingen midler til at konstruere intracellulær foldning. Antistoffer manipuleret under anvendelse indre membran display har forbedret cytoplasmatisk stabilitet og opløselighed fordi foldningen kvalitetskontrol af Tat-banen forhindrer translokation af antistoffer, der er dårligt foldede og ustabile i cytoplasmaet. Denne metode forenkler den iterative proces i engineering intracellulære antistoffer til affinitet ennd opløselighed, da de to egenskaber er udviklet i et trin. Selv om denne fremgangsmåde er designet til ingeniørarbejde antistoffer med opløselighed i den reducerende intracellulære miljø, kan den også anvendes til engineering antistoffer til at fungere i ikke-reducerende betingelser, idet proteinerne manipuleret ved hjælp af denne metode opretholde deres foldning i oxiderende miljø af periplasma.
Selv om denne teknik simplificerer processen med engineering antistoffer med høj affinitet og høj cytoplasmatisk opløselighed, flere begrænsninger er vigtigt at overveje, når du bruger denne protokol. Ved analyse af de sekundære skærm ELISA-signaler til at identificere lovende scFv varianter, tærsklen for kræsne mellem potentielt interessante varianter og dem, der ikke udviser tilstrækkelig antigen-bindende sandsynligvis ikke vil være klart før efter flere kloner er blevet yderligere karakteriseret. Det er vigtigt at se efter forbedret binding løbet stamantistoffet; imidlertid,et unormalt højt signal kunne være tegn på aviditet 29 eller aggregation effekter 30, en udfordring, som ikke er unikt for den indre membran display screening tilgang. Et centralt begrænsning at huske, når du bruger denne protokol er den manglende evne til at inddrive sfæroplaster efter panorering, da de er ikke-levedygtige (upublicerede data). Dette nødvendiggør DNA forstærkning og transformation skridt til at inddrive de antistof-koder plasmider.
Flere kritiske trin i protokollen aktiverer samtidig engineering af foldning og binding af antistoffer. Til screening skal lykkes, må den scFv-bibliotek, der screenes udtrykkes som en fusion til ssTorA signalpeptid. Uden denne sekvens, vil antistoffer ikke rettet mod Tat-banen og vil således ikke translokeres til periplasmaet 19. Desuden er det bydende nødvendigt, at en C-terminal epitop-mærke fusioneres til antistofferne at muliggøre detektion af de viste antistoffer i skraldespandending assays. Det er klart, E. coli stamme anvendes til at udtrykke scFv'er skal også have den nødvendige Tat-banen maskiner, men dette er sandt for de almindeligt anvendte E. coli-stammer.
Ændringer til denne protokol er muligt at forbedre dets potentiale til at isolere antistoffer med de ønskede egenskaber. En subtraktiv panorering trin kan være afsluttet før panorering mod målantigenet at nedbryder scFv bibliotek af ikke-ønskede bestanddele. Biblioteket sfæroplaster kan inkuberes med magnetiske perler overtrukket med BCCP alene eller overtrukket med en ikke-ønsket protein, og sfæroplasterne som binder til disse perler kan kasseres, før screening af de resterende ubundne sfæroplaster for binding til det ønskede mål. Som nævnt i Repræsentative resultater, en fremgangsmåde forbedre affiniteten af et isoleret scFv skal omfatte et opløseligt konkurrent i panning reaktion at konkurrere med de scFv'er vises på sfæroplasterne. Fordi den opløselige competitor er et oprenset protein, intet DNA er amplificeret fra det, så kun sekvenser af scFv'er vises på sfæroplasterne vil blive inddrevet i PCR-reaktionen. Derudover kan denne fremgangsmåde udvides til manipulering andre typer antistoffer eller til ikke-antistof-bindende proteiner.
E. coli indre-membran display er en kraftfuld platform for engineering antistoffer med høj affinitet og høje niveauer af intracellulær opløselighed. Denne fremgangsmåde er særligt egnet til effektiv engineering af antistoffer bestemt til at fungere i det intracellulære miljø. Disse intracellulære antistoffer undersøges allerede som potentielle lægemidler på en række områder, herunder neurodegenerative sygdomme, kræft og virusinfektioner 31. Denne teknik vil gøre det muligt mere udbredt anvendelse af intracellulære antistoffer som redskaber til forskning og medicin på disse områder og alle andre områder, hvor studere en protein-target in situ ønskes.
Materials
| Name | Company | Catalog Number | Comments |
| scFv library | Varies | A suitable scFv library should be obtained from a commercial or academic source. | |
| MC4100 E. coli cells | Coli Genetic Stock Center | 6152 | Cells need to be chemically competent or electrocompetent, depending on the selected transformation method. |
| Glycerol | Fisher Scientific | BP229-4 | |
| Difco dehydrated culture media LB Broth, Miller (Luria-Bertani) | BD | 244610 | |
| Chloramphenicol (Cm) | Fisher Scientific | BP904-100 | |
| Sodium chloride (NaCl) | Fisher Scientific | BP358-1 | |
| Potassium chloride (KCl) | Fisher Scientific | BP366-500 | |
| Sodium phosphate, dibasic (Na2HPO4) | Fisher Scientific | BP332-500 | |
| Potassium phosphate, monobasic (KH2PO4) | Fisher Scientific | BP362-500 | |
| Bovine serum albumin (BSA) | Fisher Scientific | BP9706-100 | |
| Sucrose | Fisher Scientific | BP220-1 | |
| Tris base | Fisher Scientific | BP1521 | |
| Ethylenediaminetetraacetic acid (EDTA), 0.5 M | Fisher Scientific | BP2482-500 | |
| Magnesium chloride (MgCl2) | Fisher Scientific | BP214-500 | |
| Lysozyme | Sigma Aldrich | L3790-10X1ML | |
| Vortex mixer | VWR | 97043-564 | |
| Bicine | Fisher Scientific | BP2646100 | |
| D-Biotin | Fisher Scientific | BP232-1 | |
| Isopropyl β-D-1-thiogalactopyranoside | Fisher Scientific | BP1755-1 | |
| BugBuster Master Mix (cell lysis detergent) | EMD Millipore | 71456 | |
| Vivaspin 2 MWCO, 3,000 daltons | GE Healthcare Sciences | 28932240 | |
| Target antigen | Varies | N/A | Purified target antigen may be purchased or produced/purified. |
| Dynabeads MyOne Streptavidin T1 | Invitrogen | 65601 | |
| Dynamag-2 magnet | Invitrogen | 12321D | |
| Tube rotator | VWR | 13916-822 | |
| PCR primers | IDT | N/A | Primer sequences are as described in the protocol. |
| 10x T4 DNA ligase reaction buffer | New England BioLabs | B0202S | |
| T4 Polynucelotide kinase (PNK) | New England BioLabs | M0201S | Make sure the T4 ligase buffer used in the primer phosphorylation reaction contains 1 mM ATP. |
| 5x Phusion HF buffer pack | New England BioLabs | B0518S | |
| Deoxynucleotide (dNTP) solution mix, 10 mM each dNTP | New England BioLabs | N0447L | |
| Phusion DNA polymerase | New England BioLabs | M0530S | Other high-fidelity polymerases may be used as an alternative, but the annealing temperature in Table 3 must be adjusted. |
| C1000 Touch thermal cycler with dual 48/48 fast reaction module | Bio-Rad | 185-1148 | |
| Agarose | Promega | V3121 | |
| SYBR Safe DNA gel stain | Invitrogen | S33102 | |
| Wizard SV gel and PCR clean-up system | Promega | A9281 | |
| T4 DNA ligase | New England BioLabs | M0202S | |
| Microdialysis membrane filter | EMD Millipore | VSWP04700 | |
| Agar | BD | 214030 | |
| 96-well polystyrene round-bottom cell culture plates | VWR | 10062-902 | |
| Costar general polystyrene assay plate lids | Corning | 3931 | |
| Microtitre plate shaker | VWR | 12620-926 | |
| Costar 96 well EIA/RIA Easy Wash clear flat bottom polystyrene high bind microplate | Corning | 3369 | |
| Bel-blotter polycarbonate 96-well replicating tool | Bel-Art Products | 378760002 | |
| Instant nonfat dry milk | Quality Biological | A614-1000 | |
| Tween 20 (polysorbate 20) | Fisher Scientific | BP337-500 | |
| PopCulture reagent (concentrated cell lysis detergent) | EMD Millipore | 71092-3 | |
| Monoclonal ANTI-FLAG M2-Peroxidase(HRP) antibody produced in mouse | Sigma Aldrich | A8592 | |
| SigmaFast OPD | Sigma Aldrich | P9187-50SET | |
| Sulfuric acid (H2SO4), 10 N solution | Fisher Scientific | SA200-1 | |
| Reynolds Wrap aluminum foil | VWR | 89079-075 | |
| BioTek Epoch microplate spectrophotometer | Fisher Scientific | 11120570 |
References
- Biocca, S., Pierandrei-Amaldi, P., Campioni, N., Cattaneo, A. Intracellular immunization with cytosolic recombinant antibodies. Biotechnology (NY). 12 (4), 396-399 (1994).
- Chen, S. Y., Bagley, J., Marasco, W. A. Intracellular antibodies as a new class of therapeutic molecules for gene-therapy. Hum. Gene Ther. 5 (5), 595-601 (2008).
- Gargano, N., Biocca, S., Bradbury, A., Cattaneo, A. Human recombinant antibody fragments neutralizing human immunodeficiency virus type 1 reverse transcriptase provide an experimental basis for the structural classification of the DNA polymerase family. J Virol. 70 (11), 7706-7712 (1996).
- Mhashilkar, A. M., et al. Inhibition of HIV-1 Tat-mediated LTR transactivation and HIV-1 infection by anti-Tat single chain intrabodies. Embo J. 14 (7), 1542-1551 (1995).
- Strube, R. W., Chen, S. Y. Characterization of anti-cyclin E single-chain Fv antibodies and intrabodies in breast cancer cells: enhanced intracellular stability of novel sFv-F-c intrabodies. J. Immunol. Meth. 263 (1-2), 149-197 (2002).
- Mössner, E., Koch, H., Plückthun, A. Fast selection of antibodies without antigen purification: adaptation of the protein fragment complementation assay to select antigen-antibody pairs. J. Mol. Biol. 308 (2), 115-122 (2001).
- Wörn, A., et al. Correlation between in vitro stability and in vivo performance of anti-GCN4 intrabodies as cytoplasmic inhibitors. J Biol Chem. 275 (4), 2795-2803 (2000).
- Knappik, A., Plückthun, A. Engineered turns of a recombinant antibody improve its in vivo folding. Protein Eng. 8 (1), 81-89 (1995).
- Martineau, P., Jones, P., Winter, G. Expression of an antibody fragment at high levels in the bacterial cytoplasm. J Mol Biol. 280 (1), 117-127 (1998).
- Steipe, B., Schiller, B., Plückthun, A., Steinbacher, S. Sequence statistics reliably predict stabilizing mutations in a protein domain. J. Mol. Biol. 240 (3), 188-192 (1994).
- Daugherty, P. S. Protein engineering with bacterial display. Curr Opin Struct Biol. 17 (4), 474-480 (2007).
- Lener, M., et al. Diverting a protein from its cellular location by intracellular antibodies. Eur. J. Biochem. 267 (4), 1196-1205 (2000).
- Lynch, S. M., Zhou, C., Messer, A. An scFv intrabody against the nonamyloid component of α-synuclein reduces intracellular aggregation and toxicity. J. Mol. Biol. 377 (1), 136-147 (2008).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr. Opin. Struc. Biol. 17 (4), 467-473 (2007).
- Kieke, M. C., et al. Selection of functional T cell receptor mutants from a yeast surface-display library. Proc. Natl. Acad. Sci. USA. 96 (10), 5651-5656 (1999).
- Steiner, D., Forrer, P., Stumpp, M. T., Pluckthun, A. Signal sequences directing cotranslational translocation expand the range of proteins amenable to phage display. Nat. Biotechnol. 24, 823-831 (2006).
- Pugsley, A. P. The complete general secretory pathway in gram-negative bacteria. Microbiol. Rev. 57 (1), 50-108 (1993).
- Rapoza, M. P., Webster, R. E. The filamentous bacteriophage assembly proteins require the bacterial SecA protein for correct localization to the membrane. J. Bacteriol. 175 (6), 1856-1859 (1993).
- Karlsson, A. J., et al. Engineering antibody fitness and function using membrane-anchored display of correctly folded proteins. J. Molec. Biol. 416 (1), 94-107 (2012).
- DeLisa, M. P., Tullman, D., Georgiou, G. Folding quality control in the export of proteins by the bacterial twin-arginine translocation pathway. Proc Natl Acad Sci U S A. 100 (10), 6115-6120 (2003).
- Fisher, A. C., Kim, W., DeLisa, M. P. Genetic selection for protein solubility enabled by the folding quality control feature of the twin-arginine translocation pathway. Protein Sci. 15 (3), 449-458 (2006).
- Maynard, J., Georgiou, G. Antibody engineering. Annu Rev Biomed Eng. 2, 339-376 (2000).
- Green, M. R., Sambrook, J. Molecular Cloning: A Laboratory Manual. 1, Fourth, Cold Spring Harbor Laboratory Press. (2012).
- Milo, R., Jorgensen, P., Moran, U., Weber, G., Springer, M. BioNumbers--the database of key numbers in molecular and cell biology. Nucleic Acids Res. 38, D750-D753 (2010).
- Hermanson, G. T. Bioconjugate Techniques. , Third, Elsevier/Academic Press. (2013).
- Tayapiwatana, C., Chotpadiwetkul, R., Kasinrerk, W. A novel approach using streptavidin magnetic bead-sorted in vivo biotinylated survivin for monoclonal antibody production. J Immunol Methods. 317 (1-2), 1-11 (2006).
- Zhu, G., Song, L., Lippard, S. J. Visualizing inhibition of nucleosome mobility and transcription by cisplatin-DNA interstrand crosslinks in live mammalian cells. Cancer Res. 73 (14), 4451-4460 (2013).
- Bogsch, E. G., et al. An essential component of a novel bacterial protein export system with homologues in plastids and mitochondria. J. Biol. Chem. 273, 18003-18006 (1998).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng. Des. Sel. 28 (10), 339-350 (2015).
- Garber, K. Bispecific antibodies rise again. Nat. Rev. Drug. Discov. 13 (11), 799-801 (2014).
- Marschall, A. L., Dübel, S., Böldicke, T. Specific in vivo knockdown of protein function by intrabodies. MAbs. 7 (6), 1010-1035 (2015).



