Summary
Para testar o efeito de um quimiocina no recrutamento do macrófago in vivo, a hibridação in situ da montagem inteira foi usada para detectar a expressão ectópica do quimiocina, e a imunomarcação foi usada para etiquetar macrófagos. A imagem latente viva foi usada para a observação tempo real da migração do macrófago.
Abstract
Zebrafish são amplamente utilizados na pesquisa básica e biomédica. Muitas linhas transgênicas de zebrafish estão atualmente disponíveis para rotular vários tipos de células. Devido ao corpo embrionário transparente de zebrafish, é conveniente para nós estudar o efeito de uma quimiocina sobre o comportamento de um determinado tipo de células in vivo. Aqui nós fornecemos um fluxo de trabalho para investigar a função de uma quimiocina na migração de macrófago in vivo. Nós construímos um plasmídeo tecido-específico do superexpressão para sobre-expressão Il-34 e injetamos o plasmídeo em embriões transgênicas da fase da um-pilha dos peixes cujos os macrófagos foram etiquetados especificamente por uma proteína fluorescente. Nós usamos então a hibridação in situ fluorescente da montagem inteira e a imunocoloração para detectar o teste padrão da expressão do quimiocina e o número ou a posição dos macrófagos. Os embriões de WT injetados foram levantados para gerar uma linha transgênica estável. Por fim, utilizou-se a imagem latente ao vivo confocal para observar diretamente o comportamento dos macrófagos no peixe transgênico estável para estudar a função da IL-34 sobre o macrófago in vivo.
Introduction
O zebrafish é um pequeno peixe tropical de água doce de ossos duros originado na Índia. A respeito da conservação genética, o zebrafish tem uma similaridade de 87% ao ser humano1. Pode fornecer-nos introspecções em assuntos relacionados do ser humano estudando o Regulamento do gene, a função da proteína e o comportamento da pilha tais como a migração, proliferação et.al no zebrafish. O embrião de zebrafish pode ser usado para observar o desenvolvimento de embriões adiantados em estágios diferentes após ter inibindo o pigmento. Enquanto isso, leva apenas três meses para que o zebrafish se desenvolva em maturidade sexual, então o zebrafish pode produzir centenas de ovos a cada 4 dias. O mini-tamanho, melhoramento simples, capacidade reprodutiva forte, estas vantagens faz a cultura do zebrafish muito espaço-economia, conducente à cultura em grande escala. O rato modelo tradicional de mamíferos tem um custo de manutenção mais elevado do que o zebrafish, limitando conseqüentemente a escala da elevação do rato. No aspecto do desenvolvimento embrionário precoce, o embrião de camundongo é difícil de observar em condições de viver devido às características do desenvolvimento embrionário do camundongo no útero materno. Pelo contrário, os embriões de zebrafish desenvolvem-se externamente e são transparentes, por isso são fáceis de observar um microscópio. Além disso, o zebrafish é muito fácil construir uma variedade de linhas transgênicas para a pesquisa relacionada da função do gene. Atualmente, várias linhas de zebrafish transgênicos estão disponíveis para rotular diferentes tipos de células. É muito conveniente agora para construir linhas transgênicas para sobre-expressão quimiocinas em locais específicos e estudar a função de quimiocinas no comportamento celular em zebrafish.
Aqui, fornecemos um fluxo de trabalho para a utilização da linha de zebrafish transgênica para investigar a função do Il-34 no comportamento dos macrófago in vivo2,3,4,5,6,7. Primeiramente, nós construímos um plasmídeo de superexpressão fígado-específico do gene il34 e injetamos o plasmídeo em embriões de peixes do estágio TG da um-pilha (MPEG1: GFP) que etiquetaram especificamente os macrófagos pela proteína fluorescente GFP. Então, nós usamos a hibridação in situ fluorescente da montagem inteira e a imunocoloração para detectar o teste padrão da expressão il34 e o número ou a posição dos macrófagos. Os embriões de WT injetados foram levantados para gerar uma linha transgênica estável. Nessas etapas, estabelecemos e validamos a linha produtora de citocinas e avaliamos visualmente os efeitos que podem ser observados na distribuição de macrófago. Por fim, para investigar o comportamento dos macrófago em resposta à citocina, utilizou-se a imagem viva confocal para observar diretamente a migração de macrófago para confirmar a função de il34 na migração de macrófago in vivo.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Nota: Todas as amostras foram tratadas pela água do ovo de phenylthiourea (PTU) para inibir o pigmento.
1. geração de TG (fabp10a: il34) construções transgênicas e injeção de peixe
- Clone o 2,8 KB fabp10a promotor8 e as regiões de codificação Il-34 (ensdart 00000126460.3) de zebrafish no vetor pTol2 para gerar a construção fabp10a-il34. Injete os constructos em um estádio de células TG (MPEG1: GFP) e em embriões de peixes WT juntamente com a peptídeo mRNA. Levante os embriões injetados fabp10a-il34 do WT ao adulto9 e identifique o founder transgênicas pela hibridação in situ.
Nota: A injeção do construto Tol2 diretamente em outro transgênico pode ser problemática se a outra linha transgênica for feita com o mesmo sistema de Transposon. Uma prática geral seria fazer uma linha transgênica independente e, posteriormente, cruzar a nova linha com outra linha de repórter. Isso garante que não haverá efeitos da nova transgênese em um transgene previamente inserido.
2. fluorescente toda a montagem in situ hibridação (WISH) combinar com imunocoloração
- Fixação da amostra
- Colete embriões de injeção transitória ou linha transgênica estável IL-34 que cruzou com TG (MPEG1: GFP) em estágios desejados.
Nota: Para este caso, os embriões foram coletados em 4 d pós-fertilização (DPF). (Se necessário) Retire o Chorion por seringa. - Fixar os embriões com paraformaldeído 4% (PFA) durante a noite a 4 ° c ou 2 h à temperatura ambiente (RT) (cerca de 25 ° c).
- Lave os embriões com tampão fosfato salina mais Tween 20 (PBST) 3x 5 min.
- Desidrata os embriões separadamente com 50% de metanol em PBST (50% metanol/PBST) e 100% de metanol, 1x 5 min cada. Em seguida, mude para o metanol 100% fresco e armazene a-20 ° c (pelo menos 2 h).
Nota: O protocolo pode ser pausado aqui.
- Colete embriões de injeção transitória ou linha transgênica estável IL-34 que cruzou com TG (MPEG1: GFP) em estágios desejados.
- Hibridação da sonda (dia I)
- Rehidratar os embriões nas etapas anteriores com 50% de metanol em PBST (50% metanol/PBST), em seguida, lave com PBST 3x 5 min.
- Digerir os embriões com proteinase K em PBST em RT (concentração final: 10 μg/mL; 1:2000 em PBST).
Nota: O tempo da digestão depende do estágio dos embriões: menos do que a fertilização do borne de 36 h (HPF), nenhuma necessidade; 36 HPF-2 DPF embrião, 3-5 min; 2-3 DPF embrião, 10 min; 3-4 DPF embrião, 15 min; 4-5 DPF embrião, 15-20 min; 5-6 DPF embrião, 20-27 min; > 6 DPF embrião, 25-30 min em RT (cerca de 25 ° c). - Descarte a solução de digestão e realize a fixação novamente com 4% de PFA, por 20 min em RT.
- Lave os embriões com PBST 2x 10 min.
- Descarte o PBST, realize a pré-hibridação com amortecedor aquecido da hibridação (amortecedor do HB) em 65 ° c por 5 minutos, recicl o amortecedor do HB no tubo original.
- Realize a pré-hibridação com um novo tampão de HB aquecido a 65 ° c, pelo menos, 1 h.
- Pré-aqueça a sonda9 (para este caso foi uma sonda il34 , 1 ng/ml) a 65 ° c, pelo menos, 10 min. Em seguida, recicle o tampão HB no tubo original. Realize a hibridação com a sonda pré-aquecida a 65 ° c durante a noite.
- Tratamento de anticorpos (dia II)
- Pré-aqueça o 50% formamide/2x citrato de sódio fisiológico mais Tween 20 (SSCT), 2x SSCT, 0,2 x SSCT a 65 ° c.
- Recicle a sonda no tubo original e guarde a sonda a-20 ° c.
- Lave os embriões separadamente com 50% de formamida/2x SSCT; 2x SSCT; 0.2 x SSCT, 3x 20 min ou 2x 30 min cada a 65 ° c.
- Lave os embriões com PBST 3x 5 min.
- Bloqueie as amostras com 600 μL de tampão de bloqueio (5% de soro bovino fetal filtrado (FBS) em PBST) durante 1 h na RT.
- Adicionar 400 μL de solução de anticorpos anti-Digoxigenina-HRP (1:1.000-1:2000 em tampão de bloqueio) e incubar os embriões a 4 ° c durante a noite. Se os sinais são fracos, use 1:500 diluição do anticorpo.
- Coloração e anticorpo primário incubador (dia III)
- Retire o anticorpo; Lave os embriões com PBST, 6x 20 min em RT.
- Enxague a amostra com 30 μL de diluente de amplificação 1x Plus durante 5 min em RT.
- Descarte o diluente introduzindo a pipetagem; diluir a solução de Fluorophore Tyramide (cyanine 3 Plus reagente de amplificação (Cy3) ou o reagente de amplificação de cyanine 5 Plus (Cy5), para este caso Cy3 foi utilizado) 1:50 em 1x Plus diluente de amplificação para fazer a Fluorophore Tyramide solução de trabalho. Prepare 50-100 μL de solução de trabalho para cada amostra.
- Incubar a amostra no Fluorophore Tyramide solução de trabalho para 5-15 min no escuro em RT. Se os sinais são fracos, estender o tempo de incubação para 30 min.
- Pare a reacção alterando a solução de trabalho com PBST e examine os sinais.
- Lave os embriões com PBST 3x 10 min em RT.
- Incubar a amostra com anticorpo primário a 4 ° c durante a noite. Para este caso, use o anticorpo goat-anti-GFP como o anticorpo preliminar.
- Coloração do anticorpo secundário (dia IV)
- Lave os embriões com PBST por 4x 30 min.
- Incubar os embriões com anticorpo secundário a 4 ° c durante a noite. Para este caso, use o anticorpo de Alexa 488-anti-cabra como o anticorpo secundário.
- Tirar fotos (dia V)
- Lave os embriões com PBST 3x 10 min em RT.
- Armazene os embriões em 70% de glicerol em escuro a 4 ° c durante a noite ou-20 ° c por mais tempo.
3. imagens ao vivo
-
Seleção de amostra
Nota: use a imagem ao vivo para observar diretamente se os macrófagos de TG (fabp10a: Il34; fabp10a: DSRED; MPEG1: GFP) peixes migrariam para o fígado a indução de Il-34 durante 3-3,5 DPF. Aqui a linha transgênica TG (Fabp10a-dsred) é usada para rotular a região hepática e torná-la visível, para facilitar a localização do fígado e para determinar se os macrófagos migram para o fígado. Antes da imagem latente, use um microscópio de fluorescência para selecionar os embriões positivos dobro de DsRed e de GFP. -
Montagem dos peixes
- Use um banho do metal para aquecer 1 mL do agarose de derretimento baixo de 1% a acima de 90 ° c para derretê-la completamente.
- Depois que o baixo agarose de derretimento é refrigerado à temperatura de corpo, adicione 50 μL do tricaine de 0,2%, e misture uniformemente o tricaine com o agarose.
- Mova os embriões anestesiados para um pequeno prato montado com um slide de cobertura na parte inferior, retire a água circundante, solte lentamente o baixo agarose de fusão sobre os embriões, cuidadosamente definir a posição do peixe antes que o agarose é solidificada, manter a área do fígado perto de a corrediça da tampa na parte inferior do prato.
- Depois que o baixo agarose de derretimento solidified, cubra-o com cuidado com uma outra camada de agarose para reforçá-la.
- Coloc o prato na tabela confocal do portador do microscópio, cubra os peixes com a solução E210 com tricaine e comece a imagem latente.
-
Operação do software do microscópio confocal
- Abra o software do preto 2,3 do Zen , instale a bancada viva da pilha na tabela do portador do microscópio.
- Clique em Localizar | Incubação | Temperatura para ajustar a temperatura a 29 ° c.
- Coloque o prato no centro da bancada de célula viva, cubra o peixe com a solução E210 com tricaína.
- Clique no menu aquisição , selecione o modo de digitalização e os lasers necessários no menu configuração inteligente e, em seguida, selecione Z-Stack e Position.
- Clique no menu Designer de experimento , selecione habilitar experimento de vários blocos, no primeiro bloco, para localizar a amostra a baixa ampliação e, em seguida, alterne para a alta ampliação, deixe a área observada no centro do campo visual.
- Ajuste a posição e a informação da Z-pilha, selecione a intensidade apropriada do laser, as camadas da exploração e a velocidade da imagem latente.
- Crie um novo bloco e repita os passos acima. Depois de configurar todos os blocos, defina o número apropriado de loops e inicie a gravação (Figura 1).
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
As etapas envolvidas no protocolo de zebrafish são ilustradas na Figura 2. Em primeiro lugar, geramos o construto pBLK-fabp10a-il34-SV40 no qual il34 foi conduzido pelo promotor fabp10a (Figura 2). O construto foi microinjetado em embriões de zebrafish de fase TG (MPEG1: GFP) de uma célula que pode rotular macrófagos com embriões de GFP e WT que foram levantados para adultos para gerar linha estável transgênica (Figura 2). A expressão de il34 foi analisada pela hibridação in situ da fluorescência da montagem inteira (Figura 2 e Figura 3). Os macrófagos rotulados por GFP foram analisados por imunocoloração (Figura 2 e Figura 3). Nós usamos a imagem latente viva para observar diretamente se os macrófagos migrariam no fígado a indução il34 durante 3-3.5 DPF (Figura 2, Figura 4, filme suplementar 1 e filme suplementar 2) .
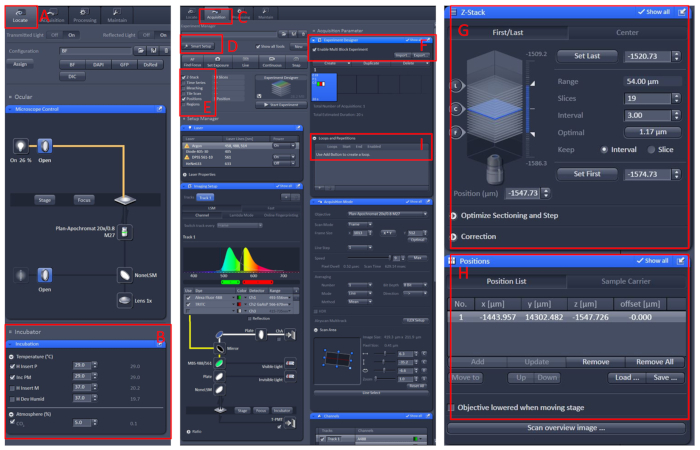
Figura 1: operação do software da imagem latente viva do microscópio confocal. Abra o software Zen black 2,3 , instale a bancada de célula viva na mesa do portador do microscópio e clique em Localizar (a) | Incubação | Temperatura (B) para ajustar a temperatura a 29 ° c. Coloque o prato no centro da bancada de célula viva, cubra o peixe com a solução E210 com tricaína. Depois de tudo isso, clique no menu aquisição (C), selecione o modo de digitalização necessário e os lasers no menu configuração inteligente (D) e, em seguida, selecione Z-Stack e Position (e). Por fim, clique no menu Designer de experimento (F), selecione a opção Habilitar experimento multibloco, no primeiro bloco, para localizar a amostra a baixa ampliação e, em seguida, alterne para a alta ampliação, deixe a área observada no centro do campo visual, definir a posição e Z-Stack (G e H) informações, selecione a intensidade do laser apropriado, as camadas de digitalização e velocidade de imagem. Crie um novo bloco e repita as etapas acima. Depois de configurar todos os blocos, defina o número apropriado de loops (I) e iniciar a gravação. Por favor clique aqui para ver uma versão maior desta figura.

Figura 2: um fluxo de trabalho para investigar a função de uma quimiocina na migração de macrófago in vivo. Nós construímos um plasmídeo do superexpressão do tecido-específico (fígado) para sobre-expressão Il-34 e injetamos o plasmídeo em uns embriões transgênicas do peixe da fase da um-pilha cujos os macrófagos fossem etiquetados especificamente por uma proteína fluorescente (TG: (MPEG1: GFP )). Os embriões de WT injetados foram levantados para gerar uma linha transgênica estável. Nós usamos então a hibridação in situ fluorescente da montagem inteira e a imunocoloração para detectar o teste padrão da expressão de gene e o número ou a posição dos macrófagos dos embriões injetados transientes ou da linha estável embriões (4 DPF). Por fim, utilizou-se a imagem latente ao vivo confocal para observar diretamente o comportamento dos macrófagos no peixe transgênico estável (3-3,5 DPF) para estudar a função de IL-34 em macroages in vivo. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3: desejo fluorescente combinar com imunocoloração. Este número foi modificado de Jiang et al.11. Um total de 1,8 nL (30 NG/μL) da construção pBLK-fabp10a-il34-SV40 foi microinjetado em embriões de zebrafish de fase TG (MPEG1: GFP) de uma célula. (A) desejo de expressão de il34 (vermelho) e coloração de anticorpos de montagem inteira de expressão de GFP (verde) em 4 embriões de DPF (6x). O retrato inteiro do corpo do peixe é compo de duas imagens separadas tomadas por confocal e costuradas junto em Photoshop. Os Insets são a ampliação elevada (20x) das regiões encaixotadas correspondentes (áreas pontilhadas laranja). (B) análise quantitativa de números de células de macrófago no fígado de embriões injetados e construto (mostrados na área pontilhada branca) e na região da cauda (aproximadamente entre os 13 e 17 somite, mostrados entre duas linhas pontilhadas brancas). Os dados foram analisados pelo teste U de Mann Whitney, * * p < 0, 1 comparado ao controle. n = 5, 5 para o 4 DPF injetado e controle de peixes. Barras: 200 μm (linha branca); 50 μm (linha amarela). (C) desejo da expressão de il34 e do anticorpo da inteiro-montagem que mancha da expressão do GFP em 4 DPF linha estável embrião (6x). O retrato inteiro do corpo do peixe é compo de duas imagens separadas tomadas por confocal e costuradas junto em Photoshop. Os Insets são a ampliação elevada (20x) das regiões encaixotadas correspondentes (áreas pontilhadas laranja). (D) análise quantitativa de números de células de macrófago em TG (MPEG1: GFP) e TG (fabp10a: il34; MPEG1: GFP) embriões ' fígado (mostrado na área pontilhada branca) e região da cauda (aproximadamente entre o 13º e 17 somite, mostrada entre duas linhas brancas pontilhadas). Por favor clique aqui para ver uma versão maior desta figura.

Figura 4: a imagem latente viva confocal para observar diretamente o comportamento do macrófago no peixe transgênicas estável. Este número foi modificado de Jiang et al.11. Os micrographs da imagem latente viva mostram o processo de um macrófago (verde, etiquetado por setas brancas) que passam pelo fígado (vermelho) dentro de 28 minutos no peixe do controle (a) e no processo de um macrófago (verde, etiquetado por setas brancas) que migram no fígado (vermelho) dentro de 28 min em IL-34 sobreexpressando peixes (B). Barras de escala = 40 μm (linha branca). Por favor clique aqui para ver uma versão maior desta figura.

Complementar filme 1: imagens ao vivo mostrando o processo de macrófagos (verde, rotulado por setas brancas) migrando para o fígado (vermelho) dentro de 2 h em Il-34 sobreexpressar peixes. Barras de escala = 20 μm (linha branca). Este filme foi republicado a partir de Jiang et al.11. Por favor clique aqui para ver este vídeo. (Clique com o botão direito do mouse para baixar.)

Complementar filme 2: imagens ao vivo mostrando o processo de macrófagos (verde, rotulado por setas brancas) vagando ao redor do fígado (vermelho) dentro de 2 h em peixes de controle. Barras de escala = 20 μm (linha branca). Este filme foi republicado a partir de Jiang et al.11. Por favor clique aqui para ver este vídeo. (Clique com o botão direito do mouse para baixar.)
Subscription Required. Please recommend JoVE to your librarian.
Discussion
O protocolo descrito aqui nos permite investigar a função de uma quimiocina sobre o comportamento do macróagein vivo e o procedimento requer algum conhecimento técnico. Em síntese, existem várias etapas críticas para evitar complicações no protocolo: 1) selecionar uma linha transgênica adequada que mostre um sinal transgênico específico e forte para rotular a célula de interesse; 2) selecionar um tecido apropriado que seja acessível para a imagem latente e o overexpression do gene transgênicas; 3) faça uma ponta de prova sensível e específica do RNA; 4) Selecione uma janela de tempo de observação apropriada para capturar com precisão o comportamento da célula.
No procedimento da hibridação in situ da fluorescência da montagem inteira combinada com a imunocoloração, a ponta de prova do RNA usada para detectar a expressão de gene deve ser sensível e o sinal precisa de ser forte bastante. A fim capturar a função do gene no comportamento da pilha, uma série de pontos do tempo deve ser testada. Por exemplo, ao observar o efeito do il34 sobre a migração de macrófagos, embora o promotor fabp10a tenha começado a expressar em 2-3 DPF, a acumulação de macrófago no fígado não era óbvia naquele momento. É somente por 4 DPF que o enriquecimento do macrófago no fígado se torna aparente. Além, após a hibridação in situ, a intensidade do sinal da imunomarcação subseqüente será afetada. Por exemplo, comparando com o GFP, DsRed é difícil de colorir na imunofluorescência que mancha após a hibridação in situ, provavelmente por causa das estruturas diferentes da proteína. De um modo geral, a intensidade do sinal de imunocoloração após a hibridação in situ da fluorescência da montagem inteira seria menos do que aquela da única imunomarcação.
Na etapa de imagem ao vivo com o microscópio confocal, é necessário manter a amostra próxima à parte inferior do prato. Quando os embriões flutuam em agarose, a distância de trabalho do objetivo pode ser insuficiente, também, a agarose entre o objetivo e a amostra afetaria a qualidade da imagem. Adicionalmente, o número de amostras para a imagem latente em cada vez deve ser ajustado corretamente. Um deve certificar-se de que o intervalo de tempo entre duas varreduras de cada peixe não seria demasiado longo perder os detalhes do comportamento da pilha. Portanto, este método não é adequado para rastreamento de células que se movem rapidamente em tecidos grossos.
Em conclusão, este protocolo pode ser usado para observar a função dos quimiocinas no comportamento de uma variedade de pilhas tais como macrófagos, neutrófilos, e T-Cells. Aqui, nósusamos Il-34, um ligante recentemente identificado da função do CSF-1R nos quimiotaxia6,7, comoum quimiocina expressado ectópica para induzir a migração dos macrófagos. A maioria dos modelos experimentais existentes de quimiotaxia celular baseiam-se em experimentos de células in vitro, mas os experimentos in vitro às vezes são muito simples para modelar o ambiente complexo in vivo. Além disso, é difícil de imagem a quimio-atração abilityin vivo quando simplesmente olhar para a situação in vitro. Este método utilizou as vantagens específicas do zebrafish para a observação direta do comportamento da pilha que é difícil para ratos. O método atual permitiu-nos de testar rapidamente as funções do quimiocina em comportamentos da pilha dentro de diversos dias e fazer a zebrafish um modelo poderoso para estudar a biologia molecular e da pilha.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Os autores não têm nada a revelar.
Acknowledgments
Agradecemos ao Dr. Jingrong Peng por compartilhar a linha transgênica TG (fabp10a: DsRed) ; Dr. Zilong Wen por compartilhar as linhas transgênicas TG (MPEG1: GFP) ; Dr. Koichi Kawakami para fornecer o vetor pTol2. Este trabalho foi apoiado pela Fundação Nacional de ciências naturais da China (31771594), Guangdong ciência e tecnologia projetos plano (2019A030317001) e os fundos de investigação fundamentais para as universidades centrais (D2191450).
Materials
| Name | Company | Catalog Number | Comments |
| Antibody | |||
| Alexa 488-Anti-Goat antibody | Invitrogen | A11055 | |
| Anti-Digoxigenin-HRP | perkinelmer | NEF832001EA | |
| Goat-Anti-GFP antibody | Abcam | ab6658 | |
| Reagent | |||
| CaCl2· 2H2O | Sigma | 21097 | |
| Cyanine 3 Plus Amplification Reagent | perkinelmer | NEL745001KT | |
| E2 solution | 15 mM NaCl +0.5 mM KCl +1.0 mM MgSO4+150 µM KH2PO4 + 50 µM Na2HPO4 +1.0 mM CaCl2 + 0.7 mM NaHCO3 | ||
| Fetal Bovine Serum (FBS) | Life | 10099-133 | |
| Formamide | Diamond | A100314 | |
| Glycerol | Sigma | V900860 | |
| Heparin sodium | Sigma | H3149 | |
| Hybridization buffer(HB) | 50% formamide+ 5×SSC+9 mM sodium citrate+50 μg/ml heparin sodium+ 500 μg/mL tRNA+ 0.1% Tween20 | ||
| KCl | Sigma | P5405 | |
| KH2PO4 | Sigma | P5655 | |
| Low melting agarose | Sigma | A9414 | |
| Methanol | GHTECH | 1.17112.023 | |
| Methylene blue | Sigma | M9140 | |
| MgSO4 | Sigma | M2643 | |
| Na2HPO4 | Sigma | S5136 | |
| NaCl | Sigma | S5886 | |
| NaHCO3 | Sigma | S5761 | |
| Paraformaldehyde (PFA) | Sigma | 158127 | Suspend 16 g of PFA in 400 ml of 1x PBS, heat at 60 °C to dissolve about 30 min. This solution can be prepared in advance and stored at -4 °C. Caution. Manipulate with mask. |
| 10×PBS | 14.2 g Na2HPO4+80 g NaCl+2 g KCl+ 2.4 g KH2PO4 in 1L ddH2O | ||
| Phenylthiourea (PTU) | Sigma | P7629 | |
| 1×Plus Amplification Diluent | perkinelmer | NEL745001KT | |
| Proteinase K | Fermentas | E00492 | |
| 20×Saline sodium citrate(SSC) | 175.3 g NaCl+ 88.2 g sodium citrate in 1 L ddH2O, PH 7.0 | ||
| Sodium citrate | Sigma | A5040 | |
| Tricaine | Sigma | E10521 | |
| tRNA | Sigma | R6625 | |
| Tween20 | Sigma | P2287 | |
| Plasmid | |||
| pBLK-fabp10a-il34-sv40 | For Tg (fab10a:il34) transgenic line generation | ||
| pBSK-il34 | For il34 probe preparation | ||
| Fish | |||
| Tg (mpeg1: GFP) | Label macrophages with GFP | ||
| Tg (fabp10a: DsRed) | Label liver cells with DsRed | ||
| Tg (fab10a:il34) | Over-expression IL-34 in liver cells |
References
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Wang, Y., et al. IL-34 is a tissue-restricted ligand of CSF1R required for the development of Langerhans cells and microglia. Nature Immunology. 13 (8), 753-760 (2012).
- Lin, H., et al. Discovery of a cytokine and its receptor by functional screening of the extracellular proteome. Science. 320 (5877), 807-811 (2008).
- Wei, S., et al. Functional overlap but differential expression of CSF-1 and IL-34 in their CSF-1 receptor-mediated regulation of myeloid cells. Journal of leukocyte biology. 88 (3), 495-505 (2010).
- Etienne, D., Foucher, S. B. L. P., Norbert Ifrah, P. G. Y. D. IL-34 Induces the Differentiation of Human Monocytes into Immunosuppressive Macrophages. Antagonistic Effects of GM-CSF and IFNc. PLoS One. 8 (2), e56045 (2013).
- Segaliny, A. I., et al. Syndecan-1 regulates the biological activities of interleukin-34. Biochimica et Biophysica Acta. 1853 (5), 1010-1021 (2015).
- Zhou, S. L., et al. miR-28-5p-IL-34-macrophage feedback loop modulates hepatocellular carcinoma metastasis. Hepatology. 63 (5), 1560-1575 (2016).
- Gordon, J. I., et al. Tissue specific expression and developmental regulation of two genes coding for rat fatty acid binding proteins. Journal of Biological Chemistry. 260 (4), 1995-1998 (1985).
- Westerfield, M. The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio rerio). , University of Oregon. Eugene, OR. (2000).
- Nüsslein-Volhard, C., Dahm, R. Zebrafish: a practical approach. , Oxford University Press. New York. (2002).
- Jiang, Y., Chen, J., Yen, K., Xu, J. Ectopically Expressed IL-34 Can Efficiently Induce Macrophage Migration to the Liver in Zebrafish. Zebrafish. 16 (2), 165-170 (2019).



