Summary
Dit is een methode om "scarless" recombinant vaccinia virussen te genereren met behulp van host-range selectie en visuele identificatie van recombinante virussen.
Abstract
Vaccinia virus (VACV) was instrumenteel in de uitroeiing van variola virus (VARV), de veroorzaker van pokken, uit de natuur. Sinds het eerste gebruik als vaccin is VACV ontwikkeld als vector voor therapeutische vaccins en als oncolytisch virus. Deze toepassingen maken gebruik van VACV's gemakkelijk gemanipuleerde genoom en brede host bereik als een uitstekend platform om recombinante virussen te genereren met een verscheidenheid aan therapeutische toepassingen. Er zijn verschillende methoden ontwikkeld om recombinant VACV te genereren, waaronder markerselectiemethoden en tijdelijke dominante selectie. Hier presenteren we een verfijning van een selectiemethode voor het hostbereik in combinatie met visuele identificatie van recombinante virussen. Onze methode maakt gebruik van selectieve druk gegenereerd door de gastheer antivirale eiwit kinase R (PKR) in combinatie met een fluorescerend fusie-gen uitdrukken mCherry-tagged E3L, een van de twee VACV PKR antagonisten. De cassette, inclusief het gen van belang en de mCherry-E3L fusie wordt geflankeerd door sequenties afgeleid van het VACV genoom. Tussen het gen van belang en mCherry-E3L is een kleiner gebied dat identiek is aan de eerste ~ 150 nucleotiden van de 3 'arm, ter bevordering van homologe recombinatie en verlies van de mCherry-E3L gen na selectie. We tonen aan dat deze methode een efficiënte, naadloze generatie rVACV mogelijk maakt in verschillende celtypen zonder dat er medicijnselectie of uitgebreide screening op gemuteerde virussen nodig is.
Introduction
Vaccinia virus (VACV) was instrumenteel voor de eerste succesvolle uitroeiing van een menselijke ziekteverwekker, variola virus (VARV), uit de natuur. Sinds de uitroeiing van het variolavirus zijn poxvirussen, waaronder VACV, nog steeds nuttige therapeutische virussen voor zowel de menselijke als de diergeneeskunde. Zo is een vaccin tegen rabiës door VACV zeer effectief geweest bij het voorkomen van overdracht van sylvatische rabiës in Europa1 en de Verenigde Staten2. Meer recent hebben recombinante poxvirussen die een verscheidenheid aan anti-tumormoleculen uitdrukken (bijvoorbeeld single-chain antilichamen of menselijke erythropoietine) bemoedigend succes gezien als oncolytische middelen3,4,5. VACV is bijzonder aantrekkelijk als vector omdat het gemakkelijk vatbaar is voor genetische manipulatie, beschikt over een breed scala aan gastheer, en het is stabiel onder een verscheidenheid van voorwaarden, waardoor gemakkelijk transport en vaccin levensvatbaarheid in het veld6,7. Terwijl meerdere technieken zijn ontwikkeld om recombinant VACV genereren voor laboratoriumexperimenten en vaccin generatie, huidige strategieën om deze virussen te genereren hebben opmerkelijke beperkingen.
Vanwege het nut van VACV zijn er meerdere strategieën ontwikkeld om recombinante virussen te genereren. De eerste strategie maakt gebruik van homologe recombinatie om een cassette met het transgene en een selecteerbaar markergen zoals een antibioticaresistentiegen te introduceren. De cassette wordt geflankeerd door twee ~ 500 nucleotiden (nt) of grotere armen die het gen naar een specifieke plaats in het virale genoom leiden, die vervolgens stabiel wordt geïntegreerd door dubbele crossover gebeurtenissen8,9,10. Deze strategie is snel en efficiënt; het resulteert echter in extra genetisch materiaal in de vorm van het markergen dat onverwachte effecten kan veroorzaken. Bovendien is er een praktische bovengrens aan het aantal transgenen dat kan worden ingevoerd beperkt door het aantal unieke selecteerbare markers beschikbaar. Voorbijgaande dominante selectie (TDS) strategieën hebben dit probleem aangepakt door het vergemakkelijken van de generatie van "littekenloze" recombinant virussen11. Met behulp van deze strategie worden een plasmide met een gemuteerd VACV-gen en een selecteerbaar markergen geïntegreerd in het virale genoom, maar zonder extra flankerend VACV-DNA. Deze aanpak resulteert in een tijdelijke integratie van het gehele plasmideen en duplicatie van het VACV-gen als gevolg van integratie door één cross-overgebeurtenis. Dit tussenproduct is stabiel zolang het onder selectiedruk wordt gehandhaafd, waardoor verrijking van deze constructie mogelijk is. Wanneer de selectie wordt verwijderd, maakt de VACV-duplicatie een tweede crossover-gebeurtenis mogelijk die resulteert in het verwijderen van het plasmilaatpunt en de daaropvolgende vorming van het wilde type (wt) of recombinantvirus in een verhouding van ongeveer 50:50. Terwijl TDS recombinante virussen genereert zonder de stabiele introductie van vreemd DNA, moeten meerdere virusklonen worden gescreend op de verwachte mutatie door sequencing analyse, een potentieel tijdrovende en kostbare stap.
Hier presenteren we een benadering voor het genereren van recombinante pokkenvirussen die de beste aspecten van elk van deze benaderingen combineren, vergelijkbaar met een benadering die is beschreven voor de replicatie incompetente gemodificeerde vaccinia Ankara12,13,14. Deze strategie combineert visuele en gastheer bereik selectie om snel recombinante virussen te genereren door dubbele crossover gebeurtenissen, en vervolgens elimineren van de selecteerbare marker gen door homologe recombinatie. Deze aanpak maakt de snelle generatie mutanten bemiddeld door homologe recombinatie, met de "littekenloze" aard van TDS benaderingen, terwijl niet vereist een latere screening stap om wilde type en mutant virussen te onderscheiden. Onze methode maakt ook gebruik van gastheer bereik selectie in plaats van antibiotica selectie, waardoor het risico van chemisch geïnduceerde fenotypische veranderingen in de cellijn. Voor deze aanpak hebben we ervoor gekozen om de gastheer antivirale eiwit kinase R (PKR) te gebruiken als het selectieve middel om recombinant VACV te genereren. PKR wordt uitgedrukt als een inactieve monomeer in de meeste celtypen15. Na het binden van dubbelstrengs RNA (dsRNA) bij de N-terminal dsRNA-bindende domeinen, dimerizes PKR en wordt autophosphorylated16. Deze actieve vorm van PKR-fosforylaatse de alfa-subeenheid van de eukaryotische initiatiefactor 2 (eIF2) remt uiteindelijk de levering van initiator methionyl-tRNA aan het rinoom, waardoor intracellulaire vertaling wordt voorkomen en de replicatie van veel virusfamilies17,18in grote lijnen wordt geremd .
In reactie op de brede en krachtige antivirale activiteit van PKR, veel virussen hebben ontwikkeld ten minste een strategie om PKR activering te voorkomen. De meeste poxvirussen drukken twee PKR antagonisten uit, gecodeerd door de E3L en K3L genen in VACV, die PKR door twee verschillende mechanismen antagonize19. E3 voorkomt PKR homodimerisatie door binding van dubbelstrengs RNA20,21, terwijl K3 fungeert als een pseudosubstraatremmer door direct te binden aan geactiveerde PKR en daarmee de interactie met het substraat eIF2α22te remmen . Belangrijk is dat deze twee PKR-antagonisten pkr niet noodzakelijkerwijs van alle soorten remmen. Zo remde de K3 homolog van het schapenpokkenvirus PKR sterk van schapen, terwijl de schapenpokken E3 homolog geen aanzienlijke PKR-remming23,24. In deze studie presenteren we een methode om PKR-gemedieerde selectieve druk te gebruiken in combinatie met fluorescentieselectie om een VACV-recombinant te genereren die is verwijderd voor E3L en K3L (VC-R4), die niet kunnen repliceren in PKR-competente cellen afkomstig van verschillende soorten. Dit recombinant virus biedt een uitstekende achtergrond voor snelle generatie van recombinante virussen uitdrukken genen onder controle van de inheemse E3L promotor.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Het genereren van de recombinatievector
- Ontwerp primers om de selectiecassette te genereren. Ontwerp elk individueel amplicon met overlappende sequenties met naburige versterkers en de vector om isothermale enzymatische assemblage van DNA-moleculen te vergemakkelijken, ook wel Gibson-assemblage genoemd, met behulp van een van de verschillende online primerontwerptools.
OPMERKING: Dit protocol kan ook worden voltooid met behulp van traditionele beperking endonuclease-gebaseerde klonen methoden. Ontwerp in dat geval primers met de juiste beperkingssites in plaats van met overlappende sequenties. - Met behulp van de primers die in stap 1.1 zijn ontworpen, versterken PCR de volgende elementen in volgorde van 5' tot 3' (Figuur 1): ~500 nucleotiden van het VACV genomische gebied 5' van E3L (5' arm), EGFP of het gen van belang, ~150 nucleotiden uit het genomische gebied VACV onmiddellijk 3' van E3L (korte 3' arm), een synthetische vroege/late poxvirus promotor25, de mCherry-E3L fusie gen, en ~ 500 nucleotiden uit de VACV genomic regio 3 ' van E3L met inbegrip van de korte 3 'arm (lange 3' arm).
- Voeg in een PCR-buis de reagentia in de volgende volgorde toe voor elke amplicon: 17 μL DNase vrij water; 1,2 μL van elke primer (initiële concentratie = 10 μM, eindconcentratie = 0,5 μM), 5 μL van 5x PCR-reactiebuffer, sjabloon-DNA (10 ng voor amplicons versterkt door plasmiden: EGFP en E/L promoter-mCherry-E3L cassette; 100 ng voor amplicons versterkt uit viraal genomisch DNA: 5' en 3' armen), en 0,5 μL DNA-polymerase. Pas het volume van het water dat wordt toegevoegd aan voor een eindreactievolume van 50 μL.
OPMERKING: De concentratie van sjabloon-DNA moet empirisch worden bepaald, maar we beginnen over het algemeen met 10 ng/reactie. - Plaats de buis(en) in een thermocycler en smelt het DNA bij 98 °C gedurende 30 s, en gebruik vervolgens 25 rondes van een 3-stappen-PCR-protocol: 98 °C voor 5 s, 55 °C voor 10 s en 72 °C gedurende 1 min.
LET OP: Bepaal de smelttemperatuur op basis van de door de fabrikant voorgestelde Tm voor elke primerset. Bepaal de juiste verlengingstijd op basis van de lengte van elke amplicon (1 minuut/kb).
- Voeg in een PCR-buis de reagentia in de volgende volgorde toe voor elke amplicon: 17 μL DNase vrij water; 1,2 μL van elke primer (initiële concentratie = 10 μM, eindconcentratie = 0,5 μM), 5 μL van 5x PCR-reactiebuffer, sjabloon-DNA (10 ng voor amplicons versterkt door plasmiden: EGFP en E/L promoter-mCherry-E3L cassette; 100 ng voor amplicons versterkt uit viraal genomisch DNA: 5' en 3' armen), en 0,5 μL DNA-polymerase. Pas het volume van het water dat wordt toegevoegd aan voor een eindreactievolume van 50 μL.
- Visualiseer de versterkingsproducten op een 1% agarose gel. Voeg 10 μL van elk DNA-product en 2 μL laadbuffer toe aan elke put en voer 1 uur lang op 8 V/cm.
- Gel zuiveren elke amplicon met behulp van een DNA-gel extractie kit en het protocol van de fabrikant. Maak de amplicons uit de kolom door 50 μL DNase vrij water toe te voegen en onmiddellijk te centrifugeren.
- Lineariseren van de pUC19 klonen vector met behulp van EcoRI endonuclease spijsvertering. Voeg aan een buis 1 μg pUC19, water toe aan een volume van 17 μL, 2 L reactiebuffer en 1 μL (20 eenheden) EcoRI. Incubeer bij 37 °C gedurende 1 uur.
- Visualiseer de versterkingsproducten op een 1% agarosegel die op 8 V/cm gedurende 1 uur loopt.
- Ligate alle van de individuele, gel gezuiverdamplicen en de gelineariseerde vector met behulp van een master mix kit.
- Voeg aan een PCR-buis 0,2 pmol van gelineariseerde pUC19 en elke amplicon (5' arm, EGFP, korte 3' arm, E/L promoter-mCherry-E3L cassette, 3'arm) toe. Voeg DNase vrij water toe aan een eindvolume van 10 μL en voeg vervolgens 10 μL DNA-assemblagemastermix toe. Incubeer monsters bij 50 °C gedurende 1 uur.
- Transformeer chemisch competente E. coli met 2 μL van het geassembleerde product vanaf stap 1.6, zoals eerder beschreven26,27. Plaat 100 μL van de getransformeerde cellen op LB agaroseplaten met 100 μg/mL ampicillin. De platen 's nachts uitbroeden bij 37 °C.
- Kies goed geïsoleerde kolonies en breng individuele kolonies over aan buizen met Luria bouillon met 100 μg/mL ampicillin. Incubeer de buizen 's nachts bij 37 °C tijdens het schudden bij 225 tpm.
- Isoleer de plasmiden van de nachtelijke cultuur met behulp van een plasmid miniprep kit. Controleer de concentratie en zuiverheid van het DNA met behulp van een spectrofotometer. Een A260/A280 verhouding tussen 1,8 en 2,0 is acceptabel.
- Dien de plasmiden in voor Sanger sequencing om te bepalen of het gewenste kloonproduct correct is. Bewaar het DNA op -20 °C.
2. Het recombinantvirus genereren
- Infecteer een confluentmonolayer van geschikte cellen met het virus dat moet worden gecombineerd bij een veelheid van infectie van 1.0 (MOI = 1.0) in een 6-well plaat. De geïnfecteerde cellen uitbroeden bij 37 °C en 5% CO2 gedurende 1 uur. Dan aanzuigen het medium en vervang het door verse DMEM. De geïnfecteerde cellen uitbroeden bij 37 °C en 5% CO2.
OPMERKING: Voor replicatie is bevoegde virussen zoals een vacciniavirus zonder K3L22, een cellijn zoals de Europese konijnenniercellijn RK13 (ATCC #CCL-37) of BSC-40 geschikt. Echter, voor replicatie gebrekkige virussen, zoals het virus beschreven in dit papier ontbreekt zowel PKR antagonisten E3L en K3L, een aanvulling cellijn uitdrukken van deze twee genen in trans of PKR knock-down of knock-out cellen zijn vereist. - Transfect de geïnfecteerde cellen met 500 ng van de vector gegenereerd en gevalideerd in stap 1.10 met behulp van een commercieel beschikbare transfection reagens volgens het protocol van de fabrikant. Incubeer de cellen bij 37 °C en 5% CO2 gedurende 48 uur.
OPMERKING: Als het gebruik van een vacciniavirus dat zowel E3L als K3L mist, zal pkr-gemedieerde selectieve druk de selectie van opnieuw gecombineerde virussen stimuleren en de expressie van het mCherry-E3L-fusieeiwit in deze cellen behouden. Indien gewenst moet het ook mogelijk zijn om PCR alleen de insert te versterken om te gebruiken voor transfection in plaats van het hele plasmide. - 48 uur na de infectie, oogst de geïnfecteerde monolayer. In sommige gevallen kunnen de cellen worden geoogst door pipetteren, maar als ze nog steeds goed worden nageleefd, oogst ze dan met een celschraper. Ontdooi de cellen drie keer en sonicate de lysaten vervolgens voor 15 s bij 50% amplitude. Bewaar deze lysate bij -80 °C tot het klaar is voor gebruik.
- Sergewijs verdunnen 10-voudig elysaat geoogst in stap 2.3 van 10-1 tot 10-6 door 120 μL van het lysaat toe te voegen aan 1080 μL DMEM (10-1), en vervolgens 120 μL van deze verdunning toe te voegen aan 1080 μL DMEM (10-2), en dit proces nog vier keer te herhalen. Voeg 1 mL van elke verdunning toe aan een individuele, confluentgoed van een PKR-cellenlijn, in dit geval RK13-cellen.
- De geïnfecteerde cellen uitbroeden bij 37 °C en 5% CO2 gedurende 1 uur. Zuig vervolgens het medium aan en vervang het door verse DMEM Incubatie de geïnfecteerde cellen bij 37 °C en 5% CO2.
- 24 tot 48 uur na infectie, recombinante virussen identificeren door fluorescentiemicroscopie. Plaques van recombinante virussen drukken rode fluorescentie als gevolg van integratie het mCherry-E3L fusiegen (Figuur 2). Als een virus zonder PKR-remmers in eerste instantie werd gebruikt, zullen alle plaques recombinantvirus bevatten.
- Plaque zuivert recombinante virussen drie keer op RK13 cellen. Na de laatste ronde van plaquezuivering moeten alle plaques rode fluorescentie uitdrukken.
- Infecteer een confluent6-well plaat van RK13 cellen uitdrukken van de VACV PKR remmers E3L en K3L (RK13+E3L +K3L cellen28) met de plaque gezuiverde rode fluorescerende virus van stap 2.6. Streven naar ongeveer 50-100 plaques per put.
OPMERKING: Deze cellen bieden de VACV PKR-antagonisten in trans en verlichten de PKR-gemedieerde selectieve druk om het mCherry-E3L-fusiegen te behouden, waardoor de "littekenloze" generatie van het recombinantvirus wordt bevorderd. - Identificeer ingestorte virussen door fluorescentiemicroscopie met behulp van een EVOS2-microscoop en een GFP-filterkubus (Excitatie: 470/22, Emissie: 525/50) en een RFP-filterkubus (Excitatie: 531/40, Emissie: 593/40).
OPMERKING: De frequentie waarmee het mCherry-E3L-fusiegen verloren gaat, bedraagt ongeveer 2,5% (Tabel 2). Als EGFP niet als markergen wordt opgenomen, zullen plaques van gemuteerde virussen die het mCherry-E3L-fusiegen hebben verloren, kleurloos zijn. - Plaque zuivert groen-only (VC-R4) of kleurloze plaques (E3L) drie keer op RK13+E3L+K3L cellen. Zorg ervoor dat er geen plaques rood fluoresceren.
- Bevestig het verlies van mCherry-E3L en de aanwezigheid van de verwachte mutatie door PCR en Sanger sequencing.
OPMERKING: Als het gen of de mutatie van belang geen PKR-remmende activiteit heeft, moeten recombinante virussen worden gekweekt op RK13+E3L+K3L-cellen of een gelijkwaardige PKR-geremde of PKR-ficiënte cellijn (figuur 3).
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
We gebruikten de procedure in figuur 1 om een VACV te genereren zonder zowel PKR-antagonisten E3L als K3L, door E3L te vervangen door EGFP in een virus dat al is verwijderd voor K3L (vP872). Figuur 2 toont rode fluorescerende plaques in PKR bevoegde RK13 cellen indicatief voor virale expressie van mCherry-E3L, evenals EGFP uitgedrukt in RK13 +E3L+K3L cellen die het verlies van E3L en ineenstorting van de mCherry-E3L selectie marker bevestigen. Figuur 3 bevestigt dat dit recombinantvirus, VC-R4, dat beide PKR-antagonisten niet kan repliceren in PKR-competente RK13-cellen, terwijl het bovenliggende virus, vP872 dat E3L uitdrukt, replicatiebevoegde is. Om te bevestigen dat dit onvermogen om te repliceren in RK13-cellen alleen te wijten was aan het verlies van E3L, hebben we EGFP in VC-R4 vervangen door E3L, om een terugkerend virus te genereren met behulp van hetzelfde selectieprotocol. Figuur 3 valideert ook dat dit revaliderende virus net zo efficiënt repliceert als vP872 in RK13-cellen. Interessant is dat kleurloze plaques in overeenstemming met de ineenstorting van de mCherry-E3L selectie marker werden geïdentificeerd voorafgaand aan de selectie in RK13 + E3 + K3 cellen die over het algemeen nodig zijn om "littekenloze" recombinanten te selecteren, waarschijnlijk vanwege de uitgebreide sequentie-identiteit tussen de mCherry-E3L recombinatie cassette en de E3L gen wordt ingevoegd in VC-R4. Daarom, om de efficiëntie van de recombinatie en de mate van instorting te bepalen hebben we ervoor gekozen om virussen te produceren die het poxvirus PKR antagonist K3L uitdrukken om het probleem van vroege ineenstorting te voorkomen23. Figuur 4 geeft het uiterlijk aan van kleurloze plaques (pijlpunten) na infectie van RK13+E3L+K3L-cellen. Tabel 1 toont de resultaten van drie onafhankelijke experimenten, waarbij gemiddeld 12,6% van de virionen recombinatie had ondergaan met de getransfecteerde plasmide, vergelijkbaar met eerder gerapporteerde frequenties29,30,31. Tabel 2 beschrijft de frequentie van kleurloze plaques ten opzichte van de totale plaques in RK13+E3L+K3L-cellen, waaruit de mate van instorting en verlies van de mCherry-E3L-selectiemarkering werd aangetoond met een frequentie van ongeveer 1,8%.
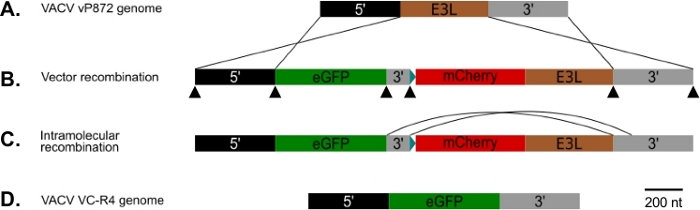
Figuur 1: Diagram van p837-GOI-mCherry-E3L, evenals de strategie voor host-range en visuele recombinatie. (A) 5' arm (zwart) en 3' arm (grijs) flankeren de E3L locus (bruin) in VACV. (B) In p837-GOI-mCherry-E3L flankeren deze armen een cassette met het gen van belang (GoI), in dit geval EGFP, (groen) gescheiden van een mCherry-E3L (rood) fusiegen onder controle van de synthetische vroege/late poxvirus promotor25 blauw) door een korte 3' (armgrijs). Deze externe armen rijden homologe recombinatie tussen VACV en de p837-GOI-mCherry-E3L. Zwarte pijlpunten geven de sites van de overlappende primers gebruikt om deze plasmide genereren door Gibson klonen. (C) Wanneer pkr selectieve druk wordt verwijderd, virussen die een intramoleculaire recombinatie tussen de korte en lange 3 'armen hebben ondergaan kunnen worden geselecteerd. (D) Resulterend in een virus (VC-R4) dat alleen het gen bevat dat van belang is in de E3L-locus. Klik hier om een grotere versie van dit cijfer te bekijken.

Figuur 2: Fluorescerende micrografen van (boven) een recombinant virusplaque 24 uur na recombinatie met p837-GOI-mCherry-E3L die zowel mCherry (links) als EGFP (rechts) uitdrukken in RK13-cellen. (Onder) Micrograaf van een recombinant virus plaque 48 uur na PKR-gemedieerde selectieve druk is verwijderd in RK13 ++ cellen, uitdrukken EGFP (rechts), maar niet mCherry (links). De schaalbalk geeft 650 μm aan voor alle panelen. Klik hier om een grotere versie van dit cijfer te bekijken.
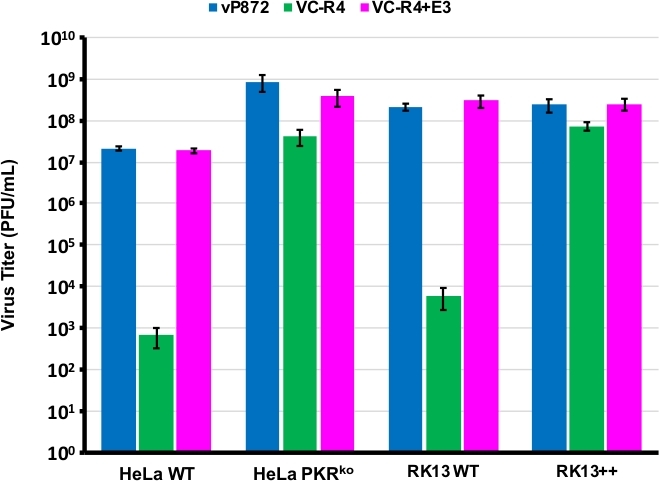
Figuur 3: VC-R4 kan niet repliceren in pkr-cellen. De aangegeven cellijnen waren geïnfecteerd met vP872 (blauw), VC-R4 (groen) of VC-R4+E3L (magenta) bij MOI = 0,1. 48 uur na infectie werden de geïnfecteerde cellen geoogst en getitered door seriële verdunning op RK13+E3L+K3L cellen. Titers worden gerapporteerd in PFU/mL, foutenbalken vertegenwoordigen de standaarddeviatie van drie replicerende experimenten. Klik hier om een grotere versie van dit cijfer te bekijken.

Figuur 4: Verlies van mCherry-E3L expressie in RK13+E3L+K3L cellen. Overlay van fluorescerende en fase contrast micrografen van VC-R4+K3L-mCherry-E3L geïnfecteerde RK13+E3L+K3L cellen. Drie plaques niet langer uitdrukken mCherry (cirkels) als gevolg van het instorten van de selectie cassette opbrengst VC-R4 + K3L. Klik hier om een grotere versie van dit cijfer te bekijken.
| Experiment 1 | Experiment 2 | Experiment 3 | |
| Rode plaquettes (RK13) | 30 | 11 | 18 |
| Totaal aantal plaques (RK13+E3L+K3L) | 225 | 64 | 249 |
| Recombinatiesnelheid | 13.30% | 17.20% | 7.20% |
Tabel 1: Recombinatie frequentie van VACV met de p837-K3L-mCherry-E3L plasmide.
| Experiment 1 | Experiment 2 | Experiment 3 | |
| Totaal aantal plaques (RK13+E3L+K3L) | 115 | 44 | 210 |
| Kleurloze plaques (RK13+E3L+K3L) | 3 | 1 | 1 |
| Recombinatiesnelheid | 2.60% | 2.30% | 0.50% |
Tabel 2: Frequentie van mCherry-E3L verlies van VC-R4+K3L-mCherry-E3L in RK13+E3+K3 cellen.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Hier presenteren we een variatie van een voorbijgaande marker selectie strategie 32 om recombinant vaccinia virussen te genereren zonder het behoud van vreemd DNA in de uiteindelijke recombinant virus. Onze strategie maakt gebruik van selectieve druk bemiddeld door de gastheer antivirale eiwit PKR in plaats van andere vormen van selectieve druk, zoals antibiotica. Het gebruik van gastheer antivirale genen elimineert de mogelijkheid van chemisch geïnduceerde fenotypische veranderingen in de cellen, of een verhoogd risico op mutatie als gevolg van selectie drugs. Bovendien is er, in tegenstelling tot bij de selectie van geneesmiddelen, geen vertragingsfase voor onze aanpak, omdat PKR constitutief wordt uitgedrukt in alle cellen. Secundaire visuele selectie op basis van mCherry expressie verbetert ook de specificiteit van deze methode door ervoor te zorgen dat alleen plaques uitdrukken van de transgene worden geplukt tijdens de eerste fase, en is even efficiënt als een negatieve selectieve marker tijdens het selecteren van volwassen recombinant virussen die het mCherry-E3L gen hebben verloren.
De meest kritieke stappen voor deze recombinatiestrategie zijn het genereren van de juiste recombinatievector en de juiste plaquezuivering om ervoor te zorgen dat het geselecteerde virus klonaal is. In dit artikel stellen we "Gibson assembly" voor om de recombinatie vector te genereren. Deze strategie is uiterst efficiënt en maakt het mogelijk om alle fragmenten die de recombinatievector in één dag omvatten, te assembleren. Echter, omdat de korte 3 'arm en de lange 3 'arm delen identieke sequenties, deze fragmenten hebben het potentieel om te worden samengevoegd tijdens het klonen reactie, en sommige vectoren kunnen niet de mCherry-E3L cassette bevatten. In onze ervaring is dit zeldzaam, maar het bevestigen van de structuur van de vector na het klonen is noodzakelijk. We hebben ook recombinatievectoren voor deze strategie gegenereerd met behulp van traditionele endonuclease- en ligasemethoden. Deze strategie vermijdt het hierboven beschreven probleem, maar kan arbeidsintensiever zijn. Plaque zuivering is over het algemeen eenvoudig en is in de eerste plaats afhankelijk van het gebruik van geschikte tolerante cellen voor de eerste recombinatie, PKR-bevoegde cellen voor de eerste plaque zuivering om ervoor te zorgen dat alleen recombinante virussen kunnen repliceren, en vervolgens tolerante cellen weer te vergemakkelijken intramoleculaire recombinatie en verlies van de selecteerbare marker. Veel aandacht voor cellijnen is daarom cruciaal voor de succesvolle en efficiënte toepassing van deze strategie.
In deze studie tonen we het gebruik van deze methode aan om een VACV recombinant te genereren die is verwijderd voor zowel PKR-antagonisten E3L als K3L en egfp onder controle van de E3L-promotor uit te drukken. In de toekomst zal dit virus dienen als een efficiënte achtergrond voor toekomstige recombinante virussen, omdat het niet in staat is om te repliceren in PKR bevoegde cellen. Daarom zal er een sterke PKR-gemedieerde selectieve druk zijn om de mCherry-E3L recombinatiecassette tot nakomelingenvirions te drijven, terwijl tegelijkertijd replicatie van niet-recombinantvirus wordt voorkomen. Bovendien is het verlies van EGFP door de opname van de recombinatiecassette een nuttige secundaire selectiemarker om ervoor te zorgen dat geplukte plaques niet co-geïnfecteerd zijn met een niet-recombinantvirus. We hebben de recombinatiepercentages waargenomen die overeenkomen met eerder gerapporteerde tarieven voor VACV, maar de visuele fluorescerende markers verhogen de efficiëntie van het genereren van recombinante virussen door ervoor te zorgen dat de kans dat de juiste recombinante virussen worden geselecteerd, toenemen. Onze observatie van kleurloze plaques na twee rondes van selectie op PKR-competente cellen, vermoedelijk te wijten aan de verhoogde lengte van identieke sequentie tussen E3L en de mCherry-E3L marker gen, suggereert dat de snelheid van mCherry-E3L verlies kan worden "afgestemd" door het verhogen of verminderen van de lengte van de 3 'korte arm. De primaire beperking van deze techniek is het gebruik van PKR als selectieve druk voor recombinanten. Het meest efficiënte gebruik van deze recombinatie strategie is het genereren van deze virussen op een achtergrond ontbreekt PKR antagonisten. Echter, de colorimetrische selectie marker maakt het mogelijk deze recombination strategie te worden gebruikt, zelfs zonder de selectie bemiddeld door PKR, gewoon door plaque zuiveren mCherry-expressing plaques. Hoewel het ontbreken van PKR-gemedieerde selectieve druk de efficiëntie van de eerste screeningstap zal verminderen, is het percentage mCherry dat plaques uitdrukt nog steeds hoog genoeg dat op kleur gebaseerde selectie levensvatbaar is. Zo kan deze methode worden gebruikt om bijna elk gen in het genoom van het poxvirus in te brengen.
Zoals blijkt uit het inbrengen van EGFP, kan met deze aanpak elk gen snel in de E3L-locus worden ingevoegd onder controle van de oorspronkelijke promotor, op voorwaarde dat PKR null-cellen of complimenterende cellijnen worden gebruikt voor downstream-experimenten als het transgeen geen PKR-antagonist is. Deze strategie, gecombineerd met het VC-R4-virus dat we hier rapporteren, voegt een nieuwe en krachtige methode toe om snel en betrouwbaar recombinantvaccinia-virussen te genereren met behulp van gastheer-gemedieerde selectieve druk en visuele identificatie van recombinanten vroeg in het proces.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
De auteurs verklaren geen concurrerende financiële belangen.
Acknowledgments
Dit project werd gefinancierd door de National Institutes of Health (AI114851) aan SR.
Materials
| Name | Company | Catalog Number | Comments |
| 2X-Q5 Master Mix | NEB | M0492L | High-fidelity polymerase used in PCR |
| Ampicillin | ThermoFisher Scientific | 11593027 | Bacterial selective agent |
| Disposable Cell Scrapers | ThermoFisher Scientific | 08-100-242 | Cell scraper to harvest infected cells |
| EVOS FL Auto 2 Cell imaging system | ThermoFisher Scientific | AMAFD2000 | Fluorescent microscope |
| EVOS Light Cube, GFP | ThermoFisher | AMEP4651 | GFP Cube |
| EVOS Light Cube, RFP | ThermoFisher | AMEP4652 | RFP Cube |
| GenJet | SignaGen Laboratories | SL100489 | Transfection reagent |
| Luria Bertani (LB) Broth | Gibco | 10855021 | Bacterial growth medium |
| Monarch DNA gel extraction kit | NEB | T1020L | Gel purification kit used to purify amplicons and linearized vectors |
| Monarch Plasmid Miniprep kit | NEB | T1010L | Miniprep kit ussed to purify plasmids |
| NanoDrop One | ThermoFisher Scientific | ND-ONE-W | Spectrophotometer used to measure RNA and DNA concentration |
| NEBuilder Master Mix | NEB | E2621L | Isothermal enzymatic assembly kit used to generate the recombination vector |
| Q500 Sonicator | Qsonica | Q500-110 | Sonicator for virus lysates |
| RK13 cells | ATCC | CCL-37 | Rabbit kidney cells |
| VWR Multiwell Cell Culture plates | VWR | 10062-892 | Cell culture plates |
References
- Brochier, B., et al. Large-scale eradication of rabies using recombinant vaccinia-rabies vaccine. Nature. 354 (6354), 520-522 (1991).
- Pastoret, P. P., Brochier, B. The development and use of a vaccinia-rabies recombinant oral vaccine for the control of wildlife rabies; a link between Jenner and Pasteur. Epidemiology and Infection. 116 (3), 235-240 (1996).
- Chan, W. M., McFadden, G. Oncolytic Poxviruses. Annual review of virology. 1 (1), 119-141 (2014).
- Nguyen, D. H., et al. Vaccinia virus-mediated expression of human erythropoietin in tumors enhances virotherapy and alleviates cancer-related anemia in mice. Molecular Therapy. 21 (11), 2054-2062 (2013).
- Frentzen, A., et al. Anti-VEGF single-chain antibody GLAF-1 encoded by oncolytic vaccinia virus significantly enhances antitumor therapy. Proceedings of the National Academy of Sciences of the United States of America. 106 (31), 12915-12920 (2009).
- Pastoret, P. P., Vanderplasschen, A. Poxviruses as vaccine vectors. Comparative Immunology, Microbiology and Infectious Diseases. 26 (5-6), 343-355 (2003).
- COLLIER, L. H. The development of a stable smallpox vaccine. The Journal of Hygiene. 53 (1), 76-101 (1955).
- Weir, J. P., Bajszár, G., Moss, B. Mapping of the vaccinia virus thymidine kinase gene by marker rescue and by cell-free translation of selected mRNA. Proceedings of the National Academy of Sciences of the United States of America. 79 (4), 1210-1214 (1982).
- Mackett, M., Smith, G. L., Moss, B. Vaccinia virus: a selectable eukaryotic cloning and expression vector. Proceedings of the National Academy of Sciences of the United States of America. 79 (23), 7415-7419 (1982).
- Nakano, E., Panicali, D., Paoletti, E. Molecular genetics of vaccinia virus: demonstration of marker rescue. Proceedings of the National Academy of Sciences of the United States of America. 79 (5), 1593-1596 (1982).
- Falkner, F. G., Moss, B. Transient dominant selection of recombinant vaccinia viruses. Journal of Virology. 64 (6), 3108-3111 (1990).
- Staib, C., Drexler, I., Ohlmann, M., Wintersperger, S., Erfle, V., Sutter, G. Transient Host Range Selection for Genetic Engineering of Modified Vaccinia Virus Ankara. BioTechniques. 28 (6), 1137-1148 (2000).
- Staib, C., Drexler, I., Sutter, G. Construction and Isolation of Recombinant MVA. Vaccinia Virus and Poxvirology. , 77-99 (2004).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Pfaller, C. K., Li, Z., George, C. X., Samuel, C. E. Protein kinase PKR and RNA adenosine deaminase ADAR1: New roles for old players as modulators of the interferon response. Current Opinion in Immunology. 23 (5), 573-582 (2011).
- Bevilacqua, P. C., George, C. X., Samuel, C. E., Cech, T. R. Binding of the protein kinase PKR to RNAs with secondary structure defects: Role of the tandem A - G mismatch and noncontigous helixes. Biochemistry. 37 (18), 6303-6316 (1998).
- Krishnamoorthy, T., Pavitt, G. D., Zhang, F., Dever, T. E., Hinnebusch, A. G. Tight Binding of the Phosphorylated Subunit of Initiation Factor 2 (eIF2) to the Regulatory Subunits of Guanine Nucleotide Exchange Factor eIF2B Is Required for Inhibition of Translation Initiation. Molecular and Cellular Biology. 21 (15), 5018-5030 (2001).
- Rothenburg, S., Georgiadis, M. M., Wek, R. C. Evolution of eIF2α kinases: Adapting translational control to diverse stresses. Evolution of the Protein Synthesis Machinery and Its Regulation. , 235-260 (2016).
- Bratke, K. A., McLysaght, A., Rothenburg, S. A survey of host range genes in poxvirus genomes. Infection, Genetics and Evolution. 14, 406-425 (2013).
- Chang, H. W., Watson, J. C., Jacobs, B. L. The E3L gene of vaccinia virus encodes an inhibitor of the interferon-induced, double-stranded RNA-dependent protein kinase. Proceedings of the National Academy of Sciences. 89 (11), 4825-4829 (1992).
- Romano, P. R., et al. Inhibition of double-stranded RNA-dependent protein kinase PKR by vaccinia virus E3: role of complex formation and the E3 N-terminal domain. Molecular and Cellular Biology. 18 (12), 7304-7316 (1998).
- Beattie, E., Tartaglia, J., Paoletti, E. Vaccinia virus-encoded eIF-2 alpha homolog abrogates the antiviral effect of interferon. Virology. 183 (1), 419-422 (1991).
- Park, C., Peng, C., Brennan, G., Rothenburg, S. Species-specific inhibition of antiviral protein kinase R by capripoxviruses and vaccinia virus. Annals of the New York Academy of Sciences. 1438 (1), 18-29 (2019).
- Rothenburg, S., Brennan, G. Species-Specific Host-Virus Interactions: Implications for Viral Host Range and Virulence. Trends in Microbiology. , (2019).
- Chakrabarti, S., Sisler, J. R., Moss, B. Compact, synthetic, vaccinia virus early/late promoter for protein expression. BioTechniques. 23 (6), 1094-1097 (1997).
- Chung, C. T., Niemela, S. L., Miller, R. H. One-step preparation of competent Escherichia coli: Transformation and storage of bacterial cells in the same solution (recombinant DNA). Biochemistry. 86, 2172-2175 (1989).
- Chung, C. T., Miller, R. H. Preparation and storage of competent Escherichia coli cells. Methods in Enzymology. 218, 621-627 (1993).
- Rahman, M. M., Liu, J., Chan, W. M., Rothenburg, S., McFadden, G. Myxoma Virus Protein M029 Is a Dual Function Immunomodulator that Inhibits PKR and Also Conscripts RHA/DHX9 to Promote Expanded Host Tropism and Viral Replication. PLOS Pathogens. 9 (7), 1003465 (2013).
- Evans, D. H., Stuart, D., McFadden, G. High levels of genetic recombination among cotransfected plasmid DNAs in poxvirus-infected mammalian cells. Journal of Virology. 62 (2), 367-375 (1988).
- Ball, L. A. High-frequency homologous recombination in vaccinia virus DNA. Journal of Virology. 61 (6), 1788-1795 (1987).
- Spyropoulos, D. D., Roberts, B. E., Panicali, D. L., Cohen, L. K. Delineation of the viral products of recombination in vaccinia virus-infected cells. Journal of Virology. 62 (3), 1046-1054 (1988).
- Liu, L., et al. Transient dominant host-range selection using Chinese hamster ovary cells to generate marker-free recombinant viral vectors from vaccinia virus. BioTechniques. 62 (4), 183-187 (2017).



