Transducción de fagos: Un método para transferir resistencia a ampicilina de un donante una E. coli receptora
English
Share
Overview
Fuente: Alexander S. Gold1, Tonya M. Colpitts1
1 Departamento de Microbiología, Escuela de Medicina de la Universidad de Boston, Laboratorios Nacionales de Enfermedades de Infecciones Emergentes, Boston, MA
La transducción es una forma de intercambio genético entre bacterias que utiliza bacteriófagos, o fagos, una clase de virus que infecta exclusivamente organismos procaarios. Esta forma de transferencia de ADN, de una bacteria a otra a través de un fago, fue descubierta en 1951 por Norton Zinder y Joshua Ledererg, quienes denominaron el proceso “transducción” (1). Los bacteriófagos fueron descubiertos por primera vez en 1915 por el bacteriólogo británico Frederick Twort, luego descubiertos de nuevo de forma independiente en 1917 por el microbiólogo franco-canadiense Felix d’Herelle (2). Desde entonces, la estructura y función de estos fagos se han caracterizado ampliamente (3), dividiendo estos fagos en dos clases. La primera de estas clases son los fagos líticos que tras la infección se multiplican dentro de la bacteria huésped, alterando el metabolismo bacteriano, lysing la célula, y liberando fago progenie (4). Como resultado de esta actividad antibacteriana y la creciente prevalencia de bacterias resistentes a los antibióticos, estos fagos líticos han demostrado ser útiles recientemente como tratamiento sustituto de los antibióticos. La segunda de estas clases son los fagos lisogénicos que pueden multiplicarse dentro del huésped a través del ciclo lítico o entrar en un estado de reposo en el que su genoma se integra en el del huésped (Figura 1), un proceso conocido como lisógeno, con la capacidad de fago producción que se inducirá en varias generaciones posteriores (4).
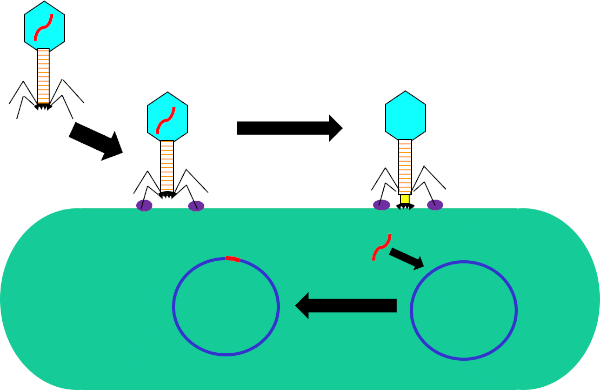
Figura 1: Infección de la célula huésped por bacteriófago. Adsorción por el fago a la pared celular bacteriana a través de interacciones entre las fibras de cola y el receptor (púrpura). Una vez en la superficie celular, el fago se une irreversiblemente a la célula bacteriana utilizando la placa base (negro) que se mueve a la pared celular por la vaina contráctea (amarillo). El genoma del fago (rojo) entra en la célula y se integra en el genoma de la célula huésped.
Mientras que la transducción bacteriana es un proceso natural, utilizando tecnología moderna ha sido manipulada para la transferencia de genes a bacterias en el entorno de laboratorio. Al insertar genes de interés en el genoma de un fago lisogénico, como el fago, uno es capaz de transferir estos genes a los genomas de las bacterias y, en consecuencia, expresarlos dentro de estas células. Mientras que otros métodos de transferencia de genes, como la transformación, utilizan un plásmido para la transferencia y expresión de genes, la inserción del genoma del fago en el de la bacteria receptora no sólo tiene el potencial de conferir nuevos rasgos a esta bacteria, sino que también permite mutaciones naturales y otros factores del entorno celular para alterar la función del gen transferido.
En comparación con otros métodos de transferencia de genes horizontales, como la conjugación, la transducción es bastante flexible en los criterios requeridos para las células donantes y receptoras. Cualquier elemento genético que pueda caber dentro del genoma del fago que se está utilizando puede transferirse de cualquier cepa de bacterias donantes a cualquier cepa de bacterias receptoras, siempre y cuando ambas sean permisivas para el fago, requiriendo la expresión de los receptores de fago necesarios en el superficies celulares. Una vez que este gen se mueve fuera del genoma del donante y se empaqueta en el fago, se puede transferir al receptor. Después de la transducción, es necesario seleccionar para las bacterias receptoras que contienen el gen de interés que deben seleccionarse. Esto podría hacerse mediante el uso de un marcador genético, como una etiqueta FLAG o una etiqueta de polihistidina, para marcar el gen de interés, o la función intrínseca del gen, en el caso de genes que codifican para la resistencia a los antibióticos. Además, la PCR podría utilizarse para confirmar aún más la transducción correcta. Mediante el uso de imprimaciones para una región dentro del gen de interés y la comparación de la señal con un control positivo, bacterias que tiene el gen de interés, y un control negativo, bacterias que se sometieron a los mismos pasos que la reacción de transducción sin fago. Si bien la transducción bacteriana es una herramienta útil en biología molecular, ha desarrollado y sigue desempeñando un papel importante en la evolución de las bacterias, particularmente en lo que respecta al reciente aumento de la resistencia a los antibióticos.
En este experimento, la transducción bacteriana se utilizó para transferir la codificación genética para la resistencia a la ampicilina antibiótica de la cepa W3110 de E.coli a la cepa J53 a través del bacteriófago P1 (5). Este experimento consistió en dos pasos principales. En primer lugar, la preparación de fago P1 que contiene el gen de resistencia a la ampicilina de la cepa del donante. En segundo lugar, la transferencia de este gen a la cepa receptora por transducción con el fago P1 (Figura 1). Una vez realizada, la transferencia exitosa del gen de resistencia a la ampicilina podría determinarse por qPCR (Figura 2). Si la transducción fuera exitosa, la cepa J53 de E. coli sería resistente a la ampicilina, y el gen que confiere esta resistencia es detectable por qPCR. De no tener éxito, no habría detección del gen de resistencia a la ampicilina y la ampicilina seguiría funcionando como un antibiótico eficaz contra la cepa J53.
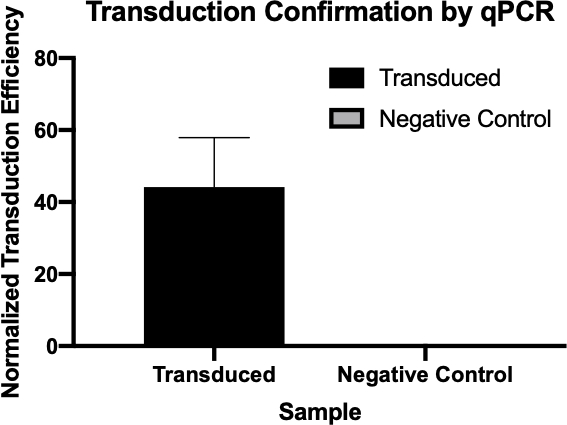
Figura 2: La confirmación de la transducción exitosa por qPCR. Al comparar los valores Cq detectados para el gen de interés de la reacción de transducción y la reacción de control negativa, y normalizar estos valores contra un gen de limpieza, se pudo confirmar que la transducción bacteriana fue exitosa.
Procedure
Applications and Summary
The transfer of genes to and from bacteria by bacteriophage, while a natural process, has proved extremely useful for a multitude of research purposes. While other methods of gene transfer such as transformation and conjugation are possible, transduction uniquely uses bacteriophages; not only allowing for gene integration into the host genome, but also for gene delivery to multiple bacteria that are not susceptible to other methods. This process, while especially useful in the laboratory, has also been used in the recently emerging field of gene therapy, more specifically in alternative gene therapy, a therapeutic strategy that utilizes bacteria to deliver therapeutics to target tissues, many of which are not susceptible to other delivery methods and have much clinical relevance (8,9).
References
- Lederberg J, Lederberg E.M., Zinder, N.D., et al. Recombination analysis of bacterial heredity. Cold Spring Harbor symposia Quantitative Biol. 1951;16:413-43.
- Duckworth DH. "Who Discovered Bacteriophage?". Bacteriology Reviews. 1976;40:793-802.
- Yap ML, Rossman, M.G. Structure and Function of Bacteriophage T4. Future Microbiol. 2014;9:1319-27.
- Sulakvelidze A, Alavidze, Z., Morris, J. G. Bacteriophage Therapy Antimicrobial Agents and Chemotherapy 2001;45(3):649-59.
- Moore S. Sauer:P1vir phage transduction 2010 [Available from: https://openwetware.org/wiki/Sauer:P1vir_phage_transduction].
- Kobayashi A, et al. Growth Phase-Dependent Expression of Drug Exporters in
- Escherichia coli and Its Contribution to Drug Tolerance. Journal of Bacteriology. 2006;188(16):5693-703.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.
- Pálffy R. et al. Bacteria in gene therapy: bactofection versus alternative gene therapy. Gene Ther. 2006 13:101-5.
- O'Neill JM, et al. Intestinal delivery of non-viral gene therapeutics: physiological barriers and preclinical models. Drug Discovery Today. 2011;16:203-2018.
Transcript
Bacteria can adapt quickly to a fast-changing environment by exchanging genetic material and one way they can do this is via transduction, the exchange of genetic material mediated by bacterial viruses. A bacteriophage, often abbreviated to phage, is a type of virus that infects bacteria by first attaching to the surface of the host and then injecting its DNA into the bacterial cell. It then degrades the host cell’s own DNA and replicates its viral genome, whilst hijacking the cell’s machinery to synthesize many copies of its proteins. These phage proteins then self-assemble and package the phage genomes to form multiple progeny. However, due to the low fidelity of the DNA packaging mechanism, occasionally, the phage packages fragments of bacterial DNA into the phage capsid. After inducing the lysis of the host, the phage progeny are released and, once such a phage infects another host cell, it transfers the DNA fragment of its previous host. This can then recombine and become permanently incorporated into the new host’s chromosome, thereby mediating gene transfer between the two bacteria.
To carry out phage transduction in the laboratory requires a donor strain that contains a gene of interest, a recipient strain that lacks it, a phage that can infect both the strains, and a method to select the transduced bacteria. In most cases, this will be a selective solid growth media that supports the growth of transduced bacteria but inhibits the growth of non-transduced ones. To begin, the donor strain that contains the gene of interest is cultured in a liquid growth medium. When all the bacteria are actively dividing in the log phase of their growth, the culture is inoculated with the target phage. After three to four hours of incubation, when nearly all the bacteria have lysed and released the phage particles, the donor phage lysate is inoculated into a freshly grown culture of the recipient bacterial strain. After a brief incubation of one hour, the culture should now contain a mixture of transduced and non-transduced bacterial cells and this is screened for the transduced cells by spreading a fraction of the suspension onto an appropriate selective solid growth media. Upon further incubation, the transduced cells should grow and multiply to yield visible colonies. These colonies can then be selected for further analysis using a variety of methods to further confirm successful transduction, such as colony PCR, DNA sequencing, or quantitative PCR.
Before starting the procedure, put on any appropriate personal protective equipment, including a lab coat and gloves. Next, sterilize the workspace with 70% ethanol and wipe down the surface.
After this, prepare three one-milliliter aliquots of LB salt solution. Now, prepare a donor strain culture by adding 100 microliters of E. coli to a 15 milliliter conical vial containing five milliliters of LB growth medium with 500 micrograms of ampicillin. Then, grow the culture overnight at 37 degrees Celsius with aeration and shaking at 220 rpm. The next day, wipe down the bench top with 70% ethanol before removing the culture from the shaking incubator. Next, dilute the overnight culture one to 100 by adding 10 microliters of donor strain to 990 microliters of fresh LB supplemented with salt solution.
Allow the bacterial dilution to grow at 37 degrees Celsius for two hours with aeration and shaking at 220 rpm. Once the cells have reached early log phase, remove the culture from the incubator, add 40 microliters of P1 phage to the culture and incubate again. Continue to monitor the cells for one to three hours until the culture has lysed. Next, add 50 to 100 microliters of chloroform to the lysate and mix by vortexing. Then, centrifuge the lysate to remove debris and transfer the supernatant to a fresh tube. Add a few drops of chloroform to the supernatant and store it at four degrees Celsius for no more than one day.
To begin the transduction procedure, obtain a one milliliter culture of recipient strain. Next, transfer 100 microliters of donor phage lysate into a 1.5 milliliter microcentrifuge tube and incubate it at 37 degrees Celsius with the cap open for 30 minutes to allow any remaining chloroform to evaporate. While the donor phage lysate incubates, pellet the recipient strain cells via gentle centrifugation. Discard the supernatant and resuspend the cell pellet in 300 microliters of fresh LB containing 100 millimolar magnesium sulfate and five millimolar calcium chloride.
Next, set up the transduction reaction by combining 100 microliters of the recipient strain and 100 microliters of the donor phage lysate in a microcentrifuge tube. Then, set up the negative control by combining 100 microliters of the recipient strain and 100 microliters of the LB with magnesium sulfate and calcium chloride. After incubation, add 200 microliters of one molar sodium citrate and one milliliter of LB to both tubes, and mix by gently pipetting up and down. Then, after the tubes have been incubated for an hour, gently pellet the cells via centrifugation.
After centrifuging, discard the supernatant and resuspend the pelleted cells in 100 microliters of LB with 100 millimolar sodium citrate. Vortex the solutions and pipette the entire transduced sample onto an LB agar plate with 1X ampicillin. Finally, pipette the entire volume of the negative control cell mixture onto an LB agar plate without ampicillin. After incubating the plates overnight at 37 degrees Celsius, use a sterile pipette tip to pick three to four colonies from the transduction plate and streak them onto a new LB agar plate containing 1X ampicillin and 100 microliters of one molar sodium citrate. Repeat this plating method for the negative control on another LB agar plate containing only 100 microliters of one molar sodium citrate. Then, incubate the plates at 37 degrees Celsius overnight to allow colonies free of phage to grow.
The next day, wipe down the bench top with 70% ethanol before removing your plates from the incubator. Using a sterile pipette tip, pick three colonies from the transduction plate and add them each to a separate tube containing five milliliters of LB media. Then, select three colonies from the negative control plate and add them to another tube containing five milliliters of LB media. Grow the cultures overnight at 37 degrees Celsius with aeration and shaking at 220 rpm. After sterilizing the bench top as previously demonstrated, use a DNA miniprep kit to isolate DNA from 4.5 milliliters of each culture according to the manufacturer’s instructions. Then, elute the DNA with 35 microliters of nuclease-free water and measure the resulting concentration by lab spectrophotometer. Finally, prepare glycerol stocks by adding the remaining 0.5 milliliters of both bacterial solutions to 0.5 milliliters of 100% glycerol.
To confirm transduction, first prepare two qPCR master mixes for 24 qPCR reactions. For the first master mix, add 150 microliters of qPCR buffer mix to a microcentrifuge tube and 12 microliters each of a forward and reverse primer designed to amplify the ampicillin resistance gene. Next, prepare a second qPCR master mix by adding 150 microliters of qPCR master mix to a microcentrifuge tube and then adding 12 microliters each of a forward primer and reverse primer designed to amplify a housekeeping gene.
For each qPCR reaction, combine 100 micrograms of experimental DNA from each reaction with 14.5 microliters of qPCR master mix. Now, prepare the remaining reactions as previously demonstrated. Transfer the reactions to a thermocycler preheated to 94 degrees Celsius and then initiate the program. Finally, use the cycle quantification, or Cq, values generated by qPCR to calculate the normalized transduction efficiency of the ampicillin resistance gene.
The cycle quantitation, or Cq, values for the genes of interest were tabulated for each of the negative controls and transduced samples. Low Cq values, typically below 29 cycles, like the transduced samples in this example indicate high amounts of the target sequence.
A housekeeping gene, also tabulated here, is used as a loading control to normalize the amount of DNA in each reaction and as a positive control to ensure the qPCR is working. Provided the same amounts of the housekeeping gene are loaded, it is found at relatively the same rate in each sample.
Next, to calculate the delta Cq value for each sample, subtract the Cq value of the housekeeping gene for each sample from the Cq value of its corresponding target gene. For example, the delta Cq of the first negative control is 13.54. Then, use this value to calculate the normalized transduction efficiency of each sample using the formula shown here. Finally, the average normalized transduction efficiency for each sample group can be calculated.
