Summary
蛋白质聚集诱发细胞氧化应激。本协议描述了一种通过流式细胞仪监测 amyloidogenic 蛋白的细胞内状态和与之相关的氧化应激的方法。该方法用于研究淀粉样β肽的可溶性和聚集易变性的行为。
Abstract
蛋白质错误折叠和聚集到淀粉样蛋白构象已经与一些神经退行性疾病的发病和进展有关。然而, 对于不溶性蛋白聚合体在体内如何发挥其毒性作用, 仍有很少的信息。简单的原核和真核模型有机体, 如细菌和酵母, 已大大有助于我们目前对细胞内淀粉样形成的机制, 聚集繁殖和毒性的理解。在本议定书中, 酵母的使用被描述为一个模型来解剖蛋白质团聚体的形成和它们对细胞氧化应激的影响之间的关系。该方法将 amyloidogenic 蛋白的胞内可溶性/聚集态检测与流式细胞术 (FC) 表达的细胞氧化损伤的量化相结合。这种方法简单、快速、定量。本研究通过将一大组淀粉样β肽变异体所引起的细胞氧化应激与它们各自的内在聚集倾向进行关联来说明该技术。
Introduction
Proteostasis 是细胞适应和衰老过程的基本决定因素。在细胞中, 蛋白质的稳态是由复杂的蛋白质质量控制网络维持的, 目的是通过伴侣和/或他们的靶向蛋白质错误折叠, 以确保正确的复性蛋白构象1 ,2,3,4,5。大量的研究为人类疾病的发生和发展与 proteostasis 的失败之间的联系提供了支持, 导致蛋白质错误折叠和聚集。例如, 蛋白质矿床的存在被认为是许多神经退行性疾病的病理特征, 如阿尔茨海默氏症、帕金森氏病、亨廷顿疾病6、7、8、prionogenic 疾病, 非退行性 amyloidoses9。有人建议, 早期寡聚和 protofibrillar 聚集反应是细胞毒性的主要诱导, 与其他蛋白质在拥挤的细胞环境中建立异常的相互作用10。此外, 蛋白质包裹体 (PI) 可以在细胞间传播, 传播其毒性效应11,12。因此, 可以说, PI 的形成实际上可能构成一种解毒机制, 它将危险的聚集物种的存在限制在细胞中的特定位置, 在那里它们可以被加工或积累而不会产生重大的副作用。13,14。
标准的体外生化方法为不同物种的聚集反应及其性质15,16提供了重要的见解。然而, 这些化验所使用的条件明显不同于细胞内发生的情况, 因此, 质疑它们的生理相关性。由于细胞通路的显著保护, 如蛋白质质量控制, 自噬, 或调节细胞氧化还原状态17,18之间的真核生物19,20,21 ,22,23, 萌芽酵母酿酒酵母 (酵母) 已成为一个特权简单的细胞模型研究蛋白质聚集的分子决定因素及其相关的细胞毒性影响在生物相关的环境24,25,26。
蛋白质聚集倾向是在主序列中固有编码的特征。因此, 可以根据对多肽27中聚集促进区域的效力的鉴定和评价, 预测淀粉样结构的形成。然而, 尽管生物信息学算法成功地预测蛋白质序列的体外聚集特性, 但它们仍远未预测这些倾向如何转化为体内的细胞毒性影响。以系统的方式解决给定蛋白质的聚合状态与其相关的细胞损伤之间的联系的研究可能有助于规避这一计算限制。这个连接在本研究中被处理, 利用一大组变异的淀粉样β肽 Aβ42不同只在单一的残余, 但显示连续的范围的聚集倾向在体内28。特别是, 本文介绍了一种基于 FC 的方法, 用于识别酵母细胞中聚集易感蛋白引起的氧化损伤的构象物种。该方法提供了许多优点, 如简单性、高通量能力和精确的定量测量。这种方法使人们有可能确认 PI 对氧化应激起着保护作用。
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1.酵母培养和蛋白质表达
注: Aβ变种表现出不同的相对聚集倾向, 由于突变在一个单一的残留在位置 19 (Phe19) 的 Aβ42肽 (图 1A)。这些肽变体被标记为绿色荧光蛋白 (GFP), 它充当聚合报告员 (图 1)29。
- 将 20 Aβ42-GFP 变种的质粒编码转化成酵母细胞, 具有 BY4741 的父母背景 (他的 3Δ1列伊2Δ0满足15Δ0 ura3 Δ0)30. 每个质粒编码的 Aβ42突变体融合到 GFP 由一个 GSSGSSG 链接器。质粒为收支处 (市建局), 包括可选标记 URA329。
- 制备无尿 (SC-市建局) 的合成完全培养基, 其成分为: 1.9 克/升酵母氮基, 无尿, 5 克/升硫酸铵, 900 毫升 ddH2O 结合100毫升 (瓦特/v) 葡萄糖。
- 将转化后的酵母细胞的一个菌落放入20毫升的 SC-市建局培养基中, 含2% 葡萄糖, 并在30摄氏度一夜之间以 210 rpm 的搅动生长文化。
- 第二天, 培养新的酵母细胞培养, 通过接种100µL 的隔夜文化成5毫升新鲜的 SC-市建局培养基。
- 在 OD590的 0.5, 离心的文化在 3000 x g 4 分钟, 丢弃上清, 并并用重悬细胞在相同数量的新鲜 SC-市建局培养基中含有2% 糖。
- 孵育在30°c 下搅动在210转每分钟30分钟。然后, 离心细胞在 3000 x g 4 分钟, 并丢弃上清。并用重悬新鲜的 SC-市建局培养基中含有2% 半乳糖的细胞, 诱导重组蛋白表达。
- 16 h 诱导蛋白在 SC-市建局培养基中含有2% 半乳糖后, 在无菌离心管中获得1毫升的诱导细胞, 离心在 3000 x g 处, 4 分钟。
注: 最相关的差异发生在诱导期为16小时的诱导酵母细胞表达 Aβ42-GFP 变种可以通过荧光显微镜可视化, 以确定重组蛋白在细胞内分布 (图 2B)。
2. 细胞染色
注意: 在 FC 分析期间, 新鲜非诱导细胞作为阴性控制需要建立荧光阈值。
- 取 16 h 诱导的酵母细胞, 并确定其光学密度在 590 nm (OD590).将其稀释在无菌磷酸盐缓冲盐水 (PBS) 到 OD590的0.1。
- 准备1x 磷酸盐缓冲盐水如下: 添加8克氯化钠, 0.2 克氯化钾, 1.44克 Na2HPO4, 和0.24 克的 ddH 2 毫升4至800 mL 2 o 在调整 pH 值到1后, 填充解决方案多达2升 ddH7.4用 HCl 过滤缓冲区通过0.2 µm 过滤器。
- 将表示细胞悬浮液转移到适当标记的管 (12 x 75 毫米圆底聚苯乙烯), 并保护它们不受光照。确保他们已经准备好被装载在流式细胞仪进行分析, 连同非诱导和非染色的电池。
- 将氧化应激探针 (例如, CellROX 深红色) 添加到每个样品中, 最终浓度为5µM, 在黑暗中孵化细胞在30摄氏度30分钟。
- 孵化后, 用 1x PBS 冲洗细胞3次, 在 FC 分析前将它们并用重悬在相同的缓冲容积内。
3. 流式细胞仪分析
注: 使用 FC 设置与适当的激光和过滤器, 以检测 GFP 和氧化应激探针荧光信号。一个流式细胞仪配备了 488 nm 蓝激光检测 GFP 和 635 nm 红色激光检测氧化应激探针荧光可以使用。采用 530/30 nm bp 滤波器和 660/20 bp 滤波器分别对 GFP 和氧化应激探针的发射荧光进行了采集 (表 1)。
- 单击 "打开新工作表" 面板并创建以下购置点图。
- 从工具栏中选择散点图工具, 然后在 x 轴上的线性刻度上, 在 y 轴与正向散射区域 ( FSC a) 上创建一个带有变量侧散射区域 (SSC a) 的图。
- 单击工具栏上的 "散点图" 工具, 并在 x 轴与 FL1-Area (FITC) 的变量之间创建一个图形。FL3-Area (APC) 在 y 轴上的对数刻度。
- 单击 "仪器设置" 图标。单击 "补偿" 选项卡并设置所有补偿级别。单击 "购置" 选项卡, 然后选择要记录的2万个事件的总数量。
- 单击 "流速" 图标可将流量切换到较低的速率。
- 单击 "获取" 选项卡, 开始运行非诱导的、无瑕的单元格, 并调整正向和侧向散射的仪器设置中的电压, 直到将人口分布到左中象限。
- 点击多边形图标, 设置一个区域 R1 周围的细胞人口, 不包括细胞碎片, 并使用这个门控人口 P1 = R1 所有荧光点图和直方图表示 (图 3)。
- 要调整荧光信号的 PMT 电压, 请在 FL1-FL3 点图中运行无瑕单元格, 调整仪表设置选项卡中的增益, 直到单元格在左下象限中分布。
- 将样品改成诱导细胞, 以测定 GFP 荧光。在 "仪器设置" 选项卡中, 在 FL1 (FITC) 中, 当人口分布在右下象限时, 设置在 FSC 中的增益。用门 (门 P2) 定义阳性细胞群。
- 将样品改为非诱导的染色细胞, 在 FSC-FL3 (APC) 点图上通过荧光显示氧化应激。调整增益, 直到细胞数量分布在左上象限。P3 阳性细胞群的门。
- 用直方图图标绘制两个直方图, 表示细胞荧光。为了表示荧光强度, 请确保代表 GFP 荧光的 FL1 (FITC) 在 x 轴中, 代表荧光的 FL3 (APC) 在 y 轴中, 分别应用 P2 和 P3 种群。
注意: 直方图叠加是一种很好的方法来说明不同 Aβ变种的细胞的荧光剖面之间的差异。
4. 重组蛋白 Immunodetection
- 为了确定蛋白质的表达水平, 通过离心 4000 x g 为6分钟, 收获16小时诱导的酵母培养, 并将细胞颗粒贮存在-80 摄氏度。
- 并用重悬在 PBS 中表达重组蛋白16小时的收集细胞。将每个 Aβ42-GFP 突变体的200µL 细胞悬浮物准备成590 20。
- 总蛋白分数分析, 以 1.4万 x g 为20分钟, 用离心法收获100µL, 并用重悬在同一体积酵母裂解缓冲液中的细胞颗粒 (50 毫米三盐酸 pH 8.0, 1% 亚砜, 200 毫米氯化钠, 1 毫米 EDTA, 含10毫克/毫升 SB3-14 (马币istyl sulfobetaine) 补充1毫米 phenylmethylsulfonyl 氟 (PMSF))。
- 在轻度搅拌的室温下, 将样品孵化20分钟。
- 用布拉德福德测定法测定蛋白质的提取浓度。在15% 丙烯酰胺 SDS 页电泳凝胶上, 将每个样品的蛋白质提取物载入5µg, 并将其涂抹到聚偏聚氟 (PVDF) 膜上100伏60分钟。
- 准备一个布拉德福德试剂与100毫克的考马斯灿烂的蓝色 G-250 溶解在50毫升95% 乙醇和添加100毫升 85% (w/v) 磷酸。然后, 当染料完全溶解, 稀释的混合物与 ddH2O 到1升和过滤它通过纤维素过滤纸刚刚使用。
注: 布拉德福德试剂应为浅褐色。 - 制备标准的牛血清白蛋白 (BSA) 范围从 5-100 µg 的蛋白质在1000µL 的布拉德福德试剂。
- 将 1-10 µL 蛋白提取物添加到布拉德福德试剂1毫升中, 在室温下孵化混合物5分钟。
- 测量标准和蛋白质提取样品在 OD595的吸光度。
- 对于分析, 用所得到的标准与所获得的吸光度值作一个图。蛋白质的µg。根据标准曲线, 确定原始样品的浓度, 从蛋白质的数量, 考虑到体积和稀释, 如果有的话。
- 准备一个布拉德福德试剂与100毫克的考马斯灿烂的蓝色 G-250 溶解在50毫升95% 乙醇和添加100毫升 85% (w/v) 磷酸。然后, 当染料完全溶解, 稀释的混合物与 ddH2O 到1升和过滤它通过纤维素过滤纸刚刚使用。
- 用1x 的三缓冲盐水 (tb), 0.1% 吐温 20 (TTBS) 冲洗膜, 并用 5% (w/v) 脱脂干牛奶在 1x TTBS 中阻断。
- 准备1升 10x tb, 溶解24克的三基和88克氯化钠在900毫升的 H2Odd和调整 pH 值为7.6。增加 ddH2O 到最后容量 1 L。对于1x 解决方案, 混合1部分10x 库存与 9 ddH2O 的一部分。
- 在室温下孵育1小时的膜, 其主要β淀粉样变性抗体6E10 稀释 1:1, 000 和 1 h 常温下与二级抗体山羊抗鼠 IgG-HRP 共轭稀释1:10,000。
- 用1毫升的 Luminata 荧光试剂和密度分析的方法来发展膜, 如步骤5.2 所述。
5. 数据分析
- 为了分析 FC 获得的数据, 创建一个表, 显示平均荧光强度和中值荧光及其相应的标准误差和/或 GFP 荧光和氧化应激水平的方差系数 (CV)。
- 单击 "检查器" 选项卡和 "统计" 图标可自定义每个通道所需的统计数据。
注意: 在比较蛋白质变体时, 将荧光值数据转换为折叠背景或分辨率度量 (RD):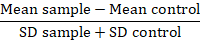
在这个度量中, 平均样本是问题样本的均值荧光, 均值控制是作为控制的细胞的均值荧光, SD 是平均荧光的标准偏差。RD 因子是一个比多边基金会价值更准确的指数, 因为它不仅计算样本之间的差异, 而且也说明数据的传播。当比较来自不同实验的数据时, 这个计算是特别相关的。一旦 RDs 被计算出来, 使用统计测试是可取的, 以确认的意义, 观察差异的蛋白质变异。
- 单击 "检查器" 选项卡和 "统计" 图标可自定义每个通道所需的统计数据。
- 对于蛋白质 immunodetection, 用 ImageJ 软件量化了西方印迹的重组蛋白水平。
- 在密度分析之前, 将所开发膜的原始原始图像转换为 JPEG 文件格式和灰度模式。
- 将 "分析" 菜单的 "设置测量" 部分调整为灰色值。
- 点击矩形工具从 ImageJ 和定义一个感兴趣的区域 (ROI) 与一致的大小为波段密度分析。在所创建的矩形框架内, 将薄膜图像上的每个波段居中, 并使用 Ctrl + M (测量命令) 逐个记录测量结果。
- 测量每个波段相邻的本地背景, 并应用相同的 ROI 区域。测量后, 从每个波段减去相应的局部背景。
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
该协议描述如何使用 Aβ42肽的20变种的集合, 其中 Phe19 已经变异为所有自然 proteinogenic 氨基酸28。这些蛋白质的理论聚集倾向可以用两种不同的生物信息学算法 (AGGRESCAN 和探戈31,32) 来分析。在这两种情况下, 这种分析呈现出聚合倾向的渐进级化, 作为一般规则, 将最高值归结为疏水性残留物和最低的电荷和极性 (图 1B, 1C)。
为了追踪不同 Aβ42变种的细胞内聚集状态, 所描述的实验需要在半乳糖诱导 GAL1 的控制下, 对质粒进行转化, Aβ42编码, 并融合到 GFP (图 1A),到S 酵母。在16小时的诱导期后表达 Aβ42变种的酵母细胞可以在荧光显微镜下可视化。这可视化使它成为可能确定 PI 的形成在10从20个变形从被分析的汇集。荧光骨料的数量和大小不同于变体 (图 2)。图 2A显示了从这两种独立文化中的每一个500个细胞中的 PI 量化。在这里, 我们可以看到一个很好的协议, 预测和体内聚集的性质。
为了解决蛋白质团聚体的形成是否能在这个模型细胞系统中引发氧化应激, 该协议使用 FC 监测细胞 GFP 荧光和荧光探针的荧光作为活性氧的指示器。种 (ROS) 生产。图 3说明了 Gln 和 Aβ42突变体的过程和获得的结果, 分别对应于均匀分布和 PI 形成的变异体。
首先, 分析的细胞数量是门控 (P1) 在 FSC-a与SSC-一个点图, 以消除细胞碎屑从分析 (图 3A)。P1 种群的荧光信号与氧化应激探针在 P3 (图 3B) 中的绿色荧光细胞被封闭的点图图中表示。图 3C和3D显示为获取每个表示的变体 (包括 CV (表 2) 的统计数据而创建的直方图, 以量化每个荧光标记的平均值和中值荧光。
所有突变体都将在同一水平上表达, 因为它们在单个氨基酸 (Aβ42-GFP 序列的 0.02%) 上不同。然而, proteostasis 机械可以以不同的方式对其特定的聚集倾向作出反应。因此, 细胞提取物中蛋白质表达水平使用 Aβ特异抗体进行量化 (图 4)。我们看到, 作为一个普遍的趋势, PI 形成的 Aβ42变种存在于较低的水平比那些保持均匀分布在细胞质。
当它们的氧化应激探针荧光、蛋白质水平和 GFP 荧光特性相对于它们形成 PI 的能力和它们内在的聚集倾向时, 可以观察到 Aβ42变种之间的重要差异 (图 5)。更容易聚集的 PI 形成变异引起的氧化应激比它们的可溶性对应物要低得多 (图 5A, 5B)。有了这些结果, 每个细胞的粒度和聚集数 (图 2) 和氧化应激水平之间没有明显的相关性。PI 形成的变种, 特别是在位置19的变形轴承, 在细胞中比那些分布在细胞质 (图 5C, 5D) 中的 homogenously 存在。对此的例外是该变种人, 其中不到10% 的细胞形成 PI (图 1B, 1C)。这对应于事实最容易聚集的变形由酵母质量控制退化机器有选择地清除28。GFP 荧光和蛋白质水平的相关性较好的可溶性 Aβ42形式, 但不为那些形成 PI (图 5E, 5F)。这可能是因为在这些细胞群中, 荧光来自两个不同的隔间, 细胞质和包裹体, 它们对总荧光的相对贡献在突变体34之间不同。

图 1.Aβ42-GFP 融合结构的聚集倾向分析, 该方法来源于 Aβ42肽中19位的残留物在所有天然氨基酸中的突变。a. 此图像显示一个链接器 (Aβ42, 灰色) 与 GFP Aβ42融合的3D 模型;GFP, 绿色), 基于 PDB 1EMA 的绿色荧光蛋白从水母维多利亚和2OTK 的阿尔茨海默 Aβ肽, 其中 Phe19 侧链 Aβ42是红色显示。模型是使用 Pymol 软件创建的。Aβ42序列中的渣19在中心疏水群中占据中心位置。条形图是用两个不同的生物信息学预测因子来创建的: B. AGGRESCAN, C. 探戈。请单击此处查看此图的较大版本.

图 2.Aβ42-GFP 在 S 酵母培养中形成的变异体,诱导为 16 h 的表达。条形图表示在两个生物复制中, 每个变种的500个荧光细胞中含有不同数量 PI 的细胞百分比。B. 这些图像是具有代表性的荧光显微图像的选定 Aβ42-GFP 变种 (菲比, 微笑, 中, 激进党)。它们是在紫外光下获得的, 使用的是 GFP (450-500 nm) 和一个发射范围 (515-560 纳米) 的励磁过滤器。刻度条代表10µm.请点击这里查看这个数字的大版本。

图 3.流式细胞术 (FC) 分析方案,表达选定 Aβ42-GFP 变种的酵母细胞。图表显示了点图中的门控酵母细胞 (P1), 其中细胞碎屑从细胞群中移除。Gln (均匀分布) 和微笑 (PI 形成) 变体的显微图像显示在 FC 点地块旁边。刻度条代表10µm. 这些散布点图图像代表 GFP-与氧化应激探针, 其中门控种群 (P3) 只包括荧光细胞, 不含背景信号。细胞频率直方图为C。GFP 信号 (FITC 振幅) 门控从 P1 和D。CellROX (APC 振幅) 从 P3 门控。用流式细胞仪进行单元采集。每个情节代表2万事件。Q 和我对应的 Gln 和 Aβ42变种人, 分别。请单击此处查看此图的较大版本.

图 4.细胞蛋白水平的量化。这一数字显示, 在 16 h 的酵母表达后, Aβ42-GFP 突变体的总蛋白组分的西方印迹。PI 形成的变形在绿色被上色, 并且那些弥漫分布在细胞质被上色以浅红色。请单击此处查看此图的较大版本.

图 5.Aβ42-GFP 突变体的细胞氧化应激、细胞内 GFP 荧光和细胞蛋白水平在 16 h 的酵母表达后测定 .在 x 轴上表示的变体根据其预测的聚集倾向, 按 AGGRESCAN (左面板) 或探戈 (右面板) 排序。pi 形成的变形在绿色被上色, 并且非 PI 形成的变形在淡红色被上色。A。这些条形图显示了 Aβ42-GFP 突变体的 FC 分析获得的氧化应激探针荧光值。C。这些条形图代表了用 ImageJ 软件对西方污点密度分析进行量化的蛋白质水平。E。F. 这些条形图显示了 FC 分析后的 GFP 荧光值。氧化应激探针和 GFP 荧光值的误差条代表 FC 门控细胞的方差系数 (CV)。蛋白质表达水平的误差条代表了 SE (n = 3)。请单击此处查看此图的较大版本.
| 通道 | 荧光 | 励磁波长 (nm) | 带通滤波器 |
| FITC | Gfp | 488 | 530/30 |
| Apc | CellRox | 635 | 660/20 |
| PerCP | Ip | 488 | 585/42 |
表1。用于流式细胞仪的激光源、带通滤波器和显影。
| GFP 荧光 | 简历 | CellROX 荧光 | 简历 | |
| 一个 | 9316 | 96。4 | 1964 | 127。5 |
| C | 9709 | 91。9 | 1275 | 172。2 |
| D | 11213 | 101。7 | 3443 | 155。8 |
| 电子邮件 | 12256 | 101。1 | 3220 | 152。2 |
| F | 3010 | 96。1 | 1245 | 146。4 |
| G | 11541 | 97。2 | 2947 | 158 |
| H | 7895 | 98。2 | 3582 | 120。8 |
| 我 | 7365 | 97。2 | 1416 | 141。3 |
| K | 10839 | 100。4 | 3102 | 122。1 |
| 我 | 7605 | 96。9 | 1401 | 161。8 |
| M | 8149 | 96 | 1308 | 170。4 |
| N | 12741 | 97。5 | 3403 | 134。9 |
| P | 9768 | 102。8 | 2629 | 143。6 |
| 问 | 13066 | 91。3 | 3354 | 169。9 |
| R | 8537 | 101。3 | 2839 | 127。5 |
| S | 12053 | 99。1 | 3313 | 174。3 |
| T | 10615 | 97。7 | 2213 | 107。7 |
| V | 9169 | 96。1 | 1878 | 121。7 |
| W | 1715 | 94。7 | 1531 | 100 |
| Y | 7574 | 94。5 | 1234 | 138 |
表 2.GFP 荧光和氧化应激探针荧光值列表 16 h 表达 Aβ42-GFP 突变体的酵母细胞的 FC 分析.该表显示了每个突变体的平均荧光强度和 CV 值。
Subscription Required. Please recommend JoVE to your librarian.
Discussion
广泛的疾病与错误折叠蛋白质的积累联系在一起6,7,8,33的细胞沉积。已作出许多努力, 以解开分子机制, 触发这些疾病的开始使用计算方法, 不考虑蛋白质浓度, 或在体外的方法, 其中蛋白质浓度在反应过程中保持恒定。然而, 在细胞内, 蛋白质在拥挤和非均匀的环境中不断地合成和降解。这解释了 amyloidogenic 蛋白在硅、体外和体内聚集特性之间的频繁差异。
简单的细胞模型, 例如酵母, 是研究与神经退行性疾病相关的蛋白质的聚集和相关毒性的一个合理选择, 有多种原因。例如, 它们使我们有可能通过快速分析 (如此处所实施的) 来分析蛋白质错误折叠聚集对完整的细胞群的影响。然而, 我们应该考虑到在酵母菌株中氧化水平可能不同。因此, 要比较菌株, 甚至在不同的蛋白质之间, 氧化剂和还原剂, 如德勤和二胺, 应用于建立氧化/还原量表。
在本文所示的例子中, 生物信息学分析, 细胞蛋白定量, 蛋白质定位成像和同时 FC 分析的 GFP 活性和氧化应激水平已被纳入识别分子由蛋白质聚集反应引起的氧化损伤的种类。结果表明, 较易溶 Aβ42变异, 而不是更容易聚集, 促进最高的氧化应激。这表明, 扩散物种是更危险的 Aβ42物种在体内。聚合变种的较低毒性似乎对这一事实作出反应, 即这些构象被隔离成 PI 和他们的优先退化的蛋白质质量机械, 导致较低的蛋白质水平比他们的可溶性同行。
所描述的方法不限于分析 Aβ42聚集/可溶性物种产生的氧化应激, 也可用于研究多种蛋白质聚集紊乱。此外, 该技术可能有助于监测聚合抑制剂对细胞氧化应激的影响, 允许放弃进一步的临床应用这些分子, 促进氧化活性蛋白的积累。最后, 只要有一个荧光探针可用, 该方法就能提供显著的机会, 以确定与蛋白质聚集有关的其他毒性作用的分子物种快速简便的方法, 提供定量数据。
Subscription Required. Please recommend JoVE to your librarian.
Acknowledgments
Materials
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
References
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration? Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast? Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).