Summary
חלבון צבירת מעורר הסלולר סטרס חמצוני. פרוטוקול זה מתאר שיטה לניטור את המדינות amyloidogenic חלבונים תאיים סטרס חמצוני המשויכים אליהן, באמצעות cytometry זרימה. הגישה משמש כדי ללמוד על אופן הפעולה של גרסאות מסיסים ונוטה צבירת של פפטיד עמילואיד-β.
Abstract
חלבון misfolding, צבירת לתוך עמילואיד הייצורים החיים קשורה עם התחלתה של התקדמות של מספר מחלות ניווניות. עם זאת, יש עדיין מעט מידע על חלבונים מסיסים איך אגרגטים להפעיל את ההשפעות הרעילות שלהם בתוך vivo. אורגניזמים פשוטים prokaryotic ו האיקריוטים מודל, כגון חיידקים, שמרים, תרמו משמעותית להבנתנו הנוכחי של המנגנונים מאחורי היווצרות עמילואיד תאיים, הפצת אגרגטים, רעילות. ב פרוטוקול זה, השימוש של שמרים מתואר כמודל לנתח את הקשר בין היווצרות של אגרגטים חלבון והשפעתם על הסלולר סטרס חמצוני. השיטה משלבת את הגילוי של המדינה מסיסים/המצטברים תאיים החלבון amyloidogenic עם כימות של נזק חמצוני הנובע ביטויה באמצעות cytometry זרימה (FC). גישה זו היא פשוטה, מהירה, כמותית. המחקר מדגים את הטכניקה על-ידי התאמת את הסלולר עקה הנגרמת על ידי קבוצה גדולה של עמילואיד-β משתנים פפטיד עם שלהם מספק שלעצמו צבירת מהותי בהתאמה.
Introduction
פרוטוסטאזיס הוא דטרמיננטה הבסיסית של כושר תא ותהליכי הזדקנות. תאים, חלבון הומאוסטזיס נשמר על ידי בקרת איכות החלבון המתוחכם רשתות שנועד להבטיח את נכונות refolding של חלבונים misfolded conformers מלווים ו/או proteolysis יישוב שלהם עם מספר מנגנונים שנשמרת היטב1 ,2,3,4,5. מספר רב של מחקרים מספקים תמיכה לקשר בין התפתחות והתקדמות של מגוון רחב של מחלות האדם וכישלון פרוטוסטאזיס, המוביל אל חלבון misfolding, צבירת. למשל, הנוכחות של חלבון פיקדונות נחשב סימן היכר פתולוגי של הפרעות ניווניות רבים, כמו אלצהיימר, פרקינסון, ו הנטינגטון מחלות6,7,8, מחלות prionogenic, amyloidoses ניוונית9. הוצע כי הרכבות oligomeric, protofibrillar מוקדמת בהתגובה צבירת הן elicitors הראשי של cytotoxicity, הקמת aberrant אינטראקציות עם חלבונים אחרים חצרו הסלולר צפוף10. בנוסף, חלבון הכללות (PI) יכול להיות מועבר בין תאים, הפצת שלהם השפעה רעילה11,12. לכן, זה יכול להיות כי היווצרות של PI אולי אכן מהווה מנגנון בעלת מגבילה את נוכחותם של מינים צבורים מסוכן למיקומים ספציפיים בתא, היכן ניתן לעבד או שנצבר ללא תופעות לוואי משמעותיות 13 , 14.
תקן במבחנה גישות הביוכימי סיפקו תובנות חשובות מינים שונים that לאכלס את צבירת תגובות ו15,שלהם מאפיינים16. עם זאת, תנאי שימוש אלה מבחני בבירור שונות מאלו המתרחשים בתוך התא ולתשאל, לכן, מאבדים רלבנטיות פיזיולוגיים. בגלל השימור הבולטים של מסלולים סלולריים כגון בקרת איכות חלבון, autophagy או ברגולציה של הסלולר חמצון-חיזור המדינה17,18 בין פרוקריוטים19,20,21 ,22,23, שמרים ניצני האפייה (cerevisiae ס) התפתחה מיוחס מודל הסלולר פשוט ללמוד על גורמים מולקולרית של צבירת חלבון והשפעתו המשויך ציטוטוקסיות סביבות ביולוגית רלוונטית24,25,26.
חלבון צבירת הנטייה היא תכונה מטבעו מקודד ברצף העיקרי. לפיכך, היווצרות עמילואיד-מבני ניתן לחזות המבוסס על זיהוי, הערכה של עוצמת קידום צבירת אזורים polypeptides27. עם זאת, למרות ההצלחה של אלגוריתמים bioinformatic כדי לחזות את המאפיינים צבירת במבחנה של רצפי חלבונים, הם עדיין רחוק מלהיות חיזוי איך לתרגם אלה מספק שלעצמו ויוו ציטוטוקסיות ההשפעה. מחקרים כתובת הקישור בין המדינה צבורים של חלבון נתון שלה נזק תאי המשויכים בצורה שיטתית עשוי לעזור כדי לעקוף מגבלה זו חישובית. חיבור זה הוא התייחס במחקר הנוכחי, ניצול של סט גדול של גרסאות של פפטיד עמילואיד-β Aβ42 שונות רק ב משקע יחיד, אך הצגת טווח רציף של צבירת מספק שלעצמו ויוו28. בפרט, מתואר גישה מבוססת-FC לזהות את הזן הסתגלותי החשבונאי נזק חמצוני שהפיק צבירת נוטה חלבונים בתאים שמרים. המתודולוגיה מספק יתרונות רבים כגון פשטות, תפוקה גבוהה יכולת מדידה כמותית מדויקת. גישה זו אפשרו לאשר את PI לשחק תפקיד מגן מפני עקה.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. תרבויות cerevisiae ס וביטוי חלבונים
הערה: גרסאות Aβ התערוכה מספק שלעצמו צבירת היחסי שונה עקב מוטציה בגן משקע אחד במיקום 19 (Phe19) של פפטיד Aβ42 (איור 1 א'). גרסאות פפטיד אלה מתויגות עם חלבון פלואורסצנטי ירוק (GFP), אשר משמש כתב (איור 1) צבירת29.
- להפוך פלסמידים קידוד עבור 20 Aβ42-GFP משתנים לתוך תאי שמרים עם BY4741 רקע הורים (מאטה שלו3Δ1 leu2Δ0 פגש את15Δ0 ura3Δ0)30. כל פלסמיד מקודד עבור מוטציה Aβ42 דבוקה GFP באמצעות מקשר GSSGSSG. פלסמידים הינם pESC(-URA) וכוללים דה מרקר לבחירה URA329.
- להכין המדיום מלאה סינתטי ללא אורציל (SC-עורה) עם הרכיבים הבאים: 1.9 גרם/ליטר חנקן שמרים בסיסי ללא אורציל, 5 g/L אמוניום סולפט, 900 מל של ddH2O בשילוב עם 100 מ של גלוקוז 20% (w/v).
- מכניסים המושבה אחד של תאי שמרים טרנספורמציה 20 מ"ל של המדיום SC-עורה המכיל 2% גלוקוז ולגדול התרבויות בין לילה ב 30 ° C תחת עצבנות-210 סל ד.
- למחרת, לגדול תרבויות חדשות של תאי שמרים מאת לחסן 100 µL של התרבות לילה לתוך מ של מדיום SC-עורה צח.
- יתר590 של 0.5, centrifuge התרבויות ב x 3000 g למשך 4 דקות, למחוק את תגובת שיקוע ולאחר resuspend התאים באותו אמצעי אחסון בינוני SC-עורה צח המכיל 2% raffinose.
- דגירה ב 30 ° C תחת עצבנות-210 סל"ד למשך 30 דקות. לאחר מכן, centrifuge את התאים ב 3000 g x במשך 4 דקות וזורקים את תגובת שיקוע. Resuspend התאים במדיום SC-עורה צח המכיל 2% גלקטוז לזירוז הביטוי חלבון רקומביננטי.
- לאחר ה' 16 של גרימת ביטוי חלבון במדיום SC-עורה המכיל 2% גלקטוז, קציר 1 מ"ל של התאים המושרה צינורות סטרילי microcentrifuge על ידי צנטריפוגה ב 3000 g x במשך 4 דקות.
הערה: ההבדלים הרלוונטיים ביותר להתרחש לאחר תקופה אינדוקציה של תאי שמרים המושרה ה 16 לבטא Aβ42-GFP משתנים, ניתן לאבחן על ידי קרינה פלואורסצנטית מיקרוסקופ כדי לקבוע את התפלגות חלבון רקומביננטי בתוך התאים (איור 2B).
2. התא מכתים
הערה: תאים שאינם-induced טריים נדרשים כפקד שלילי כדי לקבוע סף פלורסנט במהלך ניתוח FC.
- לוקחים תאי שמרים h-induced 16 ולקבוע שלהם צפיפות אופטית-590 nm (OD590). לדלל אותם בתוך תמיסת סטרילית פוספט buffered (PBS) יתר590 של 0.1.
- להכין 1 x buffered פוספט תמיסת מלח כדלקמן: להוסיף 8 גרם של NaCl, 0.2 גרם אשלגן כלורי, 1.44 גר' נה2HPO4, ולמלא 0.24 גר'2פו ח'4 800 מ של ddH2O. הפתרון עד 1 L של ddH2O לאחר התאמת רמת ה-pH ל 7.4 עם HCl. לסנן את המאגר דרך מסנן 0.2 µm.
- להעביר את המתלים תא לביטוי כראוי שכותרתו צינורות (פוליסטירן 12 ממ עגול-התחתון × 75), להגן עליהם מפני אור. ודא כי הם מוכנים להיות טעון על cytometer זרימת לניתוח, יחד עם תאים שאינם-induced ו שאינו מוכתם.
- להוסיף החללית סטרס חמצוני (למשל, אדום עמוק CellROX) כל דגימה-ריכוז הסופי של 5 מיקרומטר, דגירה התאים ב 30 מעלות צלזיוס למשך 30 דקות בחושך.
- לאחר דגירה, לשטוף את התאים 3 פעמים עם 1 x PBS, resuspend אותם באמצעי האחסון באותו מאגר לפני ניתוח שלהם על ידי מועדון הכדורגל.
3. לזרום Cytometry ניתוח
הערה: השתמש התקנה FC עם לייזרים המתאים ומסננים כדי לזהות GFP ושידור פלורסצנטיות בדיקה סטרס חמצוני. Cytometer זרימה מצוידים 488 ננומטר כחול בלייזר איתור GFP, לייזר nm אדום 635 איתור סטרס חמצוני בדיקה קרינה פלואורסצנטית יכול לשמש. רכישת פליטת קרינה פלואורסצנטית GFP, סטרס חמצוני בדיקה מבוצע עם 530/30 ננומטר BP מסנן, מסנן BP 660/20, בהתאמה (טבלה 1).
- לחץ על פתיחת החלונית ' גליון עבודה חדש וליצור החלקות נקודה הרכישה הבאה.
- בחר את פיזור כלי מגרש מסרגל הכלים וליצור מגרש עם המשתנים לצד פיזור-אזור (האס-A) על ציר ה-y לעומת קדימה פיזור-אזור (FSC-A) בסרגל ליניארי של בציר ה-x.
- לחץ על פיזור כלי מגרש מסרגל הכלים וליצור מגרש עם המשתנים באזור FL1 (FITC)-ציר ה-x vs. FL3-האזור (APC) ב לוגריתמי בציר ה-y.
- לחץ על הסמל הגדרות מכשירים . לחץ על הכרטיסיה פיצויים וקבע בכל רמות הפיצוי. לחץ על הכרטיסיה ייבוא , בחר את המספר הכולל של אירועים 20,000 שיירשמו.
- לחץ על הסמל קצב הזרימה כדי לעבור את קצב זרימה נמוכה.
- לחץ על הכרטיסיה ייבוא להתחיל לרוץ את התאים הלא-induced, מוכתם ולהתאים את המתח הגדרות המכשיר פארווערטס ואת פיזור בצד עד האוכלוסייה מופץ ברבע השמאלי האמצעי.
- לחץ על הסמל ' מצולע ' כדי להגדיר אזור R1 סביב האוכלוסייה תא למעט שאריות תאים, ולהשתמש אוכלוסייה מגודרת זו P1 = R1 עבור כל נקודה פלורסנט חלקות וייצוגי היסטוגרמה (איור 3).
- כדי להתאים את המתח PMT של האות פלורסצנטיות, להפעיל את התאים מוכתם העלילה נקודה FL1-FL3, כוונון של הרווח בכרטיסיה כלי ההגדרה , עד התאים מופצים ברבע השמאלי התחתון.
- לשנות את דגימת התאים המושרה למדידת קרינה פלואורסצנטית GFP. בכרטיסיה כלי ההגדרה , להגדיר את רווח FSC-A נ' FL1 (FITC) כאשר האוכלוסייה מופץ ברבע הימני התחתון. הגדרת האוכלוסייה תא חיובית עם שער (שער P2).
- שינוי הדוגמית להוכיח אי-induced מוכתם לתאים כדי להציג סטרס חמצוני זריחה על FSC-A נ' FL3 (APC) נקודה מגרש. מנגינה של הרווח עד האוכלוסייה תא מופץ ברבע השמאלי-עליון. שער אוכלוסיית תאים חיוביים P3.
- לעשות שני מגרשים היסטוגרמה עם סמל היסטוגרמה לייצג את פלורסצנטיות תא. כדי לייצג את עוצמת קרינה פלואורסצנטית, ודא FL1 (FITC) המייצג את פלורסצנטיות GFP ב ציר ה-x, FL3 (APC) המייצג את זריחה בציר ה-y, החלת P2, P3 אוכלוסיות בקנה מידה לוגריתמי, בהתאמה.
הערה: כיסויי היסטוגרמה הם דרך טובה כדי להמחיש את ההבדלים בין הפרופילים זריחה של תאים לבטא את Aβ וריאציות שונות.
4. Immunodetection חלבון רקומביננטי
- כדי לקבוע רמות ביטוי החלבון, לקצור את התרבויות שמרים המושרה עבור 16 h על ידי צנטריפוגה-g 4,000 x עבור 6 דקות ואחסן את כדורי תא ב- 80 ° c
- Resuspend התאים שנאספו ביטוי חלבון רקומביננטי עבור 16 h ב- PBS. להכין 200 המתלים תא µL של כל מוטציה Aβ42-GFP יתר590 של 20.
- לניתוח שבר מוחלט חלבון, לקצור 100 µL של כל מוטציה על ידי צנטריפוגה ב g 14,000 x במשך 20 דקות, resuspend כדורי תאים באותו אמצעי אחסון שמרים פירוק מאגר Y-לכל (50 מ מ טריס-HCl pH 8.0, 1% דימתיל סולפוקסיד, 200 מ"מ NaCl, המכיל 10 מ"ג/מ"ל (myr 14-SB3 EDTA 1 מ מ istyl sulfobetaine) בתוספת 1 מ"מ phenylmethylsulfonyl פלואוריד (PMSF)).
- דגירה הדגימות במשך 20 דקות בטמפרטורת החדר תחת עצבנות קלה.
- לקבוע את הריכוז תמצית חלבון באמצעות וזמינותו ברדפורד. לטעון עד 5 µg של חלבון תמצית של כל מדגם on a ג'ל אלקטרופורזה מרחביות-דף 15% אקרילאמיד כתם על גבי קרום polyvinylidene difluoride (PVDF) ב- 100 וולט עבור 60 דקות.
- הכן של ברדפורד מגיב עם 100 מ ג של G-250 כחול מבריק Coomassie התפרקה ב 50 מ של 95% אתנול, להוסיף 100 מ של חומצה זרחתית 85% (w/v). לאחר מכן, כאשר לצבוע התפרקה לחלוטין, לדלל את התערובת עם ddH2O עד 1 ליטר, דרך נייר סינון תאית רק לפני השימוש.
הערה: הכימית ברדפורד צריך להיות חום בהיר. - להכין תקן של שור אלבומין (BSA) הנע בין 5 ל- 100 µg של חלבון ב- 1,000 µL מהתרכובת ברדפורד.
- להוסיף 1-10 µL של חלבון תמצית 1 מ"ל מהתרכובת ברדפורד, דגירה התערובת בטמפרטורת החדר במשך 5 דקות.
- למדוד את ספיגת תקנים ולחלץ החלבון דגימות-OD595.
- לניתוח, הופכים מגרש עם הערכים ספיגת שהתקבלו עבור הסטנדרטים vs. µg של חלבון. בהתבסס על העקומה סטנדרטי, לקבוע את ריכוזי הדגימות המקורי כמות של חלבון, בהתחשב בכך את נפח ואת דילול, אם בכלל.
- הכן של ברדפורד מגיב עם 100 מ ג של G-250 כחול מבריק Coomassie התפרקה ב 50 מ של 95% אתנול, להוסיף 100 מ של חומצה זרחתית 85% (w/v). לאחר מכן, כאשר לצבוע התפרקה לחלוטין, לדלל את התערובת עם ddH2O עד 1 ליטר, דרך נייר סינון תאית רק לפני השימוש.
- לשטוף את הקרום עם 1 x באגירה טריס תמיסת מלח (TBS), 0.1% Tween 20 (TTBS), ולחסום אותו באמצעות חלב יבש ללא שומן 5% (w/v) ב- 1 x TTBS.
- להכנת 1 ליטר של 10 x TBS, להמיס 24 גר' טריס הבסיס ו- g 88 של NaCl ב 900 מל של2O Hdd ולהתאים את ה-pH ל 7.6. DdH2O להוסיף נפח סופי של 1 ל' פתרון 1 x, לערבב 1 חלק של מניות x 10 עם 9 חלקים של ddH2O.
- דגירה הקרום עבור h 1 עם 1:1,000 6E10 בדילול נוגדן ראשוני β-עמילואיד, לשעה בטמפרטורת החדר עם עז משני נוגדן אנטי עכבר IgG-HRP המספר המשלים מדולל 1:10, 000.
- לפתח ממברנות באמצעות 1 מ"ל של ריאגנט פלורסנט Luminata ולכמת להקות על ידי ניתוח densitometric, כפי שמתואר בשלב 5.2.
5. ניתוח נתונים
- כדי לנתח את הנתונים שבידי FC, ליצור טבלה מציג את עוצמת פלורסנט רשע (MFI), קרינה פלואורסצנטית החציוני לשגיאה הסטנדרטית המתאימים ו/או המקדם של השונות (CV) עבור קרינה פלואורסצנטית GFP ורמות סטרס חמצוני.
- לחץ על הכרטיסיה מפקח בסמל נתונים סטטיסטיים כדי להתאים אישית את נתונים סטטיסטיים הדרושים לכל ערוץ.
הערה: בעת השוואת גרסאות חלבון, להמיר פלורסצנטיות ערכי הנתונים מקפלים-over רקע או מדד רזולוציה (RD):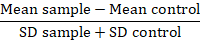
בהמדד, המדגם רשע הוא זריחה מתכוון הבעיה דוגמאות של הפקד רשע הוא זריחה אכזרי של תאים משמש פקד, הגרמנים הוא סטיית התקן של זריחה אומר. פקטור רואד הוא אינדקס מדויקת יותר מהערך MFI הוא לא רק מחשב את ההפרש בין דגימות, אלא גם חשבונות עבור הפצת הנתונים. חישוב זה רלוונטי במיוחד כאשר משווים נתונים שמקורם ניסויים שונים מתבצעת לאורך זמן. לאחר ה-RDs חושבו, השימוש של מבחן סטטיסטי מומלץ לאשר את משמעות ההבדלים שנצפה בין גרסאות חלבון.
- לחץ על הכרטיסיה מפקח בסמל נתונים סטטיסטיים כדי להתאים אישית את נתונים סטטיסטיים הדרושים לכל ערוץ.
- עבור חלבון immunodetection, לכמת את רמות חלבון רקומביננטי מאת תספיג באמצעות תוכנת ImageJ.
- לפני הניתוח densitometry, להמיר תמונות raw המקורי של קרום מפותחת תבנית קובץ JPEG ומצב בגווני אפור.
- להתאים את המקטע להגדיר מידות של התפריט נתח לערך גריי אומר.
- לחץ על הכלי מלבן של ImageJ ולהגדיר אזור בעל עניין (ROI) בגודל אחיד לניתוח densitometry הלהקה. מרכז כל הלהקה על התמונה ממברנה בתוך המסגרת מלבני נוצר והקלטה של מדידה אחת באמצעות Ctrl + M (הפקודה מדידה).
- למדוד את הרקע מקומי צמוד כל הלהקה, החלת רועי באותו האזור. לאחר המדידה, להחסיר הרקע המקומי המתאים כל הלהקה בודדים.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
פרוטוקול זה מתאר כיצד להעסיק אוסף של משתנים 20 של פפטיד Aβ42 שבו יש כבר מוטציה Phe19 proteinogenic טבעי כל חומצות אמינו28. מספק צבירת התיאורטי שלעצמו החלבונים האלה ניתן לנתח באמצעות אלגוריתמים שונים bioinformatic שני (AGGRESCAN וטנגו31,32). בשני המקרים, ניתוח זה הופך את שינוי הדרגתי פרוגרסיבי של צבירת נטיות, ומייחס, ככלל, את הערכים הגבוהים ביותר ואני את שאריות הידרופובי לאלה טעונה, קוטבי (איור 1B, 1 ג').
כדי לעקוב אחר השפעול מצב צבירה תאיים Aβ42 וריאציות שונות, הניסוי המתואר מחייבת טרנספורמציה של פלסמיד, קידוד Aβ42 ומאוחדים. עם GFP (איור 1 א') תחת השליטה של גלקטוז-inducible GAL1, לתוך cerevisiae ס. ניתן לאבחן תאי שמרים לבטא את Aβ42 וריאציות לאחר תקופה אינדוקציה של 16 h תחת מיקרוסקופ זריחה. הדמיה זו מאפשרת לך לאשר את היווצרות של פי 10 מתוך גרסאות 20 מתוך אוסף שנותחה. המספר והגודל של פלורסנט מצרפי שונים משתנה variant (איור 2). איור 2A מראה כימות PI מתוך סך של 500 תאים בכל אחד שתי התרבויות עצמאית. כאן, ניתן להבחין הסכם מעולה בין חזה וויוו צבירת נכסים.
כדי לטפל אם היווצרות של אגרגטים חלבון יכול לעורר סטרס חמצוני במערכת תא מודל, פרוטוקול זה משתמש FC כדי לפקח על זריחה GFP הסלולר יחד עם זריחה של המכשיר fluorogenic כמחוון של חמצן תגובתי ייצור מינים (ROS). איור 3 ממחיש את ההליך, השיג תוצאות עבור מוטציות אך זה לא נגמר, איל, המקבילים על גרסאות Aβ42 homogeneously מבוזרת ויוצרים PI, בהתאמה.
ראשית, האוכלוסיה תא שנותחה היא מגודרת (P1) בהנקודה האס-A FSC-A מול מגרש להסיר תאים דטריטי מניתוח (איור 3 א). האות זריחה של GFP לעומת סטרס חמצוני בדיקה של האוכלוסייה P1 מיוצג בגרפים מגרש נקודה שבה תאים ניאון ירוק הן פיקוח ב- P3 (איור 3B). 3C איור ותלת מימד מראים את היסטוגרמות נוצר כדי לקבל נתונים סטטיסטיים עבור כל ביטוי משתנה, כולל את קורות חיים (טבלה 2), כדי לכמת את הממוצע, על ידי קרינה פלואורסצנטית החציוני של כל סמן פלורסנט.
כל המוטציות צפויים לבוא לידי ביטוי ברמות באותו כיוון שהם נבדלים חומצה אמינית בודדת (0.02% של הרצף Aβ42-GFP). עם זאת, המנגנון פרוטוסטאזיס יכול להגיב באופן דיפרנציאלי שלהם מספק שלעצמו צבירה מסוים. לכן, רמות הביטוי החלבון תמציות הסלולר הן לכמת באמצעות נוגדן ספציפי Aβ (איור 4). אנו רואים, כמו כי המגמה הכללית, Aβ42 להיוות PI משתנים קיימים ברמות נמוכות יותר מאלה שנותרו homogeneously מופץ ב ציטוזול...
יכול להיות שנצפו הבדלים חשובים בין גרסאות Aβ42 כאשר שלהם סטרס חמצוני בדיקה פלורסצנטיות, רמות החלבון ומאפיינים GFP פלורסצנטיות מיוצגים באופן יחסי את היכולת שלהם ליצור PI שלהם מספק שלעצמו צבירת מהותי ( איור 5). יותר צבירת נוטה גרסאות להיוות PI להפיק הרבה סטרס חמצוני נמוכה יותר מאשר עמיתיהם יותר מסיס (איור 5A, 5B). עם התוצאות האלה, יש מתאם ברור בין הגודל והמספר של אגרגטים לכל תא בודד (איור 2) ואת רמות סטרס חמצוני. גרסאות להיוות PI, במיוחד מוטציות הנושאת Trp ו Phe במיקום 19, נמצאים בתא ברמות נמוכות יותר מאלה למשל מופץ ב- ציטוזול (איור 5C, 5 D). החריג לכך הוא חמישי החשבונאי, אשר פחות מ-10% של התאים יוצרים פאי (איור 1B, 1 ג'). זה תואם עם העובדה כי המשתנים נוטים ביותר צבירת נמחקים באופן סלקטיבי על ידי מכונות28השפלה בקרת איכות שמרים. קרינה פלואורסצנטית GFP ורמות החלבון לתאם טוב עבור הטפסים Aβ42 מסיס יותר, אך לא עבור אלו ויוצרים PI (איור 5E, 5F). זה כנראה בגלל באוכלוסיות אלה הסלולר, ידי קרינה פלואורסצנטית נובע שני תאים שונים, את ציטוזול והן תכלילים, זריחה הכולל התרומה שלהם שונים בין המוטציות34.

איור 1 . צבירת ניתוח הנטייה של האוסף של Aβ42-GFP פיוז'ן בונה נגזר המוטציה של שאריות-19 בעמדה פפטיד Aβ42 על ידי כל חומצות האמינו טבעי. א. תמונה זו מציגה מודל 3D של wt Aβ42 התמזגו ל GFP באמצעות מקשר (Aβ42, אפור; GFP, ירוק), מבוסס על 1EMA PDB עבור חלבון פלואורסצנטי ירוק, ויקטוריה Aequorea , את 2OTK עבור פפטיד אלצהיימר Aβ בו שרשרת הצד Phe19 wt Aβ42 מוצג באדום. המודל נוצר באמצעות תוכנת Pymol. שאריות 19 ברצף Aβ42 שוכן במיקום מרכזי בהאשכול המרכזי הידרופובי. מהגרפים נוצרים עם שני גורמים מנבאים bioinformatic שונים: B. AGGRESCAN, ג. טנגו. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 2 . Aβ42-GFP PI-ויוצרים גרסאות cerevisiae ס תרבויות המושרה עבור h 16 של הביטוי. א. הגרף מציין את אחוזי תאים המכילים מספר שונה של PI מחושב מתוך סך של 500 פלואורסצנט תאים עבור כל משתנה ב משכפל ביולוגית שני. B. התמונות האלו הן תמונות מיקרוסקופ פלואורסצנטי נציג של משתנים Aβ42-GFP שנבחר (Phe, איל, חמישי, Trp). הם היו רכשה תחת אור UV באמצעות מסנן עירור GFP (450-500 ננומטר) ומגוון פליטה (515-560 ננומטר). סרגל קנה מידה מייצג 10 מיקרומטר. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 3 . שיטת לזרום cytometry (FC) ניתוח of cerevisiae ס התאים לבטא גרסאות Aβ42-GFP שנבחרו. א. התרשים מראה תאי שמרים מגודרת (P1) נקודה חלקות (FSC-A לעומת האס-A) שבו תא דטריטי יוסר האוכלוסייה תא. תמונות מיקרוסקופיות של זאת שוב (homogeneously מופץ) וכל הווריאנטים איל (יוצרי PI) מוצגים ליד שהנקודה FC מגרשים. סרגל קנה מידה מייצג 10 מיקרומטר. B. הדימויים מגרש פיזור נקודה מייצגים GFP-A לעומת בדיקה סטרס חמצוני שבו האוכלוסייה מגודרת (P3) כולל רק פלואורסצנט תאים, למעט האות רקע. היסטוגרמות תדירות התא הם של C. GFP האות (FITC משרעת) מגודרת מ- P1 ו- D. CellROX (APC משרעת) מגודרת של P3. תא הרכישה בוצעה עם cytometer זרימה. כל חלקה מייצגת את האירועים 20,000. קיו, אני מתכתבת המוטנטים אך זה לא נגמר, איל Aβ42, בהתאמה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 4 . כימות של רמות החלבון הסלולר. איור זה מציג שהכלים המערבי של שברים חלבון הכולל של מוטציות Aβ42-GFP לאחר 16 h של ביטוי cerevisiae ס. יוצרי PI גרסאות צבועות בירוק, אלה המופצות diffusely ב ציטוזול נצבעים בצבע אדום בהיר. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 5 . סטרס חמצוני הסלולר תאיים GFP פלורסצנטיות, רמות החלבון הסלולר למוטאנטים Aβ42-GFP שנקבעו לאחר 16 h של ביטוי cerevisiae ס. המשתנים מיוצגים בציר ה-x כבר הורה על פי שלהם מספק שלעצמו צבירת החזוי באמצעות AGGRESCAN (פאנל שמאלי) או טנגו (לוח נכון). יוצרי PI גרסאות צבועות בירוק, גרסאות ללא PI-יוצרי נצבעים בצבע אדום בהיר. א. B. גרפים בר אלה מציגות את ערכי פלורסצנטיות בדיקה של סטרס חמצוני מתקבל על ידי ניתוח FC של המוטציות Aβ42-GFP. ג. ד. גרפים בר אלה מייצגים את רמות החלבון כפי לכמת על ידי ניתוח densitometry תספיג באמצעות תוכנת ImageJ. E. F. גרפים בר אלה מציגות את ערכי פלורסצנטיות GFP לאחר ניתוח FC. קווי השגיאה עבור סטרס חמצוני בדיקה ומייצגות ערכים פלורסצנטיות GFP המקדם של סטיית (CV) של התאים ממותגת FC. קווי השגיאה עבור רמות ביטוי חלבונים מייצגים ± SE (n = 3). אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| ערוץ | Fluorophore | גל עירור (אן אם) | הלהקה מסנן |
| FITC | GFP | 488 | 530/30 |
| APC | CellRox | 635 | 660/20 |
| PerCP | ה-IP | 488 | 585/42 |
טבלה 1. מקור לייזר, הלהקה לעבור מסננים ו fluorophores בשימוש זרימה cytometer.
| קרינה פלואורסצנטית GFP | קורות חיים | CellROX קרינה פלואורסצנטית | קורות חיים | |
| A | 9316 | 96.4 | 1964 | 127.5 |
| C | 9709 | 91.9 | 1275 | 172.2 |
| D | 11213 | 101.7 | 3443 | 155.8 |
| E | 12256 | 101.1 | 3220 | 152.2 |
| F | 3010 | 96.1 | 1245 | 146.4 |
| G | 11541 | 97.2 | 2947 | 158 |
| H | 7895 | 98.2 | 3582 | 120.8 |
| . אני | 7365 | 97.2 | 1416 | 141.3 |
| K | 10839 | 100.4 | 3102 | 122.1 |
| L | 7605 | 96.9 | 1401 | 161.8 |
| M | 8149 | 96 | 1308 | 170.4 |
| N | 12741 | 97.5 | 3403 | 134.9 |
| P | 9768 | 102.8 | 2629 | 143.6 |
| Q | 13066 | 91.3 | 3354 | 169.9 |
| R | 8537 | 101.3 | 2839 | 127.5 |
| S | 12053 | 99.1 | 3313 | 174.3 |
| T | 10615 | 97.7 | 2213 | 107.7 |
| V | 9169 | 96.1 | 1878 | 121.7 |
| W | 1715 | 94.7 | 1531 | 100 |
| Y | 7574 | 94.5 | 1234 | 138 |
בטבלה 2. רשימת הערכים GFP פלורסצנטיות, סטרס חמצוני בדיקה קרינה פלואורסצנטית מתקבל על ידי ניתוח FC של תאי שמרים לבטא Aβ42-GFP מוטציות עבור ה 16 טבלה זו מציגה את עוצמת קרינה פלואורסצנטית רשע (MFI), את קורות חיים עבור כל מוטציה.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
מגוון רחב של מחלות קשורה להצטברות של חלבונים misfolded לתוך הסלולר פיקדונות6,7,8,33. מאמצים רבים נעשו כדי לפענח את המנגנונים המולקולריים המפעילות את התפרצות של מחלות אלו באמצעות גישות חישובית, אשר לא עשה חשבון חלבון ריכוזים, או במבחנה מתקרב, שבו ריכוז חלבון נשארת קבועה במהלך התגובה. עם זאת, בתוך התא, חלבונים נמצאים כל הזמן מסונתז והשפילו בסביבה צפופה, אי-הומוגניות. זה מסביר את הסתירות תכופים בין סיליקו, חוץ גופית בתוך, ויוו צבירת ומאפייני של חלבונים amyloidogenic.
ישנן מספר סיבות מדוע דגמי הסלולר פשוטים, כגון שמרים, הם בחירה סבירה ללמוד הצבירה משויך רעילות של חלבונים הקשורים למחלות ניווניות בהקשר ביולוגי יותר. למשל, הם נותנים לנו את האפשרות לנתח את השפעת חלבון צבירה misfolding על אוכלוסיה הסלולר שלם באמצעות ניתוח מהיר כמו זו מיושמת כאן. עם זאת, אנחנו צריכים לשקול כי רמות חמצון עשויים להיות שונים בין זני שמרים. לפיכך, כדי להשוות בין זנים או אפילו בין חלבונים שונים, חמצון ו reductants, כגון DTT diamine ב, אמור לשמש להקים בקנה מידה של חמצון/צמצום.
בדוגמה המומחשות במאמר זה, ניתוח bioinformatic, חלבון תאית כמת, חלבון לוקליזציה הדמיה וניתוח FC בו זמנית של פעילות ה-GFP רמות סטרס חמצוני משולבים כדי לזהות את מולקולרית מינים אחראי לנזק חימצוני שהפיק תגובות צבירת חלבון. התוצאות להפגין על גרסאות Aβ42 מסיס יותר, יותר מאשר את יותר צבירת מועדת, לקדם את סטרס חמצוני הגבוהה ביותר. זה מצביע על המין diffusible להיות מסוכן יותר Aβ42 מינים בתוך vivo. רעילות נמוכה יותר של צבירה גרסאות נראה להגיב גם על העובדה כי אלה הייצורים החיים הן מבודדות לתוך PI וגם כדי שפלותם מועדף על ידי המנגנון באיכות החלבון, וכתוצאה מכך רמות החלבון נמוכה יותר מזו של שלהם מסיסים עמיתיהם.
השיטה המתוארת אינה מוגבלת לניתוח של סטרס חמצוני המיוצר על ידי מינים המצטברים/מסיסים Aβ42 ולא ניתן להחיל גם ללמוד מגוון רחב של הפרעות צבירת חלבון. יתר על כן, הטכניקה עשוי להיות שימושי לנטר את ההשפעה של צבירת מעכבי על הסלולר סטרס חמצוני, המאפשר להתעלם ליישומים קליניים נוספים אלה מולקולות המקדמות ההצטברות של חלבון פעיל חמצוני מינים בסופו של דבר, כל עוד בדיקה fluorogenic זמין, הגישה מציעה הזדמנויות יוצא דופן כדי לזהות את הזן המולקולרי האחראי על אפקטים רעילים אחרים הקשורים צבירת חלבון ב מהיר דרך קלה, מתן נתונים כמותיים.
Subscription Required. Please recommend JoVE to your librarian.
Acknowledgments
Materials
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
References
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration? Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast? Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).