Summary
蛋白質の集合は、細胞の酸化ストレスを引き出します。このプロトコルでは、アミロイド蛋白質の細胞内の状態およびフローサイトメトリーを使用して、それらに関連付けられている酸化ストレスを監視する方法について説明します。アミロイド β ペプチドの水溶性で集計しやすい亜種の挙動を調べるためのアプローチが使用されます。
Abstract
タンパク質のフォールディングとアミロイドの立体構造に集約は、発症といくつかの神経変性疾患の進行に関連しています。しかし、少し情報はまだどのように不溶性タンパク質について集計はその毒性で生体を発揮します。細菌や酵母などの単純な原核生物と真核生物のモデル生物は、細胞内アミロイド線維形成、集計の伝達、および毒性の背後にあるメカニズムの私達の現在の理解に大きく貢献しています。このプロトコルでは、酵母の使用は、タンパク質凝集体の形成と細胞の酸化ストレスに与える影響との関係を分析するモデルとして記述されます。メソッドは、フローサイトメトリー (FC) を使用して、式から生じる細胞の酸化損傷の定量化とアミロイド蛋白質の細胞内の可溶性/集計状態の検出を兼ね備えています。この方法は、簡単、迅速、かつ定量的です。研究は、彼らのそれぞれの組み込み集計性癖とアミロイド β ペプチド亜種の大規模なセットによって引き起こされる細胞の酸化ストレスを関連付けることによってテクニックを示しています。
Introduction
プロテオステイシスはセルのフィットネスと老化のプロセスの基本的な決定要因です。ネットワーク、正しいリフォールディング conformers の誤って折りたたまれたタンパク質のシャペロンおよびその対象となるタンパク質がいくつか保存されているメカニズム1 を確保することを目的と高度な品質管理によって細胞の蛋白質の恒常性を維持します。 ,2,3,4,5。多数の調査では、発症および人間の病気の広い範囲とのプロテオステイシス、タンパクにつながる障害の進行の間のリンクをサポートします。例えば、タンパク質の堆積物の存在の多くの神経変性疾患、アルツハイマー病、パーキンソン病などとハンチントン病6,7、8、病理学的特徴であります。prionogenic 病と非変性アミロイドーシス9。それは、凝集反応の初期のオリゴマーと protofibrillar のアセンブリが混雑した細胞環境の10の他の蛋白質と異常な相互作用を確立する細胞毒性の主な成分エリシターであることが示唆されています。また、その毒性効果11,12を伝播する、細胞間タンパク質介在物 (PI) を送信できます。したがって、それは、PI の形成可能性があります確かに危険な集約された種の存在を処理または主要な副作用のない蓄積があるなどこのセルに特定の場所に制限する解毒機構を構成するかもしれない13,14。
標準的な体外生化学的アプローチは、凝集反応およびそのプロパティ15,16種に重要な洞察を提供しています。しかし、これらの試金で使用される条件は、細胞内で発生する明確に異なると、したがって、その生理学的な妥当性を質問します。タンパク質品質管理、オートファジーは、細胞の酸化還元状態の17,18真核生物19,20,21 の規制など携帯電話経路の注目すべき保全のため ,22,23, 出芽酵母酵母(出芽酵母) 蛋白質の集合の分子決定要因を研究する特権を持つ単純な細胞モデルとして浮上しています。関連する生物学的環境24,25,26で関連付けられた細胞毒性影響。
タンパク質凝集傾向は、本質的に主なシーケンスでエンコード機能です。識別と集計推進の力の評価に基づくアミロイド様構造の形成を予測できるため、ポリペプチド27の地域。しかし、タンパク質の体外集計プロパティを予測するバイオインフォマティクス アルゴリズムの成功にもかかわらずこれらの性癖が生体内で細胞毒性影響に変換する方法を予測には程遠いです。体系的に特定の蛋白質の集合の状態とその関連付けられている細胞の損傷間のリンクに対応する研究はこの計算の制限を回避するために役立つことがあります。この接続は、単一の残渣でのみが異なるが、凝集体内性癖28の連続した範囲を表示するアミロイド β ペプチド Aβ42 の亜種の大規模なセットを利用して、本研究で扱われます。特に、酵母細胞の凝集を起こしやすい蛋白質によって誘発される酸化的損傷の会計構造の種を識別するために FC ベースのアプローチを説明します。方法論は、シンプルさ、高処理能力と正確な定量的測定など多くの利点を提供します。このアプローチでは、PI プレイ酸化ストレスに対する保護の役割を確認可能。
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1酵母文化や蛋白発現。
注: a β 亜種展示位置 19 (Phe19) Aβ42 ペプチド (図 1 a) の 1 つ残基変異による異なる相対集計性癖。これらのペプチドの亜種は緑色蛍光タンパク質 (GFP) 集計記者 (図 1)29として機能するとタグ付けされます。
- BY4741 親の背景 (マタ彼3Δ1ルー2Δ0に会った15Δ0 ura3Δ0)30酵母に 20 Aβ42 GFP 亜種のプラスミドを変換します。GSSGSSG リンカーによって GFP の融合 Aβ42 変異体の各プラスミドをエンコードします。プラスミッドは pESC(-URA) であり、選択可能なマーカー URA329を含んでいます。
- ウラシル (SC 浦) で以下のコンポーネントなし合成の完全なメディアの準備: 1.9 グラム/L の 20% (w/v) ブドウ糖の 100 mL と組み合わせて酵母窒素ウラシル、硫酸アンモニウム 5 g/L、900 mL ddH2O のも基本。
- 変換後の酵母細胞のコロニーの SC 裏培 2% ブドウ糖 20 mL を入れ、210 rpm で攪拌下 30 ° C で一晩の文化を育てます。
- 次の日、一晩かけて培養の 100 μ L 接種培 SC 裏の 5 mL に新しい酵母細胞文化を成長します。
- 0.5 の590を OD、4 分間 3,000 x g で文化を遠心、上澄みを廃棄し、2% ラフィノース培 SC 裏の同じ容積の細胞を再懸濁します。
- 210 rpm で 30 分間攪拌下 30 ° C で孵化させなさい。4 分間 3,000 x g で細胞を遠心分離し、上澄みを廃棄します。組換えタンパク質の発現を誘導するために 2% ガラクトースを含む新鮮な SC 裏中細胞を再懸濁します。
- SC 裏中 2% ガラクトースを含むタンパク質の発現を誘導する 16 時間後 4 分 3,000 × g で遠心分離によって滅菌遠心管の細胞の 1 つの mL を収穫します。
注: 最も関連性の違いは、(図 2 b) 細胞内遺伝子組換え蛋白質の分布を決定するための蛍光顕微鏡で目視できる Aβ42 GFP のバリエーションを表現する 16 h. による酵母細胞の誘導期後に発生します。
2. 細胞染色
Note: 新鮮な非誘導細胞は FC 解析中に蛍光のしきい値を確立するネガティブ コントロールとして必要です。
- 16 h による酵母細胞を取るし、590 で彼らの光学密度を決定する nm (外径590).0.1 の外径590に滅菌リン酸緩衝生理食塩水 (PBS) でそれらを希釈します。
- リン酸緩衝生理食塩水 x 1 を次のように準備: NaCl、KCl、Na2HPO4、1.44 g 0.2 g の 8 g を追加し、KH2PO4 ddH2o. の 800 mL の 0.24 g pH 7.4 に調整後最大 1 L ddH2O のソリューションを入力塩酸フィルター 0.2 μ m のフィルターを通してバッファー。
- 適切にラベル付けされた管 (12 × 75 mm 丸底ポリスチレン) を表現する細胞懸濁液を転送し、光から身を守る。彼らは非誘導し、非染め色細胞と一緒に分析の流れの cytometer でロードする準備ができていることを確認します。
- 最終濃度 5 μ M で各サンプルに酸化ストレスのプローブ (例えば、 CellROX 深い赤) を追加し、暗闇の中で 30 分間 30 ° C で細胞をインキュベートします。
- インキュベーション後、1x PBS で 3 回細胞を洗浄し、FC で分析する前にバッファーの同じボリュームにそれらを再懸濁します。
3. フローのフローサイトメトリー解析
注: は、GFP や酸化ストレス プローブ蛍光シグナルを検出するのに適切なレーザーとフィルターを FC セットアップを使用します。流れの cytometer GFP の検出のための 488 nm の青色レーザーと酸化ストレス プローブの蛍光性の検出の 635 nm の赤色レーザーを使用ことができます。GFP および酸化ストレス プローブの発光蛍光の買収がそれぞれ 530/30 nm BP フィルターと 660/20 BP フィルターで実行されます (表 1)。
- 新しいワークシート パネルを開くをクリックし、次の取得ドット プロットを作成します。
- 散布プロット ツールをツールバーから選択し、作成プロット変数側散布領域 (SSC-A)、y 軸対前方の散布面積の x 軸上線形スケールで (FSC A) します。
- ツールバーから散布プロット ツールをクリックし、変数 FL1 エリア (FITC) x 軸対上にプロットを作成します。FL3 エリア (APC) y 軸に対数スケールで。
- 楽器の設定アイコンをクリックします。[補正] タブをクリックし、すべての補正レベルを設定します。[取得] タブをクリックし、記録される 20,000 イベントの合計数を選択します。
- 低流量を切り替える流量アイコンをクリックします。
- 非誘導、無染色の細胞を実行を開始する [取得] タブをクリックし、人口は中央左の象限に分布するまでの前方の機器の設定および側方散乱で電圧を調整します。
- 細胞の残骸を除くセル人口のまわりの領域 R1 を設定するポリゴンアイコンをクリックし、使用するこのゲート人口 P1 = R1 すべて蛍光ドット プロット、ヒストグラム表現 (図 3)。
- 蛍光信号の PMT 電圧を調整するには、左下腹部に分布しているセルまで計測器の設定タブで、利得をチューニング、FL1 FL3 ドット プロットで無染色の細胞を実行します。
- GFP の蛍光を測定する誘導された細胞にサンプルを変更します。計測器の設定タブで、人口は右下の象限に分布するとき、FSC A対FL1 (FITC) のゲインを設定します。ゲート (ゲート P2) 肯定的な細胞集団を定義します。
- 変更非誘導サンプル染色 FSC A対FL3 (APC) ドット プロット蛍光による酸化ストレスを表示するセルです。左上腹部にセル人口を分散までゲインを調整します。ゲート P3 で肯定的な細胞の集団。
- 細胞蛍光を表すヒストグラムアイコンと 2 つのヒストグラム プロットを作る。蛍光強度を表す、FL1 (FITC) GFP 蛍光を表す x 軸にあり FL3 (APC) 蛍光性を表すは、y 軸に対数目盛で P2 と P3 の人口をそれぞれ適用を確認します。
メモ: ヒストグラムのオーバーレイは、バリエーション a β を発現する細胞の蛍光プロファイルの違いを説明するために良い方法です。
4. 組換え蛋白質バイオアセッテイ
- バイオマーカ発現量を調べるには、16 時間 6 分、4,000 x g で遠心分離によるイースト文化を収穫し、-80 ° C で細胞ペレットを格納
- PBS の 16 h の組換え蛋白を発現する収集した細胞を再懸濁します。20 の外径590に各 Aβ42 GFP 変異体の 200 μ L の細胞懸濁液を準備します。
- 総蛋白分画分析のため 20 分間 14,000 × g で遠心分離によって各変異体の 100 μ L を収穫し、酵母の換散バッファー Y の同じボリュームで細胞ペレットを再懸濁します-(50 mM トリス塩酸 pH 8.0、1 %dmso、200 mM の NaCl、1 mM EDTA を含む 10 mg/mL SB3 14 (myr あたりistyl スルホベタイン) を添加した 1 mM 特異フッ化物 (PMSF))。
- 軽度の動揺の下で室温で 20 分間サンプルをインキュベートします。
- ブラッドフォードの試金を使用して蛋白質の抽出濃度を決定します。15% アクリルアミド SDS-PAGE の電気泳動のゲルや、100 V 60 分のポリフッ化ビニリデン (PVDF) 二フッ化膜にしみの各サンプルのタンパク質抽出物の最大 5 μ g をロードします。
- 100 mg Coomassie ブリリアント ブルー G-250 の試薬 95% エタノール 50 mL に溶解し、85% (w/v) リン酸の 100 mL を追加ブラッドフォードを準備します。その後、染料が完全に解散したとき ddH21 L O との混合物を希釈し、使用直前のセルロース フィルター ペーパーを通してフィルターします。
注: ブラッドフォード試薬は茶色光する必要があります。 - ウシ血清アルブミン (BSA) ブラッドフォード試薬の 1,000 μ L 中のタンパク質の 5-100 μ g に至る標準を準備します。
- 1-10 μ L タンパク質のブラッドフォード試薬 1 ml を抽出し、5 分間室温で混合物をインキュベートを追加します。
- 基準及び外径595タンパク質抽出試料の吸光度を測定します。
- 分析に標準対で得られた吸光度値でプロットを作る。蛋白質の μ g。標準曲線に基づいて、あればボリュームと、希釈を考慮したタンパク質の量から元のサンプルの濃度を決定します。
- 100 mg Coomassie ブリリアント ブルー G-250 の試薬 95% エタノール 50 mL に溶解し、85% (w/v) リン酸の 100 mL を追加ブラッドフォードを準備します。その後、染料が完全に解散したとき ddH21 L O との混合物を希釈し、使用直前のセルロース フィルター ペーパーを通してフィルターします。
- 0.1% 含有トリス緩衝生理食塩水 (TBS) x 1 と膜をすすぎトゥイーン 20 (TTBS) とブロック 1 x TTBS で 5% (w/v) 脱脂乾燥したミルクを使用しては。
- 10 x TBS の 1 L を準備、基本トリスの 24 g と H2Oddの 900 mL の塩化ナトリウムの 88 g を溶解、7.6 に pH を調整します。DdH2O 1 L の最終巻を追加します。1 x ソリューションの ddH2o. の 9 の部分と 10 x の在庫の 1 部分をミックスします。
- 一次 β-アミロイド抗体希釈 6E10 縮尺で 1 h の膜をインキュベートし、二次抗体のヤギと室温で 1 時間抗マウス IgG HRP 共役希釈 1:10, 000。
- Luminata 蛍光試薬 1 mL を使用して膜を開発して手順 5.2 デンシトメトリー分析によってバンドを定量化します。
5. データの解析
- FC によって得られたデータを解析する (MFI) 平均蛍光強度を表示するテーブルを作成して、対応する標準のエラーおよび/または GFP 蛍光および酸化ストレス レベルの差異 (CV) の係数と平均蛍光。
- インスペクターのタブと各チャネルに必要な統計データをカスタマイズするのには [統計] アイコンをクリックします。
注: 蛋白質の変形を比較すると、折り畳み式の背景または解像度指標 (RD) に蛍光値データに変換します。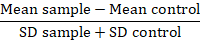
この指標で平均サンプル問題サンプルの平均蛍光、平均のコントロールは、コントロールとして使用するセルの平均蛍光あり SD は平均蛍光の標準偏差。RD 要因は MFI 値よりもより正確な指標だけでなくサンプルもデータの広がりのためのアカウントの違いを計算します。時間をかけて行ったさまざまな実験からのデータを比較する場合、この計算は具体的に関連します。RDs が計算されると、統計的検定の使用は蛋白質の変形の観察の違いの重要性を確認することをお勧め。
- インスペクターのタブと各チャネルに必要な統計データをカスタマイズするのには [統計] アイコンをクリックします。
- タンパク質バイオアセッテイ用ウエスタンブロット ImageJ ソフトウェアを使用して組換え蛋白質のレベルを定量化します。
- デンシトメトリーの分析の前に開発された膜のオリジナルの raw 画像を JPEG ファイル形式とグレースケール モードに変換します。
- 平均グレー値を [分析] メニューの設定測定セクションを調整します。
- ImageJ から長方形ツールをクリックし、バンドのデンシトメトリーの分析のための一貫性のあるサイズの利益 (率 ROI) の領域を定義します。作成された長方形フレーム内膜イメージの各バンドのセンターし、Ctrl + M (測定コマンド) を使用して 1 つずつ測定値を記録します。
- 同じ ROI 領域を適用すること、各バンドに隣接したローカルのバック グラウンドを測定します。測定後、各バンドは個別から対応するローカル背景を減算します。
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
このプロトコルでは、Phe19 がすべて天然蛋白質構成アミノ酸28変異されてどこ Aβ42 ペプチドの 20 個の商品のコレクションを使用する方法について説明します。これらの蛋白質の集計の理論的な性癖は、2 つの異なるバイオ情報アルゴリズム (AGGRESCAN とタンゴ31,32) を使用して分析できます。両方のケースでこの分析を配し、一般的なルールとして疎水性残基と最低最高値荷電と極ものに集約傾向の進歩的なグラデーションをレンダリングします (図 1 b、 1 C)。
実験的 Aβ42 のバリエーションの細胞内凝集状態を追跡するため記述されている実験のプラスミド、Aβ42 のエンコーディングと GFP を融合した (図 1 a) ガラクトース誘導 GAL1 の制御の下での変換が必要酵母。16 h の誘導期間の後 Aβ42 の亜種を発現する酵母細胞は、蛍光顕微鏡の下で視覚化できます。この可視化分析のコレクションから 20 個の商品から 10 に PI の形成を確認することが可能になります。数と蛍光の凝集体のサイズは異なります (図 2) バリアントからバリアント型です。図 2 aは、それぞれ 2 つの独立した文化の 500 セルの合計から PI の定量化を示しています。ここでは、我々 は優れた契約予測を観察できるおよび生体内の集計プロパティ。
タンパク質凝集体の形成は、この細胞系における酸化ストレスをトリガーできるかどうかに対処するため、このプロトコルは活性酸素の指標として蛍光発生プローブの蛍光と共に細胞の GFP 蛍光をモニターする FC を使用してください。種 (ROS) 生産。図 3は、手順を示しています、Gln とイルの突然変異、均一分散と PI 形成 Aβ42 のバリエーションには、それぞれ対応する結果が得られました。
まず、分析セル人口はゲート (P1) FSC A対SSC のドット プロット解析 (図 3 a) から細胞の残骸を削除します。P1 の人口の酸化ストレス プローブ対GFP の蛍光信号が p3 の緑の蛍光細胞のゲート、ドット プロット グラフで表される (図 3 b)。図 3および3 Dヒストグラムの平均と各蛍光マーカーの中央蛍光性を定量化するために CV (表 2) を含む表現された各々 の統計データを取得するために作成を示します。
すべての突然変異体は、単一のアミノ酸 (Aβ42 GFP シーケンスの 0.02%) が異なりますので、同じレベルで表現されると予想されます。ただし、プロテオステイシス機械は、その特定の集計性癖に差動方法で応答できます。したがって、細胞抽出物の蛋白質の表現のレベルは、a β 抗体 (図 4) を用いて定量化が。我々 は、一般的な傾向として PI 形成 Aβ42 の亜種が存在するそれらの残りの細胞質で均一分散よりも低いレベルで参照してください。
その酸化ストレス プローブ蛍光、蛋白質のレベル、および GFP 蛍光特性は PI とその組み込み集計性癖 (を形成する能力を基準にして表されるとき、Aβ42 の亜種間で重要な違いを観察できます。図 5)。集約より受けやすい PI 形成の亜種が (図 5 a、 5 b) より水溶性に比べて多くの低い酸化ストレスを引き出します。これらの結果は、サイズと個々 のセル (図 2) と酸化ストレス レベルごとの集計数と明白な相関関係はありません。(図 5、 5 D) 細胞質に分布してゆくものよりも低いレベルにあるセルの PI 形成変形、特に変異位置 19、トリプトファンとフェニルアラニンを軸受があります。これに例外は Thr 変異株細胞の 10% 未満が PI を形成(図 1 b、 1 C)。これは実際に最も凝集を起こしやすい亜種が酵母の品質管理の低下機械28によって選択的にクリアされていることに対応します。GFP 蛍光および蛋白質レベルより水溶性の Aβ42 フォームのためによくはなく、PI (図 5E 5 階) を形作るそれらを関連付けます。これはおそらく、これらの細胞集団で蛍光に由来する 2 つの異なるコンパートメント、細胞質および介在物, および変異体34間合計蛍光相対的貢献度が異なるためにです。

図 1.すべて天然アミノ酸による Aβ42 ペプチドで 19 位の残基の変異から派生した Aβ42 GFP 融合コンストラクトのコレクションの集約傾向分析。A. この画像 (Aβ42、グレーのリンカーによって GFP に wt Aβ42 融合の 3 D モデルを示しています。GFP、緑)、オワンクラゲから緑色蛍光タンパク質の PDB 1EMA と wt Aβ42 の Phe19 の側鎖は赤で示されているアルツハイマー病 a β ペプチドの 2OTK に基づきます。ソフトフェアのソフトウェアを使用してモデルを作成します。残留 19 Aβ42 シーケンスは、中央疎水性クラスターの中央位置を占めています。バー グラフ、2 つの異なるバイオ情報予測で作成されます: B。 AGGRESCAN、 C. タンゴ。この図の拡大版を表示するのにはここをクリックしてください。

図 2.Aβ42 GFP PI 形成亜種の酵母誘導式の 16 h の文化。A. バー グラフが 2 つの生物学的複製のバリエーションごとに 500 蛍光セルの合計から計算された円周率の異なる番号を含む細胞の割合を示します。B. これらのイメージは、選択された Aβ42 GFP バリアント (Phe、イル、スレオニン、トリプトファン) の代表的な蛍光顕微鏡画像。彼らは紫外線励起フィルターを用いた GFP (450-500 nm) と発光範囲 (515 560 nm) の下で買収されました。スケール バーを表す 10 μ m.この図の拡大版を表示するのにはここをクリックしてください。

図 3.フロー (FC) フローサイトメトリー解析 o 方式f出芽酵母 Aβ42 GFP の選択されたバリアントを発現する細胞。A。 ドット プロットでゲート酵母 (P1) の表 (FSC A対SSC A) セル人口から細胞残骸を除去します。FC のドット プロット (均一分散) Gln とイル (PI 加工) 亜種の顕微鏡画像は横に表示されます。スケール バーは、10 μ m. Bを表します。これらの散布ドット プロット イメージを表す GFP A対酸化ストレス プローブ (P3) のゲートの人口が含まれているのみ蛍光セルには背景信号を除きます。携帯周波数ヒストグラムは、 Cです。P1 とDからゲート GFP の信号 (振幅 FITC)。CellROX (APC 振幅) P3 からゲートします。流れの cytometer 細胞取得を行った。各プロットは、20,000 イベントを表します。Q、私はそれぞれ Gln とイル Aβ42 変異体に対応します。この図の拡大版を表示するのにはここをクリックしてください。

図 4.細胞のタンパク質レベルの定量化。出芽酵母における発現の 16 時間後の Aβ42 GFP 変異体の総蛋白の西部のしみを示します。PI 形成変形は緑色で着色されているし、これらの細胞質に分布するびまん性は薄い赤で着色されています。この図の拡大版を表示するのにはここをクリックしてください。

図 5.細胞の酸化ストレス、細胞内の GFP 蛍光、16 h での式の後に決定 Aβ42 GFP 変異体の細胞のタンパク質レベル酵母。X 軸で表される亜種は、AGGRESCAN (左のパネル)、タンゴ (右側のパネル) の予測集計性癖によると命ぜられています。緑色で着色されている PI 形成変形と非 PI 形成変形が薄い赤で着色されています。A。B. これらのバーグラフ表示 Aβ42 GFP 変異体の FC 解析によって得られた酸化ストレス プローブ蛍光値。C。D. これらのバー グラフ タンパク質レベルを表すは、ImageJ ソフトウェアを使用して西部のしみのデンシトメトリーの分析量。E。F. これらの棒グラフが FC 分析後 GFP 蛍光値を表示します。プローブの酸化ストレスの誤差範囲と GFP 蛍光値は FC ゲート細胞の差異 (CV) の係数を表します。タンパク質の発現レベルの誤差範囲は ± SE を表す (n = 3)。この図の拡大版を表示するのにはここをクリックしてください。
| チャネル | 蛍光体 | 励起波長 (nm) | 帯域通過フィルター |
| FITC | GFP | 488 | 530/30 |
| APC | CellRox | 635 | 660/20 |
| PerCP | IP | 488 | 585/42 |
表 1。レーザー ソース、バンドパス フィルター、およびフルオロ流れの cytometer で使用します。
| GFP 蛍光 | CV | CellROX 蛍光 | CV | |
| A | 9316 | 96.4 | 1964 | 127.5 |
| C | 9709 | 91.9 | 1275 | 172.2 |
| D | 11213 | 101.7 | 3443 | 155.8 |
| E | 12256 | 101.1 | 3220 | 152.2 |
| F | 3010 | 96.1 | 1245 | 146.4 |
| G | 11541 | 97.2 | 2947 | 158 |
| H | 7895 | 98.2 | 3582 | 120.8 |
| 私 | 7365 | 97.2 | 1416 | 141.3 |
| K | 10839 | 100.4 | 3102 | 122.1 |
| L | 7605 | 96.9 | 1401 | 161.8 |
| M | 8149 | 96 | 1308 | 170.4 |
| N | 12741 | 97.5 | 3403 | 延長 134.9 |
| P | 9768 | 102.8 | 2629 | 143.6 |
| Q | 13066 | 91.3 | 3354 | 169.9 |
| R | 8537 | 101.3 | 2839 | 127.5 |
| S | 12053 | 99.1 | 3313 | 1,743億 |
| T | 10615 | 97.7 | 2213 | 107.7 |
| V | 9169 | 96.1 | 1878 | 121.7 |
| W | 1715 | 94.7 | 1531 | 100 |
| Y | 7574 | 94.5 | 1234 | 138 |
表 2.GFP 蛍光および 16 のため Aβ42 GFP 変異体を発現する酵母細胞の FC 解析によって得られる酸化ストレス プローブ蛍光の値のリストこの表は、平均蛍光強度 (MFI) と各変異体の CV。
Subscription Required. Please recommend JoVE to your librarian.
Discussion
病気の広い範囲は、携帯電話預金6,7,8,33への誤って折りたたまれたタンパク質の蓄積にリンクされます。アカウント タンパク質濃度を考慮していない、計算のアプローチを使用してこれらの病気の発症を引き起こす分子メカニズムを解明するため多くの努力がなされたまたは体外に近づくと、タンパク質濃度反応中に一定している残る。細胞内タンパク質が常に合成し、混雑と非均質環境で劣化します。これは頻繁にアミロイド蛋白質のインシリコ、 in vitroとin vivoの集計プロパティの不一致を説明します。
複数の理由は酵母などのシンプルな携帯電話モデル集計を勉強する合理的な選択より生物学的文脈で神経変性疾患関連タンパク質の毒性を関連付けられているがあります。例えば、彼らは私たちにここで 1 つ実装など高速解析を使用してそのまま携帯電話人口のタンパク質もって集合の影響を分析する可能性を与えます。しかし、我々 は酵母の菌株の中で酸化のレベルが異なる場合があることを検討してください。したがって、系統、もしくは異なる蛋白質の間の比較、酸化剤、還元剤、DTT やジアミン類などが使用する必要が酸化/還元スケールを確立しますある。
分子を識別するこの資料で、バイオ情報解析、細胞タンパク質定量例では統合されている、タンパク質局在イメージングと GFP 活動と酸化ストレス レベルの同時 FC 分析タンパク質凝集反応によって誘発される酸化的損傷に関与する種。結果より水溶性の Aβ42 亜種ではなくより集約しやすい、最高の酸化ストレスを促進することを示します。これより危険な Aβ42 種の生体をされている拡散性の種を指しています。亜種を集約する低い毒性はその可溶性のものより低い蛋白質レベルで生じるタンパク質品質機械によって PI にこれらの構造を隔離する事実とその優遇の劣化の両方で対応するよう対応。
説明メソッドは、Aβ42 集計/水溶性種によって生成される酸化ストレスの解析に限定しない、さまざまなタンパク質凝集障害を研究にも適用することができます。さらに、技術はさらに臨床応用の酸化活性蛋白質種の蓄積を促進するそれらの分子を破棄できるように、細胞の酸化ストレスに及ぼす凝集阻害剤を監視する役に立つかもしれない。最後に、蛍光プローブが使用できる限り、アプローチは定量的なデータを提供する、簡単な方法の高速タンパク質凝集にリンクされている他の毒性作用に関与する分子種を識別するために素晴らしい機会を提供します。
Subscription Required. Please recommend JoVE to your librarian.
Acknowledgments
Materials
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
References
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration? Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast? Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).