Summary
Protein aggregering utløser mobilnettet oksidativt stress. Denne protokollen beskriver en metode for overvåking intracellulær statene amyloidogenic proteiner og oksidativt stress forbundet med dem, ved hjelp av flowcytometri. Tilnærmingen til å studere atferden til løselig og aggregering utsatt varianter av amyloid-β peptid.
Abstract
Protein misfolding og aggregering i amyloid konformasjonen har vært knyttet til utbruddet og progresjon av flere nevrodegenerative sykdommer. Men det er fortsatt litt informasjon om hvordan uløselig protein aggregater utøve deres toksiske effekter i vivo. Enkel prokaryotic og eukaryotic modell organismer, som bakterier og gjær, har bidratt betydelig til vår nåværende forståelse av mekanismene bak intracellulær amyloid dannelse, aggregater forplantning, og toksisitet. I denne protokollen, er bruk av gjær beskrevet som en modell for å analysere forholdet mellom dannelsen av protein aggregater og deres innvirkning på mobilnettet oksidativt stress. Metoden kombinerer påvisning av intracellulær løselig/samlet status for en amyloidogenic protein med kvantifisering av mobilnettet oksidative skader som følge av sitt uttrykk ved hjelp av flowcytometri (FC). Denne tilnærmingen er enkel, rask og kvantitative. Studien viser teknikken ved å samkjøre mobilnettet oksidativt stress forårsaket av et stort sett av amyloid-β peptid varianter med sine respektive iboende samling propensities.
Introduction
Proteostasis er en grunnleggende determinant av celle helse og aldring prosesser. I celler vedlikeholdes protein homeostase av sofistikert protein kvalitetskontroll nettverk rettet å sikre den riktige refolding av misfolded protein conformers chaperones og/eller deres målrettede proteolyse med flere godt bevarte mekanismer1 ,2,3,4,5. En rekke studier gir støtte til koblingen mellom utbruddet og utviklingen av en rekke menneskelige sykdommer og svikt i proteostasis, fører til protein misfolding og aggregering. For eksempel tilstedeværelsen av protein innskudd er ansett som en patologisk kjennetegner mange nevrodegenerative sykdommer, som Alzheimers, Parkinsons, og Huntingtons sykdommer6,7,8, prionogenic sykdommer og ikke-degenerative amyloidoses9. Det har blitt foreslått at tidlig oligomeric og protofibrillar samlinger i samling reaksjonen er de viktigste elicitors av cytotoksisitet, etablere avvikende interaksjoner med andre proteiner i overfylte mobilnettet miljø10. I tillegg kan protein Inneslutninger (PI) overføres mellom celler, spre sine toksisk effekt11,12. Derfor kan det være at dannelsen av PI kan faktisk utgjør en detoxifying mekanisme som begrenser tilstedeværelsen av farlig aggregert arter til bestemte steder i cellen, der de kan behandles eller akkumulert uten store bivirkninger 13 , 14.
Standard i vitro biokjemiske tilnærminger har gitt viktig innsikt i de ulike artene som fyller aggregering reaksjoner og deres egenskaper15,16. Men betingelsene i disse analyser er klart forskjellig fra de som forekommer i cellen, og derfor spørsmålet relevansen fysiologiske. På grunn av kjente bevaring av mobilnettet som protein kvalitetskontroll, autophagy eller regulering av mobilnettet redoks staten17,18 blant eukaryoter19,20,21 ,22,23, spirende gjær Saccharomyces cerevisiae (S. cerevisiae) har dukket opp som en privilegert enkel mobilnettet modell å studere molekylære determinanter av protein aggregering og tilknyttede cytotoksiske innvirkning på biologisk relevante miljøer24,25,26.
Protein aggregering tilbøyelighet er en funksjon iboende kodet i primære sekvensen. Dermed dannelsen av amyloid-lignende strukturer kan forutsies basert på identifisering og evaluering av styrken på aggregering-fremme regioner i polypeptides27. Men til tross for bioinformatic algoritmer å forutsi egenskapene for aggregering i vitro protein sekvenser, er de fortsatt langt fra prognoser hvordan disse propensities oversette til i vivo cytotoksiske innvirkning. Studier som tar koblingen mellom et bestemt protein aggregert staten og dens tilknyttede cellular skade på en systematisk måte kan bidra til å omgå denne beregningsorientert begrensningen. Denne tilkoblingen behandles i studien, drar nytte av en rekke varianter av amyloid-β peptid Aβ42 forskjellig fra en enkelt rester, men viser en kontinuerlig rekke aggregering propensities i vivo28. Spesielt er en FC-basert tilnærming til til conformational arten regnskap for oksidative skader brakt frem av aggregering utsatt proteiner i gjærceller beskrevet. Metodikken gir mange fordeler som enkelhet, høy gjennomstrømming evne og nøyaktig kvantitativ måling. Dette gjorde det mulig å bekrefte som PI spiller en beskyttende rolle mot oksidativt stress.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. S. cerevisiae kulturer og Protein uttrykk
Merk: Aβ varianter forevise annet relative aggregering propensities på grunn av en mutasjon i en enkelt rester i posisjon 19 (Phe19) av Aβ42 peptid (figur 1A). Disse peptid variantene er merket med grønne fluorescerende protein (GFP), som fungerer som en samling reporter (figur 1)29.
- Transformere plasmider koding for 20 Aβ42-GFP varianter i gjærceller med BY4741 foreldrenes bakgrunn (MATa hans3Δ1 leu2Δ0 møtte15Δ0 ura3Δ0)30. Hver plasmider koder for en Aβ42 mutant smeltet til GFP av et GSSGSSG linker. Plasmider er pESC(-URA) og inkluderer valgbar markøren URA329.
- Forberede det syntetiske komplett mediet uten uracil (SC-URA) med følgende komponenter: 1,9 finans gjær nitrogen base uten uracil, 5 finans ammonium sulfate og 900 mL ddH2O kombinert med 100 mL 20% (w/v) glukose.
- One koloni av de transformerte gjærceller inn 20 mL SC-URA mediet som inneholder 2% glukose og vokse kulturer overnatting på 30 ° C under omrøring på 210 rpm.
- Neste dag, vokse nye gjær celler kulturer av vaksinere 100 µL av natten kultur i 5 mL av fersk SC-URA medium.
- På et OD590 på 0,5, sentrifuge kulturer 3000 x g for 4 min, forkaste nedbryting og resuspend cellene i samme volum av fersk SC-URA medium som inneholder 2% raffinose.
- Ruge på 30 ° C under omrøring på 210 rpm for 30 min. Deretter sentrifuge cellene på 3000 x g for 4 min og forkaste nedbryting. Resuspend cellene i frisk SC-URA medium som inneholder 2% galaktose å indusere rekombinante proteiner uttrykket.
- Etter 16 h for å indusere protein uttrykk i SC-URA medium som inneholder 2% galaktose, høste 1 mL indusert cellene i sterilt microcentrifuge rør med sentrifugering 3000 x g for 4 min.
Merk: Den mest relevante forskjellen oppstår etter at en induksjon periode 16 h. utførte gjærceller uttrykke Aβ42-GFP varianter kan visualiseres fluorescens mikroskopi til fordelingen rekombinante proteiner i cellene (figur 2B).
2. celle flekker
Merk: Friske ikke-indusert celler kreves som en negativ kontroll etablere en fluorescerende terskel under en FC analyse.
- Ta 16 h-indusert gjær cellene og bestemme deres optisk densitet ved 590 nm (OD590). Fortynne dem i et sterilt fosfat bufret saltvann (PBS) til en OD590 0,1.
- Forberede 1 x fosfat bufret saline slik: Legg til 8 g NaCl, 0.2 g KCl, 1,44 g Na2HPO4, og 0,24 g KH2PO4 800 mL av ddH2O. fylle løsningen 1 L ddH2O etter justere pH 7,4 HCl. filter bufferen gjennom en 0,2 µm.
- Overføre uttrykke celle suspensjoner til riktig merket rør (12 × 75 mm runde bunn polystyren) og beskytte dem mot lyset. Kontroller at de er klare til å lastes inn på flyt cytometer for analyse, samt ikke-indusert og ikke-farget celler.
- Legg oksidativt stress sonden (f.eks CellROX dyp rød) til hvert utvalg i en siste konsentrasjon av 5 µM og ruge cellene på 30 ° C i 30 min i mørket.
- Etter inkubasjon vaskes cellene 3 ganger med 1 x PBS og resuspend dem i samme volum bufferen før deres analyse av FC.
3. flyt cytometri analyse
Merk: Bruk en FC oppsett med passende lasere og filtre for å oppdage GFP og oksidativt stress sonde fluorescens signal. En flyt cytometer utstyrt med en 488 nm blå laser for påvisning av GFP og en 635 nm rød laser for påvisning av oksidativt stress sonde fluorescens kan brukes. Oppkjøpet av utslipp fluorescens GFP og oksidativt stress sonde er utført med et 530/30 nm BP filter og 660/20 BP filter, henholdsvis (tabell 1).
- Klikk på åpne nye regneark og opprette følgende oppkjøpet dot tomter.
- Velg Scatter Plot verktøy på verktøylinjen og skape et plott med variablene Side Scatter-området (SSC-A) på den y vs frem Scatter-området (FSC-A) i en lineær skala på x-aksen.
- Klikk Punktdiagram Plot verktøyet fra verktøylinjen og opprette et plott med variablene FL1-området (FITC) på x-aksen vs. FL3-området (APC) i en logaritmisk skala på y-aksen.
- Klikk ikonet Instrumenter . Klikk kategorien kompensasjon og angi alle kompensasjonsnivåer. Klikk på fanebladet Kjøp og velg et totalt antall av 20.000 hendelser som skal registreres.
- Klikk Flow Rate -ikonet for å bytte infusjonshastigheten til lav.
- Klikk kategorien anskaffe å kjøre ikke-indusert, unstained kvinne cellene og justere spenningen i Instrument-innstillingene til fram og siden scatter til befolkningen er distribuert i midten venstre kvadrant.
- Klikk på ikonet Polygon til å angi et område R1 rundt celle befolkningen unntatt celle rusk og denne gated befolkningen P1 = R1 for alle fluorescerende dot tomter og histogrammet representasjoner (Figur 3).
- For å justere avdrag spenningen av fluorescens, kjører du unstained kvinne cellene i FL1-FL3 dot tomten, tuning gevinst i kategorien Instrument innstillingen til i nedre venstre kvadrant distribueres cellene.
- Endre utvalget til indusert cellene til å måle GFP fluorescens. Angi forsterkningen i den FSC-A vs. FL1 (FITC) i kategorien Instrument innstillingen når befolkningen distribueres i kvadranten nederst til høyre. Definere positiv celle befolkningen med gate (Gate P2).
- Endre utvalget til den ikke-indusert farget celler skal vise oksidativt stress av fluorescens på en FSC-A vs. FL3 (APC) dot-tomt. Tune gevinsten til celle befolkningen er distribuert i venstre-øvre kvadranten. Gate positiv celle befolkningen i P3.
- Gjøre to histogrammet tomter med ikonet histogrammet representerer cellen fluorescens. Representerer fluorescens intensiteten, sørg for at FL1 (FITC) som representerer GFP fluorescens i x-aksen, og FL3 (APC) som representerer fluorescensen er i y-aksen, bruke P2 og P3 bestander på en logaritmisk skala, henholdsvis.
Merk: Histogrammet overlegg er en god måte å illustrere forskjellene mellom fluorescens profiler av celler som uttrykker ulike Aβ varianter.
4. rekombinante proteiner Immunodetection
- For å bestemme protein uttrykk nivå, høste gjær kulturer indusert 16 h av sentrifugering 4000 x g for 6 min, og lagre celle pellets på-80 ° C.
- Resuspend det avhentet celler uttrykke rekombinante proteiner 16 h i PBS. Forberede 200 µL celle suspensjoner av hver Aβ42-GFP mutert til en OD590 20.
- For total protein fraksjon analyse, høste 100 µL av hver mutant med sentrifugering 14.000 x g for 20 min og resuspend celle pellets i samme volum av gjær lyseringsbuffer Y-PER (50 mM Tris-HCl pH 8.0, 1% DMSO 200 mM NaCl, 1 mM EDTA inneholder 10 mg/mL SB3-14 (myr istyl sulfobetaine) supplert med 1 mM phenylmethylsulfonyl fluor (PMSF)).
- Inkuber prøvene for 20 min ved romtemperatur under mild agitasjon.
- Bestemme protein ekstrakt konsentrasjonen ved hjelp av Bradford analysen. Laste opp til 5 µg av protein ekstrakt av hvert utvalg på 15% akrylamid SDS-side geleelektroforese gel og klatt på en polyvinylidene difluoride (PVDF) membran 100 V for 60 min.
- Forberede en Bradford reagens med 100 mg av Coomassie briljant blå G-250 oppløst i 50 mL av 95% etanol og legge til 100 mL 85% (w/v) fosforsyre. Når fargestoff er fullstendig oppløst, fortynne blandingen med ddH2O til 1 L og filtrere det gjennom cellulose filter papir like før bruk.
Merk: Bradford reagensen bør være lys brun. - Forberede en standard av bovin serum albumin (BSA) fra 5 til 100 µg protein i 1000 µL av Bradford reagensen.
- Legge til 1-10 µL av protein pakke til 1 mL av Bradford reagensen og Inkuber blandingen ved romtemperatur for 5 min.
- Måle absorbansen av standarder og protein ekstra samples ved OD595.
- For analyse, kan du gjøre et plott med absorbansen verdiene hentes for standarder vs. µg protein. Basert på standardkurven, bestemme konsentrasjonen av det opprinnelige prøvene fra mengden protein, vurderer volum og fortynning, eventuell.
- Forberede en Bradford reagens med 100 mg av Coomassie briljant blå G-250 oppløst i 50 mL av 95% etanol og legge til 100 mL 85% (w/v) fosforsyre. Når fargestoff er fullstendig oppløst, fortynne blandingen med ddH2O til 1 L og filtrere det gjennom cellulose filter papir like før bruk.
- Skyll membranen med 1 x Tris-bufret saltvann (SS), 0,1% mellom 20 (TTBS), og blokkere det med 5% (w/v) nonfat tørr melk i 1 x TTBS.
- For å forberede 1 L 10 x TBS, oppløse 24 g Tris base og 88 g av NaCl i 900 mL av H2Odd og justere pH til 7,6. Legg ddH2O til en endelig mengde 1 L. For en 1 x løsning, bland 1 del av 10 x lager med 9 deler av ddH2O.
- Inkuber membranen 1t med en primær β-amyloid antistoff 6E10 fortynnet 1:1,000 og 1t ved romtemperatur med en sekundær antistoff geit anti-musen IgG-HRP-konjugert fortynnet 1:10, 000.
- Utvikle membraner bruker 1 mL av Luminata fluorescerende reagens og kvantifisere band av densitometric analyse som beskrevet i trinn 5.2.
5. analyse
- Analysere dataene innhentet av FC, opprette en tabell som viser gjennomsnittlig fluorescerende intensiteten (MFI) og median fluorescens med sin tilsvarende standardfeil og/eller koeffisient av varians (CV) GFP fluorescens og oksidativt stress nivåer.
- Klikk kategorien inspektør og statistikk -ikonet for å tilpasse de statistiske dataene som kreves for hver kanal.
Merk: Når du sammenligner protein varianter, konvertere fluorescens verdier data til en folde-over bakgrunn eller oppløsning beregning (RD):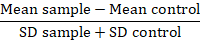
I denne beregningen, mener prøven er den bety fluorescensen problem prøver, mener kontrollen er den bety fluorescensen celler som brukes som en kontroll og SD er standardavviket for de mener fluorescensen. RD faktor er en mer nøyaktig indeks enn MFI verdien når det ikke bare beregner forskjellen mellom prøvene, men også står for spredning av data. Denne beregningen er spesielt relevant når sammenligne data fra forskjellige eksperimenter utført over tid. Når RDs er beregnet, er bruk av en statistisk test tilrådelig å anerkjenne betydningen av de observerte forskjellene mellom protein varianter.
- Klikk kategorien inspektør og statistikk -ikonet for å tilpasse de statistiske dataene som kreves for hver kanal.
- For protein immunodetection, kvantifisere rekombinante proteiner nivåene ved Western blot bruke ImageJ programvare.
- Før densitometry analyse, kan du konvertere de opprinnelige raw-bildene av utviklet membranen til JPEG fil format og gråskala-modus.
- Justere delen Angi målinger i rullegardinmenyen analyser mener gråverdi.
- Klikk rektangelverktøyet fra ImageJ og definere et område av interesse (ROI) med en enhetlig størrelse for bandet densitometry analyse. Sentrum hvert band på membran bildet inne i den opprettede rammen og Registrer målingen enkeltvis bruke Ctrl + M (måling-kommandoen).
- Måle lokale bakgrunnen ved siden av hvert band bruke samme ROI område. Etter måling, trekk tilsvarende lokale bakgrunnen fra hvert individuelle band.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Denne protokollen beskriver hvordan å ansette en samling av 20 varianter av Aβ42 peptid der Phe19 er mutert til alle naturlige proteinogenic aminosyrer28. De teoretiske aggregering propensities av disse proteinene kan analyseres ved hjelp av to forskjellige bioinformatic algoritmer (AGGRESCAN og TANGO31,32). I begge tilfeller denne analysen gjengir en progressiv gradering av aggregering tendenser, tillegge, som regel de høyeste verdiene hydrofobe rester og den laveste verdien til ladet og polar seg (figur 1B, 1 C).
For å spore eksperimentelt intracellulær aggregering delstaten ulike Aβ42 varianter, krever beskrevet eksperimentet en endring av en plasmider, koding for Aβ42 og smeltet til GFP (figur 1A) under kontroll av den galaktose-induserbart GAL1, i S. cerevisiae. Gjærceller uttrykke Aβ42 varianter etter en induksjon 16 h kan visualiseres under fluorescens mikroskop. Denne effekten gjør det mulig å bekrefte dannelsen av PI i 10 ut av 20 variantene fra samlingen analysert. Antall og størrelsen på de fluorescerende aggregatene varierer fra variant til variant (figur 2). Figur 2A viser PI kvantifiseringen fra totalt 500 celler i hver av de to uavhengige kulturene. Her vi kan observere en utmerket avtale mellom spådd og i vivo aggregering egenskaper.
For å løse om dannelsen av protein aggregater kan utløse oksidativt stress i denne modellen celle systemet, bruker denne protokollen FC overvåke mobilnettet GFP fluorescens sammen med fluorescens av fluorogenic sonden som en indikator på reaktive oksygen arter (ROS) produksjon. Figur 3 illustrerer fremgangsmåten og fikk resultatene for Gln og Ile mutanter, som tilsvarer homogent distribuert og PI-forming Aβ42 varianter, henholdsvis.
Først analysert celle befolkningen er gated (P1) i FSC-A vs. SSC-en prikk plot fjerne celle detritus fra analysis (figur 3A). Fluorescens signalet av GFP vs oksidativt stress probe av befolkningen P1 er representert i dot plottet grafer der grønne fluorescerende celler er gated in P3 (figur 3B). Figur 3 c og 3D viser histogrammer for å få statistiske data for hver uttrykt variant, inkludert CV (tabell 2), for å kvantifisere gjennomsnittet og median fluorescens av hver fluorescerende indikator.
Alle mutanter forventes å bli uttrykt på samme nivå siden de varierer i en enkelt aminosyre (0,02% av den Aβ42-GFP). Men kan proteostasis maskiner svare på en annerledes måte å deres bestemt aggregering propensities. Derfor er protein uttrykk nivåer i mobilnettet ekstrakter kvantifisert ved hjelp av et Aβ-spesifikke antistoff (Figur 4). Vi ser at, som en generell trend, PI-forming Aβ42 varianter finnes på lavere nivåer enn de resterende homogent distribueres stoffer.
Viktige forskjeller blant Aβ42 varianter kan observeres når deres oksidativt stress sonde fluorescens, protein nivå og GFP fluorescens egenskaper er representert i forhold til deres evne til å danne PI og deres iboende samling propensities ( Figur 5). Den mer aggregering-utsatt PI-forming varianter framprovosere mye lavere oksidativ stress enn sine mer løselig kolleger (figur 5A, 5B). Med disse resultatene er det ingen åpenbar sammenheng mellom størrelsen og antall aggregater per enkeltcelle (figur 2) og oksidativt stress nivåer. PI-forming varianter, spesielt mutanter bærer Trp og Phe i posisjon 19, finnes i cellen på lavere nivåer enn de homogenously fordelt på stoffer (figur 5C, 5 D). Unntaket er Thr mutant, som utgjør mindre enn 10% av cellene PI (figur 1B, 1 C). Dette samsvarer med det faktum at de mest aggregering utsatt variantene fjernes selektivt av gjær kvalitetskontroll fornedrelse maskiner28. GFP fluorescens og protein nivå relatere for mer løselige Aβ42 former, men ikke for de danner PI (figur 5E, 5F). Dette er sannsynligvis fordi disse mobilnettet bestander, fluorescensen kommer fra to ulike avdelinger, stoffer og inclusions og deres relative bidrag til den totale fluorescensen varierer mellom mutanter34.

Figur 1 . Aggregering tilbøyelighet analyse for innsamling av Aβ42-GFP fusion konstruksjoner avledet fra mutasjon av rester i posisjon 19 i Aβ42 peptid ved alle naturlige aminosyrer. A. dette bildet viser en 3D-modell av wt Aβ42 smeltet GFP av et linker (Aβ42, grå; GFP, grønn), basert på PDB-1EMA for grønne fluorescerende protein fra Aequorea victoria og 2OTK for Alzheimers Aβ peptid der Phe19 side kjede av wt Aβ42 vises i rødt. Modellen laget med Pymol. Rester 19 i Aβ42 serien har en sentral beliggenhet i sentrale hydrofobe klyngen. Søylediagrammer opprettes med to forskjellige bioinformatic prediktorer: B. AGGRESCAN, C. TANGO. Klikk her for å se en større versjon av dette tallet.

Figur 2 . Aβ42-GFP PI-forming varianter i S. cerevisiae kulturer indusert for 16 h uttrykk. A. stolpediagrammet viser prosentandelen av celler som inneholder et annet antall PI beregnes ut fra totalt 500 fluorescerende celler for hver variant i to biologiske gjentak. B. disse bildene er representativt fluorescerende mikroskopi bilder av valgte Aβ42-GFP varianter (Phe, Ile, Thr, Trp). De ble anskaffet under UV-lys med et eksitasjon filter for GFP (450-500 nm) og et utslipp utvalg (515-560 nm). Baren skala representerer 10 µm. Klikk her for å se en større versjon av dette tallet.

Figur 3 . Ordningen flyt cytometri (FC) analyse of S. cerevisiae celler uttrykke valgte Aβ42-GFP varianter. A. diagrammet viser gated gjærceller (P1) dot tomter (FSC-A vs. SSC-A) der cellen detritus fjernes fra celle befolkningen. Mikroskopiske bilder av Gln (homogent distribuert) og Ile (PI-danne) varianter vises nærmest FC dot tomter. Baren skala representerer 10 µm. B. Bildene scatter dot tomten representerer GFP-A vs. oksidativt stress sonde der befolkningen gated (P3) inneholder bare fluorescerende celler, unntatt bakgrunn signalet. Cellen frekvens histogrammer er c. GFP signalet (FITC amplitude) gated P1 og D. CellROX (APC amplituden) gated 3 vr. Cellen oppkjøpet ble utført med en flyt cytometer. Hver tomt representerer 20.000. Q og jeg svarer til Gln og Ile Aβ42 mutanter, henholdsvis. Klikk her for å se en større versjon av dette tallet.

Figur 4 . Kvantifisering av mobilnettet protein nivå. Denne illustrasjonen viser vestlige blots av de totale protein fraksjonene av Aβ42-GFP mutanter etter 16 h for uttrykk i S. cerevisiae. PI-forming variantene er farget i grønt, og de diffusely fordelt på stoffer er farget i rødt. Klikk her for å se en større versjon av dette tallet.

Figur 5 . Mobilnettet oksidativt stress og intracellulær GFP fluorescens mobilnettet protein nivåer for Aβ42-GFP mutanter bestemmes etter 16 h for uttrykk i S. cerevisiae. Variantene representert på x-aksen er bestilt ifølge deres spådd aggregering propensities av AGGRESCAN (venstre panel) eller TANGO (høyre panel). PI-forming variantene er farget i grønt og ikke-PI-forming variantene er farget i rødt. A. B. disse søylediagrammer viser oksidativt stress sonde fluorescens verdier ved en FC analyse av Aβ42-GFP mutanter. C. D. disse søylediagrammer representerer protein nivåene som kvantifisert ved en Western blot densitometry analyse bruker ImageJ programvare. E. F. disse søylediagrammer viser GFP fluorescens verdier etter en FC analyse. Feilfelt for oksidativt stress sonde og GFP fluorescens verdiene representerer koeffisient av varians (CV) FC-gated cellene. Feilfelt for protein uttrykk nivåer representerer ± SE (n = 3). Klikk her for å se en større versjon av dette tallet.
| Kanal | Fluorophore | Eksitasjon bølgelengde (nm) | Band Pass Filter |
| FITC | GFP | 488 | 530/30 |
| APC | CellRox | 635 | 660/20 |
| PerCP | IP | 488 | 585/42 |
Tabell 1. Laser kilde, båndpass filtre og fluorophores brukes i flyt cytometer.
| GFP fluorescens | CV | CellROX fluorescens | CV | |
| A | 9316 | 96.4 | 1964 | 127.5 |
| C | 9709 | 91.9 | 1275 | 172.2 |
| D | 11213 | 101.7 | 3443 | 155.8 |
| E | 12256 | 101.1 | 3220 | 152.2 |
| F | 3010 | 96.1 | 1245 | 146.4 |
| G | 11541 | 97,2 | 2947 | 158 |
| H | 7895 | 98,2 | 3582 | 120.8 |
| Jeg | 7365 | 97,2 | 1416 | 141.3 |
| K | 10839 | 100,4 | 3102 | 122.1 |
| L | 7605 | 96.9 | 1401 | 161.8 |
| M | 8149 | 96 | 1308 | 170.4 |
| N | 12741 | 97,5 | 3403 | 134.9 |
| P | 9768 | 102,8 | 2629 | 143.6 |
| Q | 13066 | 91,3 | 3354 | 169.9 |
| R | 8537 | 101.3 | 2839 | 127.5 |
| S | 12053 | 99,1 | 3313 | 174.3 |
| T | 10615 | 97.7 | 2213 | 107.7 |
| V | 9169 | 96.1 | 1878 | 121.7 |
| W | 1715 | 94,7 | 1531 | 100 |
| Y | 7574 | 94,5 | 1234 | 138 |
Tabell 2. Liste over verdier for GFP fluorescens og oksidativt stress sonde fluorescens ved FC analyse av gjærceller uttrykke Aβ42-GFP mutanter for 16 h. Denne tabellen viser mener fluorescens intensiteten (MFI) og CV for hver mutant.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
En rekke sykdommer knyttet til opphopning av misfolded proteiner i mobilnettet innskudd6,7,8,33. Mange forsøk har vært gjort å rakne molekylære mekanismer som utløser utbruddet av disse sykdommer med computational tilnærminger, som ikke tar hensyn til kontoen protein konsentrasjoner, eller i vitro tilnærminger, som protein konsentrasjonen forblir konstant under reaksjonen. Men i cellen, er proteiner stadig syntetiseres og degradert i et overfylt og ikke-homogene miljø. Dette forklarer hyppige avvik mellom i sili, i vitro, og vivo aggregering egenskaper amyloidogenic proteiner.
Det er flere grunner Hvorfor enkle mobilnettet modeller, som gjær, er et fornuftig valg å studere aggregering og tilhørende toksisitet av proteiner relatert til nevrodegenerative sykdommer i en mer biologisk sammenheng. For eksempel, gir de oss muligheten til å analysere virkningen av protein misfolding Oppsamling på en intakt mobilnettet befolkning med rask analyser som den implementert her. Men bør vi vurdere at oksidativt nivåer kan variere mellom gjær påkjenningen. Således, for å sammenligne mellom stammer, eller selv mellom ulike proteiner, oksidanter og reductants, som DTT og diamine, skal brukes til å etablere en oksidasjon/reduksjon skala.
Eksemplet i denne artikkelen, bioinformatic analyse, mobilnettet protein kvantifisering, er protein lokalisering bildebehandling og samtidige FC analyse av GFP aktivitet og oksidativt stress nivåer integrert for å identifisere den molekylære arter ansvarlig for oksidative skader brakt frem av protein aggregering reaksjoner. Resultatene viser at mer løselig Aβ42 variantene, snarere enn den mer aggregering utsatt, fremme det høyeste oksidativt stresset. Dette peker på diffusible arter blir mer farlig Aβ42 arter i vivo. Lavere toksisitet av aggregering varianter synes å svare til det faktum at disse konformasjonen er sequestered i PI og deres fortrinnsrett nedbrytning av protein kvalitet maskiner, som resulterer i lavere protein nivåer enn deres løselig kolleger.
Metoden beskrevet er ikke begrenset til analyse av oksidativt stress produsert av Aβ42 samlet/løselig arter og kan også brukes for å studere en rekke protein aggregering lidelser. Videre er kan teknikken være nyttig å overvåke effekten av aggregering hemmere på mobilnettet oksidativt stress, slik at forkaste for ytterligere klinisk bruk disse molekyler som fremmer akkumulering av oksidativt aktive protein arter. Til slutt, så lenge en fluorogenic sonde er tilgjengelig, tilbyr tilnærming bemerkelsesverdige muligheter til å identifisere molekylær arter ansvarlig for andre giftige effekter knyttet til protein samling i en rask en enkel måte, gir kvantitative data.
Subscription Required. Please recommend JoVE to your librarian.
Acknowledgments
Materials
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
References
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration? Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast? Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).