Summary
Protein toplama hücresel oksidatif stres ortaya çıkarır. Bu iletişim kuralı amyloidogenic proteinlerin hücre içi Birleşik ve onlarla akış sitometresi kullanarak ilişkili oksidatif stres izlemek için bir yöntem açıklanır. Yaklaşım β-amiloid peptid çözünür ve toplama eğilimli türevleri davranışını incelemek için kullanılır.
Abstract
Protein misfolding ve toplama amiloid biçimler içine başlangıçlı ve ilerleme çeşitli nörodejeneratif hastalıklar ile ilgili olduğu. Ancak, hala çok az bilgi var hakkında nasıl çözünmez protein toplamları onların toksik etkileri içinde vivouygulamayın. Basit prokaryotik ve ökaryotik canlılar, bakteri ve Maya, gibi önemli ölçüde mevcut anlayışımız mekanizmaları hücre içi amiloid dil oluşumu, toplamları yayma ve toksisite arkasında katkıda bulunmuştur. Bu protokol için maya kullanımı protein toplamları oluşumu ve hücresel oksidatif stres üzerindeki etkileri arasındaki ilişkiyi incelemek için bir model olarak tanımlanıyor. Yöntem bir amyloidogenic protein çözünür/toplanan hücre içi durumunu algılama akış sitometresi (FC) kullanarak kendi ifadeden kaynaklanan hücresel oksidatif hasar miktar ile birleştirir. Bu basit, hızlı ve nicel yaklaşımdır. Çalışma tekniği β-amiloid peptid türevleri ile onların anılan sıraya göre iç Toplama eğilimlerini büyük bir set neden hücresel oksidatif stres birleştiriliyor tarafından gösterilmektedir.
Introduction
Proteostasis hücre fitness ve yaşlanma süreçlerinin temel bir belirleyici olduğunu. Hücrelerde, protein homeostazı sofistike protein kalite kontrol doğru misfolded protein conformers chaperones ve/veya onların hedeflenen proteolizis tarafından birkaç iyi korunmuş mekanizmalarıyla1 refolding emin olmak için ağlar amaçlayan tarafından yapılmaktadır ,2,3,4,5. Çok sayıda çalışmalar başlangıçlı ve geniş bir insan hastalıkları ve proteostasis, protein misfolding ve toplama için önde gelen başarısızlık arasındaki bağlantı için destek sağlar. Örneğin, protein mevduat varlığı çok nörodejeneratif hastalıkları, Alzheimer, Parkinson, gibi ve Huntington hastalıkları6,7,8patolojik damgasını olarak kabul edilir, prionogenic hastalıklar ve sigara dejeneratif amyloidoses9. Toplama tepki erken oligomeric ve protofibrillar derlemelerde diğer proteinler ile anormal etkileşim içinde kalabalık hücresel ortamı10kurulması ana elicitors, sitotoksisite, çoğu sürülmüştür. Buna ek olarak, protein kapanımlar (PI) onların toksik etkisi11,12yayılıyor hücreler arasında aktarılabilir. Bu nedenle, PI oluşumu gerçekten tehlikeli toplanan türlerin varlığı belirli konumlara hücresinde, nerede onlar olabilir işlenen veya önemli yan etkileri olmadan birikmiş kısıtlar detoxifying bir mekanizma teşkil olabilir 13 , 14.
Standart vitro biyokimyasal yaklaşımlar toplama reaksiyonlar ve onların özellikleri15,16doldurmak farklı türlerin önemli anlayışlar hazırladık. Ancak, bu deneyleri içinde kullanılan koşulları açıkça farklı hücre içinde meydana gelen ve bu nedenle, kendi fizyolojik alaka soru. Protein kalite kontrol, autophagy veya Düzenleme Ökaryotlar19,20,21 arasında hücresel redoks devlet17,18 , gibi hücresel yolları önemli korunması nedeniyle ,22,23, tomurcuklanma Maya Saccharomyces cerevisiae (S. cerevisiae) protein toplama moleküler belirleyicileri çalışmaya ayrıcalıklı basit bir hücresel model çıkmıştır ve ilişkili sitotoksik etkisini de biyolojik olarak uygun ortamlar24,25,26.
Protein toplama eğilimi doğal olarak birincil sırada kodlanmış bir özelliktir. Böylece, amiloid benzeri yapıların oluşumu kimlik ve toplama teşvik potens değerlendirilmesi dayalı tahmin edilebilir polipeptitler27bölgelerde. Ancak, protein sequences vitro toplama özelliklerini tahmin etmek için bioinformatic algoritmaları başarı rağmen bunlar hala bu eğilimlerini vivo içinde sitotoksik etki nasıl tercüme tahmini çok uzak. Belirli bir protein toplanan durumunu ve onun ilişkili hücresel hasarı arasındaki bağlantıyı sistematik bir şekilde ele alan çalışmalar Hesaplamalı bu sınırlamayı aşmak için yardımcı olabilir. Bu bağlantı yalnızca tek bir kalıntı farklı ama toplama eğilimlerini vivo içinde28sürekli bir dizi görüntüleme β-amiloid peptid Aβ42 türevleri büyük bir set yararlanarak bu da çalışmanın, yöneliktir. Maya hücreleri toplama eğilimli proteinler tarafından elde edildi oksidatif hasar için muhasebe konformasyon türler tanımlamak için FC dayalı bir yaklaşım özellikle de açıklanmıştır. Metodoloji basitlik, yüksek üretilen iş yetenekleri ve doğru nicel ölçüm gibi birçok avantaj sağlar. Bu yaklaşım o PI oksidatif strese karşı koruyucu bir rol oynar teyit mümkün kıldı.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. S. cerevisiae kültür ve Protein ifade
Not: Aβ türevleri farklı göreli toplama eğilimlerini de Aβ42 peptid (Şekil 1A) 19 (Phe19) konumunu tek bir kalıntı bir mutasyon nedeniyle sergi. Bu peptid çeşitleri bir toplama muhabir (Şekil 1)29davranan floresan yeşil protein (GFP), ile etiketlenir.
- Maya hücreleri BY4741 ebeveyn arka plan (MATa onun3Δ1 leu2Δ0 bir araya geldi15Δ0 ura3Δ0)30ile 20 Aβ42-GFP türevleri için kodlama plazmid dönüşümü. Her plazmid GFP için GSSGSSG bağlayıcı tarafından erimiş bir Aβ42 mutant için kodlar. Plazmid pESC(-URA) ve seçilebilir işaret URA329içerir.
- Sentetik tam orta urasil (SC-URA) aşağıdaki bileşenleri içeren olmadan hazırlamak: 1.9 g/L Maya azot urasil, 5 g/L amonyum sülfat ve 900 mL GKD2O temel kombine ile 100 mL % 20 (w/v) glikoz.
- Dönüştürülmüş Maya hücreleri bir koloni % 2 glikoz içeren SC-URA orta 20 mL koymak ve kültürler gecede 30 ° C'de ajitasyon 210 devirde altında büyür.
- Ertesi gün, yeni Maya hücreleri kültürleri 5 mL taze SC-URA orta de aşı 100 µL gecede kültür tarafından büyümek.
- Bir OD590 0,5, 3000 x g 4 dk için de kültürler santrifüj kapasitesi, süpernatant atmak ve aynı birimin taze SC-URA Orta % 2 raffinose içeren hücrelerde resuspend.
- 30 dk için 210 devirde ajitasyon altında 30 ° C'de kuluçkaya. Sonra 4 dk 3000 x g de hücreleri santrifüj kapasitesi ve süpernatant atın. Taze SC-URA orta Rekombinant protein ifade ikna etmek için % 2 galaktoz içeren hücrelerde resuspend.
- 16 h SC-URA Orta % 2 galaktoz içeren protein ifadede inducing sonra Santrifüjü 3000 x g 4 dk için de tarafından 1 mL steril microcentrifuge tüpler indüklenen hücre hasat.
Not: 16 h. Induced Maya hücreleri Aβ42-GFP türevleri ifade bir indüksiyon döneminde Rekombinant protein dağıtım hücrelere (Şekil 2B) belirlemek için floresans mikroskobu tarafından görüntülenmiştir sonra en alakalı farklılıklar ortaya.
2. hücre boyama
Not: Taze sigara kaynaklı hücreler bir FC çözümleme sırasında bir floresan eşiği belirlemek için bir negatif kontrol olarak gereklidir.
- 16 h kaynaklı Maya hücreleri almak ve 590 optik onların yoğunluğuna belirlemek nm (OD590). Onları steril fosfat tamponlu tuz (PBS) 0.1 OD590 için sulandırmak.
- 1 fosfat tamponlu tuz x aşağıdaki gibi hazırlamak: NaCl, KCl, Na2HPO4, 1.44 g 0.2 g 8 g ekleyin ve KH2PO4 GKD2O. 800 ml 0,24 g doldurun 1 L-GKD2O kadar çözüm pH 7.4 için ayarladıktan sonra HCl. ile arabellek 0.2 µm filtreden filtre.
- Uygun şekilde etiketlenmiş tüpler (12 × 75 mm yuvarlak alt Polistiren) ifade hücre süspansiyonlar aktarmak ve onları ışıktan korumak. Akış Sitometresi Analizi, Sigara kaynaklı ve lekeli olmayan hücreleri ile birlikte için üzerinde yüklü olması için hazır olduklarından emin olun.
- Her örnek 5 mikron son bir konsantrasyon, oksidatif stres sonda (Örneğin, CellROX derin kırmızı) ekleyin ve karanlıkta 30 dk 30 ° C'de hücreler kuluçkaya.
- Kuluçka sonra 3 kez 1 x PBS içeren hücreleri yıkama ve onları arabellek onların analiz FC tarafından daha önce aynı hacmine resuspend.
3. Akış Sitometresi Analizi
Not: bir FC Kur uygun lazerler ve filtreler ile GFP ve oksidatif stres sonda floresans sinyal algılamak için kullanır. GFP tespiti için 488 nm mavi lazer ve oksidatif stres sonda floresans tespiti için 635 nm kırmızı lazer ile donatılmış bir akış sitometresi kullanılabilir. Emisyon floresans GFP ve oksidatif stres sonda edinimi yapılır 530/30 nm BP filtre ve 660/20 BP filtrenin sırasıyla (Tablo 1).
- Yeni çalışma sayfası panelini açma tıklayın ve aşağıdaki edinme nokta araziler oluşturun.
- Araç çubuğundan Dağılım çizim aracı seçin ve bir komplo ile yan dağılım-alan (SSC-A) y ekseni vs üzerinde ileri dağılım alanı (FSC-A) x ekseni üzerinde doğrusal bir ölçek değişkenlerinin.
- Araç çubuğundan Dağılım çizim aracını tıklatın ve x ekseni vsFL1-alan (FITC) değişkenleri ile bir çizim oluşturma. Fl3-alan (APC) y ekseni Logaritmik bir ölçekte.
- Aletleri ayarları simgesini tıklatın. Tazminat sekmesini tıklatın ve tüm tazminat düzeyleri ayarlayın. Satın alma sekmesinde tıklatın ve kaydedilecek 20.000 olayların toplam sayısı seçin.
- Akış hızı düşükolarak geçmek için Akış hızı simgesini tıklatın.
- Sigara kaynaklı, günahı hücreleri yayınlanmaya başlaması için Al sekmesini tıklatın ve nüfus orta-sol çeyreğinde dağıtılır kadar ileri Araç ayarları ve yan dağılım gerilim ayarlayın.
- Hücre popülasyonu hücre artıkları dışında bir bölgesini R1 ayarlamak için Çokgen ikonuna tıklayın ve bu geçişli nüfus P1 kullanın R1 = tüm floresan nokta araziler ve çubuk grafik gösterimleri (Şekil 3).
- Floresans sinyalinin Devresel_ödeme gerilimi ayarlamak için hücreleri içinde sol alt çeyrekte dağıtılır kadar kazanç Araç ayarı sekmesinde ayarlama FL1-FL3 nokta Arsa günahı hücreleri çalıştırın.
- Örnek GFP floresans ölçmek için indüklenen hücrelere değiştirin. Nüfus sağ alt çeyreğinde dağıtıldığında Araç ayarı sekmesinde kazanç FSC-A vs FL1 (FITC) olarak ayarlayın. Bir kapı (Gate P2) ile pozitif hücre nüfus tanımlayın.
- Değişiklik olmayan indüklenen için örnek bir FSC-A vs FL3 (APC) nokta arsa üzerinde floresans tarafından oksidatif stres görüntülemek için hücreleri lekeli. Hücre popülasyon sol üst çeyreğinde dağıtılır kadar kazanç ayarlayın. P3 pozitif hücre kalabalıkta bir kapı.
- Hücre floresans temsil etmek için iki çubuk grafik bir cenaze dört nikah çubuk grafik simge olun. Floresan yoğunluğu temsil etmek için FL1 (FITC GFP floresans temsil eden) x ekseni ise FL3 (APC) Floresan temsil eden y ekseni, P2 ve P3 nüfusu sırasıyla logaritmik bir ölçekte uygulama emin olun.
Not: Histogram bindirmeleri floresans profilleri farklı Aβ türevleri ifade hücre arasındaki farkları göstermek için iyi bir yoldur.
4. rekombinant Protein Immunodetection
- Protein ifade düzeylerini belirlemek için Santrifüjü 4.000 x g 6 min için de 16 h için indüklenen maya kültürleri hasat ve hücre granül-80 ° C'de depolayın
- 16 h PBS için Rekombinant protein ifade kendine hakim hücre resuspend. 200 µL hücre süspansiyonlar, her Aβ42-GFP mutant bir OD590 20 için hazırlayın.
- Toplam protein fraksiyonu analiz, her mutant 100 µL Santrifüjü 14.000 x g 20 dk için de tarafından hasat ve Maya lizis arabellek Y aynı birim hücre granül resuspend-(50 mM Tris-HCl pH 8.0, % 1 DMSO, 200 mM NaCl, 1 mM EDTA içeren 10 mg/mL SB3-14 (en düşük ücret istyl sulfobetaine) takıma 1 mM phenylmethylsulfonyl florür (PMSF)).
- Örnekleri hafif ajitasyon altında oda sıcaklığında 20 dk için kuluçkaya.
- Protein özü konsantrasyon Bradford tahlil kullanarak belirleyin. % 15 akrilamid SDS-sayfa Elektroforez jel ve leke üzerine polivinilidin difluoride (PVDF) membran 100 V 60 dk için her örneğinin protein ekstresinin 5 µg yükleyin.
- Bir Bradford reaktifi ile Coomassie parlak mavi G-250 100 mg 50 mL % 95 etanol çözünmüş ve 100 mL % 85 (w/v) fosforik asit ekleyin hazırlayın. O zaman, boya tamamen çözüldüğünde, GKD2O 1 m'ye karışımla sulandırmak ve selüloz filtre kağıdı kullanmadan önce aracılığıyla filtre.
Not: Bradford reaktifi olmak hafif kahverengi. - Sığır serum albumin (BSA) 1.000 µL Bradford reaktif proteinin 5-100 µg arasında değişen bir standartta hazırlayın.
- 1-10 µL protein Bradford reaktifi 1 mL için ayıklamak ve 5 min için oda sıcaklığında karışımı kuluçkaya ekleyin.
- Standartları ve protein özü örnekler, OD595absorbans ölçmek.
- Analiz için bir arsa için standartlar vselde absorbans değerleriyle olun. µg protein. Standart eğri dayalı, protein, miktarı üzerinden orijinal örnekleri konsantrasyonları hacmi ve seyreltme, varsa göz önünde bulundurarak belirleyin.
- Bir Bradford reaktifi ile Coomassie parlak mavi G-250 100 mg 50 mL % 95 etanol çözünmüş ve 100 mL % 85 (w/v) fosforik asit ekleyin hazırlayın. O zaman, boya tamamen çözüldüğünde, GKD2O 1 m'ye karışımla sulandırmak ve selüloz filtre kağıdı kullanmadan önce aracılığıyla filtre.
- Membran ile 1 x Tris arabelleğe alınmış serum fizyolojik (TBS), % 0,1 durulama ara 20 (TTBS) ve %5 (w/v) yağsız Kuru süt 1 x TTBS kullanarak engelleyiniz.
- 10 x TBS 1 litre hazırlamak için 24 g Tris temel ve NaCl 88 g H2Odd 900 ml dağıtılması ve pH 7,6 için ayarlayın. GKD2O 1 L. son bir birime ekleme 1 x çözüm için 10 x hisse senedi, 1 kısım GKD2O. 9 parçaları ile karıştırın.
- Membran bir birincil β-amiloid antikor seyreltilmiş 6E10 1:1,000 ile 1 h için kuluçkaya ve ikincil antikor keçi ile Oda sıcaklığında 1 h için 1:10, 000 Anti-fare IgG-HRP eşlenik seyreltilmiş.
- 1 mL Luminata floresan reaktif kullanarak membranlar geliştirmek ve grup tarafından densitometric analiz 5.2 adımda anlatıldığı gibi ölçmek.
5. veri analizi
- FC tarafından elde edilen verileri analiz etmek için Ortalama floresan yoğunluğu (MFI) görüntüleyen bir tablo oluşturmak ve onun karşılık gelen standart hata ve/veya GFP floresans ve oksidatif stres düzeyleri farkı (MF) katsayısı ile ortalama Floresans.
- Müfettiş sekmesini ve her kanal için gerekli olan istatistiksel verileri özelleştirmek için İstatistikleri simgesini tıklatın.
Not: protein türevleri karşılaştırılırken, katlanır arka plan veya çözünürlük metrik (RD) Floresan veri değerleri dönüştürür: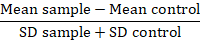
Bu ölçüm, sorun örneklerinin ortalama floresans kötü örnek olduğunu, bir denetim olarak kullanılan hücre ortalama floresans ortalama denetimidir ve SD ortalama floresans standart sapmasıdır. Sadece örnekleri aynı zamanda veri yaymak için hesapları arasındaki farkı hesaplar gibi RD faktör MFI değerden daha doğru bir dizindir. Zaman içinde farklı deneyler gelen verilere karşılaştırma yürütülen bu hesaplama özellikle uygundur. RDs hesaplanan bir kez bir istatistiksel test kullanımı protein çeşitleri arasında gözlenen farklılıklar önemini onaylamak için tavsiye edilir.
- Müfettiş sekmesini ve her kanal için gerekli olan istatistiksel verileri özelleştirmek için İstatistikleri simgesini tıklatın.
- Protein immunodetection için ImageJ yazılım kullanarak Western blot tarafından Rekombinant protein düzeyleri ölçmek.
- Dansitometresi analiz daha önce geliştirilen membran orijinal raw görüntüleri JPEG dosya biçimi ve gri tonlama moduna dönüştürün.
- Çözümle menüsünde demek grideğerine Ayarla ölçümler kısmında ayarlayın.
- ImageJ dikdörtgen aracını tıklatın ve grup Dansitometresi analiz için tutarlı boyutu ile bir bölgesi (ROI) ilgi tanımlamak. Membran görüntü oluşturulan dikdörtgen çerçeve içindeki her band merkezi ve ölçüm Ctrl + M (ölçü komutu) kullanarak tek tek kaydedin.
- Aynı YG alan uygulama her grup için bitişik yerel arka plan ölçmek. Ölçüm sonra tek tek her gruptan karşılık gelen yerel arka plan çıkarma.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Bu iletişim kuralı 20 değişik-in nereye Phe19 için tüm doğal proteinogenic amino asitler28mutasyona uğrattı Aβ42 peptid topluluğu istihdam edileceğini açıklar. Bu proteinlerin teorik toplama eğilimlerini iki farklı bioinformatic algoritmaları (AGGRESCAN ve TANGO31,32) kullanılarak analiz edilebilir. Her iki durumda da, bu analiz ascribing, genel bir kural olarak, en yüksek değerleri hidrofobik kalıntıları ve en düşük ücret ve kutup olanlar toplama eğilimler, aşamalı bir geçiş oluşturur (Şekil 1B, 1 C).
Deneysel olarak farklı Aβ42 değişik hücre içi toplama durumunu izlemek için Aβ42 için kodlama ve GFP (Şekil 1A) galaktoz-indüklenebilir GAL1 kontrolü altında erimiş bir plazmid dönüşümün açıklanan deneme gerektirir, S. cerevisiae. 16 h bir indüksiyon süre sonra Aβ42 türevleri ifade Maya hücreleri floresan mikroskop altında görüntülenmeyecektir. Bu görselleştirme 10 dışarı-in analiz koleksiyonundan 20 değişik PI oluşumunda onaylamanızı sağlar. Floresan toplamları boyutunu ve sayısını variant variant (Şekil 2) farklı. Şekil 2A iki bağımsız kültür içinde 500 hücrelerin tümü PI miktar gösterilir. Burada, biz mükemmel bir anlaşma arasında tahmin gözlemleyebilirsiniz ve in vivo toplama özellikleri.
Protein toplamları oluşumu bu modeli hücre sisteminde oksidatif stres tetikleyebilir olup olmadığını gidermek amacıyla bu iletişim kuralı ile birlikte fluorogenic sonda floresans reaktif oksijen bir göstergesi olarak hücresel GFP floresans izlemek için FC kullanır türler (ROS) üretim. Şekil 3 yordamı göstermektedir ve homojen dağıtılmış ve PI-şekillendirme Aβ42 türevleri için sırasıyla tekabül eden Gln ve Ile mutantlar için sonuçlar elde.
İlki, bu analiz hücre nüfus geçişli (P1) FSC-A vs SSC-A nokta içinde Arsa hücre zarar analizi (Şekil 3A) kaldırmak. GFP vs oksidatif stres sonda P1 nüfusunun floresans sinyalinin nerede yeşil floresan hücreleri içinde P3 geçişli nokta çizim grafiklerde temsil edilir (Şekil 3B). Şekil 3 c ve 3D ortalama ve floresan her işaretçisi ortalama floresans ölçmek için CV (Tablo 2) de dahil olmak üzere her ifade varyant için istatistiksel verileri elde etmek için oluşturulan çubuk grafikler göster.
Tüm mutantlar bir tek amino asit (Aβ42-GFP sıra %0,02) farklıdırlar beri aynı düzeyde ifade bekleniyor. Ancak, proteostasis makine onların belirli toplama eğilimlerini farklı şekilde yanıt verebilir. Bu nedenle, hücresel özleri protein ifade seviyelerinde bir Aβ özgü antikor (Şekil 4) kullanarak sayısal. Biz genel bir eğilim, PI şekillendirme Aβ42 türevleri daha homojen sitozol içinde dağıtılmış kalan daha alt düzeylerde bulunduğundan, bakın.
Onların oksidatif stres sonda floresan, protein düzeyleri ve GFP floresans özellikleri PI ve kendi içsel toplama eğilimlerini ( oluşturmak için yeteneklerini göreli olarak temsil edilen zaman Aβ42 çeşitleri arasında önemli farklılıklar görülebilir Şekil 5). Daha toplama eğilimli PI oluşturan değişik temin fazla alt oksidatif stres daha çözünür meslektaşlarına (Şekil 5A, 5B). Bu sonuçlar ile boyutu ve tek hücre (Şekil 2) ve oksidatif stres düzeyleri başına toplamları sayısı arasında bariz hiçbir bağlantı vardır. PI oluşturmayan türevleri, özellikle mutantlar Trp ve Phe 19, konumda taşıyan bu rastlanılmaması sitozol (Şekil 5C, 5 D) dağıtılmış daha alt seviyelerde hücre mevcut. Bunun istisnası için hücrelerin % 10'dan daha az PI formu Thr mutant olduğunu (Şekil 1B, 1 C). Bu gerçeği en toplama eğilimli türevleri tarafından Maya kalite kontrol bozulması makine28seçmeli olarak temizlenir ile karşılık gelir. GFP floresans ve protein düzeyleri daha fazla çözünür Aβ42 formları için iyi, ama bu PI (Şekil 5E, 5F) şekillendirme için aralarındaki ilişkileri belirlemektir. Bu çünkü bu hücresel nüfus, iki farklı bölmeleri, sitozol ve kapanımlar floresans gelir ve toplam floresans göreli onların katkıları mutantlar34arasında farklılık gösterir.

Resim 1 . Toplama eğilimi analiz Aβ42-GFP füzyon yapıları topluluğu için elde edilen Aβ42 peptid 19 pozisyonda kalıntısı mutasyon tüm doğal amino asitler tarafından. A. GFP (Aβ42, gri; bir bağlayıcı tarafından için wt Aβ42 erimiş bir 3D model bu görüntü gösterir GFP, yeşil), yeşil flüoresan protein Aequorea Victoria için PDB 1EMA ve 2OTK için wt Aβ42 Phe19 yan zincirinin kırmızıyla gösterilen Alzheimer Aβ peptid dayalı. Model Pymol yazılım kullanılarak oluşturulur. Kalıntı 19 Aβ42 sırayla merkezi hidrofobik küme içinde merkezi bir konuma sahiptir. Çubuk grafikler ile iki farklı bioinformatic Öngördürücüleri oluşturulur: B. AGGRESCAN, C. TANGO. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Resim 2 . Aβ42 GFP PI oluşturan versiyonlarında S. cerevisiae kültürler için 16 h ifade indüklenen. A. çubuk grafiği iki biyolojik çoğaltır her varyant için 500 floresan hücreleri toplam hesaplanan PI sayısının farklı bir sayı içeren hücreler yüzdesini gösterir. B. bu görüntüler seçili Aβ42-GFP türevleri (Phe, Ile, Thr, Trp) temsilcisi floresan mikroskopi görüntülerdir. GFP (450-500 nm) ve bir emisyon aralığı (515-560 nm) için bir uyarma filtresi kullanarak UV ışık altında satın alınan. Ölçek çubuğu 10 µm. temsil eden Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Şekil 3 . Akış Sitometresi (FC) analiz o düzenif S. cerevisiae hücreleri seçili Aβ42-GFP türevleri ifade. A. grafik geçişli Maya hücreleri (P1) nokta araziler gösterir (FSC-A vs SSC-A) nerede hücre zarar hücre popülasyondan kaldırılır. (Homojen dağıtılan) Gln ve Ile (PI-şekillendirme) türevleri mikroskobik görüntülerini FC nokta çizer yanında gösterilir. Ölçek çubuğu 10 µm. Btemsil eder. Bu dağılım nokta Arsa görüntüler hangi geçişli nüfus (P3) arka plan sinyal hariç sadece floresan hücreleri içeren GFP-A vs oksidatif stres sonda temsil eder. Chücre frekans çubuk grafikler vardır. P1 ve Dgeçişli GFP sinyal (FITC genlik). CellROX (APC genlik) P3 geçişli. Hücre edinme akış sitometresi ile gerçekleştirildi. Her arsa 20.000 olayları temsil eder. Q ve Gln ve Ile Aβ42 mutantlar için sırasıyla karşılık gelir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Şekil 4 . Hücresel protein düzeyleri miktar. S. cerevisiaeifadede 16 h sonra Aβ42-GFP mutant toplam protein kesirler Batı lekesi bu şekilde gösterilmektedir. PI oluşturan değişik yeşille renklendirilir ve bu havza sitozol içinde dağıtılmış hafif kırmızı renkte görünür. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Şekil 5 . Hücresel oksidatif stres, hücre içi GFP floresans ve hücresel protein düzeyleri Aβ42-GFP mutantlar ifade içinde 16 h sonra belirlenen S. cerevisiae. X ekseni üzerinde temsil değişik onların öngörülen toplama eğilimlerini göre AGGRESCAN (sol panelde) ya da TANGO (doğru kapı aynası) tarafından sipariş edilmiş. PI oluşturan değişik yeşille renklendirilir ve PI oluşturan değişik ışık kırmızı olarak renklendirilir. A. B. bu bar grafikler Aβ42-GFP mutantlar FC analizini tarafından elde edilen oksidatif stres sonda floresans değerleri gösterir. C. D. bu bar grafikler protein düzeyleri ImageJ yazılımını kullanarak bir Western blot Dansitometresi analiz tarafından sayısal olarak temsil eder. E. F. bu bar grafikler GFP floresans değerleri bir FC analizden sonra göster. Hata çubukları oksidatif stres için yoklama ve farkı (MF) FC-gated hücre katsayısı GFP floresans değerleri temsil eder. Protein ifade düzeyleri için hata çubuklarını temsil ± SE (n = 3). Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
| Kanal | Fluorophore | Uyarma dalga boyu (nm) | Bant geçiren filtre |
| FITC | GFP | 488 | 530/30 |
| APC | CellRox | 635 | 660/20 |
| PerCP | IP | 488 | 585/42 |
Tablo 1. Lazer kaynak, bant geçiren filtre ve akış sitometresi içinde kullanılan fluorophores.
| GFP floresan | CV | CellROX floresan | CV | |
| A | 9316 | 96.4 | 1964 | 127.5 |
| C | 9709 | 91,9 | 1275 | 172.2 |
| D | 11213 | 101.7 | 3443 | 155.8 |
| E | 12256 | 101.1 | 3220 | 152.2 |
| F | 3010 | 96.1 | 1245 | 146.4 |
| G | 11541 | 97,2 | 2947 | 158 |
| H | 7895 | 98,2 | 3582 | 120.8 |
| Ben | 7365 | 97,2 | 1416 | 141.3 |
| K | 10839 | 100.4 | 3102 | 122.1 |
| L | 7605 | 96,9 | 1401 | 161.8 |
| M | 8149 | 96 | 1308 | 170.4 |
| N | 12741 | 97.5 | 3403 | 134.9 |
| P | 9768 | 102.8 | 2629 | 143.6 |
| Q | 13066 | 91.3 | 3354 | 169.9 |
| R | 8537 | 101.3 | 2839 | 127.5 |
| S | 12053 | 99,1 | 3313 | 174.3 |
| T | 10615 | 97,7 | 2213 | 107.7 |
| V | 9169 | 96.1 | 1878 | 121.7 |
| W | 1715 | 94.7 | 1531 | 100 |
| Y | 7574 | 94.5 | 1234 | 138 |
Tablo 2. GFP floresans ve oksidatif stres sonda floresans FC analiz Aβ42-GFP mutantlar için 16 h. ifade Maya hücrelerinin elde için bir değerler listesi Bu tablo ortalama floresans yoğunluğu (MFI) gösterir ve her mutant için CV.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Çok çeşitli hastalıklar misfolded proteinleri birikimi hücresel mevduat6,7,8,33içine bağlıdır. Birçok çabaları rapor protein konsantrasyonları almayın, Hesaplamalı yaklaşımları kullanarak bu hastalıkların başlangıcı tetiklemek moleküler mekanizmaları çözmeye gelmiş yapılmış veya vitro yaklaşımları, içinde protein konsantrasyonu reaksiyon sırasında sabit kalır. Ancak, hücre içinde proteinler sürekli sentezlenmiş ve kalabalık ve homojen olmayan bir ortamda bozulmuş. Bu sık farklılıklar arasında amyloidogenic proteinlerin silico, içinde in vitro ve in vivo toplama özelliklerini açıklar.
Neden Maya gibi basit hücresel modeller toplama çalışmaya makul bir seçimdir ve toksisite nörodejeneratif hastalıklar bir daha biyolojik bağlamda ilgili proteinlerin ilişkili birden çok nedeni vardır. Örneğin, protein misfolding toplama burada uygulanan bir gibi hızlı analizleri kullanarak sağlam bir hücresel nüfusu etkisini analiz imkanı verirler. Ancak, oksidatif düzeyleri maya suşları arasında farklı olabilir düşünmelisiniz. Böylece, suşları arasında veya farklı proteinler arasında bile karşılaştırmak için oksidan ve reductants, DTT ve diamin, gibi bir oksidasyon/redüksiyon ölçek kurmak için kullanılmalıdır.
Resimli bu makale, bioinformatic analiz, hücresel protein miktar örnek, moleküler tanımlamak için protein yerelleştirme görüntüleme ve eşzamanlı FC analiz GFP faaliyet ve oksidatif stres düzeyleri getirilmiştir tür protein toplama reaksiyonlar tarafından elde edildi oksidatif zarar için sorumlu. Sonuçlar daha çözünür Aβ42 türevleri, yerine daha toplama eğilimli, en yüksek oksidatif stres teşvik gösterilmektedir. Bu diffusible türlerin daha tehlikeli Aβ42 türler içinde vivobesinlerini puan. Türevleri toplayarak alt toksisite onların çözünür daha düşük protein düzeyleri sonuçlanan protein kalite makine tarafından Bu biçimler PI gizlediği konusunda gerçeği ve Tercihli onların yıkımı cevap gibi görünüyor meslektaşları.
Açıklanan yöntemi Aβ42 toplanan/çözünür türlerin tarafından üretilen oksidatif stres analizini sınırlı değildir ve protein toplama bozuklukları çeşitli eğitim için de uygulanabilir. Ayrıca, teknik toplama inhibitörleri etkisi hücresel oksidatif stres, daha fazla klinik uygulamalar için oksidatif aktif protein türler birikimi teşvik bu molekülleri atmak için izin izlemek yararlı olabilir. Son olarak, bir fluorogenic sonda kullanılabilir olduğu sürece, nicel veri sağlayan kolay bir yolu hızlı bir protein toplama moleküler türlerin diğer toksik etkileri için sorumlu tanımlamak için olağanüstü fırsatlar bağlı yaklaşımı sunmaktadır.
Subscription Required. Please recommend JoVE to your librarian.
Acknowledgments
Materials
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
References
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration? Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast? Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).