ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Dette papir beskriver protokollerne for prøveforberedelse, datareduktion og dataanalyse i neutron spin echo (NSE) undersøgelser af lipidmembraner. Velovervejet deuteriummærkning af lipider giver adgang til forskellige membrandynamik på mesoskopisk længde og tidsskalaer, over hvilke vitale biologiske processer forekommer.
Abstract
Lipid bilayers danner hovedmatrixen af cellemembraner og er den primære platform for næringsstofudveksling, proteinmembraninteraktioner og viral spirende blandt andre vitale cellulære processer. For effektiv biologisk aktivitet bør cellemembraner være stive nok til at opretholde cellens integritet og dens rum, men alligevel flydende nok til, at membrankomponenter, såsom proteiner og funktionelle domæner, kan spredes og interagere. Denne fine balance mellem elastiske og flydende membranegenskaber og deres indvirkning på biologisk funktion kræver en bedre forståelse af kollektiv membrandynamik over mesoskopisk længde og tidsskalaer af vigtige biologiske processer, f.eks. membrandeformationer og proteinbindingshændelser. Blandt de teknikker, der effektivt kan sonde dette dynamiske område er neutron spin ekko (NSE) spektroskopi. Kombineret med deuteriummærkning kan NSE bruges til direkte at få adgang til bøjnings- og tykkelsesudsving samt mesoskopisk dynamik ved udvalgte membranfunktioner. Dette dokument giver en kort beskrivelse af NSE-teknikken og skitserer procedurerne for udførelse af NSE-eksperimenter på liposomale membraner, herunder detaljer om prøveforberedelses- og deuterationsordninger sammen med instruktioner til dataindsamling og -reduktion. Papiret introducerer også dataanalysemetoder, der bruges til at udtrække centrale membranparametre, såsom bøjningsstivhedsmodulus, områdekomprimeringsmodulus og viskositet i plan. For at illustrere den biologiske betydning af NSE-undersøgelser diskuteres udvalgte eksempler på membranfænomener, der undersøges af NSE, nemlig virkningen af tilsætningsstoffer på membranbøjningsstivhed, domænedannelsens indvirkning på membranudsving og den dynamiske signatur af membranproteininteraktioner.
Introduction
Forståelsen af cellemembraner og deres funktion har udviklet sig bemærkelsesværdigt i løbet af de sidste par årtier. Den tidligere opfattelse af cellemembraner som passive lipid bilayers, der definerer cellegrænser og husmembranproteiner1, er gradvist omdannet til en dynamisk model, hvor lipid bilayers spiller en vigtig rolle i reguleringen af vitale biologiske processer, herunder cellulær signalering, molekylær udveksling og proteinfunktion - for at nævne nogle få2,3,4,5,6. Denne erkendelse af, at cellemembraner er meget dynamiske, konstant under ombygning og molekylær omfordeling, har opfordret til videnskabelige udforskninger ud over ligevægtsstrukturer af membraner7,8,9. Derfor er der udviklet flere tilgange til at studere de forskellige dynamiske tilstande i biologiske og bioinspirerede lipidmembraner. Til dato har de fleste af disse undersøgelser primært fokuseret på diffusive molekylære bevægelser10,11,12,13 og makroskopiske formudsving14,15,16, hvilket efterlader et betydeligt hul i forståelsen af mellemliggende membrandynamik, dvs. kollektive udsving i lipidsamlinger bestående af få 10-100'ere lipidmolekyler. Disse dynamikker forekommer over længdeskalaer på få tiere til få 100 Å og over tidsskalaer af under ns til få hundrede ns (se figur 1), her benævnt mesoskopiske skalaer. Det er faktisk på disse skalaer , at centrale biologiske aktivitet finder sted på membran niveau17. Dette omfatter viral spirende18, kanal gating19, og membran-protein interaktioner20. Det er også vigtigt at påpege, at energilandskabet af membranproteiner21,22 viser, at kropsbygningsændringer i proteiner - der er nødvendige for deres regulerende rolle - sker over ns-tidsskalaerne23 af kollektive membranudsving, hvilket yderligere understreger betydningen af mesoskopisk dynamik i cellemembranernes biologiske funktion og deres bioinspirerede analoger20. Dette papir fokuserer på de to primære mesoskopiske dynamiske tilstande i lipidmembraner, nemlig bøjningsudsving og tykkelsesudsving.
Den største udfordring ved direkte at undersøge disse udsvingstilstande er vanskeligheden ved samtidig at få adgang til deres rumlige og tidsmæssige skalaer ved hjælp af standardspektroskopimetoder. Den anden udfordring er , at direkte kontaktteknikker kan påvirke de samme udsving , som de er beregnet til at måle16. Dette forværres yderligere af den kompositoriske og strukturelle kompleksitet af biologiske membraner24,25, hvilket resulterer i ikke-homogene membranfunktioner, herunder lipiddomsdannelse26,27,28,29,30 og membranasymme asymmetri31,32,33- krævende selektive sonder for at forstå dynamikken i forskellige membranfunktioner. Heldigvis kan disse udfordringer overvindes med ikke-invasive neutronspektroskopimetoder, såsom neutron spin echo (NSE), som i sagens natur får adgang til den krævede længde og tidsskalaer, og yderligere muliggøre undersøgelser af selektive membranfunktioner uden at ændre deres fysiskkemiske miljø34. Faktisk har NSE-spektroskopi i løbet af de sidste par år udviklet sig til en unik og kraftfuld sonde af kollektiv membrandynamik35. Resultater fra NSE undersøgelser af lipidmembraner har produceret ny indsigt i mekaniske36,37 og viskoelastiske38,39 egenskaber lipidmembraner og har kastet nyt lys over deres potentielle rolle i biologisk funktion40,41.
NSE spektroskopi teknikken er baseret på en interferometrisk instrument design, først foreslået af Mezei42, ved hjælp af en række spin-flippers og magnetiske spoler til at styre præcessionen af neutron spin som neutroner krydse instrumentet. Designet hviler på magnetisk spejling af magnetfeltelementerne med hensyn til prøvepositionen (Figur 1A). Dette indebærer, at neutronen i mangel af energiudveksling mellem neutronen og prøven udfører det samme antal spinforuøgelser i modsatte retninger i første og anden halvdel af instrumentet (bemærk π-flipperen mellem de to præcessionsspoler). Som følge heraf forbliver neutronens endelige spintilstand uændret i forhold til den oprindelige tilstand - et fænomen, der omtales som spin-echo (se gennemsigtig neutron i figur 1A). Men når neutronen interagerer energisk med prøven, ændrer energiudvekslingen antallet af spinforudvælgelser i instrumentets anden halvdel, hvilket fører til en anden endelig spintilstand (se figur 1A). Dette er eksperimentelt opdaget som et tab i polarisering, som vil blive vist senere i dette papir. For flere detaljer om NSE-teknikken henvises læseren til dedikerede tekniske papirer42,43,44,45.
Her præsenteres en forenklet beskrivelse for at give et groft skøn over længden og tidsskalaerne, der er tilgængelige med NSE. Længdeskalaerne bestemmes af rækkevidden af opnåelige bølgevektoroverførsler, Q = 4π sin θ/λ, hvor 2θ er spredningsvinklen, og λ er neutronbølgelængden. Man kan se, at Q er indstillet af bølgelængdeområdet og omfanget af rotationen af spektrometerets anden arm (se figur 1A). Et typisk Q-områdepå NSE-spektrometre er ~0,02-2 Å-146,47og op til 0,01-4 Å-1 med nylige opgraderinger48,49, svarende til rumlige skalaer på ~1-600 Å. På den anden side beregnes den tilgængelige tidsskala ud fra den samlede præcessionsvinkel (eller fase), som neutronen har erhvervet inden for de magnetiske præcessionsspoler, og det konstateres, at den er50:  . I dette udtryk er t Fourier-tiden defineret som
. I dette udtryk er t Fourier-tiden defineret som  , hvor er det
, hvor er det  neutron gyromagnetiske forhold,
neutron gyromagnetiske forhold,  er spolens længde og
er spolens længde og  er styrken af spolens magnetfelt. Det er værd at påpege, at Fourier-tiden er en mængde, der er strengt afhængig af instrumentgeometrien, magnetfeltstyrken og neutronbølgelængden. Ved hjælp af neutroner af bølgelængde
er styrken af spolens magnetfelt. Det er værd at påpege, at Fourier-tiden er en mængde, der er strengt afhængig af instrumentgeometrien, magnetfeltstyrken og neutronbølgelængden. Ved hjælp af neutroner af bølgelængde  = 8 Å og instrumentindstillinger på
= 8 Å og instrumentindstillinger på  = 1,2 m og
= 1,2 m og  = 0,4 T beregnes Fourier-tiden f.eks. Eksperimentelt, fourier tiden er indstillet ved at ændre strømmen i præcession spoler (dvs. magnetfelt styrke) eller ved hjælp af forskellige neutron bølgelængder, hvilket resulterer i typiske NSE tidsskalaer på ~ 1 ps til 100 ns. Nylige opgraderinger i NSE-spektrometre har imidlertid gjort det muligt at få adgang til længere Fourier-op til ~ 400 ns på J-NSE-Phoenix spektrometeret på Heinz Maier-Leibnitz Zentrum51 og SNS-NSE spektrometeret på Oak Ridge National Lab48, og op til ~ 1.000 ns på IN15 NSE spektrometer på Institut Laue-Langevin (ILL)49.
= 0,4 T beregnes Fourier-tiden f.eks. Eksperimentelt, fourier tiden er indstillet ved at ændre strømmen i præcession spoler (dvs. magnetfelt styrke) eller ved hjælp af forskellige neutron bølgelængder, hvilket resulterer i typiske NSE tidsskalaer på ~ 1 ps til 100 ns. Nylige opgraderinger i NSE-spektrometre har imidlertid gjort det muligt at få adgang til længere Fourier-op til ~ 400 ns på J-NSE-Phoenix spektrometeret på Heinz Maier-Leibnitz Zentrum51 og SNS-NSE spektrometeret på Oak Ridge National Lab48, og op til ~ 1.000 ns på IN15 NSE spektrometer på Institut Laue-Langevin (ILL)49.
Udover direkte adgang til længden og tidsskalaen af membrandynamik har NSE de iboende muligheder for neutronisotopfølsomhed52. Specifikt, evne til neutroner til at interagere forskelligt med isotoper af brint, det mest rigelige element i biologiske systemer, resulterer i en anden neutron spredning længde tæthed,34 eller NSLD (svarende til det optiske indeks for brydning50), når protium erstattes af deuterium. Dette muliggør en tilgang kendt som kontrastvariation, som almindeligvis bruges til at fremhæve specifikke membranfunktioner eller skjule andre - sidstnævnte scenario kaldes kontrastmatchning. En hyppig anvendelse af kontrastvariation/matchning er substitutionen af vand (NSLD = -0,56 × 10-6 Å-2) med tungt vand eller D2O (NSLD = 6.4 × 10-6 Å-2) for at forstærke neutronsignalet fra protiated lipidmembraner (NSLD ~ 0 × 10-6 Å-2). Denne tilgang er meget effektiv i undersøgelser af membranstruktur, fordi indtrængen af D2O i membranens hovedgrupperegion giver mulighed for nøjagtig bestemmelse af membranetykkelserne (se figur 2A, venstre panel) og af placeringen af forskellige lipidundergrupper, når der anvendes mere sofistikerede modeller53,54. Dette papir fremhæver nogle eksempler på brugen af kontrastvariation til undersøgelser af kollektiv dynamik i biomimetiske membraner og udvalgte membranfunktioner.
Her illustreres NSE's effektivitet med hensyn til at give unik indsigt i dynamiske og funktionelle membranegenskaber gennem håndgribelige eksempler på NSE-undersøgelser af model- og biologisk relevante lipidmembransystemer med vægt på mesoskaldynamik i fritstående membraner i form af liposomale suspensioner. Til NSE-målinger af membrandynamik i flyet henvises læseren til dedikerede publikationer om neutronspektroskopi (GINSES)55,56 og andre undersøgelser af justerede multilamellare membranstakke57,58,59,60.
For nemheds skyld fremhæver dette papir tre forskellige ordninger for membrandeuteration illustreret på en velunderbyrdet domæneformning, eller faseadskillelse, lipid bilayersystem på 1,2-dimyristoyl-sn-glycero-3-phosphocholin (DMPC) og 1,2-distearoyl-sn-glycero-3-phosphocholin (DSPC) blandinger61,62. De to lipider er kendetegnet ved et misforhold i deres kulbrintekædelængde (14 carbons/tail i DMPC vs. 18 carbons/tail i DSPC) og deres gelvæskeovergangstemperatur (Tm, DMPC = 23 °C vs. Tm, DSPC = 55 °C). Dette resulterer i lateral faseadskillelse i DMPC:DSPC-membraner ved temperaturer mellem blandingens øvre og nedre overgangstemperaturer63. De her overvejede deuterationsordninger er valgt til at demonstrere de forskellige dynamiske tilstande, der er tilgængelige i NSE-målinger på liposomale membraner, nemlig bøjningsudsving, tykkelsesudsving og selektive udsving i bøjning/tykkelse af laterale domæner. Alle lipidsammensætninger rapporteres for DMPC:DSPC-bilayere, der fremstilles ved en molefraktion på 70:30, ved hjælp af kommercielt tilgængelige protiated og perdeutererede varianter af DMPC og DSPC. Alle prøveforberedelsestrin er baseret på 4 mL liposomal suspension, i D2O, med en lipidkoncentration på 50 mg/mL, for en total lipidmasse på Mtot = 200 mg pr. prøve.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Deuterationsordning, der kræves til forsøget
- Til måling af svingudsving foretages fuldt protiated liposomer i D2O (D 99,9%) eller D2O-buffer (f.eks. fosfatbuffer tilberedt med D2O i stedet for H2O). Brug fuldt protiated DMPC (C36H72NO8P) og DSPC (C44H88NO8P) med
 133,4 mg, hvor XDMPC og XDSPC er molefraktionerne af DMPC og DSPC, her indstillet til henholdsvis 0,7 og 0,3, og MwDMPC og MwDSPC er de molvægte, der er angivet med henholdsvis 677,9 g/mol og 790,1 g/mol. Tilsvarende mDSPC = 66,6 mg. Denne deuterationsordning øger spredningskontrasten mellem membranen (NSLD ~ 0 × 10-6 Å-2) og den deutererede buffer (NSLD ~ 6,4 × 10-6 Å-2) og forstærker signalet fra membranoverkommninger (se figur 2A venstre panel).
133,4 mg, hvor XDMPC og XDSPC er molefraktionerne af DMPC og DSPC, her indstillet til henholdsvis 0,7 og 0,3, og MwDMPC og MwDSPC er de molvægte, der er angivet med henholdsvis 677,9 g/mol og 790,1 g/mol. Tilsvarende mDSPC = 66,6 mg. Denne deuterationsordning øger spredningskontrasten mellem membranen (NSLD ~ 0 × 10-6 Å-2) og den deutererede buffer (NSLD ~ 6,4 × 10-6 Å-2) og forstærker signalet fra membranoverkommninger (se figur 2A venstre panel). - At måle bøjningsdynamikken i udvalgte laterale membranfunktioner, f.eks.83,Mw873,7g/mol),således at mDMPC = 128,8 mg og mDSPC-d83 = 71,2 mg. Denne ærværdighedsordning minimerer spredningen fra de uønskede DSPC-rige domæner, hvilket muliggør selektive målinger af bøjningsudsving fra DMPC-rige matrix (se figur 2B midten).
BEMÆRK: For at finde den optimale lipid deuteration, der kræves for en bestemt kontrast matchende ordning, udnytte tilgængelige web-baserede spredning længde tæthed (SLD) regnemaskiner, som den, der er udviklet af NIST Center for Neutron Research64. Disse web-baserede grænseflader er udstyret med brugervenlige værktøjer til nem beregning af SLD af lipider med forskellige grader af deuteration, samt af lipid blandinger. - Til NSE-målinger af gennemsnitlige udsving i membrantykkelsen (uden sidekontrast) skal du bruge tail-deuterated varianter af de bestanddele lipider, dvs. DMPC-d54 (C36H18NO8PD54, 732,3 g/mol) og DSPC-d70 (C44H18NO8PD70, 860,1 g/mol)35,38, således at mDMPC-d54 = 133,0 mg og mDSPC-d70 = 67,0 mg. Denne kontrastordning(Figur 2A, højre panel) forstærker spredningssignalet fra lipidhovedgrupperne (NSLD ~ 4,5 × 10-6 Å-2) ved kontrastmatchende halegruppen (NSLD ~ 6,4 × 10-6 Å-2) til den deutererede buffer, der gør det muligt at opdage udsving i membrantykkelsen.
- Til undersøgelser af udsving i tykkelsen af udvalgte membranrum f.eks DMPC-rige matrix, bruge den samme strategi, der er beskrevet i trin 1,2 ved at erstatte protiated DMPC lipider med deres hale-deuterated analoger, dvs DMPC-d54, således at DSPC-rige domæner er kontrast-matchede til den deutererede buffer og den primære spredning signal er fra hovedgruppe regionen hale-deuterated DMPC-rige matrix.
2. Forberedelse af lipid suspension til ekstrudering
- Massen af hver enkelt bestanddel i prøven beregnes, afhængigt af prøvesammensætningen. Som tommelfingerregel, for prøver med flere molekylære komponenter, massen af en komponent er givet ved sin molar masse, Mwi, vægtet af sin muldvarp fraktion, Xi, og normaliseret over alle komponenter, således at: hvor
 Mtot er den samlede masse, der er her til 200 mg. Se eksemplet ovenfor for DMPC-DSPC lipid bilayers med forskellige deuteration ordninger.
Mtot er den samlede masse, der er her til 200 mg. Se eksemplet ovenfor for DMPC-DSPC lipid bilayers med forskellige deuteration ordninger. - Ved hjælp af en digital semimikrobielbalance vejes de beregnede masser af lipider (og andre prøvebestanddele, f.eks. proteiner, nanopartikler osv.), og de tilsættes til en hætteglas eller rundbundskolbe – husk at veje hætteglasset eller kolben på forhånd. Der tilsættes 1 mL opløsningsmiddel for at opløse de vejede komponenter ved manuelt at blande inde i en emhætte. Til rene lipidprøver skal du bruge kloroform eller ethanol. For prøver med yderligere, ikke-lipidkomponenter (f.eks. nanopartikler) skal du vælge et almindeligt opløsningsmiddel, der spreder alle komponenter.
- For små lipidmængder (<10 mg) skal du forberede en stamopløsning og pipette det krævede volumen i blandingen.
BEMÆRK: Tilsæt ikke for store mængder opløsningsmiddel, da det vil bremse tørringstrinnet for opløsningsmidler, der er beskrevet nedenfor.
- For små lipidmængder (<10 mg) skal du forberede en stamopløsning og pipette det krævede volumen i blandingen.
- Lipidopløsningen tørres inde i en hætte ved forsigtigt at streame en inert gas (f.eks. nitrogen, argon) i hætteglasset, mens hætteglasset langsomt roteres i en vinkel. Hold hætteglassene i vippet position for at skabe en tynd film af tørret lipid på hætteglasvæggene, hvilket giver mulighed for jævn tørring. Placer hætteglasset periodisk i et vandbad ved 35 °C for at omgå fordampningsmedieret køling, hvilket vil bremse fordampningen af opløsningsmidlet.
- Tag hætteglassene natten over i en vakuumovn ved ~35 °C for at fjerne restopløsningsmidlet helt. For umættede lipider renses vakuum med en inert gas for at minimere oxidation.
- For at sikre fuld fjernelse af opløsningsmidler skal hætteglasset vejes efter tørring af lipider, og det bekræftes, at der ikke er nogen overskydende masse ud over de målte mængder materialer. Gør dette ved at trække hætteglassets masse fra den målte masse efter tørring. Hvis der er overskydende masse, tørres prøven under vakuum i yderligere 6 timer. Gentag denne proces efter behov.
- Hydrat lipidfilmen med 4 mL D2O eller D2O-buffer for at opnå en lipidkoncentration på 50 mg/mL. For lipider med høje overgangstemperaturer, såsom DMPC-DSPC-blandinger, opvarmes bufferen til over overgangstemperaturen (60 °C) for at sikre jævn blanding.
BEMÆRK: Da NSE-forsøg kræver relativt store prøvemængder (~4 mL), skal du overveje at hydrere prøven ved hjælp af halvdelen af den krævede buffer, dvs. I dette tilfælde tilsættes den resterende halvdel af bufferposten ekstrudering. Bemærk, at kapaciteten af sprøjter, der anvendes i ekstrudering, er begrænset til 1 mL. Således ville hydrering med 4 mL buffer kræve fire sæt ekstrudering. - Vortex-mix den hydrerede lipidopløsning, indtil lipidfilmen er helt opløst og ikke længere er synlig på hætteglassets vægge. På dette stadium danner de hydrerede lipider multilamellare vesikler og mikron mellemstore multilamellare stakke, og suspensionen vises mælkehvid.
- For at lette brud på lipidstakkene og reducere multilamellariteten skal du udføre fem fryse-/optøningscyklusser ved at placere hætteglasset med hydreret lipidopløsning i en fryser af laboratoriekvalitet (helst -80 °C fryser), indtil det er helt frosset, og derefter overføre hætteglasset til et 35 °C vandbad, indtil lipidopløsningen er helt optøet. Hvirvel den optøede opløsning indtil homogen. Gentag fire gange mere.
BEMÆRK: Alternativt kan et tørisbad tilberedes til hurtig frysning ved at kombinere acetone og tøris.
3. Ekstrudering af den hydrerede lipidopløsning
- Saml ekstruderopsætningen ved hjælp af en polycarbonatmembran mellem to membranstøtter og tilsæt to papirfiltre på hver side for at give yderligere støtte. Brug en polycarbonatmembran med en porestørrelse, der matcher målmaosomal størrelse (almindelige porestørrelser til NSE-eksperimenter er 50 nm og 100 nm – typisk giver 100 nm diameter liposomer mulighed for mindre begrænsede membranudsving, men mindre 50 nm liposomer kan bruges til krumningsundersøgelser). Sørg for, at polycarbonatmembranen er fuldt strakt, inden samlingen færdiggøres, og at det udvendige ekstruderhus strammes.
- Polycarbonatmembranen hydreres ved at passere ~0,3 mL D2O eller D2O-buffer et par gange gennem membransamlingen ved hjælp af lufttætte glassprøjter. Udnyt den samme buffer, der bruges i prøveforberedelsen. Lad det stå i mindst 10 minutter, og sug derefter bufferen helt ud, før prøven introduceres.
- Fyld en 1 ml gastæt sprøjte med den forberedte lipidopløsning og sæt den i den ene ende af ekstruderapparatet. Sæt derefter en tom sprøjte i den modsatte ende. Når sprøjterne er forbundet til ekstrudersamlingen, skal du placere den i ekstruderblokken.
- Hvis der er behov for forhøjede temperaturer til ekstrudering, som det er tilfældet med mættede lipider med høje overgangstemperaturer (f.eks.
BEMÆRK: Dette trin er afgørende for at sikre homogen blanding af lipider og for at undgå at udøve ekstremt tryk under ekstrudering, hvilket kan sprænge polycarbonatmembranen. For lipidprøver med lave overgangstemperaturer (<25 °C) udføres ekstrudering ved stuetemperatur. - For at ekstrudere lipidopløsningen skal du fastgøre ekstruderen til en programmerbar sprøjtepumpe med en aluminiums-/stålramme som vist i figur 3A. Til temperaturstyrede ekstruderinger tilsættes en specialbygget ekstruderbase med en væskekanal og fastgøres til et cirkulerende vandbad.
- Programmer sprøjtepumpen til at udføre 15-20 ekstruderingscyklusser efter producentens manual. Når lipidopløsningens farve ekstruderes, ændres den fra mælkehvid til gennemsigtig opalblå (Figur 3B, C), hvilket angiver en endelig liposomal størrelse, der er mindre end bølgelængden af synligt lys som forventet. For den type sprøjtepumpe, der er vist i figur 3A, skal du følge nedenstående trin.
- Start med at justere pumpeindstillingerne. Hold knappen Hastighed nede, og gå ind i ekstruderingshastigheden (50,99 mL/h), tryk derefter på knappen Diameter, og indtast sprøjtediameteren (4,606 mm). Brug pil op under hvert ciffer på skærmen til at ændre den pågældende cifferværdi.
- Udstø mig-sættet anbringes sammen med prøvesprøjten til højre (se figur 3A). Tryk på knappen Træk ud, indtil træk lyset tændes. Tryk på Start, og vent på, at prøven dispenserer ind i venstre (tomme) sprøjte.
- Tryk på Stop-knappen lige før prøvesprøjten (højre) er helt tom. Optag den udleverede diskenhed, og brug den til at programmere ekstruderingscyklussen. Hold knappen Hastighed nede, indtil fase 1 (PH:01) vises på skærmen. Tryk på knappen Lydstyrke for at angive den dispenserede diskenhed, der blev optaget tidligere. I denne fase skal du sørge for, at træklyset er slukket – dette dispenserer prøven i den rigtige retning.
- Tryk på knappen Hastighed igen, og brug pil op længst til højre for at få adgang til fase 2 (PH:02). Tryk på Lydstyrke for at angive den samme værdi af den diskenhed, der er optaget tidligere. I denne fase skal du trykke på knappen Træk ud, indtil træk lyset er tændt – dette dispenserer prøven til venstre.
- Hvis du vil gentage denne cyklus, skal du trykke på knappen Hastighed igen og bruge pil op længst til højre for at få adgang til fase 3 (PH:03). Tryk på knappen Lydstyrke, indtil LP:SE vises på skærmen, og indstil den til 20. Dette er antallet af sløjfer eller gentagelser, som pumpen vil udføre. Til sidst skal du trykke på knappen Hastighed, få adgang til fase 4 (PH:04) og trykke på lydstyrkeknappen for at komme til stopfunktionen. Pumpen er nu sat op til automatiseret ekstrudering.
- Tryk på Start for at starte ekstruderingscyklussen.
- Tøm sprøjten med den ekstruderede lipidaffjedring i et rent hætteglas, og forbered dig på opbevaring eller målinger. For lipidprøver med høj smeltetemperatur opbevares prøven over væskefaseovergangen, indtil den måles. Ellers skal prøverne opbevares ved stuetemperatur.
- Må ikke fryse ekstruderede prøver som frysning vil medføre, at vesikler til at briste (suspensionen vil vende mælkehvid igen).
4. NSE-målinger for prøven/prøverne og reduktion af de indsamlede data
- Før NSE-eksperimentet karakteriseres den ekstruderede liposomale prøve fra trin 3.7 ved hjælp af tilgængelige metoder for at sikre tilstrækkelig prøvekvalitet. En liste over mulige karkateriseringsmetoder, der kan bruges til at vurdere kvaliteten af liposomale suspensioner til NSE-eksperimenter, f.eks. størrelsesfordeling, multilamellaritet, lateral membranstruktur, er inkluderet i diskussionsafsnittet.
- Bestem de Q-område og tilsvarende instrumentindstillinger, der kræves til eksperimentet. Til bøjning af stivhedsmålinger af lipid bilayers skal du bruge et Q-område på ~(0,04 - 0,2) Å-1. Til undersøgelser af udsving i membrantykkelse skal du bruge et Q-område på ~(0,04 - 0,2) Å-1 svarende til membrantykkelsen35,66,67.
BEMÆRK: Diskuter den eksperimentelle opsætning med instrumentforskeren inden eksperimentets start. Som tidligere nævnt er sans-karakterisering af prøven nødvendig, især hvis der ikke foreligger forudgående oplysninger om spredningssignalet, som i selektivt deutererede membraner. Alternativt kan du køre statiske (også kendt som diffraktion) målinger over et begrænset Q-område på NSE-instrumentet med det forbehold, at sådanne målinger tager meget længere tid sammenlignet med SANS. - Ved hjælp af en sprøjte eller en overførselspipette skal du indlæse de ekstruderede liposomale suspensioner i de udpegede prøveceller, der er tilgængelige på NSE-strålelinjer. Bemærk, at standard NSE-prøveceller fås i tykkelser på 1, 2, 3 og 4 mm. Vælg celletykkelsen på en sådan måde, at spredningssignalet optimeres, samtidig med at det usammenhængende baggrundssignal holdes til en rimelig intensitet.
BEMÆRK: Som tommelfingerregel skal du bruge prøveceller med en fremspringende stilængde på 1 eller 2 mm til protiated liposomer i deutereret buffer – tykkere celler kan resultere i flere spredningseffekter, der er vanskelige at korrigere for. For liposomer med højere niveauer af deuterationer (f.eks. halekontrastmatchede liposomer eller asymemetriske liposomer med enkelte fremspringede foldere) kan du overveje at bruge en tykkere prøvecelle (f.eks. 3 eller 4 mm pathlength) til at forbedre optællingsstatistikkerne, hvis prøven er tilgængelig i større mængder – nogle gange kan dette være omkostningsoverkommeligt. - Forbered en identisk eksempelcelle til bufferen. Brug den samme buffer som i den liposomale affjedring. Målinger på bufferen er nødvendige for intensitetsnormalisering og baggrundskorrektioner (BKG).
- Placer prøvecellerne i prøveholderen på NSE-spektrometeret, programmer målekørslerne, og indsaml ekkodata. Rådfør dig med instrumentforskeren om programmering af målingerne, hvis en første gang NSE-bruger.
- Udfør to ekstra målesæt, der er nødvendige for datareduktionen: opløsningsmålinger (R) og transmissionsmålinger (T).
- Udføre opløsningsmåling (R) på en elastisk spredningsreference (f.eks. kulstof) — der skal køres under de samme indstillinger samme bølgevektor og Fourier gange som prøve- og buffermålingerne.
- Udfør transmissionsmålinger (T) på prøven og bufferen for at beregne intensiteten af den transmitterede neutronstråle (se trin 4.9 nedenfor). Transmissionen beregnes som forholdet mellem neutrontællinger fra prøven eller bufferen divideret med neutrontællingerne for en åben stråle (dvs. med en tom prøveposition).
- Brug den dedikerede datareduktionssoftware til det NSE-spektrometer, hvor målingerne udføres, til at reducere de indsamlede data.
BEMÆRK: Forskellige spektrometre kan bruge forskellige software- eller brugergrænseflader. Nedenfor er et eksempel på NSE datareduktion ved hjælp af DataAnalyse og Visualisering Miljø (DAVE)68 software specielt skrevet til NSE spektrometeret på NIST Center for Neutron Research.- Åbn DAVE-softwaren, og vælg Reducer NSE-data i datareduktionsmenuen. Der vises flere pop op-vinduer.
- Overfør datafilerne over forskellige Q-værdier ved hjælp af Open .echo-filerne fra filmenuen. Disse filer svarer til de rå datafiler med spin-ekkosignalerne og har filtypenavnet .echo i filnavnet. Når filoverførslen er fuldført, vises filerne under de tilgængelige datasæt.
- Højreklik på den valgte fil, og mærk den i overensstemmelse med den måling, den svarer til. dvs. Eksempel, Celle (for tom celle eller buffer) eller Opløsning.
- Grupper detektorpxlerne i 2 x 2 for at forbedre signal-til-støj-forholdet ved hjælp af fanen Datasæt. Anvend den samme placering på alle filer. opløsning, celle og prøve.
- Undersøg dataene over alle pixelgrupper, og maskér dem med dårlige signaler (se figur 4B) ved at trykke på m-tasten på tastaturet. Tryk på Enter for at få adgang til et pop op-vindue for at anvende den samme maske på alle Fourier-tider eller efterfølgende Fourier-tider. Dette kan også anvendes på individuelle pixel på ethvert tidspunkt under datareduktion. Maskerede pixel bliver grønne.
- Sørg for, at de indsamlede data er i form af et ekkosignal, dvs.
BEMÆRK: Fasestrømmen er proportional med neutronspinets præcessionsvinkel; Det er derfor almindeligt at repræsentere fasestrømmen som en fasevinkel som vist i figur 4A. Ved målinger på pulserende kilder anvendes yderligere tid til flyveberegninger på dataene for at opnå ekkosignalerne som funktion af hændelses neutronbølgelængde inden for en neutronpuls. - Start med at tilpasse opløsningsfilen. Vælg en løsningsfil på listen over overførte filer, og højreklik på filen. Vælg Tilpas handlinger: Tilpas ekkoer (opløsning) i pop op-menuen.
- Sørg for, at ekkosignalernes pasform giver en række tilpasningsparametre, herunder parameteren A, der kræves i trin 4.8. Anfaldene udføres automatisk ved hjælp af følgende udtryk.

Her er ζ den periode, hvor ekkosignalet (dvs. cosinusfunktionen i figur 4A), σ er bredden af den gaussiske kuvert bestemt af den gennemsnitlige bølgelængde og bølgelængdespredning af den indfaldende neutronstråle, Φc er fasestrømmen, og Φ0 er ekkopunktet, der afhænger af den feltsti, som neutronerne50 oplever. Fysiske oplysninger om prøven er kodet i amplituden, A, af cosinusfunktionen i ligningen (1).
BEMÆRK: Bredden af den gaussiske kuvert er baseret på værdier, der er forudbestemt af instrumentforskeren og bør ikke ændres. De andre paramters er variabler, der er monteret på det specifikke ekkosignal over hver pixel. - Undersøg fit-resultaterne ved at klikke på hver pixel for at vise de resulterende monteringsparametre, kvaliteten af pasformen og den gennemsnitlige firkantede afvigelse af pasformen. Hvis du vil undersøge den fejl, der er forbundet med hver monteringsparameter over hele detektoren, skal du vælge Billedindstillinger og derefter vælge den passende parameter af interesse. Dette vil generere et kort med værdien af montering paramter over hver pixel. Højreklik på detektorbilledet. Der vises et pop op-vindue, der viser et fejllinjekort over den valgte tilpasningsparameter.
- Hvis pasformen over en bestemt pixel er utilfredsstillende (f.eks. passer parameetere med store fejllinjer), skal signalet omstås over den specifikke pixel. Marker denpågældende pixel , tryk på fanen Tilpasning , og tryk derefter på Tilpas pixel. Indtast nye startparametre for fasen (Φ0) og punktum (ζ) i monteringsfanen for at opnå en mere tilfredsstillende pasform.
BEMÆRK: Det er nyttigt at afbilde den monterede fase som en funktion af Fourier Time. Det kan du gøre ved at gå til hovedafbildningsvinduet og vælge Tilpas fase v. Fourier Time. Dette plot skal være glat og kontinuerligt. Undersøg afbrydelser i dette plot, og tilpas de pixel, de svarer til.
- Reducer eksempel- eller cellefilen ved at vælge den tilsvarende fil på listen over overførte og navngivne filer.
- Undersøg alle pixels, og mask dem med dårlige statistikker som beskrevet i trin 4.7.5.
- Højreklik på filen, og vælg Tilpas til handlinger: Importer faser (eksempel, celle). Dette importerer faserne og den anvendte maske fra løsningsfilen.
- Tilpas ekkosignalerne ved hjælp af den samme procedure, der er beskrevet før for løsningsfilen (trin 4.7.8-4.7.10). Når du tilpasser eksempel- og cellefilerne, må du ikke ændre værdierne for den periode og det ekkofasepunkt, der importeres fra opløsningstilpasningen. Disse parametre afhænger af de instrumentale indstillinger og bør ikke variere med prøver.
- Før du går videre til datareduktion, skal du indtaste strålecentret for alle datafiler. Marker datafilen, gå til fanen Generelt, og angiv X- og Y-strålecenterværdier. Disse værdier registreres under eksperimentet.
- Når filerne Eksempel, Celle og Opløsning er fuldført, skal du beregne den normaliserede mellemliggende spredningsfunktion, der skal bruges senere i dataanalyse og fortolkning. Det kan du gøre ved at højreklikke på eksempelfilen for at blive reduceret fra listen over monterede filer og vælge Beregn I(Q) i pop op-menuen. Der vises et vindue med adgangsvalg for filerne Opløsning og Celle (dvs. buffer) og antallet af Q-buer (se trin 4.9). Når du har indtastet alle de nødvendige informaiton, skal du trykke på OK-knappen. Resultaterne vises i et nyt vindue.
BEMÆRK: Datareduktionen udføres i henhold til følgende ligning for at opnå den normaliserede mellemliggende spredningsfunktion69.
hvor t er Fourier-tid, er Nop og Nned neutrontællingerne i ikke-spin-flip- og spin-flip-konfigurationerne (målt med π/2-flippers slukket og π-flipper slukket og tændt, henholdsvis) og hævet skrift, BKG og R, svarer til henholdsvis baggrunds- og opløsningsmålingerne som defineret i henholdsvis trin 4.4 og 4.6. Bemærk, at strålepolariseringen , således ændringer i spintilstanden på grund af energiudveksling mellem neutronen og prøven, opdages som et fald i polariseringen (fra enhed).
, således ændringer i spintilstanden på grund af energiudveksling mellem neutronen og prøven, opdages som et fald i polariseringen (fra enhed).
- Til sidst skal detektorens pixel grupperes i Q-buersom vist i figur 4B for at opnå Q-afhængighedenaf den normaliserede mellemliggende spredningsfunktion, S(Q,t) / S (Q,0). Dette kaldes teknisk set data binning og bør gøres med omtanke, dvs.
- Ved stærk spredning af prøver opdeles detektoren i flere Q-buer, samtidig med at der opretholdes rimelige fejllinjer på den resulterende mellemliggende spredningsfunktion, S(Q,t) / S(Q,0). Dette giver flere Q-datapunkter og er vigtigt for den dataanalyseprocedure, der er beskrevet nedenfor. Vær opmærksom på, at for svagt spredning prøver, overdreven binning resulterer i dårlig henfald signaler, dvs store fejl barer på S(Q,t) / S(Q, 0), hvilket kan resultere i store usikkerheder.
5. Dataanalyse og -fortolkning
- Tilpas de normaliserede mellemliggende spredningsfunktioner, S(Q,t) / S(Q,0), der er opnået fra datareduktionen ovenfor, til en strakt eksponentiel funktion med en strækker eksponent på 2/370.

BEMÆRK: Et eksempel på disse anfald findes i figur 5B. Anfald af S(Q,t) / S(Q, 0) til ligning (3) giver Q-afhængige afslapning satser Γ (Q). - Plot Γ(Q) som en funktion af Q og passer til en passende model til at udtrække relevante membranparametre.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
NSE undersøgelser adgang til bøjning udsving er typisk udføres over en Q-range på ~ (0,04 - 0,2) Å-1. Dette Q-område svarer til mellemliggende længdeskalaer mellem membrantykkelsen og den liposomale radius, hvor bøjningsdynamikken dominerer. Måling over et udvidet Q-område kan give adgang til yderligere dynamiske tilstande, herunder liposomal diffusion og intramembrane dynamik. For flere detaljer om cross-over i membran dynamik adgang til NSE, tjek disse relevante publikationer25,71. Det er vigtigt at understrege, at NSE-signaler er proportionale med:  , hvor jegcoh og Iinc er henholdsvis den sammenhængende og usammenhængende spredningsintensitet fra prøven. Det anbefales derfor at forberede NSE-liposomale prøver i deutererede buffere (dvs. buffere tilberedt med D2O i stedet for H2O) for at minimere det usammenhængende spredningssignal, hovedsageligt bidraget med prøvens hydrogenindhold. I nogle tilfælde kan det dog være nødvendigt at anvende blandinger af D2O og H2O for at opnå optimale kontrastforhold. Typisk udføres NSE-målinger af membranbøjningsudsving på fuldt protiated liposomer i deutereret buffer, benævnt fuldt kontrasterede liposomer i figur 5. Denne deuterationsordning resulterer i en stor NSLD-forskel mellem membrankernen (~0 × 10-6 Å-2) og dens deutererede væskemiljø (~6,4 × 10-6 Å-2), hvilket væsentligt forbedrer spredningssignalet fra liposomale membraner og forbedrer målestatistikken for bøjningsdynamik. Denne kontrastordning(figur 2A venstre panel) anvendes ofte i undersøgelser af bøjningsstivhed af lipidmembraner med enkelt38,72 og flere39,66 lipidkomponenter og i undersøgelser af membranblødning/stivhed ved biologiske indeslutninger (f.eks. kolesterol, lægemiddelmolekyler, peptider/proteiner)36,37,73,74,75og syntetiske tilsætningsstoffer (f.eks. nanopartikler)76,77.
, hvor jegcoh og Iinc er henholdsvis den sammenhængende og usammenhængende spredningsintensitet fra prøven. Det anbefales derfor at forberede NSE-liposomale prøver i deutererede buffere (dvs. buffere tilberedt med D2O i stedet for H2O) for at minimere det usammenhængende spredningssignal, hovedsageligt bidraget med prøvens hydrogenindhold. I nogle tilfælde kan det dog være nødvendigt at anvende blandinger af D2O og H2O for at opnå optimale kontrastforhold. Typisk udføres NSE-målinger af membranbøjningsudsving på fuldt protiated liposomer i deutereret buffer, benævnt fuldt kontrasterede liposomer i figur 5. Denne deuterationsordning resulterer i en stor NSLD-forskel mellem membrankernen (~0 × 10-6 Å-2) og dens deutererede væskemiljø (~6,4 × 10-6 Å-2), hvilket væsentligt forbedrer spredningssignalet fra liposomale membraner og forbedrer målestatistikken for bøjningsdynamik. Denne kontrastordning(figur 2A venstre panel) anvendes ofte i undersøgelser af bøjningsstivhed af lipidmembraner med enkelt38,72 og flere39,66 lipidkomponenter og i undersøgelser af membranblødning/stivhed ved biologiske indeslutninger (f.eks. kolesterol, lægemiddelmolekyler, peptider/proteiner)36,37,73,74,75og syntetiske tilsætningsstoffer (f.eks. nanopartikler)76,77.
Målinger af bøjningsudsving resulterer i afslapningsrater, der følger en Q3-afhængighed, som forudsagt af Zilman og Granek for termisk bølgende elastiske tynde plader70. En raffineret form af denne Q-afhængigheder opnået fra teoretiske korrektioner af Watson og Brown78, som tager hensyn til virkningerne af intermonolayer friktion foreslået af Seifert og Langer79. Ved desuden at definere det neutrale plan, der skal være på grænsefladen mellem de hydrofile hovedgrupper og membranens hydrofobe haler, kan de bøjningsafslapningshastigheder derefter monteres på følgende udtryk38.

hvor ηbuff er bufferviskositeten, er kBT den termiske energi, κ og er den målte membrans bøjningsstivhed (eller den kontrasterede del af membranen i selektivt deutererede systemer). Denne type måling muliggør direkte beregning af membranelastiske egenskaber i form af de bøjningsstivhedsmoduler. Bemærk , at κ udvindes fra hældningen af den lineære pasform af Γ vs. Q3, som vist i figur 5C.
På den anden side viser NSE-målinger af membranetykkelsesudsving afvigelser fra Q3-afhængighedeni Γ(Q)omkring Q-værdier, der svarer til membrantykkelsen (se figur 2 i ref.66). For at isolere udsvingssignalet for tykkelse kan man dividere Γ(Q) med 3 . De resulterende data viser, at den overskydende dynamik på grund af tykkelsesudsving følger en lorentzisk funktion i Q, som for nylig bekræftet i grovkornede molekylære dynamik (MD)simuleringer 67. For at passe til den observerede overskydende dynamik udviklede Nagao et al.38 et udtryk baseret på den teoretiske ramme for membranudsving med Bingham et al.80 som følger.

I dette udtryk er Q0 den maksimale Q-værdi,der svarer til membrantykkelsen (som kan opnås uafhængigt af SANS-målinger), μ er membranens viskositet i flyet, AL er arealet pr. lipid (målt med SANS/ SAXS), og KA er områdets komprimerbarhedsmodulus. Hvis det antages, at KA kan beregnes ud fra κ ved hjælp af polymerbørstemodellen, reduceres dette udtryk til én egnet parameter, nemlig membranens viskositet μ, hvilket præsenterer en ny metode til måling af membranviskositet uden behov for fluorescensmærkning eller partikelbinding/sporing13. Forudsætningen er, at ifølge deformationsmodeller af elastiske tynde plader81, κ og KA er indbyrdes afhængige, således at: ,  hvor tm er den mekaniske (eller deformerbare) membrantykkelse og β er en konstant, der beskriver interleaflet kobling. Antagelsen er, at β = 12 for fuldt koblede foldere, β = 48 for helt afkoblede foldere, og β = 24 for mellemkoblede foldere. Sidstnævnte kaldes polymerbørstemodellen81 og har vist sig at gælde i enkeltkomponent- og binære flydende lipidmembraner39. Dette skal dog gribes an med forsigtighed. For eksempel, seneste simuleringer af Doktorova et al. 82 viste, at for at polymerbørstemodellen kan holde i umættede lipidmembraner, der indeholder kolesterol, skal der anvendes et modificeret udtryk for den mekaniske membrantykkelse. Ideelt set, hvis en uafhængig måling af KA er mulig, f.eks. ved hjælp af micropipette aspiration83, ville en kombination af KA-resultater med NSE-bøjningsstivhedsmålinger være en unik mulighed for at undersøge interleafletkobling i model og biologiske membraner – et langvarigt spørgsmål i membranbiofysik og strukturel biologi. Når værdierne af KA er valideret, kan de bruges i ligning 5 for at opnå den mesoskopiske membran viskositet.
hvor tm er den mekaniske (eller deformerbare) membrantykkelse og β er en konstant, der beskriver interleaflet kobling. Antagelsen er, at β = 12 for fuldt koblede foldere, β = 48 for helt afkoblede foldere, og β = 24 for mellemkoblede foldere. Sidstnævnte kaldes polymerbørstemodellen81 og har vist sig at gælde i enkeltkomponent- og binære flydende lipidmembraner39. Dette skal dog gribes an med forsigtighed. For eksempel, seneste simuleringer af Doktorova et al. 82 viste, at for at polymerbørstemodellen kan holde i umættede lipidmembraner, der indeholder kolesterol, skal der anvendes et modificeret udtryk for den mekaniske membrantykkelse. Ideelt set, hvis en uafhængig måling af KA er mulig, f.eks. ved hjælp af micropipette aspiration83, ville en kombination af KA-resultater med NSE-bøjningsstivhedsmålinger være en unik mulighed for at undersøge interleafletkobling i model og biologiske membraner – et langvarigt spørgsmål i membranbiofysik og strukturel biologi. Når værdierne af KA er valideret, kan de bruges i ligning 5 for at opnå den mesoskopiske membran viskositet.

Figur 1: NSE-instrumentdesign og synergistisk overlapning med længde/tidsskalaer af mesoskopisk membrandynamik. Den fremhævede neutron indikerer ændring i spinorientering (eller polariseringstab) på grund af energiudveksling mellem neutronen og prøven, mens den gennemsigtige neutron repræsenterer spin-ekko, dvs. ingen ændring i neutronspin på grund af nul energiudveksling. Den grå pil angiver muligheden for at dreje spektrometerets anden arm for at få adgang til større spredningsvinkler. (B) Billedlig repræsentation af hierarkisk dynamik i lipidmembraner, der viser forskellige dynamiske tilstande, der strækker sig over flere længde- og tidsskalaer. Det skraverede område repræsenterer den længde og de tidsskalaer, som NSE har adgang til, og som overlapper mesoskalaerne for kollektive membranudsving, nemlig udsving i bøjning og tykkelse. Klik her for at se en større version af dette tal.

Figur 2: Eksempler på mulige deuterationsordninger i NSE-forsøg med lipidmembraner. Forskellen i NSLD mellem haleregionen (~0 × 10-2 Å-2) og hovedgruppeområdet (~4,5 × 10-6 Å-2) af membranen skyldes hovedgruppens hydrering med deutereret buffer. Til højre: Halekontrast matchede membraner, således at membranens kulbrintehaleregion har samme NSLD som bufferen, som vist i den tilsvarende NSLD-profil langs membranen normal. (B) Domænedannende membraner med to neutronkontrastskemaer, hvor domænerne (i midten) eller matrixen (til venstre) er kontrastmatchede med bufferen, hvilket muliggør selektive undersøgelser af henholdsvis matrix- eller domænedynamik. Dette tal er blevet ændret fra Nickels et al., JACS 201541. (C) Asymmetriske membraner fremstillet ved cyclodextrinudveksling mellem protiated og deutererede lipidvesikler, hvilket resulterer i deuteration af den ene membranbrochure, samtidig med at den anden indlægsseddel opbevares protieret. Dette gør det muligt at studere den beslapserede folders bøjningsdynamik og giver indsigt i den mekaniske kobling mellem modsatrettede foldere i asymmetriske membraner. Dette tal er blevet ændret fra Rickeard et al., Nanoscale 202040. Klik her for at se en større version af dette tal.

Figur 3: Illustration af opsætningen tilautomatiseret ekstrudering af liposomer. (A) Specialbygget automatiseret ekstruder ved hjælp af en sprøjtepumpe, et mini ekstrudersæt og en aluminiums-/stålramme, der muliggør cykliske ekstruderinger. (B) og (C) viser forskellen i det visuelle udseende af lipidaffjedring før (mælkehvid) og efter (gennemsigtig opalblå) ekstrudering. Dette skyldes den første dannelse af mikron mellemstore lipid stakke eller gigantiske vesikler, som er på rækkefølgen af, eller større end, bølgelængden af synligt lys. Efter ekstrudering vil suspensionen bestå af nanoskopiske vesikler (~100 nm), som er mindre end bølgelængden af synligt lys, hvilket giver en gennemsigtig affjedring. Klik her for at se en større version af dette tal.

Figur 4: Repræsentative data fra NSE-eksperimenter med liposomale suspensioner. (A) Eksempel på et ekkosignal over en enkelt detektorpixel (markeret pixel i panel B), der viser ekkosignalets anfald ved hjælp af ligning (1) med en illustration af de forskellige parametre, der kræves i ekkoets pasform. Bemærk, at ekkosignalet afbildes som en funktion af fasevinklen i stedet for fasestrømmen som beskrevet i trin 4.7 i protokollen. (B) NSE-detektorbillede, der viser variationen i neutrontal pr. pixel. Billedet viser også elimineret detektor pixels (grøn) på grund af dårlige ekko signaler. Placeringen af detektorpixelerne i Q-buer (også kendt som Debye-Scherrer-ringe) giver Q-afhængigheden af den mellemliggende spredningsfunktion, der er nødvendig for at analysere og fortolke NSE-data. Dette tal blev ændret fra Ashkar, J. Appl. Phys. 202050. Klik her for at se en større version af dette tal.

Figur 5: Repræsentative resultater fra NSE-forsøg med liposomale suspensioner med forskellige deuterationsordninger. (A) Spredning af geometri af en neutron, der interagerer med et liposom, der viser spredningsvinklen, 2θ, og bølgevektoroverførslen.  (B) Mellemliggende spredningsfunktioner, S(Q,t) / S(Q,0), udviser henfald som en funktion af Fourier-tiden. Fit af de målte henfald til en strakt eksponentiel funktion givet ved ligning 3 giver afslapning satser, Γ(Q). (C) For fuldt protiated liposomer i deuterated buffer, Γ(Q) følger en Q3 afhængighed, typisk forbøjning dynamik. Den lineære pasform af de opnåede data til en Zilman-Granek model giver den bøjning stivhed modulus af membranen. (D) For tail deuterated liposomer observeres overskydende dynamik ud over bøjningsudsving og er mest udtalt ved Q-værdier, der svarer til membrantykkelsen. Montering af den overskydende dynamik til en lorentzisk funktion (ligning 5) tillader ekstraktion af membranens viskositet. Der blev indsamlet datasæt på NSE-spektrometeret ved NIST. Klik her for at se en større version af dette tal.
(B) Mellemliggende spredningsfunktioner, S(Q,t) / S(Q,0), udviser henfald som en funktion af Fourier-tiden. Fit af de målte henfald til en strakt eksponentiel funktion givet ved ligning 3 giver afslapning satser, Γ(Q). (C) For fuldt protiated liposomer i deuterated buffer, Γ(Q) følger en Q3 afhængighed, typisk forbøjning dynamik. Den lineære pasform af de opnåede data til en Zilman-Granek model giver den bøjning stivhed modulus af membranen. (D) For tail deuterated liposomer observeres overskydende dynamik ud over bøjningsudsving og er mest udtalt ved Q-værdier, der svarer til membrantykkelsen. Montering af den overskydende dynamik til en lorentzisk funktion (ligning 5) tillader ekstraktion af membranens viskositet. Der blev indsamlet datasæt på NSE-spektrometeret ved NIST. Klik her for at se en større version af dette tal.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
NSE er en kraftfuld og unik teknik til måling af mesoskopiske dynamik lipidmembraner under forskellige forhold. Den effektive udnyttelse af NSE afhænger af prøvekvaliteten, neutronkontrasten og det udvalg af tilgængelig dynamik, der kan undersøges for en given prøve. Der kræves således flere kritiske trin for at udføre vellykkede NSE-eksperimenter og indsamle data af høj kvalitet. Et vigtigt skridt i at sikre en effektiv brug af neutronstråletid under et NSE-eksperiment er at karakterisere de liposomale suspensioner med laboratoriebaserede metoder forud for NSE-eksperimentet. For exmaple kan størrelsesfordelingen (eller diffusionskonstanten) af ekstruderede liposomer bestemmes ved dynamisk lysspredning (DLS), let tilgængelig i individuelle laboratorier eller i fælles faciliteter84. Kryo-elektronmikroskopi er en anden charcateriseringsmetode, der for nylig blev valideret på liposomale prøver, hvor mikroskopibilleder i høj opløsning på kryokroterede dele af liposomal suspension effektivt kan bruges til at undersøge liposomal unilamellaritet65, domænedannelse85,86eller inkorporering af tilsætningsstoffer som nanopartikler76 og proteiner87. Alternativt kan småvinklet røntgenspredning (SAXS) bruges til at karakterisere membranstrukturen88, vurdere liposomal multilamellaritet65eller evaluere tilsætningsstoffernes virkninger på membran strukturelle egenskaber89. Ud over disse lab-baserede teknikker, er det meget tilrådeligt, at NSE målinger på liposomal prøver er parret med strukturelle undersøgelser ved hjælp af små-vinkel neutron spredning (SANS)54,90. SANS er et glimrende supplement til NSE, ikke kun til at erhverve strukturelle membranoplysninger, men også til at undersøge intensiteten af neutronspredningssignalet fra prøven, bekræfte kontrastordningen og træffe et informeret valg om det Q-interval, som NSE-målingerne skal udføres over. Derfor anbefales det, at NSE-brugere anmoder om SANS-stråletid, når de ansøger om NSE-eksperimenter.
NSE lider imidlertid af prøvebegrænsninger i undersøgelser af biologiske membraner. En af de vigtigste begrænsende faktorer ved sådanne forsøg er den standardmængde af prøve, der kræves til NSE-målinger (2-4 mL) og de høje prøvekoncentrationer på 100-200 mg membranmateriale (lipider og proteiner) for at opnå data af høj kvalitet. I mange tilfælde er produktion af sådanne mængder biologisk materiale ikke mulig eller er omkostningsoverkommelig. I sådanne scenarier er det muligt at reducere koncentrationen til 20-25 mg/mL, men dette ville kræve en stigning på mindst fire gange i anskaffelsestiden for at opnå sammenlignelige statistikker til prøver med 50 mg/mL-koncentrationer. Disse strenge krav til prøvevolumen og koncentration kan afhjælpes med den næste generation af NSE-spektrometre på neutronkilder med højere flux, såsom den anden målstation ved Oak Ridge National Lab og den europæiske spallationskilde. En anden kritisk begrænsning i udførelsen af NSE eksperimenter på lipid membraner kræver selektiv deuteration ordninger er manglen på kommerciel tilgængelighed af nogle deuterated varianter af lipid molekyler eller deres ublu priser, hvis de findes. I nogle tilfælde kan disse begrænsninger omgås ved at anmode om syntese af deutererede lipider (eller kolesterol, proteiner) gennem brugerdeuterationsfaciliteter, såsom biodeuterationslaboratoriet på Oak Ridge National Lab, den nationale deuterationsanlæg på ANSTO eller deuterationsfaciliteten ved ISIS Neutron og Muon Source. Adgang til disse faciliteter og deres syntesekapacitet er tilgængelig gennem indsendte brugerforslag, som gennemgås på grundlag af den videnskabelige værdi af den foreslåede materialesyntese og dens tilsigtede anvendelse i isotopfølsomme undersøgelser.
På trods af disse begrænsninger har anvendelsen af NSE-spektroskopi i undersøgelser af membranmekanik ført til bestemmelse af membranernes bøjningsstivhedsmoduli med forskellige grader af kompleksitet, fra enkeltkomponent lipidmembraner35,38 til multikomponent biomimetiske membraner41,66,91, som alle har avanceret vores forståelse af lipidmembranernes dynamiske natur. For eksempel har NSE bøjning stivhedsmålinger af lipidmembraner med forskellige molekylære enheder, f.eks. lipider af forskellige acylkædelængder og kædemætning38,72,92, givet vigtige oplysninger om molekylær kemis rolle i membranmekanik. Når disse målinger kombineres med strukturelle oplysninger, såsom membrantykkelse eller molekylær pakning93, begynder de at give nye perspektiver på den indbyrdes afhængighed mellem membranstruktur og dynamik, og hvordan de påvirker membranfunktionen. NSE's mesoskopiske skalaer placerer det unikt til sådanne grundlæggende undersøgelser af struktur-ejendomsforhold, mest relevante på længden af molekylære forsamlinger. Dette emne blev for nylig undersøgt i to NSE-undersøgelser af kolesterolrige lipidmembraner36 og i binære lipidmembraner med hydrofobisk mismatch mellem de to lipidkomponenter39. Begge undersøgelser fandt stærke beviser for, at membranmekanik skaleres med området pr. Lipid, hvilket underbygger konklusionerne fra en nylig all-atom MD-simuleringer af Doktorova et al.82. Disse fund understreger lipidmembranernes selvsamlede natur og giver et samlende billede af molekylær pakning som en nøgleparameter i definitionen af membrandynamiske og funktionelle egenskaber.
Andre anvendelser af NSE omfatter undersøgelser af membranernes mekaniske respons på små tilsætningsstoffer, herunder biologiske molekyler som kolesterol36,37, trehalose92, og melittin73,94eller uorganiske tilsætningsstoffer såsom nanopartikler til lægemiddelleveringsapplikationer76. NSE er også blevet brugt til at forstå, hvordan membranmekanik reagerer på ændringer i deres miljø, herunder temperatur92, pH74og tilstedeværelsen af fortrængning af makromolekyler96. Sådanne undersøgelser bidrager til en bedre forståelse af de faktorer, der påvirker blødgøring eller stivhed af lipidmembraner, under biologiske forhold relateret til sundhed og sygdom og i kontrollerede indstillinger til terapeutiske anvendelser. Især er NSE-målinger også blevet brugt til at undersøge effekten af antimikrobielle peptider på membrandynamik73,94,95. Yderligere eksempler på NSE applikationer på biomembraner omfatter undersøgelser af dynamikken i fladede membranstrukturer, kaldet thylakoider, som huser fotosyntetiske maskiner i cyanobakterielle celler97,98.
Man kan også bruge selektiv lipid deuteration i NSE undersøgelser til at undersøge dynamikken i specifikke membran funktioner, der er relevante for biologisk funktion. For eksempel brugte Nickels et al. selektiv lipiddeuteration i domænedannende lipidmembraner til at generere lateral kontrast i membranen, som tidligere illustreret af Heberle et al.28. Denne deuterationsordning muliggjorde uafhængige målinger af lipiddomænernes bøjningsstivhed og værts lipidmatrix41 (se figur 2B). Resultaterne bekræftede, at de to membranrum har forskellige bøjningsstivhedsmoduli, hvilket kunne være en køremekanisme for domænedannelse i cellulære membraner. I en nyere undersøgelse brugte Rickeard et al. cyclodextrinudveksling mellem protiated og deuterated liposomer for at opnå asymmetriske liposomer med isotopisk mærkede foldere40 (Figur 2C). Deres ende liposomer havde en protiated folder og en deuterated folder, der er kontrast matchet til bufferen, så undersøgelser af de enkelte folder dynamik og give en første direkte eksperimentel redegørelse for effekten af asymmetri og folder kobling på membran bøjning udsving.
Selektiv membrandeuteration er også blevet brugt i NSE-undersøgelser af membrantykkelsesudsving, en lang forudsagt dynamisk tilstand i lipidmembraner99, der først for nylig blev observeret med fremkomsten af NSE-spektroskopi35,100. Disse målinger bruger hale-deutererede membraner til at forstærke signalet fra membranens hovedgrupperegioner og løse tykkelsesudsvingssignalet. Denne type NSE-eksperimenter er relativt nye, men det er effektivt blevet brugt til at forstå den indbyrdes afhængighed af membranelastiske og viskøse egenskaber38, for at udforske skaleringen af bøjningsstivhed og viskositet med molekylær pakning i blandede lipidmembraner39, og for at undersøge de lokale virkninger af kolesterol på membranviskositet36. Et andet område af biologisk betydning, hvor denne dynamiske tilstand kan have vidtrækkende konsekvenser, er mesoskopiske membranproteininteraktioner95. Det er kendt, at funktionen af membranproteiner er tæt forbundet med hydrofobisk matchning mellem proteinet og værtsmembranen. Således kan variationer i membrantykkelse på grund af tykkelsesudsving fungere som en reguleringsmekanisme for membraneproteinernes funktion. NSE er yderst velegnet til sådanne undersøgelser, da det direkte kan sonde virkningerne af proteinbinding og indsættelse på membrantykkelse udsving. Nylige NSE-målinger fra vores gruppe (ikke offentliggjort) tyder på, at transmembraneproteinindsættelse i væsentlig grad kan undertrykke udsving i membrantykkelse og kan udgøre en potentiel mekanisme til regulering af signalhændelser. Dette er et presserende, men underudviklet forskningsområde, hvor NSE kan have betydelig indflydelse på forståelsen af membranernes dynamiske reaktioner på proteinbinding og indsættelse på længden og tidsskalaerne af vigtige biologiske funktioner, der formidles af interaktioner mellem proteiner og cellemembraner.
Sammenfattende har NSE udviklet sig i løbet af de sidste par år som et kraftfuldt værktøj til at afhøre membrandynamik over rumlige og tidsmæssige skalaer af vitale biologiske funktioner. Teknikken er hurtigt ved at vinde udbredt interesse og dens potentiale i at besvare centrale spørgsmål i membran funktion er ved at blive anerkendt. Kontrastvariationsfunktionerne i NSE har placeret det som en unik tilgang til måling af mesoskopiske membranegenskaber, der ellers ville være udfordrende at opnå. En anden væsentlig fordel ved NSE i forhold til traditionelle spektroskopimetoder i undersøgelser af membrandynamik er dens overlapning med den længde og tidsskala, der er tilgængelig med MD-simuleringer, hvilket giver mulighed for synergistiske eksperimentelle / beregningsundersøgelser for at få en molekylær niveauforståelse af de forskellige molekylære komponenter, der udgør membraner. På trods af sit løfte er der stadig visse begrænsninger i brugen af NSE i biologiske membranundersøgelser, herunder kravet om store prøvemængder, vanskeligheden ved selektiv deuteration i biologiske systemer og den relativt lave neutronflux på NSE-spektrometre, hvilket resulterer i længere måletider og begrænset tilgængelighed af stråletid. Disse mangler kunne imidlertid afhjælpes i den nærmeste fremtid med en konstant udvikling inden for neutronkilder og instrumentering sammen med fremskridt inden for deuterationsfaciliteter.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne erklærer ingen interessekonflikter og har intet at afsløre.
Acknowledgments
R. Ashkar takker M. Nagao, L.-R. Stingaciu, og P. Zolnierczuk for mange nyttige diskussioner og for deres hyppige bistand med NSE eksperimenter på deres respektive strålelinjer. Forfatterne anerkender brugen af neutron spin ekko spektrometre på NIST og ORNL. NSE spektrometeret på NIST støttes af Center for High Resolution Neutron Scattering, et partnerskab mellem National Institute of Standards and Technology og National Science Foundation i henhold til aftale nr. DMR-1508249. NSE-spektrometeret ved ORNL's Spallation Neutron Source understøttes af Scientific User Facilities Division, Office of Basic Energy Sciences, US Department of Energy. Oak Ridge National Laboratory administreres af UT-Battelle, LLC under US DOE Contract No. DE-AC05-00OR22725.
Materials
| Name | Company | Catalog Number | Comments |
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. Ø, Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes--a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich's model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a "modeling manifesto". Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D. Neutrons in Soft Matter. Imae, T., Kanaya, T., Furusaka, M., Torikai, N. , Wiley. ch6 147-182 (2011).
- Pynn, R. Neutron Spin Echo. Mezei, F., Pappas, C., Gutberlet, T. , Springer. Berlin Heidelberg. 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , Oxford University Press. (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- Neutron activation and scattering calculator. , Available from: https://www.ncnr.nist.gov/resources/activation/ (2021).
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , Walter de Gruyter, Inc. 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles - as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O'Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -R., O'Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).
Tags
Biologi Problem 171 kollektiv lipiddynamik bøjningsmodulus områdekomprimering tykkelsesudsving membranviskositet selektiv deuteration liposome forberedelseErratum
Formal Correction: Erratum: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions
Posted by JoVE Editors on 08/06/2021.
Citeable Link.
An erratum was issued for: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions. The Introduction, Protocol, and Representative Results sections have been updated.
In the Introduction, the fith pargraph was updated from:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 2 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
to:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 0 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
In the Protocol, step 1.1 was updated from:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 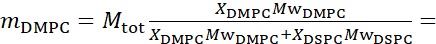 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
to:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 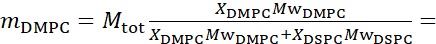 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
In the Representative Results, the fist pagargaph was updted from:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 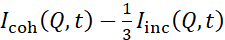 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
to:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 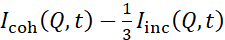 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
In the Representative Reults, Figure 2 was updated from:
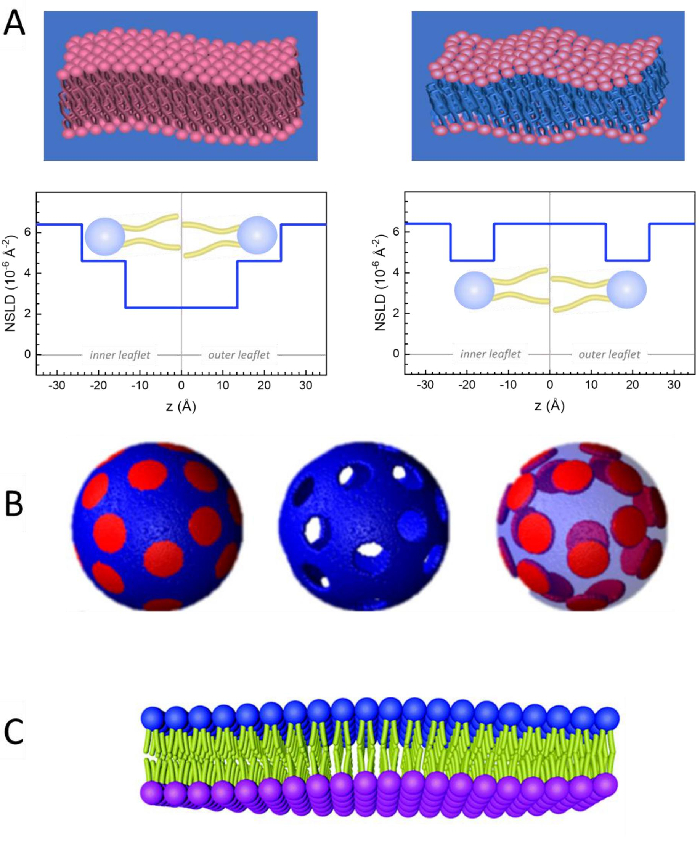
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the headgroup (~2 × 10-2 Å-2) and tail region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
to:
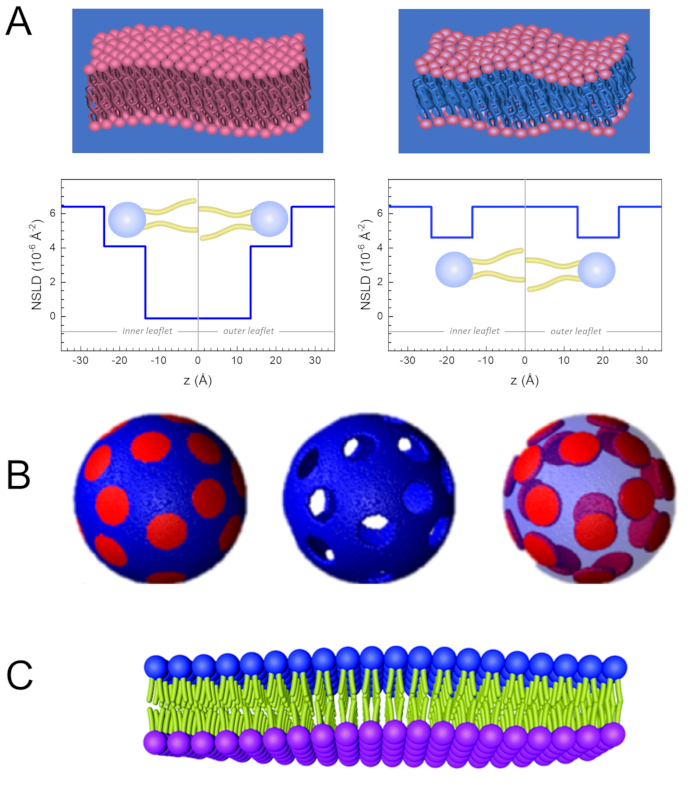
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the tail region (~0 × 10-2 Å-2) and headgroup region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.



 133,4 mg, hvor XDMPC og XDSPC er molefraktionerne af DMPC og DSPC, her indstillet til henholdsvis 0,7 og 0,3, og MwDMPC og MwDSPC er de molvægte, der er angivet med henholdsvis 677,9 g/mol og 790,1 g/mol. Tilsvarende mDSPC = 66,6 mg. Denne deuterationsordning øger spredningskontrasten mellem membranen (NSLD ~ 0 × 10-6 Å-2) og den deutererede buffer (NSLD ~ 6,4 × 10-6 Å-2) og forstærker signalet fra membranoverkommninger (se figur 2A venstre panel).
133,4 mg, hvor XDMPC og XDSPC er molefraktionerne af DMPC og DSPC, her indstillet til henholdsvis 0,7 og 0,3, og MwDMPC og MwDSPC er de molvægte, der er angivet med henholdsvis 677,9 g/mol og 790,1 g/mol. Tilsvarende mDSPC = 66,6 mg. Denne deuterationsordning øger spredningskontrasten mellem membranen (NSLD ~ 0 × 10-6 Å-2) og den deutererede buffer (NSLD ~ 6,4 × 10-6 Å-2) og forstærker signalet fra membranoverkommninger (se figur 2A venstre panel). Mtot er den samlede masse, der er her til 200 mg. Se eksemplet ovenfor for DMPC-DSPC lipid bilayers med forskellige deuteration ordninger.
Mtot er den samlede masse, der er her til 200 mg. Se eksemplet ovenfor for DMPC-DSPC lipid bilayers med forskellige deuteration ordninger.

 , således ændringer i spintilstanden på grund af energiudveksling mellem neutronen og prøven, opdages som et fald i polariseringen (fra enhed).
, således ændringer i spintilstanden på grund af energiudveksling mellem neutronen og prøven, opdages som et fald i polariseringen (fra enhed).
