ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Dit artikel beschrijft de protocollen voor monstervoorbereiding, gegevensreductie en gegevensanalyse in neutronenspinecho (NSE) studies van lipidemembranen. Verstandige deuteriumetikettering van lipiden maakt toegang tot verschillende membraandynamiek op mesoscopische lengte- en tijdschalen mogelijk, waarover vitale biologische processen plaatsvinden.
Abstract
Lipide bilayers vormen de belangrijkste matrix van celmembranen en zijn het primaire platform voor nutriëntenuitwisseling, eiwit-membraan interacties, en virale ontluikende, onder andere vitale cellulaire processen. Voor een efficiënte biologische activiteit moeten celmembranen stijf genoeg zijn om de integriteit van de cel en zijn compartimenten te behouden, maar toch vloeibaar genoeg om membraancomponenten, zoals eiwitten en functionele domeinen, te laten diffuus en interactief te zijn. Deze delicate balans van elastische en vloeibare membraaneigenschappen, en hun impact op de biologische functie, vereist een beter begrip van de collectieve membraandynamiek over mesoscopische lengte- en tijdschalen van belangrijke biologische processen, zoals membraanvervormingen en eiwitbindende gebeurtenissen. Een van de technieken die dit dynamisch bereik effectief kan onderzoeken, is neutronenspinecho (NSE) spectroscopie. In combinatie met deuteriumetikettering kan NSE worden gebruikt om direct toegang te krijgen tot buig- en dikteschommelingen, evenals mesoscopische dynamiek van bepaalde membraankenmerken. Dit document geeft een korte beschrijving van de NSE-techniek en schetst de procedures voor het uitvoeren van NSE-experimenten op liposomale membranen, inclusief details van monstervoorbereidings- en deuteratieschema's, samen met instructies voor het verzamelen en verminderen van gegevens. Het artikel introduceert ook gegevensanalysemethoden die worden gebruikt om belangrijke membraanparameters te extraheren, zoals de buigstijfheidsmodulus, gebiedsdrukmodulus en viscositeit in het vlak. Om het biologische belang van NSE-studies te illustreren, worden enkele voorbeelden van membraanverschijnselen besproken die door NSE zijn onderzocht, namelijk het effect van additieven op de stijfheid van membraanbuiging, de impact van domeinvorming op membraanschommelingen en de dynamische signatuur van membraan-eiwitinteracties.
Introduction
Het begrip van celmembranen en hun functie is de afgelopen decennia opmerkelijk geëvolueerd. De vroegere visie van celmembranen als passieve lipide-bilayers die celgrenzen definiëren en huismembraaneiwitten1 is geleidelijk veranderd in een dynamisch model waarin lipide-tweelagen een belangrijke rol spelen bij het reguleren van vitale biologische processen, waaronder cellulaire signalering, moleculaire uitwisseling en eiwitfunctie - om er maar een paar2,3,4,5,6te noemen . Dit besef dat celmembranen zeer dynamisch zijn en voortdurend een remodellering en moleculaire herverdeling ondergaan, heeft wetenschappelijke verkenningen aangedrongen die verder gaan dan evenwichtsstructuren van membranen7,8,9. Dienovereenkomstig zijn meerdere benaderingen ontwikkeld om de verschillende dynamische modi in biologische en bio-geïnspireerde lipidenmembranen te bestuderen. Tot op heden hebben de meeste van deze studies zich voornamelijk gericht op diffusieve moleculaire bewegingen10,11,12,13 en macroscopische vormschommelingen14,15,16, waardoor een aanzienlijke kloof is achtergelaten in het begrijpen van de intermediaire membraandynamiek, d.w.z. collectieve fluctuaties van lipidenassemblages bestaande uit enkele 10-100s lipidemoleculen. Deze dynamieken komen voor over lengteschalen van enkele tientallen tot enkele 100 Å en in de loop van de tijd schalen van sub-ns tot enkele honderden ns (zie figuur 1), hier aangeduid als mesoscopische schalen. Het is inderdaad op deze schalen dat belangrijke biologische activiteit plaatsvindt op membraanniveau17. Dit omvat virale ontluikende18, kanaal gating19, en membraan-eiwit interacties20. Het is ook belangrijk erop te wijzen dat het energielandschap van membraaneiwitten21,22 aantoont dat conformationele veranderingen in eiwitten — noodzakelijk voor hun regulerende rol — plaatsvinden in de tijdschalen23 van collectieve membraanschommelingen , waarbij het belang van mesoscopische dynamiek in de biologische functie van celmembranen en hun bio-geïnspireerde analogen verder wordt benadrukt20. Dit artikel richt zich op de twee primaire mesoscopische dynamische modi in lipidemembranen, namelijk buigschommelingen en dikteschommelingen.
De grootste uitdaging bij het direct onderzoeken van deze fluctuatiemodi is de moeilijkheid om tegelijkertijd toegang te krijgen tot hun ruimtelijke en temporele schalen met behulp van standaardspectroscopiemethoden. De andere uitdaging is dat directe contacttechnieken dezelfde fluctuaties kunnen beïnvloeden als die welke bedoeld zijn om16te meten . Dit wordt nog verergerd door de compositorische en structurele complexiteit van biologische membranen24,25, wat resulteert in niet-homogene membraankenmerken, waaronder lipidedomeinvorming26,27,28,29,30 en membraanasymmetrie31,32,33- die selectieve sondes vereisen om de dynamiek van verschillende membraankenmerken te begrijpen. Gelukkig kunnen deze uitdagingen worden overwonnen met niet-invasieve neutronenspectroscopiemethoden, zoals neutronenspinecho (NSE), die inherent toegang hebben tot de vereiste lengte- en tijdschalen, en verder studies van selectieve membraankenmerken mogelijk maken zonder hun fysisch-chemische omgeving te veranderen34. Inderdaad, in de afgelopen jaren is NSE spectroscopie geëvolueerd tot een unieke en krachtige sonde van collectieve membraandynamica35. Resultaten van NSE-studies over lipidemembranen hebben nieuwe inzichten opgeleverd in mechanische36,37 en visco-elastische38,39 eigenschappen van lipidemembranen en hebben nieuw licht geworpen op hun potentiële rol in biologische functie40,41.
De NSE-spectroscopietechniek is gebaseerd op een interferometrisch instrumentontwerp, voor het eerst voorgesteld door Mezei42, met behulp van een reeks spin-flippers en magnetische spoelen om de precessie van de neutronenspin te regelen terwijl neutronen het instrument doorkruisen. Het ontwerp berust op magnetische spiegeling van de magnetische veldelementen ten opzichte van de monsterpositie (figuur 1A). Dit houdt in dat bij gebrek aan energie-uitwisseling tussen het neutron en het monster, het neutron hetzelfde aantal spinprecessies uitvoert, in tegengestelde richting, in de eerste en tweede helft van het instrument (let op de π-flipper tussen de twee precessiespoelen). Als gevolg hiervan blijft de uiteindelijke spintoestand van het neutron ongewijzigd ten opzichte van de begintoestand - een fenomeen dat spin-echo wordt genoemd (zie transparant neutron in figuur 1A). Wanneer het neutron echter energetisch interageert met het monster, wijzigt de energie-uitwisseling het aantal spinprecessies in de tweede helft van het instrument, wat leidt tot een andere eindspintoestand (zie figuur 1A). Dit wordt experimenteel gedetecteerd als een verlies in polarisatie, zoals later in dit artikel zal worden getoond. Voor meer informatie over de NSE-techniek wordt de lezer verwezen naar speciale technische documenten42,43,44,45.
Hier wordt een vereenvoudigde beschrijving gepresenteerd om een ruwe schatting te geven van de lengte- en tijdschalen die toegankelijk zijn met NSE. De lengteschalen worden bepaald door het bereik van haalbare wavevectortransfers, Q = 4π sin θ/λ, waarbij 2θ de verstrooiingshoek is en λ de neutronengolflengte. Men kan zien dat Q wordt ingesteld door het golflengtebereik en de mate van rotatie van de tweede arm van de spectrometer (zie figuur 1A). Een typisch Q-bereikop NSE-spectrometers is ~0.02-2 Å-146,47en tot 0.01-4 Å-1 met recente upgrades48,49, overeenkomend met ruimtelijke schalen van ~1-600 Å. Aan de andere kant wordt de toegankelijke tijdschaal berekend op basis van de totale precessiehoek (of fase) die het neutron in de magnetische precessiespoelen heeft verkregen, en blijktdeze 50te zijn :  . In deze uitdrukking is t de Fouriertijd gedefinieerd als
. In deze uitdrukking is t de Fouriertijd gedefinieerd als  , waar is de
, waar is de  neutronen gyromagnetische verhouding,
neutronen gyromagnetische verhouding,  is de spoellengte, en
is de spoellengte, en  is de sterkte van het magnetisch veld van de spoel. Het is de moeite waard erop te wijzen dat de Fouriertijd een hoeveelheid is die strikt afhankelijk is van de instrumentgeometrie, de magnetische veldsterkte en de neutronengolflengte. Bijvoorbeeld, met behulp van neutronen van golflengte
is de sterkte van het magnetisch veld van de spoel. Het is de moeite waard erop te wijzen dat de Fouriertijd een hoeveelheid is die strikt afhankelijk is van de instrumentgeometrie, de magnetische veldsterkte en de neutronengolflengte. Bijvoorbeeld, met behulp van neutronen van golflengte  = 8 Å en instrumentinstellingen van
= 8 Å en instrumentinstellingen van  = 1,2 m en
= 1,2 m en  = 0,4 T, wordt de Fouriertijd berekend op t ~ 50 ns. Experimenteel wordt de Fouriertijd afgestemd door de stroom in de precessiespoelen (d.w.z. magnetische veldsterkte) te veranderen of verschillende neutronengolflengten te gebruiken, wat resulteert in typische NSE-tijdschalen van ~ 1 ps tot 100 ns. Recente upgrades in NSE-spectrometers hebben echter toegang gegeven tot langere Fourier-tijden, tot ~ 400 ns op de J-NSE-Phoenix spectrometer op de Heinz Maier-Leibnitz Zentrum51 en de SNS-NSE spectrometer in Oak Ridge National Lab48, en tot ~ 1.000 ns op de IN15 NSE spectrometer in het Institut Laue-Langevin (ILL).
= 0,4 T, wordt de Fouriertijd berekend op t ~ 50 ns. Experimenteel wordt de Fouriertijd afgestemd door de stroom in de precessiespoelen (d.w.z. magnetische veldsterkte) te veranderen of verschillende neutronengolflengten te gebruiken, wat resulteert in typische NSE-tijdschalen van ~ 1 ps tot 100 ns. Recente upgrades in NSE-spectrometers hebben echter toegang gegeven tot langere Fourier-tijden, tot ~ 400 ns op de J-NSE-Phoenix spectrometer op de Heinz Maier-Leibnitz Zentrum51 en de SNS-NSE spectrometer in Oak Ridge National Lab48, en tot ~ 1.000 ns op de IN15 NSE spectrometer in het Institut Laue-Langevin (ILL).
Naast directe toegang tot de lengte- en tijdschaal van membraandynamica heeft NSE de inherente mogelijkheden van neutronenisotopengevoeligheid52. Met name het vermogen van neutronen om verschillend te interageren met de isotopen van waterstof, het meest voorkomende element in biologische systemen, resulteert in een andere neutronenverstrooiingslengtedichtheid,34 of NSLD (het equivalent van de optische brekingsindex50), wanneer protium wordt vervangen door deuterium. Dit maakt een benadering mogelijk die bekend staat als contrastvariatie, die vaak wordt gebruikt om specifieke membraankenmerken te benadrukken of anderen te verbergen - het laatste scenario wordt contrastmatching genoemd. Een frequente toepassing van contrastvariatie/matching is de vervanging van water (NSLD = -0,56 × 10-6 Å-2) door zwaar water of D2O (NSLD = 6,4 × 10-6 Å-2) om het neutronensignaal van geprotieerde lipidemembranen (NSLD ~ 0 × 10-6 Å-2)te versterken. Deze aanpak is zeer effectief in studies van membraanstructuur omdat de penetratie van D2O in het hoofdgroepgebied van het membraan een nauwkeurige bepaling van de membraandikten mogelijk maakt (zie figuur 2A, linkerpaneel) en van de locatie van verschillende lipidesubgroepen wanneer meer geavanceerde modellen worden toegepast53,54. Dit artikel belicht enkele voorbeelden over het gebruik van contrastvariatie voor studies naar collectieve dynamiek in biomimetische membranen en bepaalde membraankenmerken.
Hier wordt de effectiviteit van NSE bij het bieden van unieke inzichten in dynamische en functionele membraaneigenschappen geïllustreerd aan de hand van tastbare voorbeelden van NSE-studies naar model- en biologisch relevante lipidenmembraansystemen met de nadruk op mesoschaaldynamiek in vrijstaande membranen, in de vorm van liposomale suspensuspensuspensussen. Voor NSE-metingen van membraandynamica in het vlak wordt de lezer verwezen naar speciale publicaties over beweidingsincidentie neutronenspin-echospectroscopie (GINSES)55,56 en andere studies van uitgelijnde multilamellar membraanstapels57,58,59,60.
Voor de eenvoud belicht dit artikel drie verschillende schema's van membraandeuteratie geïllustreerd op een goed bestudeerd domeinvormend, of fasescheidend, lipidenbilayer-systeem van 1,2-dimyristoyl-sn-glycero-3-phosphocholine (DMPC) en 1,2-distearoyl-sn-glycero-3-phosphocholine (DSPC) mengsels61,62. De twee lipiden worden gekenmerkt door een mismatch in hun koolwaterstofketenlengte (14 carbons/tail in DMPC vs 18 carbons/tail in DSPC) en hun gel-fluid overgangstemperatuur (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C). Dit resulteert in laterale fasescheiding in DMPC:DSPC-membranen bij temperaturen tussen de bovenste en onderste overgangstemperaturen van het mengsel63. De hier overwogen deuteratieschema's zijn gekozen om de verschillende dynamische modi aan te tonen die toegankelijk zijn in NSE-metingen op liposomale membranen, namelijk buigschommelingen, dikteschommelingen en selectieve buig-/dikteschommelingen van laterale domeinen. Alle lipidesamenstellingen worden gerapporteerd voor DMPC:DSPC-bilayers bereid met een molfractie van 70:30, met behulp van in de handel verkrijgbare protiated en perdeuterated varianten van DMPC en DSPC. Alle monstervoorbereidingsstappen zijn gebaseerd op 4 ml liposomale suspensie, in D2O, met een lipideconcentratie van 50 mg/ml, voor een totale lipidenmassa van Mtot = 200 mg per monster.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Deuteratieschema vereist voor het experiment
- Voor buigfluctuatiemetingen, maak volledig geprotieerde liposomen in D2O (D 99,9%) of D2O-buffer (bijv. fosfaatbuffer bereid met D2O in plaats van H2O). Gebruik volledig geprotiteerd DMPC (C36H72NO8P) en DSPC (C44H88NO8P) met
 133,4 mg, waarbij XDMPC en XDSPC de molfracties van DMPC en DSPC zijn, hier ingesteld op respectievelijk 0,7 en 0,3, en MwDMPC en MwDSPC zijn de molaire gewichten gegeven door 67.7 en 0.3. Evenzo, mDSPC = 66,6 mg. Dit deuteratieschema verhoogt het strooicontrast tussen het membraan (NSLD ~ 0 × 10-6 Å-2) en de gedeutereerde buffer (NSLD ~ 6,4 × 10-6 Å-2) en versterkt het signaal van membraangolvingen (zie figuur 2A linkerpaneel).
133,4 mg, waarbij XDMPC en XDSPC de molfracties van DMPC en DSPC zijn, hier ingesteld op respectievelijk 0,7 en 0,3, en MwDMPC en MwDSPC zijn de molaire gewichten gegeven door 67.7 en 0.3. Evenzo, mDSPC = 66,6 mg. Dit deuteratieschema verhoogt het strooicontrast tussen het membraan (NSLD ~ 0 × 10-6 Å-2) en de gedeutereerde buffer (NSLD ~ 6,4 × 10-6 Å-2) en versterkt het signaal van membraangolvingen (zie figuur 2A linkerpaneel). - Om de buigdynamiek van bepaalde laterale membraankenmerken te meten, bijvoorbeeld matrixdynamica in fasescheidende DMPC:DSPC-membranen, gebruikt u protiated DMPC (C36H72NO8P) en gedeutereerd, DSPC-d83 (C44H5NO 8 PD 83 , Mw 873,7 g/mol), zodat mDMPC = 128,8 mg en mDSPC Dit deuteratieschema minimaliseert de verstrooiing van de ongewenste DSPC-rijke domeinen, waardoor selectieve metingen van buigschommelingen uit de DMPC-rijke matrix mogelijk zijn (zie figuur 2B midden).
OPMERKING: Om de optimale lipidendeuteratie te vinden die nodig is voor een specifiek contrastmatchingschema, gebruikt u beschikbare webgebaseerde verstrooiingslengtedichtheidscalculators (SLD), zoals die ontwikkeld door het NIST Center for Neutron Research64. Deze webgebaseerde interfaces zijn uitgerust met gebruiksvriendelijke tools voor eenvoudige berekening van de SLD van lipiden met verschillende graden van deuteratie, evenals die van lipidenmengsels. - Gebruik voor NSE-metingen van gemiddelde membraandikteschommelingen (zonder zijdelings contrast) staart-gedeutereerde varianten van de samenstellende lipiden; d.w.z. DMPC-d54 (C36H18NO8PD54, 732.3 g/mol) en DSPC-d70 (C44H18NO8PD70,860.1g/mol)35,38 , zodat mDMPC-d54 = 133,0 mg en mDSPC-d70 Dit contrastschema (figuur 2A, rechterpaneel) versterkt het verstrooiingssignaal van de lipidekopgroepen (NSLD ~ 4,5 × 10-6 Å -2) door de staartgroep (NSLD ~ 6,4 × 10-6 Å-2) te matchen met de gedeutereerde buffer die de detectie van fluctuaties in membraandikte mogelijk maakt.
- Voor diktefluctuatiestudies van bepaalde membraancompartimenten, bijvoorbeeld DMPC-rijke matrix, gebruikt u dezelfde strategie die in stap 1.2 wordt beschreven door protiated DMPC-lipiden te vervangen door hun staart-gedeutereerde analogen, d.w.z. DMPC-d54, zodat de DSPC-rijke domeinen contrast-afgestemd zijn op de gedeutereerde buffer en het primaire verstrooiingssignaal afkomstig is uit het hoofdgroepgebied van de staart-gedeutereerde DMPC-rijke matrix.
2. Bereiding van lipidensuspensie voor extrusie
- Bereken de massa van elk bestanddeel in het monster, afhankelijk van de samenstelling van het monster. Als vuistregel geldt dat voor monsters met meerdere moleculaire componenten de massa van een component wordt gegeven door de molaire massa, Mwi, gewogen door de molfractie, Xi, en genormaliseerd over alle componenten, zodat: waarbij
 Mtot de totale massa is, hier ingesteld op 200 mg. Zie het voorbeeld hierboven voor DMPC-DSPC lipide bilayers met verschillende deuteratieschema's.
Mtot de totale massa is, hier ingesteld op 200 mg. Zie het voorbeeld hierboven voor DMPC-DSPC lipide bilayers met verschillende deuteratieschema's. - Weeg met behulp van een digitale semi-microbalans de berekende massa's lipiden (en andere monsterbestanddelen, bijvoorbeeld eiwitten, nanodeeltjes, enz.) en voeg ze toe aan een flacon of kolf met ronde bodem – vergeet niet om de flacon of kolf van tevoren te wegen. Voeg 1 ml oplosmiddel toe om de gewogen componenten op te lossen door handmatig in een afzuigkap te mengen. Gebruik chloroform of ethanol voor zuivere lipidenmonsters. Kies voor monsters met extra, niet-lipidecomponenten (bijv. nanodeeltjes) een gemeenschappelijk oplosmiddel dat alle componenten verspreidt.
- Voor kleine lipidehoeveelheden (<10 mg), bereid een stamoplossing en pipetteer het vereiste volume in het mengsel.
OPMERKING: Voeg geen overmatige hoeveelheden oplosmiddel toe, omdat dit de hieronder beschreven droogstap van het oplosmiddel aanzienlijk zal vertragen.
- Voor kleine lipidehoeveelheden (<10 mg), bereid een stamoplossing en pipetteer het vereiste volume in het mengsel.
- Droog de lipideoplossing in een kap door voorzichtig een inert gas (bijv. stikstof, argon) in de flacon te gieten terwijl u de flacon langzaam onder een hoek draait. Houd de flacons in gekanteld om een dunne film gedroogd lipide op de wanden van de flacon te creëren, waardoor ze gelijkmatig kunnen drogen. Plaats de flacon met tussenpozen in een waterbad bij 35 °C om verdampingsgemedieerde koeling te omzeilen, wat de verdamping van oplosmiddelen zal vertragen.
- Plaats de flacons een nacht in een vacuümoven op ~35 °C om het resterende oplosmiddel volledig te verwijderen. Voor onverzadigde lipiden, zuiver vacuüm met een inert gas om oxidatie te minimaliseren.
- Om volledige verwijdering van oplosmiddelen te garanderen, weegt u de flacon na het drogen van lipiden en bevestigt u dat er geen overtollige massa is die groter is dan de gemeten hoeveelheden materialen. Doe dit door de massa van de flacon af te trekken van de gemeten massa na het drogen. Als er een overmaat aan massa is, droogt u het monster nog 6 uur onder vacuüm. Herhaal dit proces indien nodig.
- Hydrateer de lipidenfilm met 4 ml D2O of D2O-buffer om een lipideconcentratie van 50 mg/ml te verkrijgen. Voor lipiden met hoge overgangstemperaturen, zoals DMPC-DSPC-mengsels, verwarmt u de buffer tot boven de overgangstemperatuur (60 °C) om een gelijkmatige menging te garanderen.
OPMERKING: Aangezien NSE-experimenten relatief grote monstervolumes (~4 ml) vereisen, kunt u overwegen het monster te hydrateren met de helft van de vereiste buffer, d.w.z. 2 ml, om het aantal extrusies per monster te minimaliseren (zie rubriek 3). Voeg in dit geval de resterende helft van de buffer na extrusie toe. Merk op dat de capaciteit van spuiten die bij extrusie worden gebruikt, beperkt is tot 1 ml. Hydrateren met 4 ml buffer zou dus vier sets extrusie vereisen. - Vortex-mix de gehydrateerde lipideoplossing totdat de lipidefilm volledig is opgelost en niet langer zichtbaar is op de wanden van de flacon. In dit stadium vormen de gehydrateerde lipiden multilamlaire blaasjes en micron-formaat multilamellar-stapels en lijkt de suspensie melkachtig wit.
- Om het breken van de lipidenstapels te vergemakkelijken en multilamellariteit te verminderen, voert u vijf vries-/dooicycli uit door de flacon gehydrateerde lipidenoplossing in een laboratoriumvriezer (bij voorkeur -80 °C vriezer) te plaatsen totdat deze volledig bevroren is en vervolgens de flacon over te brengen naar een waterbad van 35 °C totdat de lipideoplossing volledig ontdooid is. Draai de ontdooide oplossing homogeen. Herhaal dit nog vier keer.
OPMERKING: Als alternatief kan een droogijsbad worden voorbereid op snelvriezen door aceton en droog ijs te combineren.
3. Extrusie van de gehydrateerde lipidenoplossing
- Monteer de extruderopstelling met behulp van een polycarbonaatmembraan tussen twee membraansteunen en voeg twee papierfilters aan elke kant toe om extra ondersteuning te bieden. Gebruik een polycarbonaatmembraan met een poriegrootte die overeenkomt met de doelliposomale grootte (gemeenschappelijke poriegroottes voor NSE-experimenten zijn 50 nm en 100 nm - meestal zorgen liposomen met een diameter van 100 nm voor minder beperkte membraanschommelingen, maar kleinere 50 nm-liposomen kunnen worden gebruikt voor krommingsstudies). Zorg ervoor dat het polycarbonaatmembraan volledig is uitgerekt voordat u de montage voltooit en de externe extruderbehuizing aandraait.
- Hydrateer het polycarbonaatmembraan door ~0,3 ml D2O of D2O-buffer een paar keer door de membraanassemblage te laten gaan met luchtdichte glazen spuiten. Gebruik dezelfde buffer die wordt gebruikt bij de monstervoorbereiding. Laat het minstens 10 minuten staan en zuig de buffer er vervolgens volledig uit voordat u het monster introduceert.
- Vul een gasdichte spuit van 1 ml met de bereide lipideoplossing en steek deze in één uiteinde van het extruderapparaat. Steek vervolgens een lege spuit in het tegenovergestelde uiteinde. Zodra de spuiten zijn aangesloten op de extruderassemblage, plaatst u deze in het extruderblok.
- Indien verhoogde temperaturen nodig zijn voor extrusie, zoals bij verzadigde lipiden met hoge overgangstemperaturen (bijv. DSPC, Tm = 55 °C), verwarmt u het extruderverwarmingsblok voor boven de lipideovergangstemperatuur (bijv. 60 °C), door het verwarmingsblok op een kookplaat te plaatsen of door een circulatiebad te gebruiken zoals weergegeven in figuur 3A.
OPMERKING: Deze stap is cruciaal om een homogene menging van lipiden te garanderen en om extreme druk tijdens extrusie te voorkomen, waardoor het polycarbonaatmembraan kan scheuren. Voor lipidenmonsters met lage overgangstemperaturen (<25 °C) voert u de extrusie uit bij kamertemperatuur. - Om de lipideoplossing te extruderen, bevestigt u de extruderset aan een programmeerbare spuitpomp met een aluminium/stalen frame zoals weergegeven in figuur 3A. Voeg voor temperatuurgecontroleerde extrusies een op maat gemaakte extruderbasis met een vloeistofkanaal toe en bevestig deze aan een circulerend waterbad.
- Programmeer de spuitpomp om 15-20 extrusiecycli uit te voeren volgens de handleiding van de fabrikant. Wanneer geëxtrudeerd, de kleur van de lipide oplossing verandert van melkachtig wit naar transparant opaal blauw(Figuur 3B,C),wat wijst op een uiteindelijke liposomale grootte die kleiner is dan de golflengte van zichtbaar licht, zoals verwacht. Volg de onderstaande stappen voor het type spuitpomp in figuur 3A.
- Begin met het aanpassen van de pompinstellingen. Houd de rateknop ingedrukt en voer de extrusiesnelheid (50,99 ml/h) in, druk vervolgens op de diameterknop en voer de spuitdiameter (4,606 mm) in. Gebruik de pijl-omhoog onder elk cijfer op het scherm om die cijferwaarde te wijzigen.
- Plaats de extruderset met de monsterspuit naar rechts (zie figuur 3A). Druk op de knop Terugtrekken totdat het optreklampje gaat branden. Druk op Start en wacht tot het monster in de linker (lege) spuit is afgegeven.
- Druk op de stopknop net voordat de monsterspuit (rechts) volledig leeg is. Noteer het gedoseerde volume en gebruik het om de extrusiecyclus te programmeren. Houd de rateknop ingedrukt totdat fase 1 (PH:01) op het scherm verschijnt. Druk op de volumeknop om het eerder opgenomen afgegeven volume in te voeren. Zorg er in deze fase voor dat het treklicht uit staat – dit geeft het monster in de juiste richting af.
- Druk nogmaals op de toets Snelheid en gebruik de meest rechtse pijl-omhoog om toegang te krijgen tot fase 2 (PH:02). Druk op Volume om dezelfde waarde in te voeren als het eerder geregistreerde afgegeven volume. Druk in deze fase op de terugtrekknop totdat het optreklampje is ingeschakeld – hiermee wordt het monster naar links afgegeven.
- Om deze cyclus te herhalen, drukt u nogmaals op de toets Snelheid en gebruikt u de meest rechtse pijl-omhoog om toegang te krijgen tot fase 3 (PH:03). Druk op de volumeknop totdat LP:SE op het scherm verschijnt en stel deze in op 20. Dit is het aantal lussen of herhalingen dat de pomp zal uitvoeren. Druk ten slotte op de knop Snelheid, toegang tot fase 4 (PH:04) en druk op de volumeknop om bij de stopfunctie te komen. De pomp is nu ingesteld voor geautomatiseerde extrusie.
- Druk op Start om de extrusiecyclus te starten.
- Leeg de spuit met de geëxtrudeerde lipidensuspensie in een schone flacon en bereid u voor op opslag of metingen. Voor lipidemonsters met een hoge smelttemperatuur slaat u het monster boven de vloeistoffaseovergang op totdat het is gemeten. Bewaar monsters anders op kamertemperatuur.
- Vries geëxtrudeerde monsters niet in, omdat bevriezing ervoor zorgt dat de blaasjes barsten (de suspensie wordt weer melkwit).
4. NSE-metingen voor de steekproef(en) en vermindering van de verzamelde gegevens
- Voorafgaand aan het NSE-experiment, karakteriseer het geëxtrudeerde liposomale monster uit stap 3.7 met behulp van beschikbare methoden om een adequate monsterkwaliteit te garanderen. Een lijst van mogelijke charcaterisatiemethoden die kunnen worden gebruikt om de kwaliteit van liposomale suspensuspensussingen voor NSE-experimenten te beoordelen, bijvoorbeeld grootteverdeling, multilamellariteit, laterale membraanstructuur, is opgenomen in de discussiesectie.
- Bepaal het Q-bereik en de bijbehorende instrumentinstellingen die nodig zijn voor het experiment. Gebruik voor buigstijfheidsmetingen van lipide-tweelagen een Q-bereik van ~(0,04 - 0,2) Å-1. Gebruik voor studies van membraandikteschommelingen een Q-bereik van ~(0,04 - 0,2) Å-1 dat overeenkomt met de membraandikte35,66,67.
OPMERKING: Bespreek de experimentele opstelling met de instrumentwetenschapper voor het begin van het experiment. Zoals eerder vermeld, is SANS-karakterisering van het monster noodzakelijk, vooral als voorafgaande informatie over het verstrooiingssignaal niet beschikbaar is, zoals in selectief gedeutereerde membranen. U kunt ook statische (ook bekend als diffractie) metingen uitvoeren over een beperkt Q-bereik op het NSE-instrument, met het voorbehoud dat dergelijke metingen veel langer duren in vergelijking met SANS. - Laad met behulp van een spuit of een transferpipet de geëxtrudeerde liposomale suspensie(s) in de aangewezen monstercellen die beschikbaar zijn op NSE-straallijnen. Merk op dat standaard NSE-monstercellen verkrijgbaar zijn in diktes van 1, 2, 3 en 4 mm. Kies de celdikte op zo'n manier om het strooisignaal te optimaliseren en tegelijkertijd het onsamenhangende achtergrondsignaal op een redelijke intensiteit te houden.
OPMERKING: Gebruik als vuistregel monstercellen met 1 of 2 mm pathlength voor geprotieerde liposomen in gedeutereerde buffer - dikkere cellen kunnen resulteren in meerdere verstrooiingseffecten die moeilijk te corrigeren zijn. Voor liposomen met hogere niveaus van deuteraties (bijv. staartcontrastgematchte liposomen of asymemetrische liposomen met enkele protiated bijsluiters), overweeg dan om een dikkere monstercel (bijv. 3 of 4 mm pathlength) te gebruiken om de telstatistieken te verbeteren als het monster in grotere hoeveelheden beschikbaar is - soms kan dit onbetaalbaar zijn. - Bereid een identieke monstercel voor de buffer voor. Gebruik dezelfde buffer als in de liposomale suspensie. Metingen aan de buffer zijn nodig voor intensiteitsnormalisatie en achtergrondcorrecties (BKG).
- Plaats de monstercel(en) in de monsterhouder van de NSE-spectrometer, programmeer de meetruns en verzamel echogegevens. Overleg met de instrumentwetenschapper over het programmeren van de metingen als een nieuwe NSE-gebruiker.
- Voer twee extra sets metingen uit die nodig zijn voor de gegevensreductie: resolutie (R) en transmissie (T) metingen.
- Voer resolutiemeting (R) uit op een elastische strooireferentie (bijv. koolstof) — die onder dezelfde instellingen moet worden uitgevoerd; d.w.z. dezelfde wavevector en Fourier keer als de monster- en buffermetingen.
- Voer transmissiemetingen (T) uit op het monster en de buffer om de intensiteit van de overgedragen neutronenbundel te berekenen (zie stap 4.9. hieronder). De transmissie wordt berekend als de verhouding van neutronentellingen ten opzichte van het monster of de buffer gedeeld door de neutronentellingen voor een open bundel (d.w.z. met een lege monsterpositie).
- Gebruik de speciale software voor gegevensreductie voor de NSE-spectrometer waarop de metingen worden uitgevoerd om de verzamelde gegevens te verminderen.
OPMERKING: Verschillende spectrometers kunnen verschillende software of gebruikersinterfaces gebruiken. Hieronder vindt u een voorbeeld van NSE-gegevensreductie met behulp van de Data Analysis and Visualization Environment (DAVE)68 software die speciaal is geschreven voor de NSE-spectrometer in het NIST Center for Neutron Research.- Open de DAVE-software en selecteer NSE-gegevens verminderen in het menu gegevensreductie. Er verschijnen verschillende pop-upvensters.
- Upload de gegevensbestanden over verschillende Q-waarden met behulp van de Open .echo Files vanuit het bestandsmenu. Deze bestanden komen overeen met de onbewerkte gegevensbestanden met de spin echo signalen en hebben de extensie .echo in de bestandsnaam. Zodra het uploaden van bestanden is voltooid, worden de bestanden weergegeven onder de beschikbare gegevenssets.
- Klik met de rechtermuisknop op het geselecteerde bestand en label het op basis van de meting waaraan het overeenkomt; d.w.z. Monster, Cel (voor lege cel of buffer) of Resolutie.
- Groepeer de detectorpixles in 2 x 2 om de signaal-ruisverhouding te verbeteren met behulp van het tabblad Gegevensset. Pas dezelfde binning toe op alle bestanden; d.w.z. resolutie, cel en monster.
- Inspecteer de gegevens over alle pixelgroepen en maskeer personen met slechte signalen (zie figuur 4B)door op de m-toets op het toetsenbord te drukken. Druk op Enter om toegang te krijgen tot een pop-upvenster om hetzelfde masker toe te passen op alle Fourier-tijden of volgende Fourier-tijden. Dit kan ook op elk moment tijdens gegevensreductie op afzonderlijke pixels worden toegepast. Gemaskeerde pixels worden groen.
- Zorg ervoor dat de verzamelde gegevens de vorm hebben van een echosignaal, d.w.z. cosinusfunctie in termen van fasestroom, over elke detectorpixel (zie figuur 4A).
OPMERKING: De fasestroom is evenredig met de precessiehoek van de neutronenspin; daarom is het gebruikelijk om de fasestroom weer te geven als een fasehoek zoals weergegeven in figuur 4A. Voor metingen op gepulseerde bronnen wordt extra tijd van vluchtberekeningen toegepast op de gegevens om de echosignalen te verkrijgen als functie van de neutronengolflengte in een neutronenpuls. - Begin met het aanpassen van het resolutiebestand. Selecteer een resolutiebestand in de geüploade bestandslijst en klik met de rechtermuisknop op het bestand. Selecteer Bewerkingen aanpassen: Echo's (resolutie) aanpassen in het pop-upmenu.
- Zorg ervoor dat de pasvormen van de echosignalen een aantal montageparameters opleveren, waaronder de parameter A,vereist in stap 4.8. De fits worden automatisch uitgevoerd met behulp van de volgende expressie.

Hier is ζ de periode van het echosignaal (d.w.z. cosinusfunctie in figuur 4A),σ de breedte van de Gaussiaanse envelop wordt bepaald door de gemiddelde golflengte en golflengtespreiding van de neutronenbundel van het incident, Φc de fasestroom is en Φ0 het echopunt dat afhankelijk is van het veldpad dat neutronenervaren 50. Fysische informatie over het monster wordt gecodeerd in de amplitude, A, van de cosinusfunctie in vergelijking (1).
OPMERKING: De breedte van de Gaussiaanse envelop is gebaseerd op waarden die vooraf zijn bepaald door de instrumentwetenschapper en mag niet worden gewijzigd. De andere paramters zijn variabelen die over elke pixel op het specifieke echosignaal zijn gemonteerd. - Inspecteer de pasvormresultaten door op elke pixel te klikken om de resulterende pasparameters, de kwaliteit van de pasvorm en de gemiddelde vierkante afwijking van de pasvorm weer te geven. Als u de fout wilt inspecteren die aan elke montageparameter over de hele detector is gekoppeld, selecteert u Afbeeldingsopties en selecteert u vervolgens de fittingparameter van belang. Dit genereert een kaart met de waarde van de passende paramter over elke pixel. Klik met de rechtermuisknop op de detectorafbeelding. Er verschijnt een pop-upvenster met een foutbalktoewijzing van de geselecteerde montageparameter.
- Als de pasvorm over een specifieke pixel onbevredigend is (bijvoorbeeld parameeters met grote foutbalken passen), monteert u het signaal opnieuw op die specifieke pixel. Selecteer die pixel, druk op het tabblad Passend en druk vervolgens op Pixel aanpassen. Voer nieuwe startparameters in voor de fase (Φ0) en periode (ζ) in het tabblad Montage om een meer bevredigende pasvorm te verkrijgen.
OPMERKING: Het is handig om de gemonteerde fase uit te stippelen als functie van Fourier Time. Ga hiervoor naar het hoofdplotvenster en selecteer Fase v. Fouriertijd aanpassen. Dit perceel moet soepel en continu zijn. Inspecteer discontinuïteiten in deze plot en monteer de pixels waarmee ze overeenkomen opnieuw.
- Verklein het voorbeeld- of celbestand door het bijbehorende bestand te selecteren in de lijst met geüploade en gelabelde bestanden.
- Inspecteer alle pixels en maskeer pixels met slechte statistieken zoals beschreven in stap 4.7.5.
- Klik met de rechtermuisknop op het bestand en selecteer Bewerkingen aanpassen: Fasen importeren (Voorbeeld, Cel). Hiermee worden de fasen en het toegepaste masker geïmporteerd uit het resolutiebestand.
- Monteer de echosignalen volgens dezelfde procedure die eerder is beschreven voor het resolutiebestand (stappen 4.7.8-4.7.10). Wijzig bij het aanpassen van de voorbeeld- en celbestanden de waarden van het periode- en echofasepunt dat is geïmporteerd uit de resolutie past niet. Deze parameters zijn afhankelijk van de instrumentele instellingen en mogen niet variëren met samples.
- Voordat u doorgaat met gegevensreductie, voert u het straalcentrum in voor alle gegevensbestanden. Selecteer het gegevensbestand, ga naar het tabblad Algemeen en voer de middenwaarden X en Y-balk in. Deze waarden worden tijdens het experiment vastgelegd.
- Zodra de aanvragen voor de bestanden Sample, Cell en Resolution zijn voltooid, berekent u de genormaliseerde tussenliggende verstrooiingsfunctie die later in gegevensanalyse en -interpretatie moet worden gebruikt. Om dat te doen, klikt u met de rechtermuisknop op het voorbeeldbestand dat moet worden verkleind uit de lijst met aangesloten bestanden en selecteert u Berekenen I(Q) in het pop-upmenu. Er verschijnt een venster met invoerkeuzes voor de resolutie- en celbestanden (d.w.z. bufferbestanden) en het aantal Q-bogen (zie stap 4.9). Druk na het invoeren van alle vereiste informatie op de OK-knop. De resultaten worden in een nieuw venster weergegeven.
OPMERKING: De gegevensreductie wordt uitgevoerd volgens de volgende vergelijking om de genormaliseerde tussenliggende verstrooiingsfunctie te verkrijgen69.
waarbij t fouriertijd is, Nomhoog en Nomlaag, zijn de neutronentellingen in de niet-spin-flip- en spin-flipconfiguraties (gemeten met respectievelijk de π/2-flippers uit en de π-flipper uit en aan), en de superscripts BKG en R, overeenkomen met de achtergrond- en resolutiemetingen, zoals gedefinieerd in de stappen 4.4 en 4.6. Merk op dat de bundelpolarisatie , dus verandert in de spintoestand als gevolg van energie-uitwisseling tussen het neutron en het monster wordt gedetecteerd als een daling van de polarisatie (van eenheid).
, dus verandert in de spintoestand als gevolg van energie-uitwisseling tussen het neutron en het monster wordt gedetecteerd als een daling van de polarisatie (van eenheid).
- Groeper ten slotte de detectorpixels in Q-bogenzoals weergegeven in figuur 4B om de Q-afhankelijkheidvan de genormaliseerde tussenstrooifunctie, S(Q,t) / S(Q, 0 ) te verkrijgen. Dit wordt technisch gegevens binning genoemd en moet verstandig worden gedaan, d.w.z. rekening houdend met de telstatistieken van de steekproef en de verwachte standaardafwijking van de gegevens over de gegroepeerde pixels.
- Voor sterk verstrooiende monsters verdeelt u de detector in meer Q-bogen met behoud van redelijke foutbalken op de resulterende tussenliggende verstrooiingsfunctie, S(Q,t) / S(Q, 0). Dit levert meer Q-datapunten op en is belangrijk voor de hieronder beschreven data-analyseprocedure. Houd er rekening mee dat bij zwak verstrooiende monsters overmatige binning resulteert in slechte vervalsignalen, d.w.z. grote foutbalken op S(Q,t) / S(Q, 0), wat kan leiden tot grote onzekerheden.
5. Gegevensanalyse en -interpretatie
- Pas de genormaliseerde tussenliggende verstrooiingsfuncties, S(Q,t) / S(Q, 0), verkregen uit de bovenstaande gegevensreductie tot een uitgerekt exponentieel functioneren met een uitrekkende exponent van 2/370.

OPMERKING: Een voorbeeld van deze pasvormen is te vinden in figuur 5B. Pasvormen van S(Q,t) / S(Q, 0) tot vergelijking (3) leveren de Q-afhankelijke ontspanningspercentages op Γ(Q). - PlotΓ(Q)als functie van Q en passen op een geschikt model om relevante membraanparameters te extraheren.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
NSE-studies die toegang krijgen tot buigschommelingen worden meestal uitgevoerd over een Q-bereik van ~ (0,04 - 0,2) Å-1. Dit Q-bereik komt overeen met tussenliggende lengteschalen tussen de membraandikte en de liposomale radius, waar buigdynamiek domineert. Meting over een uitgebreid Q-bereik kan toegang geven tot extra dynamische modi, waaronder liposomale diffusie en intramembrane dynamica. Voor meer informatie over de cross-over in membraandynamiek die NSE heeft geraadpleegd , raadpleegt u deze relevante publicaties25,71. Het is belangrijk om te benadrukken dat NSE-signalen evenredig zijn met:  , waar Icoh en Iinc respectievelijk de coherente en onsamenhangende verstrooiingsintensiteit van het monster zijn. Daarom is het raadzaam om NSE-liposomale monsters te bereiden in gedeutereerde buffers (d.w.z. buffers bereid met D2O in plaats van H2O) om het onsamenhangende verstrooiingssignaal te minimaliseren, voornamelijk bijgedragen door het waterstofgehalte van het monster. In sommige gevallen kunnen echter tussentijdse deuteratieschema's (d.w.z. het gebruik van mengsels van D2O en H2O) nodig zijn om optimale contrastomstandigheden te verkrijgen. Typisch, NSE metingen van membraan buigen fluctuaties worden uitgevoerd op volledig protiated liposomen in gedeutereerde buffer, aangeduid als volledig contrasterende liposomen in figuur 5. Dit deuteratieschema resulteert in een groot NSLD-verschil tussen de membraankern (~ 0 × 10-6 Å-2)en de gedeutereerde vloeistofomgeving (~ 6,4 × 10-6 Å-2),wat het strooisignaal van de liposomale membranen aanzienlijk verbetert en de meetstatistieken van buigdynamiek verbetert. Dit contrastschema(figuur 2A linkerpaneel) wordt vaak gebruikt in studies naar buigstijfheid van lipidenmembranen met enkelvoudige38,72 en meerdere39,66 lipidecomponenten en in studies naar membraanverzachting/verstijving door biologische insluitsels (bijv. cholesterol, medicijnmoleculen,peptiden/eiwitten)36 , 37,73,74 , 75,en synthetischeadditieven(bijv. nanodeeltjes).
, waar Icoh en Iinc respectievelijk de coherente en onsamenhangende verstrooiingsintensiteit van het monster zijn. Daarom is het raadzaam om NSE-liposomale monsters te bereiden in gedeutereerde buffers (d.w.z. buffers bereid met D2O in plaats van H2O) om het onsamenhangende verstrooiingssignaal te minimaliseren, voornamelijk bijgedragen door het waterstofgehalte van het monster. In sommige gevallen kunnen echter tussentijdse deuteratieschema's (d.w.z. het gebruik van mengsels van D2O en H2O) nodig zijn om optimale contrastomstandigheden te verkrijgen. Typisch, NSE metingen van membraan buigen fluctuaties worden uitgevoerd op volledig protiated liposomen in gedeutereerde buffer, aangeduid als volledig contrasterende liposomen in figuur 5. Dit deuteratieschema resulteert in een groot NSLD-verschil tussen de membraankern (~ 0 × 10-6 Å-2)en de gedeutereerde vloeistofomgeving (~ 6,4 × 10-6 Å-2),wat het strooisignaal van de liposomale membranen aanzienlijk verbetert en de meetstatistieken van buigdynamiek verbetert. Dit contrastschema(figuur 2A linkerpaneel) wordt vaak gebruikt in studies naar buigstijfheid van lipidenmembranen met enkelvoudige38,72 en meerdere39,66 lipidecomponenten en in studies naar membraanverzachting/verstijving door biologische insluitsels (bijv. cholesterol, medicijnmoleculen,peptiden/eiwitten)36 , 37,73,74 , 75,en synthetischeadditieven(bijv. nanodeeltjes).
Metingen van buigschommelingen resulteren in ontspanningssnelheden die volgen op een afhankelijkheid van Q3, zoals voorspeld door Zilman en Granek voor thermisch golvende elastische dunne platen70. Een verfijnde vorm van deze Q-afhankelijkheidwordt verkregen uit theoretische correcties door Watson en Brown78, die rekening houden met de effecten van intermodererende wrijving voorgesteld door Seifert en Langer79. Door bovendien het neutrale vlak te definiëren dat zich op het raakvlak tussen de hydrofiele hoofdgroepen en de hydrofobe staarten van het membraan bevindt, kunnen de buigontspanningssnelheden vervolgens op de volgende uitdrukking worden aangebracht38.

waarbij ηbuff de bufferviscositeit is, is kBT de thermische energie, κ en is de buigstijfheid van het gemeten membraan (of van het contrasterende deel van het membraan in selectief gedeutereerde systemen). Dit type meting maakt directe berekening van membraanelastiek eigenschappen mogelijk in de vorm van de buigstijfheidsmodulus. Merk op dat κ wordt geëxtraheerd uit de helling van de lineaire pasvorm van Γ vs. Q3, zoals weergegeven in figuur 5C.
Daarentegen laten NSE-metingen van membraandikteschommelingen afwijkingen zien van de Q3-afhankelijkheidin Γ(Q) rond Q-waarden die overeenkomen met de membraandikte (zie figuur 2 in ref.66). Om het diktefluctuatiesignaal te isoleren, kan men Γ(Q) delen door Q3, zoals weergegeven in figuur 5D. De resulterende gegevens tonen aan dat de overtollige dynamiek als gevolg van dikteschommelingen een Lorentziaanse functie in Qvolgt , zoals onlangs bevestigd in grofkorrelige moleculaire dynamica (MD) simulaties67. Om aan de waargenomen overdynamiek te voldoen, ontwikkelden Nagao et al.38 een uitdrukking gebaseerd op het theoretische kader van membraanschommelingen door Bingham et al.80 als volgt.

In deze uitdrukking is Q0 de piek Q-waardedie overeenkomt met de membraandikte (die onafhankelijk kan worden verkregen uit SANS-metingen), μ de in-plane membraanviscositeit is, AL het gebied per lipide (gemeten met SANS / SAXS) en KA de gebiedsdrukbaarheidsmodulus. Ervan uitgaande dat KA kan worden berekend op basis van κ met behulp van het polymeerborstelmodel, vermindert deze expressie tot één geschikte parameter, namelijk de membraanviscositeit μ, die een nieuwe benadering presenteert om de viscositeit van het membraan te meten zonder dat fluorescentieetikettering of deeltjestenthering / tracking nodig is13. Het uitgangspunt is dat volgens vervormingsmodellen van elastische dunne platen81, κ en KA onderling afhankelijk zijn, zodat: ,  waarbij tm de mechanische (of vervormbare) membraandikte is en β een constante is die interleafletkoppeling beschrijft. De veronderstelling is dat β = 12 voor volledig gekoppelde bijsluiters, β = 48 voor volledig ontkoppelde bijsluiters en β = 24 voor tussenliggende gekoppelde bijsluiters. Dit laatste wordt aangeduid als het polymeerborstelmodel81 en is aangetoond dat het van toepassing is in eencomponenten- en binaire vloeistoflipidemembranen39. Dit moet echter met voorzichtigheid worden benaderd. Bijvoorbeeld recente simulaties door Doktorova et al. 82 toonde aan dat om het polymeerborstelmodel in onverzadigde lipidenmembranen met cholesterol te houden, een gewijzigde uitdrukking van de mechanische membraandikte moet worden gebruikt. Idealiter, als een onafhankelijke meting van KA mogelijk is, bijvoorbeeld met behulp van micropipet aspiratie83, dan zou het combineren van KA-resultaten met NSE-buigstijfheidsmetingen een unieke kans bieden om interleafletkoppeling in model en biologische membranen te onderzoeken - een al lang bestaande vraag in membraanbiofysica en structurele biologie. Zodra de waarden van KA zijn gevalideerd, kunnen ze worden gebruikt in vergelijking 5 om de mesoscopische membraanviscositeit te verkrijgen.
waarbij tm de mechanische (of vervormbare) membraandikte is en β een constante is die interleafletkoppeling beschrijft. De veronderstelling is dat β = 12 voor volledig gekoppelde bijsluiters, β = 48 voor volledig ontkoppelde bijsluiters en β = 24 voor tussenliggende gekoppelde bijsluiters. Dit laatste wordt aangeduid als het polymeerborstelmodel81 en is aangetoond dat het van toepassing is in eencomponenten- en binaire vloeistoflipidemembranen39. Dit moet echter met voorzichtigheid worden benaderd. Bijvoorbeeld recente simulaties door Doktorova et al. 82 toonde aan dat om het polymeerborstelmodel in onverzadigde lipidenmembranen met cholesterol te houden, een gewijzigde uitdrukking van de mechanische membraandikte moet worden gebruikt. Idealiter, als een onafhankelijke meting van KA mogelijk is, bijvoorbeeld met behulp van micropipet aspiratie83, dan zou het combineren van KA-resultaten met NSE-buigstijfheidsmetingen een unieke kans bieden om interleafletkoppeling in model en biologische membranen te onderzoeken - een al lang bestaande vraag in membraanbiofysica en structurele biologie. Zodra de waarden van KA zijn gevalideerd, kunnen ze worden gebruikt in vergelijking 5 om de mesoscopische membraanviscositeit te verkrijgen.

Figuur 1: NSE-instrumentontwerp en synergetische overlap met lengte/tijdschalen van mesoscopische membraandynamica. (A) Schematisch van de verschillende magnetische elementen van een NSE-instrument, gebruikt om de spin van neutronen die het instrument van links naar rechts doorkruisen te manipuleren. Het gemarkeerde neutron duidt op een verandering in de centrifugeoriëntatie (of polarisatieverlies) als gevolg van de energie-uitwisseling tussen het neutron en het monster, terwijl het transparante neutron spin-echo vertegenwoordigt, d.w.z. geen verandering in de neutronenspin als gevolg van energie-nuluitwisseling. De grijze pijl geeft de mogelijkheid aan om de tweede arm van de spectrometer te draaien om toegang te krijgen tot grotere verstrooiingshoeken. (B) Picturale weergave van hiërarchische dynamiek in lipidemembranen, met verschillende dynamische modi die meerdere lengte- en tijdschalen omvatten. Het gearceerde gebied vertegenwoordigt de lengte- en tijdschalen waartoe NSE toegang heeft, die overlappen met de mesoma's van collectieve membraanschommelingen, namelijk buig- en dikteschommelingen. Klik hier om een grotere versie van deze afbeelding te bekijken.

Figuur 2: Voorbeelden van mogelijke deuteratieschema's in NSE-experimenten op lipidemembranen. (A) Links: Volledig contrasterende membranen, bijv. geprotieerde membranen in gedeutereerde buffer, met het NSLD-profiel langs het normale naar het membraanoppervlak. Het verschil in de NSLD tussen het staartgebied (~0 × 10-2 Å-2)en de kopgroepregio (~4,5 × 10-6 Å-2) van het membraan is te wijten aan de hydratatie van de hoofdgroep met gedeutereerde buffer. Rechts: Staartcontrast komt overeen met membranen zodat het koolwaterstofstaartgebied van het membraan dezelfde NSLD heeft als de buffer, zoals weergegeven in het overeenkomstige NSLD-profiel langs het membraan normaal. (B) Domeinvormende membranen met twee neutronencontrastschema's waarbij de domeinen (midden) of de matrix (links) contrastafhankelijk zijn met de buffer, waardoor selectieve studies van respectievelijk matrix- of domeindynamica mogelijk zijn. Dit cijfer is gewijzigd van Nickels et al., JACS 201541. (C) Asymmetrische membranen bereid door cyclodextrine uitwisseling tussen geprotieerde en gedeutereerde lipide blaasjes, resulterend in de deuteratie van de ene membraanbijsluiter terwijl de andere bijsluiter protiated blijft. Dit maakt studies van de buigdynamiek van de geprotieerde bijsluiter mogelijk en geeft inzicht in de mechanische koppeling tussen tegengestelde folders in asymmetrische membranen. Dit cijfer is gewijzigd van Rickeard et al., Nanoschaal 202040. Klik hier om een grotere versie van deze afbeelding te bekijken.

Figuur 3: Illustratie van de opstelling voor geautomatiseerde extrusie van liposomen. (A) Op maat gemaakte geautomatiseerde extruder met behulp van een spuitpomp, een mini-extruderset en een aluminium/stalen frame om cyclische extrusies mogelijk te maken. (B) en (C) tonen het verschil in visuele verschijning van lipidensuspensuspensingen vóór (melkachtig wit) en na (transparante opaalblauwe) extrusie. Dit komt door de eerste vorming van micron-sized lipide stapels of gigantische blaasjes die in de orde van, of groter dan, de golflengte van zichtbaar licht. Na extrusie zal de suspensie bestaan uit nanoscopische blaasjes (~ 100 nm), die kleiner zijn dan de golflengte van zichtbaar licht, wat een transparante suspensie oplevert. Klik hier om een grotere versie van deze afbeelding te bekijken.

Figuur 4: Representatieve gegevens uit NSE-experimenten met liposomale suspensusingen. (A) Voorbeeld van een echosignaal over een enkele detectorpixel (gemarkeerde pixel in paneel B), die de pasvormen van het echosignaal laat zien met behulp van vergelijking (1), met een illustratie van de verschillende parameters die nodig zijn in de echo-fit. Merk op dat het echosignaal wordt uitgezet als functie van de fasehoek in plaats van de fasestroom zoals besproken in stap 4.7 van het protocol. (B) NSE-detectorbeeld dat de variatie in neutronentellingen per pixel weergeeft. De afbeelding toont ook geëlimineerde detectorpixels (groen) als gevolg van slechte echosignalen. Het binning van de detectorpixels in Q-bogen (ook bekend als Debye-Scherrer ringen) levert de Q-afhankelijkheid op van de tussenliggende verstrooiingsfunctie, die nodig is voor het analyseren en interpreteren van NSE-gegevens. Dit cijfer is gewijzigd van Ashkar, J. Appl. Phys. 202050. Klik hier om een grotere versie van deze afbeelding te bekijken.

Figuur 5: Representatieve resultaten van NSE-experimenten met liposomale suspensuspensus met verschillende deuteratieschema's. (A) Verstrooiingsgeometrie van een neutron dat interageert met een liposoom, dat de verstrooiingshoek, 2θ, en de wavevectoroverdracht toont,  . (B) Tussenliggende verstrooiingsfuncties, S(Q,t) / S(Q,0), vertonen verval als functie van de Fourier-tijd. De pasvorm van de gemeten vervalsen aan een uitgerekt exponentieel functie gegeven door vergelijking 3 levert de ontspanningssnelheden op, Γ(Q). (C) Voor volledig geprotieerde liposomen in gedeutereerde buffer volgt Γ(Q) een Q3 afhankelijkheid, typisch voor buigdynamiek. De lineaire pasvorm van de verkregen gegevens op een Zilman-Granek-model levert de buigstijfheidsmodulus van het membraan op. (D) Voor staart gedeutereerde liposomen wordt naast buigschommelingen ook een overdynamiek waargenomen die het meest uitgesproken is bij Q-waarden die overeenkomen met de membraandikte. Door de overtollige dynamiek aan te passen aan een Lorentziaanse functie (vergelijking 5) kan de membraanviscositeit worden afzuigd. Op de NSE spectrometer bij NIST zijn datasets verzameld. Klik hier om een grotere versie van deze afbeelding te bekijken.
. (B) Tussenliggende verstrooiingsfuncties, S(Q,t) / S(Q,0), vertonen verval als functie van de Fourier-tijd. De pasvorm van de gemeten vervalsen aan een uitgerekt exponentieel functie gegeven door vergelijking 3 levert de ontspanningssnelheden op, Γ(Q). (C) Voor volledig geprotieerde liposomen in gedeutereerde buffer volgt Γ(Q) een Q3 afhankelijkheid, typisch voor buigdynamiek. De lineaire pasvorm van de verkregen gegevens op een Zilman-Granek-model levert de buigstijfheidsmodulus van het membraan op. (D) Voor staart gedeutereerde liposomen wordt naast buigschommelingen ook een overdynamiek waargenomen die het meest uitgesproken is bij Q-waarden die overeenkomen met de membraandikte. Door de overtollige dynamiek aan te passen aan een Lorentziaanse functie (vergelijking 5) kan de membraanviscositeit worden afzuigd. Op de NSE spectrometer bij NIST zijn datasets verzameld. Klik hier om een grotere versie van deze afbeelding te bekijken.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
NSE is een krachtige en unieke techniek in het meten van mesoscopische dynamiek van lipidenmembranen onder verschillende omstandigheden. Het effectieve gebruik van NSE hangt af van de monsterkwaliteit, het neutronencontrast en het bereik van de toegankelijke dynamiek die voor een bepaald monster kan worden onderzocht. Er zijn dus verschillende kritieke stappen nodig voor het uitvoeren van succesvolle NSE-experimenten en het verzamelen van hoogwaardige gegevens. Een belangrijke stap in het waarborgen van het effectieve gebruik van neutronenstraaltijd tijdens een NSE-experiment is het karakteriseren van de liposomale suspensuspensussingen met laboratoriumgebaseerde methoden voorafgaand aan het NSE-experiment. Voor exmaple kan de grootteverdeling (of diffusieconstante) van geëxtrudeerde liposomen worden bepaald door dynamische lichtverstrooiing (DLS), direct beschikbaar in individuele laboratoria of in gedeelde faciliteiten84. Cryo-elektronenmicroscopie is een andere charcaterisatiemethode die onlangs is gevalideerd op liposomale monsters, waarbij microscopiebeelden met hoge resolutie op cryomicrotomed secties van liposomale suspensie effectief kunnen worden gebruikt om liposomale unilamellaliteit65, domeinvorming85,86of de integratie van additieven zoals nanodeeltjes76 en eiwitten87te onderzoeken . Als alternatief kan x-ray scattering (SAXS) met een kleine hoek worden gebruikt om de membraanstructuur88te karakteriseren, liposomale multilamellariteit65te beoordelen of de effecten van additieven op membraanstructuureigenschappen te evalueren89. Naast deze lab-gebaseerde technieken is het zeer raadzaam dat NSE-metingen op liposomale monsters worden gecombineerd met structurele studies met behulp van small-angle neutron scattering (SANS)54,90. SANS is een uitstekende aanvulling op NSE, niet alleen voor het verkrijgen van structurele membraaninformatie, maar ook voor het onderzoeken van de intensiteit van het neutronenverstrooiingssignaal uit het monster, het bevestigen van het contrastschema en het maken van een weloverwogen keuze over het Q-bereik waarover de NSE-metingen moeten worden uitgevoerd. Daarom wordt aanbevolen dat NSE-gebruikers SANS-beamtime aanvragen bij het aanvragen van NSE-experimenten.
NSE lijdt echter aan steekproefbeperkingen in studies van biologische membranen. Een van de belangrijkste beperkende factoren van dergelijke experimenten is de standaardhoeveelheid monster die nodig is voor NSE-metingen (2-4 ml) en de hoge monsterconcentraties van 100-200 mg membraanmateriaal (lipiden en eiwitten) om gegevens van hoge kwaliteit te verkrijgen. In veel gevallen is de productie van dergelijke hoeveelheden biologisch materiaal niet haalbaar of is het kosten onbetaalbaar. In dergelijke scenario's is het mogelijk om de concentratie te verlagen tot 20-25 mg/ml, maar dit zou ten minste een verviervoudiging van de verwervingstijd vereisen om vergelijkbare statistieken te verkrijgen als monsters met concentraties van 50 mg/ml. Deze strenge eisen inzake monstervolume en concentratie kunnen worden verlicht met de volgende generatie NSE-spectrometers op neutronenbronnen met een hogere flux, zoals het tweede doelstation in Oak Ridge National Lab en de European Spallation Source. Een andere kritische beperking bij het uitvoeren van NSE-experimenten op lipidemembranen die selectieve deuteratieschema's vereisen, is het gebrek aan commerciële beschikbaarheid van sommige gedeutereerde varianten van lipidemoleculen of hun exorbitante prijzen, indien beschikbaar. In sommige gevallen kunnen deze beperkingen worden omzeild door de synthese van gebiteerde lipiden (of cholesterol, eiwitten) aan te vragen via gebruikersdeuteratiefaciliteiten, zoals het bio-deuteratielab in Oak Ridge National Lab, de nationale deuteratiefaciliteit bij ANSTO of de deuteratiefaciliteit bij de ISIS Neutron and Muon Source. Toegang tot deze faciliteiten en hun synthesemogelijkheden is beschikbaar via ingediende gebruikersvoorstellen die peer-reviewed zijn op basis van de wetenschappelijke verdienste van de voorgestelde materiaalsynthese en het beoogde gebruik ervan in isotopengevoelige studies.
Ondanks deze beperkingen heeft de toepassing van NSE-spectroscopie in studies van membraanmechanica geleid tot de bepaling van de buigstijfheidsmoduli van membranen met verschillende gradaties van complexiteit, van eencomponenten lipidemembranen35,38 tot multicomponent biomimetische membranen41,66,91, die allemaal ons begrip van de dynamische aard van lipidemembranen hebben bevorderd. NSE-buigstijfheidsmetingen van lipidemembranen met verschillende moleculaire eenheden, bijvoorbeeld lipiden van verschillende acylkettinglengten en kettingverzadiging38,72,92,hebben essentiële informatie verstrekt over de rol van moleculaire chemie in membraanmechanica. In combinatie met structurele informatie, zoals membraandikte of moleculaire verpakking93,beginnen deze metingen nieuwe perspectieven te bieden op de onderlinge afhankelijkheid tussen membraanstructuur en dynamiek en hoe ze de membraanfunctie beïnvloeden. De mesoscopische schalen van NSE positioneren het op unieke wijze voor dergelijke fundamentele onderzoeken naar structuur-eigendomsrelaties, het meest relevant op de lengteschaal van moleculaire assemblages. Dit onderwerp werd onlangs onderzocht in twee NSE-studies over cholesterolrijke lipidenmembranen36 en in binaire lipidemembranen met hydrofobe mismatch tussen de twee lipidecomponenten39. Beide studies vonden sterk bewijs dat membraanmechanica schaalt met het gebied per lipide, wat de conclusies bevestigt van een recente MD-simulaties met alle atomen door Doktorova et al.82. Deze bevindingen benadrukken de zelfgeassembleerde aard van lipidemembranen en geven een verenigend beeld van moleculaire verpakking als een belangrijke parameter bij het definiëren van dynamische en functionele eigenschappen van het membraan.
Andere toepassingen van NSE omvatten studies naar de mechanische respons van membranen op kleine additieven, waaronder biologische moleculen zoals cholesterol36,37, trehalose92en melittine73,94, of anorganische additieven zoals nanodeeltjes voor toepassingen van medicijnafgifte76. NSE is ook gebruikt om te begrijpen hoe membraanmechanica reageert op veranderingen in hun omgeving, waaronder temperatuur92, pH74, en de aanwezigheid van verdringing macromoleculen96. Dergelijke studies dragen bij aan een beter begrip van de factoren die van invloed zijn op de verzachting of verstijving van lipidemembranen, onder biologische omstandigheden die verband houden met gezondheid en ziekte, en in gecontroleerde omgevingen voor therapeutische toepassingen. Met name zijn NSE-metingen ook gebruikt om het effect van antimicrobiële peptiden op membraandynamica73,94,95te onderzoeken . Andere voorbeelden van NSE-toepassingen op biomembranen omvatten studies naar de dynamiek van afgeplatte membraanstructuren, thylakoïden genaamd, die de fotosynthetische machines huisvesten in cyanobacteriële cellen97,98.
Men kan ook selectieve lipide deuteratie gebruiken in NSE-studies om de dynamiek van specifieke membraankenmerken te onderzoeken die relevant zijn voor de biologische functie. Nickels et al. gebruikten bijvoorbeeld selectieve lipidedeuteratie in domeinvormende lipidemembranen om lateraal contrast in het membraan te genereren, zoals eerder geïllustreerd door Heberle et al.28. Dit deuteratieschema maakte onafhankelijke metingen mogelijk van de buigstijfheid van de lipidendomeinen en de gastheerlipidematrix41 (zie figuur 2B). De bevindingen bevestigden dat de twee membraancompartimenten een duidelijke buigstijfheidsmoduli hebben, wat een drijfmechanisme zou kunnen zijn voor domeinvorming in cellulaire membranen. In een recentere studie gebruikten Rickeard et al. cyclodextrine-uitwisseling tussen geprotieerde en gedeutereerde liposomen om asymmetrische liposomen te verkrijgen met isotopisch gelabelde bijsluiters40 (Figuur 2C). Hun eindliposomen hadden een geprotieerde bijsluiter en een gedeutereerde bijsluiter die contrasterend is met de buffer, waardoor studies van individuele bijsluiterdynamiek mogelijk werden en een eerste direct experimenteel verslag werd verstrekt van het effect van asymmetrie en bijsluiterkoppeling op membraanbuigschommelingen.
Selectieve membraandeuteratie is ook gebruikt in NSE-studies naar membraandikteschommelingen, een lang voorspelde dynamische modus in lipidemembranen99 die pas onlangs werd waargenomen met de komst van NSE-spectroscopie35,100. Deze metingen maken gebruik van staart-gedeutereerde membranen om het signaal van de membraan hoofdgroep regio's te versterken en het dikte fluctuatie signaal op te lossen. Dit type NSE-experimenten is relatief recent, maar het is effectief gebruikt om de onderlinge afhankelijkheid van membraanelastiek en viskeuze eigenschappen38te begrijpen , om de schaalvergroting van buigstijfheid en viscositeit met moleculaire verpakking in gemengde lipidemembranen te onderzoeken39, en om de lokale effecten van cholesterol op membraanviscositeit36te onderzoeken . Een ander gebied van biologische betekenis waarin deze dynamische modus verstrekkende implicaties zou kunnen hebben, zijn mesoscopische membraan-eiwitinteracties95. Het is bekend dat de functie van membraaneiwitten nauw verbonden is met hydrofobe matching tussen het eiwit en het gastheermembraan. Variaties in membraandikte, als gevolg van dikteschommelingen, kunnen dus fungeren als een regulerend mechanisme voor de functie van membraaneiwitten. NSE is uitermate geschikt voor dergelijke studies omdat het direct de effecten van eiwitbinding en inbrengen op membraandikteschommelingen kan onderzoeken. Recente NSE-metingen van onze groep (niet gepubliceerd) suggereren dat het inbrengen van transmembrane-eiwitten de fluctuaties in de membraandikte aanzienlijk kan onderdrukken en een potentieel mechanisme kan vormen voor het reguleren van signaleringsgebeurtenissen. Dit is een urgent, maar onderontwikkeld onderzoeksgebied waar NSE een aanzienlijke impact kan hebben bij het begrijpen van de dynamische reacties van membranen op eiwitbinding en het inbrengen op de lengte- en tijdschalen van belangrijke biologische functies die worden gegeven door de interacties van eiwitten met celmembranen.
Kortom, NSE is de afgelopen jaren geëvolueerd als een krachtig instrument voor het ondervragen van membraandynamiek over ruimtelijke en temporele schalen van vitale biologische functies. De techniek wint snel aan brede belangstelling en het potentieel ervan in het beantwoorden van belangrijke vragen in de membraanfunctie wordt goed erkend. De contrastvariatiemogelijkheden binnen NSE hebben het gepositioneerd als een unieke benadering om mesoscopische membraaneigenschappen te meten die anders een uitdaging zouden zijn om te verkrijgen. Een ander belangrijk voordeel van NSE ten opzichte van traditionele spectroscopiemethoden in studies van membraandynamica is de overlap met de lengte- en tijdschalen die toegankelijk zijn met MD-simulaties, waardoor synergetische experimentele / berekeningsstudies een moleculair begrip kunnen krijgen van de verschillende moleculaire componenten die membranen vormen. Ondanks zijn belofte zijn er nog steeds enkele beperkingen in het gebruik van NSE in biologische membraanstudies, waaronder de vereiste voor grote monstervolumes, de moeilijkheid om selectieve deuteratie in biologische systemen te deutereren en de relatief lage neutronenflux op NSE-spectrometers, wat resulteert in langere meettijden en beperkte beschikbaarheid van bundeltijd. Deze tekortkomingen kunnen echter in de nabije toekomst worden overwonnen met constante ontwikkelingen in neutronenbronnen en instrumentatie, samen met vooruitgang in deuteratiefaciliteiten.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
De auteurs verklaren geen belangenconflicten en hebben niets openbaar te maken.
Acknowledgments
R. Ashkar bedankt M. Nagao, L.-R. Stingaciu, en P. Zolnierczuk voor vele nuttige besprekingen en voor hun frequente hulp met NSE experimenten op hun respectieve beamlines. De auteurs erkennen het gebruik van neutronenspinechospectrometers bij NIST en ORNL. De NSE spectrometer bij NIST wordt ondersteund door het Center for High Resolution Neutron Scattering, een samenwerkingsverband tussen het National Institute of Standards and Technology en de National Science Foundation in het kader van overeenkomst nr. Dmr-1508249. De NSE spectrometer bij ORNL's Spallation Neutron Source wordt ondersteund door de Scientific User Facilities Division, Office of Basic Energy Sciences, Us Department of Energy. Oak Ridge National Laboratory wordt beheerd door UT-Battelle, LLC onder US DOE Contract No. DE-AC05-00OR22725.
Materials
| Name | Company | Catalog Number | Comments |
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. Ø, Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes--a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich's model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a "modeling manifesto". Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D. Neutrons in Soft Matter. Imae, T., Kanaya, T., Furusaka, M., Torikai, N. , Wiley. ch6 147-182 (2011).
- Pynn, R. Neutron Spin Echo. Mezei, F., Pappas, C., Gutberlet, T. , Springer. Berlin Heidelberg. 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , Oxford University Press. (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- Neutron activation and scattering calculator. , Available from: https://www.ncnr.nist.gov/resources/activation/ (2021).
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , Walter de Gruyter, Inc. 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles - as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O'Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -R., O'Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).
Tags
Biologie Nummer 171 collectieve lipidendynamica buigmodulus gebiedsdrukbaarheid dikteschommelingen membraanviscositeit selectieve deuteratie liposoompreparaatErratum
Formal Correction: Erratum: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions
Posted by JoVE Editors on 08/06/2021.
Citeable Link.
An erratum was issued for: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions. The Introduction, Protocol, and Representative Results sections have been updated.
In the Introduction, the fith pargraph was updated from:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 2 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
to:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 0 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
In the Protocol, step 1.1 was updated from:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 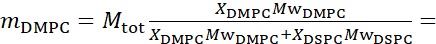 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
to:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 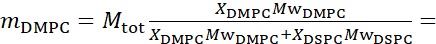 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
In the Representative Results, the fist pagargaph was updted from:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 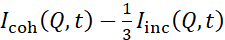 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
to:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 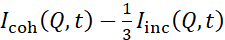 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
In the Representative Reults, Figure 2 was updated from:
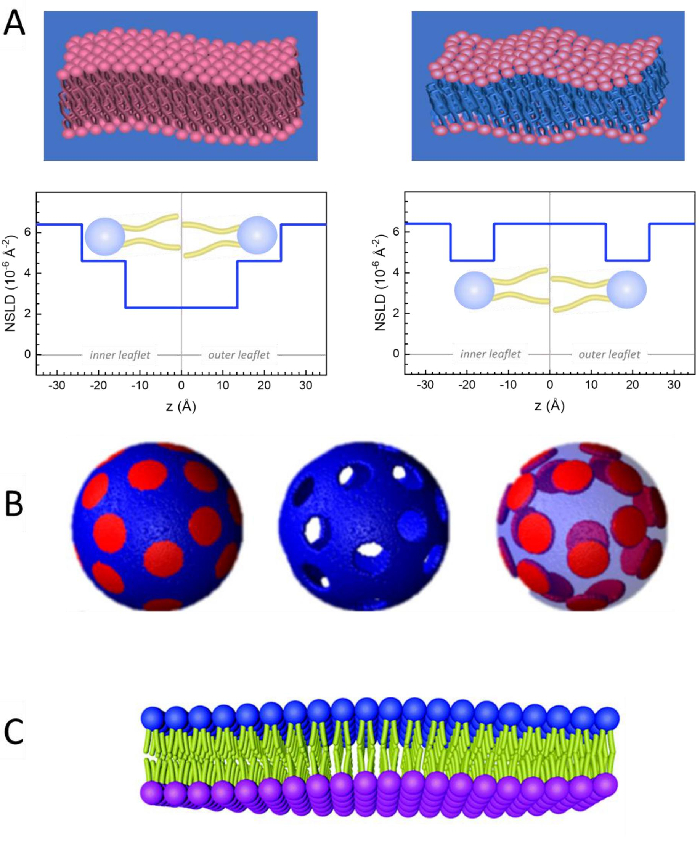
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the headgroup (~2 × 10-2 Å-2) and tail region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
to:
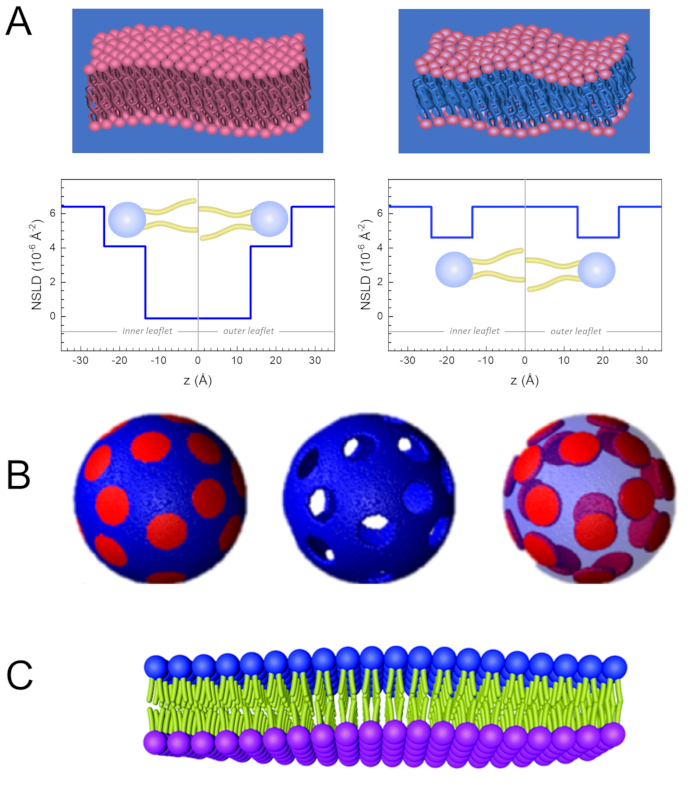
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the tail region (~0 × 10-2 Å-2) and headgroup region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.



 133,4 mg, waarbij XDMPC en XDSPC de molfracties van DMPC en DSPC zijn, hier ingesteld op respectievelijk 0,7 en 0,3, en MwDMPC en MwDSPC zijn de molaire gewichten gegeven door 67.7 en 0.3. Evenzo, mDSPC = 66,6 mg. Dit deuteratieschema verhoogt het strooicontrast tussen het membraan (NSLD ~ 0 × 10-6 Å-2) en de gedeutereerde buffer (NSLD ~ 6,4 × 10-6 Å-2) en versterkt het signaal van membraangolvingen (zie figuur 2A linkerpaneel).
133,4 mg, waarbij XDMPC en XDSPC de molfracties van DMPC en DSPC zijn, hier ingesteld op respectievelijk 0,7 en 0,3, en MwDMPC en MwDSPC zijn de molaire gewichten gegeven door 67.7 en 0.3. Evenzo, mDSPC = 66,6 mg. Dit deuteratieschema verhoogt het strooicontrast tussen het membraan (NSLD ~ 0 × 10-6 Å-2) en de gedeutereerde buffer (NSLD ~ 6,4 × 10-6 Å-2) en versterkt het signaal van membraangolvingen (zie figuur 2A linkerpaneel). Mtot de totale massa is, hier ingesteld op 200 mg. Zie het voorbeeld hierboven voor DMPC-DSPC lipide bilayers met verschillende deuteratieschema's.
Mtot de totale massa is, hier ingesteld op 200 mg. Zie het voorbeeld hierboven voor DMPC-DSPC lipide bilayers met verschillende deuteratieschema's.

 , dus verandert in de spintoestand als gevolg van energie-uitwisseling tussen het neutron en het monster wordt gedetecteerd als een daling van de polarisatie (van eenheid).
, dus verandert in de spintoestand als gevolg van energie-uitwisseling tussen het neutron en het monster wordt gedetecteerd als een daling van de polarisatie (van eenheid).
