ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Questo documento descrive i protocolli per la preparazione dei campioni, la riduzione dei dati e l'analisi dei dati negli studi NSE (Neutron Spin Echo) delle membrane lipidiche. L'etichettatura giudiziosa dei lipidi al deuterio consente l'accesso a diverse dinamiche di membrana su scale di lunghezza e tempo mesoscopiche, su cui si verificano processi biologici vitali.
Abstract
I doppi strati lipidici formano la matrice principale delle membrane cellulari e sono la piattaforma primaria per lo scambio di nutrienti, le interazioni proteina-membrana e il germogliamento virale, tra gli altri processi cellulari vitali. Per un'attività biologica efficiente, le membrane cellulari dovrebbero essere abbastanza rigide da mantenere l'integrità della cellula e dei suoi compartimenti ma abbastanza fluide da consentire ai componenti della membrana, come proteine e domini funzionali, di diffondersi e interagire. Questo delicato equilibrio delle proprietà elastiche e fluide della membrana, e il loro impatto sulla funzione biologica, richiedono una migliore comprensione delle dinamiche collettive della membrana su scale mesoscopiche di lunghezza e tempo dei principali processi biologici, ad esempio deformazioni della membrana ed eventi di legame proteico. Tra le tecniche che possono sondare efficacemente questa gamma dinamica c'è la spettroscopia neutron spin echo (NSE). In combinazione con l'etichettatura al deuterio, nSE può essere utilizzato per accedere direttamente alle fluttuazioni di flessione e spessore, nonché alla dinamica mesoscopica di caratteristiche selezionate della membrana. Questo documento fornisce una breve descrizione della tecnica NSE e delinea le procedure per l'esecuzione di esperimenti NSE su membrane liposomiali, compresi i dettagli della preparazione del campione e degli schemi di deuterazione, insieme alle istruzioni per la raccolta e la riduzione dei dati. Il documento introduce anche metodi di analisi dei dati utilizzati per estrarre i parametri chiave della membrana, come il modulo di rigidità di flessione, il modulo di comprimibilità dell'area e la viscosità nel piano. Per illustrare l'importanza biologica degli studi NSE, vengono discussi esempi selezionati di fenomeni di membrana sondati da NSE, vale a dire, l'effetto degli additivi sulla rigidità di flessione della membrana, l'impatto della formazione del dominio sulle fluttuazioni della membrana e la firma dinamica delle interazioni membrana-proteina.
Introduction
La comprensione delle membrane cellulari e della loro funzione si è notevolmente evoluta negli ultimi decenni. La precedente visione delle membrane cellulari come doppi strati lipidici passivi che definiscono i confini cellulari e le proteine di membrana1 si è gradualmente trasformata in un modello dinamico in cui i doppi strati lipidici svolgono un ruolo importante nella regolazione dei processi biologici vitali, tra cui la segnalazione cellulare, lo scambio molecolare e la funzione proteica - per citarne alcuni2,3,4,5,6. Questa consapevolezza che le membrane cellulari sono altamente dinamiche, costantemente in fase di rimodellamento e ridistribuzione molecolare, ha sollecitato esplorazioni scientifiche oltre le strutture di equilibrio delle membrane7,8,9. Di conseguenza, sono stati sviluppati diversi approcci per studiare le varie modalità dinamiche nelle membrane lipidiche biologiche e bioispira. Ad oggi, la maggior parte di questi studi si è concentrata principalmente sui moti molecolari diffusivi10,11, 12,13 e sulle fluttuazioni macroscopiche della forma14,15,16,lasciando un vuoto significativo nella comprensione della dinamica intermedia della membrana, cioè fluttuazioni collettive di assemblaggi lipidici costituiti da pochi 10-100 di molecole lipidiche. Queste dinamiche si verificano su scale di lunghezza da poche decine a poche 100 Å e su scale temporali di sub-ns a poche centinaia di ns (vedi Figura 1), qui indicate come scale mesoscopiche. È infatti su queste scale che l'attività biologica chiave si svolge a livello di membrana17. Ciò include il germogliamento virale18,il gating del canale19e le interazioni membrana-proteina20. È anche importante sottolineare che il panorama energetico delle proteine di membrana21,22 mostra che i cambiamenti conformazionali nelle proteine - necessari per il loro ruolo regolatore - avvengono sulle scale temporali ns23 delle fluttuazioni collettive della membrana, sottolineando ulteriormente l'importanza della dinamica mesoscopica nella funzione biologica delle membrane cellulari e dei loro analoghi bioispirati20. Questo documento si concentra sulle due principali modalità dinamiche mesoscopiche nelle membrane lipidiche, vale a dire, fluttuazioni di flessione e fluttuazioni di spessore.
La sfida principale nel sondare direttamente queste modalità di fluttuazione è la difficoltà di accedere simultaneamente alle loro scale spaziali e temporali utilizzando metodi di spettroscopia standard. L'altra sfida è che le tecniche di contatto diretto potrebbero avere un impatto sulle stesse fluttuazioni che dovrebbero misurare16. Ciò è ulteriormente esacerbato dalla complessità composizionale e strutturale delle membrane biologiche24,25, che si traduce in caratteristiche di membrana non omogenee, tra cui la formazione del dominio lipidico26,27,28,29,30 e l'asimmetria di membrana31,32,33- che richiedono sonde selettive per comprendere la dinamica delle diverse caratteristiche della membrana. Fortunatamente, queste sfide possono essere superate con metodi di spettroscopia neutronica non invasivi, come l'eco di spin neutronico (NSE), che accedono intrinsecamente alle scale di lunghezza e tempo richieste e consentono ulteriormente studi sulle caratteristiche selettive della membrana senza modificare il loro ambiente fisico-chimico34. Infatti, negli ultimi anni la spettroscopia NSE si è evoluta in una sonda unica e potente di dinamica collettiva della membrana35. I risultati degli studi NSE sulle membrane lipidiche hanno prodotto nuove intuizioni sulle proprietà meccaniche36,37 e viscoelastiche38,39 delle membrane lipidiche e hanno gettato nuova luce sul loro potenziale ruolo nella funzione biologica40,41.
La tecnica di spettroscopia NSE si basa su un progetto di strumento interferometrico, proposto per la prima volta da Mezei42, utilizzando una serie di pinne di spin e bobine magnetiche per controllare la precessione dello spin neutronico mentre i neutroni attraversano lo strumento. Il design si basa sulla specchiatura magnetica degli elementi del campo magnetico rispetto alla posizione del campione (Figura 1A). Ciò implica che in assenza di scambio di energia tra il neutrone e il campione, il neutrone esegue lo stesso numero di precessione di spin, in direzioni opposte, nella prima e nella seconda metà dello strumento (si noti la π-flipper tra le due bobine di precessione). Di conseguenza, lo stato di spin finale del neutrone rimane invariato rispetto allo stato iniziale - un fenomeno denominato spin-eco (vedi neutrone trasparente in Figura 1A). Tuttavia, quando il neutrone interagisce energeticamente con il campione, lo scambio di energia modifica il numero di precessione di spin nella seconda metà dello strumento, portando ad un diverso stato di spin finale (vedi Figura 1A). Questo è sperimentalmente rilevato come una perdita di polarizzazione, come verrà mostrato più avanti in questo articolo. Per maggiori dettagli sulla tecnica NSE, si rimanda al lettore gli articoli tecnici dedicati42,43,44,45.
Qui viene presentata una descrizione semplificata per fornire una stima approssimativa della lunghezza e delle scale temporali accessibili con NSE. Le scale di lunghezza sono determinate dall'intervallo di trasferimenti di motovettori realizzabili, Q = 4π sin θ/λ, dove 2θ è l'angolo di diffusione e λ è la lunghezza d'onda del neutrone. Si può vedere che Q è impostato dall'intervallo di lunghezze d'onda e dall'estensione della rotazione del secondo braccio dello spettrometro (vedi Figura 1A). Un tipico intervallo Qsugli spettrometri NSE è ~ 0,02-2 Å-146,47e fino a 0,01-4 Å-1 con recenti aggiornamenti48,49, corrispondenti a scale spaziali di ~ 1-600 Å. D'altra parte, la scala temporale accessibile è calcolata dall'angolo di precessione totale (o fase) acquisito dal neutrone all'interno delle bobine di precessione magnetica, e si trova ad essere50:  . In questa espressione, t è il tempo di Fourier definito come
. In questa espressione, t è il tempo di Fourier definito come  , dove è il rapporto
, dove è il rapporto  giromagnetico neutronico,
giromagnetico neutronico,  è la lunghezza della bobina e è la forza del campo
è la lunghezza della bobina e è la forza del campo  magnetico della bobina. Vale la pena sottolineare che il tempo di Fourier è una quantità che dipende strettamente dalla geometria dello strumento, dall'intensità del campo magnetico e dalla lunghezza d'onda dei neutroni. Ad esempio, usando neutroni di lunghezza d'onda
magnetico della bobina. Vale la pena sottolineare che il tempo di Fourier è una quantità che dipende strettamente dalla geometria dello strumento, dall'intensità del campo magnetico e dalla lunghezza d'onda dei neutroni. Ad esempio, usando neutroni di lunghezza d'onda  = 8 Å e impostazioni dello strumento di
= 8 Å e impostazioni dello strumento di  = 1,2 m e
= 1,2 m e  = 0,4 T, il tempo di Fourier è calcolato per essere t ~ 50 ns. Sperimentalmente, il tempo di Fourier viene regolato modificando la corrente nelle bobine di precessione (cioè l'intensità del campo magnetico) o utilizzando diverse lunghezze d'onda dei neutroni, con conseguente scala temporale tipica di NSE da ~ 1 ps a 100 ns. Tuttavia, i recenti aggiornamenti degli spettrometri NSE hanno consentito l'accesso a tempi di Fourier più lunghi, fino a ~ 400 ns sullo spettrometro J-NSE-Phoenix presso lo spettrometro Heinz Maier-Leibnitz Zentrum51 e lo spettrometro SNS-NSE presso l'Oak Ridge National Lab48e fino a ~ 1.000 ns presso lo spettrometro NSE IN15 presso l'Institut Laue-Langevin (ILL)49.
= 0,4 T, il tempo di Fourier è calcolato per essere t ~ 50 ns. Sperimentalmente, il tempo di Fourier viene regolato modificando la corrente nelle bobine di precessione (cioè l'intensità del campo magnetico) o utilizzando diverse lunghezze d'onda dei neutroni, con conseguente scala temporale tipica di NSE da ~ 1 ps a 100 ns. Tuttavia, i recenti aggiornamenti degli spettrometri NSE hanno consentito l'accesso a tempi di Fourier più lunghi, fino a ~ 400 ns sullo spettrometro J-NSE-Phoenix presso lo spettrometro Heinz Maier-Leibnitz Zentrum51 e lo spettrometro SNS-NSE presso l'Oak Ridge National Lab48e fino a ~ 1.000 ns presso lo spettrometro NSE IN15 presso l'Institut Laue-Langevin (ILL)49.
Oltre all'accesso diretto alla scala di lunghezza e tempo della dinamica di membrana, NSE ha le capacità intrinseche della sensibilità isotopica dei neutroni52. In particolare, la capacità dei neutroni di interagire in modo diverso con gli isotopi dell'idrogeno, l'elemento più abbondante nei sistemi biologici, si traduce in una diversa densità di lunghezza di scattering neutronico,34 o NSLD (l'equivalente dell'indice ottico di rifrazione50), quando il prozio è sostituito dal deuterio. Ciò consente un approccio noto come variazione di contrasto, che viene comunemente utilizzato per evidenziare specifiche caratteristiche della membrana o nascondene altre - quest'ultimo scenario è indicato come corrispondenza di contrasto. Un'applicazione frequente di variazione/corrispondenza del contrasto è la sostituzione dell'acqua (NSLD = -0,56 ×10 -6 Å -2) con acqua pesante o D2O (NSLD = 6,4 × 10-6 Å-2) per amplificare il segnale neutronico dalle membrane lipidiche protiate (NSLD ~ 0 × 10-6 Å-2). Questo approccio è altamente efficace negli studi sulla struttura della membrana perché la penetrazione di D2O nella regione del gruppo di testa della membrana consente una determinazione accurata degli spessori della membrana (vedi Figura 2A,pannello di sinistra) e della posizione di diversi sottogruppi lipidici quando vengono applicati modelli più sofisticati53,54. Questo documento evidenzia alcuni esempi sull'uso della variazione di contrasto per studi di dinamica collettiva in membrane biomimetiche e caratteristiche selezionate della membrana.
Qui, l'efficacia di NSE nel fornire approfondimenti unici sulle proprietà dinamiche e funzionali della membrana è illustrata attraverso esempi tangibili di studi NSE su sistemi di membrana lipidica modello e biologicamente rilevanti con particolare attenzione alla dinamica della mesoscala nelle membrane autoportanti, sotto forma di sospensioni liposomiali. Per le misure NSE della dinamica della membrana in-plane, il lettore è rimandato a pubblicazioni dedicate sulla spettroscopia spin-eco neutronica ad incidenza radente (GINSES)55,56 e altri studi su pile di membrane multilamellari allineate57,58,59,60.
Per semplicità, questo articolo evidenzia tre diversi schemi di deuterazione della membrana illustrati su un sistema a doppio strato lipidico ben studiato di formazione di dominio, o separazione di fase, di miscele di 1,2-dimiristoil-sn-glicero-3-fosfocolina (DMPC) e 1,2-distearoil-sn-glicero-3-fosfocolina (DSPC)61,62. I due lipidi sono caratterizzati da una discrepanza nella lunghezza della catena idrocarburica (14 atomi di carbonio/coda in DMPC vs 18 atomi di carbonio/coda in DSPC) e dalla loro temperatura di transizione gel-fluido (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C). Ciò si traduce in una separazione di fase laterale nelle membrane DMPC:DSPC a temperature comprese tra le temperature di transizione superiore e inferiore della miscela63. Gli schemi di deuterazione qui considerati sono scelti per dimostrare le diverse modalità dinamiche accessibili nelle misurazioni NSE su membrane liposomiali, vale a dire, fluttuazioni di flessione, fluttuazioni di spessore e fluttuazioni selettive di flessione / spessore di domini laterali. Tutte le composizioni lipidiche sono riportate per i doppi strati DMPC:DSPC preparati a una frazione molare di 70:30, utilizzando varianti proziate e perdeuterate disponibili in commercio di DMPC e DSPC. Tutte le fasi di preparazione del campione si basano su 4 mL di sospensione liposomiale, in D2O, con una concentrazione lipidica di 50 mg/mL, per una massa lipidica totale di Mtot = 200 mg per campione.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Schema di deuterazione richiesto per l'esperimento
- Per le misurazioni delle fluttuazioni di flessione, effettuare liposomi completamente proziati in D2O (D 99,9%) o D2O-buffer (ad esempio, tampone fosfato preparato con D2O invece di H2O). Utilizzare DMPC completamente proziato (C36H72NO8P) e DSPC (C44H88NO8P) con
 133,4 mg, dove XDMPC e XDSPC sono le frazioni molari di DMPC e DSPC, qui impostate rispettivamente a 0,7 e 0,3, e MwDMPC e MwDSPC sono i pesi molari dati rispettivamente da 677,9 g / mol e 790,1 g / mol. Allo stesso modo, mDSPC = 66,6 mg. Questo schema di deuterazione aumenta il contrasto di scattering tra la membrana (NSLD ~ 0 × 10-6 Å-2) e il tampone deuterato (NSLD ~ 6,4 × 10-6 Å -2) e amplifica il segnale dalle indulazioni di membrana (vedi Figura 2A pannello di sinistra).
133,4 mg, dove XDMPC e XDSPC sono le frazioni molari di DMPC e DSPC, qui impostate rispettivamente a 0,7 e 0,3, e MwDMPC e MwDSPC sono i pesi molari dati rispettivamente da 677,9 g / mol e 790,1 g / mol. Allo stesso modo, mDSPC = 66,6 mg. Questo schema di deuterazione aumenta il contrasto di scattering tra la membrana (NSLD ~ 0 × 10-6 Å-2) e il tampone deuterato (NSLD ~ 6,4 × 10-6 Å -2) e amplifica il segnale dalle indulazioni di membrana (vedi Figura 2A pannello di sinistra). - Per misurare la dinamica di flessione di caratteristiche selezionate della membrana laterale, ad esempio la dinamica della matrice nelle membrane DMPC:DSPC a separazione di fase, utilizzare DMPC proziato (C36H72NO8P) e deuterato, DSPC-d83 (C44H5NO8PD83, Mw 873,7 g/mol), in modo tale che mDMPC = 128,8 mg e mDSPC-d83 = 71,2 mg. Questo schema di deuterazione riduce al minimo la dispersione dai domini indesiderati ricchi di DSPC, consentendo misurazioni selettive delle fluttuazioni di flessione dalla matrice ricca di DMPC (vedere la Figura 2B centrale).
NOTA: Per trovare la deuterazione lipidica ottimale necessaria per uno specifico schema di corrispondenza del contrasto, utilizzare i calcolatori di densità di lunghezza di dispersione (SLD) basati sul web disponibili, come quello sviluppato dal NIST Center for Neutron Research64. Queste interfacce basate sul web sono dotate di strumenti user-friendly per un facile calcolo del DSA dei lipidi con vari gradi di deuterazione, così come quello delle miscele lipidiche. - Per le misurazioni NSE delle fluttuazioni medie dello spessore della membrana (senza contrasto laterale), utilizzare varianti deuterate a coda dei lipidi costituenti, cioè DMPC-d54 (C36H18NO8PD54, 732,3 g / mol) e DSPC-d70 (C44H18NO8PD70, 860,1 g / mol)35,38, tale chemPC-d54 = 133,0 mg e m DSPC-d70 = 67,0 mg. Questo schema di contrasto(Figura 2A,pannello di destra) amplifica il segnale di scattering dai gruppi di testa lipidica (NSLD ~ 4,5 × 10-6 Å-2) abbinando in contrasto il gruppo di coda (NSLD ~ 6,4 × 10-6 Å -2) al tampone deuterato che consente il rilevamento delle fluttuazioni dello spessore della membrana.
- Per gli studi di fluttuazione dello spessore di compartimenti di membrana selezionati, ad esempio la matrice ricca di DMPC, utilizzare la stessa strategia descritta nel passaggio 1.2 sostituendo i lipidi DMPC proziati con i loro analoghi deuterati della coda, cioè DMPC-d54, in modo tale che i domini ricchi di DSPC siano abbinati al tampone deuterato e il segnale di scattering primario provenga dalla regione del gruppo di testa della matrice ricca di DMPC deuterata dalla coda.
2. Preparazione della sospensione lipidica per l'estrusione
- Calcolare la massa di ciascun componente nel campione, a seconda della composizione del campione. Come regola generale, per i campioni con più componenti molecolari, la massa di un componente è data dalla sua massa molare, Mwi, ponderata dalla sua frazione molare, Xi, e normalizzata su tutti i componenti in modo tale che: dove
 Mtot è la massa totale, impostata qui a 200 mg. Vedi l'esempio sopra per i doppi strati lipidici DMPC-DSPC con diversi schemi di deuterazione.
Mtot è la massa totale, impostata qui a 200 mg. Vedi l'esempio sopra per i doppi strati lipidici DMPC-DSPC con diversi schemi di deuterazione. - Utilizzando una semi-microbilanza digitale, pesare le masse calcolate di lipidi (e altri costituenti del campione, ad esempio proteine, nanoparticelle, ecc.) e aggiungerle a una fiala o a un pallone a fondo tondo - ricordarsi di pesare la fiala o il pallone in anticipo. Aggiungere 1 mL di solvente per sciogliere i componenti pesati mescolando manualmente all'interno di una cappa. Per i campioni di lipidi puri, utilizzare cloroformio o etanolo. Per i campioni con componenti aggiuntivi non lipidici (ad esempio, nanoparticelle), scegliere un solvente comune che disperda tutti i componenti.
- Per piccole quantità lipidiche (<10 mg), preparare una soluzione stock e pipettare il volume richiesto nella miscela.
NOTA: non aggiungere quantità eccessive di solvente in quanto rallenterà significativamente la fase di asciugatura del solvente descritta di seguito.
- Per piccole quantità lipidiche (<10 mg), preparare una soluzione stock e pipettare il volume richiesto nella miscela.
- Asciugare la soluzione lipidica, all'interno di un cappuccio, facendo scorrere delicatamente un gas inerte (ad esempio, azoto, argon) nel flaconcino mentre ruota lentamente il flaconcino ad angolo. Tenere i flaconcini in posizione inclinata per creare un sottile film di lipidi secchi sulle pareti del flaconcino, che consentirà un'asciugatura uniforme. Posizionare il flaconcino a intermittenza a bagnomaria a 35 °C per evitare il raffreddamento mediato dall'evaporazione, che rallenterà l'evaporazione del solvente.
- Posizionare i flaconcini durante la notte in un forno sottovuoto a ~35 °C per rimuovere completamente il solvente residuo. Per i lipidi insaturi, eliminare il vuoto con un gas inerte per ridurre al minimo l'ossidazione.
- Per garantire la completa rimozione del solvente, pesare il flaconcino dopo l'essiccazione dei lipidi e confermare che non vi sia massa in eccesso oltre le quantità misurate di materiali. Farlo sottraendo la massa del flaconcino dalla massa misurata dopo l'essiccazione. Se c'è massa in eccesso, asciugare il campione sotto vuoto per altre 6 ore. Ripetere questo processo in base alle esigenze.
- Idratare il film lipidico con 4 mL di D2O o D2O-tampone per ottenere una concentrazione lipidica di 50 mg/mL. Per i lipidi con alte temperature di transizione, come le miscele DMPC-DSPC, riscaldare il tampone al di sopra della temperatura di transizione (60 °C) per garantire una miscelazione uniforme.
NOTA: Poiché gli esperimenti NSE richiedono volumi di campione relativamente grandi (~4 mL), prendere in considerazione l'idratazione del campione utilizzando metà del buffer richiesto, cioè 2 mL, per ridurre al minimo il numero di estrusi per campione (vedere paragrafo 3). In questo caso, aggiungere la metà rimanente del buffer dopo l'estrusione. Si noti che la capacità delle siringhe utilizzate nell'estrusione è limitata a 1 mL. Pertanto, l'idratazione con 4 ml di tampone richiederebbe quattro serie di estrusione. - Mescolare a vortice la soluzione lipidica idratata fino a quando il film lipidico non è completamente disciolto e non è più visibile sulle pareti del flaconcino. In questa fase, i lipidi idratati formano vescicole multilamellari e pile multilamellari di dimensioni micron e la sospensione appare bianco latte.
- Per facilitare la rottura delle pile lipidiche e ridurre la multilamellarità, eseguire cinque cicli di congelamento/scongelamento posizionando il flaconcino di soluzione lipidica idratata in un congelatore di livello di laboratorio (preferibilmente un congelatore a -80 °C) fino a completo congelamento e quindi trasferendo il flaconcino a un bagno d'acqua a 35 °C fino a quando la soluzione lipidica non è completamente scongelata. Vortice la soluzione scongelata fino a quando omogenea. Ripeti altre quattro volte.
NOTA: In alternativa, un bagno di ghiaccio secco può essere preparato per il congelamento rapido combinando acetone e ghiaccio secco.
3. Estrusione della soluzione lipidica idratata
- Assemblare la configurazione dell'estrusore utilizzando una membrana in policarbonato tra due supporti a membrana e aggiungendo due filtri di carta su ciascun lato per fornire ulteriore supporto. Utilizzare una membrana in policarbonato con una dimensione dei pori che corrisponda alla dimensione liposomiale target (le dimensioni dei pori comuni per gli esperimenti NSE sono 50 nm e 100 nm - in genere, i liposomi di 100 nm di diametro consentono fluttuazioni di membrana meno vincolate, ma i liposomi più piccoli da 50 nm potrebbero essere utilizzati per studi di curvatura). Assicurarsi che la membrana in policarbonato sia completamente tesa prima di completare l'assemblaggio e serrare l'involucro esterno dell'estrusore.
- Idratare la membrana in policarbonato facendo passare ~ 0,3 mL di D2O o D2O-buffer alcune volte attraverso il gruppo membrana utilizzando siringhe di vetro eermetiche. Utilizzare lo stesso buffer utilizzato nella preparazione del campione. Lasciarlo per almeno 10 minuti, quindi aspirare completamente il buffer prima di introdurre il campione.
- Riempire una siringa a tenuta di gas da 1 mL con la soluzione lipidica preparata e inserirla in un'estremità dell'apparecchio di estrusore. Quindi, inserire una siringa vuota all'estremità opposta. Una volta che le siringhe sono collegate al gruppo estrusore, posizionarle nel blocco dell'estrusore.
- Se sono necessarie temperature elevate per l'estrusione, come nel caso di lipidi saturi con alte temperature di transizione (ad esempio, DSPC, Tm = 55 °C), preriscaldare il blocco riscaldante dell'estrusore al di sopra della temperatura di transizione lipidica (ad esempio, 60 °C), posizionando il blocco riscaldante su una piastra calda o utilizzando un bagno di circolazione come mostrato nella Figura 3A.
NOTA: Questo passaggio è fondamentale per garantire una miscelazione omogenea dei lipidi e per evitare di esercitare una pressione estrema durante l'estrusione, che potrebbe rompere la membrana in policarbonato. Per campioni lipidici con basse temperature di transizione (<25 °C), eseguire l'estrusione a temperatura ambiente. - Per estrudere la soluzione lipidica, collegare il set di estrusori a una pompa a siringa programmabile con un telaio in alluminio/acciaio, come mostrato nella Figura 3A. Per gli estrusi a temperatura controllata, aggiungere una base di estrusore personalizzata con un canale del fluido e collegarla a un bagno d'acqua circolante.
- Programmare la pompa a siringa per eseguire 15-20 cicli di estrusione seguendo il manuale del produttore. Quando estruso, il colore della soluzione lipidica cambia da bianco latte a blu opale trasparente (Figura 3B,C), indicando una dimensione liposomiale finale che è più piccola della lunghezza d'onda della luce visibile, come previsto. Per il tipo di pompa per siringhe mostrato nella Figura 3A,attenersi alla seguente procedura.
- Inizia regolando le impostazioni della pompa. Tenere premuto il pulsante Velocità e immettere la velocità di estrusione (50,99 ml/h), quindi premere il pulsante Diametro e immettere il diametro della siringa (4,606 mm). Utilizzare le frecce su sotto ogni cifra sullo schermo per modificare il valore della cifra.
- Posizionare il set di estrusori con la siringa campione a destra (vedere figura 3A). Premere il pulsante Preleva finché la spia di prelievo non si accende. Premere Start e attendere che il campione si diserti nella siringa sinistra (vuota).
- Premere il pulsante Stop poco prima che la siringa campione (a destra) sia completamente vuota. Registrare il volume erogato e utilizzarlo per programmare il ciclo di estrusione. Tenere premuto il pulsante Rate fino a quando sullo schermo non viene visualizzata la fase 1 (PH:01). Premere il pulsante Volume per immettere il volume erogato registrato in precedenza. In questa fase, assicurarsi che la luce di prelievo sia spenta - questo eroga il campione nella giusta direzione.
- Premere nuovamente il pulsante Rate e utilizzare la freccia su più a destra per accedere alla fase 2 (PH:02). Premere Volume per immettere lo stesso valore del volume erogato registrato in precedenza. In questa fase, premere il pulsante Prelievo fino a quando la spia di prelievo non è accesa, che eroga il campione a sinistra.
- Per ripetere questo ciclo, premere nuovamente il pulsante Rate e utilizzare la freccia verso l'alto più a destra per accedere alla fase 3 (PH:03). Premere il pulsante Volume finché LP:SE non viene visualizzato sullo schermo e impostarlo su 20. Questo è il numero di loop o ripetizioni che la pompa eseguirà. Infine, premi il pulsante Rate, accedi alla fase 4 (PH: 04) e premi il pulsante Volume per accedere alla funzione Stop. La pompa è ora configurata per l'estrusione automatica.
- Premere Start per avviare il ciclo di estrusione.
- Svuotare la siringa contenente la sospensione lipidica estrusa in un flaconcino pulito e prepararla per la conservazione o le misurazioni. Per i campioni lipidici con temperatura di fusione elevata, conservare il campione al di sopra della transizione di fase fluida fino a quando non viene misurato. Altrimenti, conservare i campioni a temperatura ambiente.
- Non congelare i campioni estrusi poiché il congelamento causerà lo scoppio delle vescicole (la sospensione diventerà di nuovo bianco latte).
4. Misurazioni NSE per il campione o i campioni e riduzione dei dati raccolti
- Prima dell'esperimento NSE, caratterizzare il campione liposomiale estruso dal passo 3.7 utilizzando i metodi disponibili per garantire un'adeguata qualità del campione. Un elenco di potenziali metodi di charcaterizzazione che possono essere utilizzati per valutare la qualità delle sospensioni liposomiali per esperimenti NSE, ad esempio distribuzione dimensionale, multilamellarità, struttura della membrana laterale, è incluso nella sezione di discussione.
- Determinare l'intervallo Q e le impostazioni dello strumento corrispondenti necessarie per l'esperimento. Per le misure di rigidità di flessione dei doppi strati lipidici, utilizzare un intervallo Q di ~(0,04 - 0,2) Å-1. Per gli studi sulle fluttuazioni dello spessore della membrana, utilizzare un intervallo Q di ~(0,04 - 0,2) Å-1 corrispondente allo spessore della membrana35,66,67.
NOTA: Discutere la configurazione sperimentale con lo scienziato dello strumento prima dell'inizio dell'esperimento. Come accennato in precedenza, la caratterizzazione SANS del campione è necessaria, soprattutto se non sono disponibili informazioni preliminari del segnale di scattering, come nelle membrane deuterate selettivamente. In alternativa, eseguire misurazioni statiche (note anche come diffrazione) su un intervallo Q limitato sullo strumento NSE, con l'avvertenza che tali misurazioni richiedono molto più tempo rispetto al SANS. - Utilizzando una siringa o una pipetta di trasferimento, caricare la sospensione liposomiale estrusa o le sospensioni liposomiali estruse nelle celle campione designate disponibili presso le linee di fascio NSE. Si noti che le celle campione NSE standard sono disponibili in spessori di 1, 2, 3 e 4 mm. Scegli lo spessore della cella in modo tale da ottimizzare il segnale di scattering mantenendo il segnale di fondo incoerente a un'intensità ragionevole.
NOTA: come regola generale, utilizzare celle campione con una lunghezza di percorso di 1 o 2 mm per i liposomi proziati nel tampone deuterato - cellule più spesse potrebbero causare effetti di scattering multipli difficili da correggere. Per i liposomi con livelli più elevati di deuterazioni (ad esempio, liposomi a contrasto di coda o liposomi asimmetrici con foglietti singoli proziati), prendere in considerazione l'utilizzo di una cellula campione più spessa (ad esempio, 3 o 4 mm di lunghezza del percorso) per migliorare le statistiche di conteggio se il campione è disponibile in quantità maggiori - a volte questo può essere proibitivo in termini di costi. - Preparare una cella campione identica per il buffer. Utilizzare lo stesso tampone della sospensione liposomiale. Le misurazioni sul buffer sono necessarie per la normalizzazione dell'intensità e le correzioni di fondo (BKG).
- Posizionare le celle campione nel portacampioni dello spettrometro NSE, programmare le esecuzioni di misurazione e raccogliere i dati dell'eco. Consultare lo scienziato dello strumento sulla programmazione delle misurazioni se un utente NSE per la prima volta.
- Eseguire due serie aggiuntive di misurazioni necessarie per la riduzione dei dati: misurazioni di risoluzione (R) e trasmissione (T).
- Eseguire la misurazione della risoluzione(R)su un riferimento di dispersione elastico (ad esempio, carbonio) — da eseguire con le stesse impostazioni; vale a.d. stessovettore d'onda e tempi di Fourier delle misurazioni del campione e del tampone.
- Eseguire misurazioni di trasmissione (T) sul campione e sul tampone per calcolare l'intensità del fascio di neutroni trasmesso (vedere il punto 4.9. di seguito). La trasmissione è calcolata come il rapporto tra i conteggi dei neutroni dal campione o dal buffer diviso per i conteggi dei neutroni per un fascio aperto (cioè con una posizione di campione vuota).
- Utilizzare il software di riduzione dei dati dedicato per lo spettrometro NSE su cui vengono eseguite le misurazioni per ridurre i dati raccolti.
NOTA: spettrometri diversi potrebbero utilizzare software o interfacce utente diversi. Di seguito è riportato un esempio di riduzione dei dati NSE utilizzando l'ambiente di analisi e visualizzazione dei dati (DAVE)68 software scritto appositamente per lo spettrometro NSE presso il NIST Center for Neutron Research.- Aprire il software DAVE e selezionare Riduci dati NSE dal menu di riduzione dei dati. Appariranno diverse finestre pop-up.
- Caricare i file di dati su valori Q diversi utilizzando l'opzione Apri file .echo dal menu file. Questi file corrispondono ai file di dati grezzi con i segnali di eco di spin e hanno l'estensione .echo nel nome del file. Una volta completato il caricamento del file, i file verranno visualizzati sotto i set di dati disponibili.
- Fare clic con il pulsante destro del mouse sul file selezionato ed etichettarlo in base alla misurazione a cui corrisponde; ad esempio, Campione, Cella (per cella vuota o buffer) o Risoluzione.
- Raggruppare i pixel del rilevatore in 2 x 2 per migliorare il rapporto segnale-rumore utilizzando la scheda Set di dati. Applicare lo stesso binning a tutti i file; cioè, Risoluzione, Cella e Campione.
- Ispeziona i dati su tutti i gruppi di pixel e maschera quelli con segnali scarsi (vedi Figura 4B) premendo il tasto m sulla tastiera. Premere Invio per accedere a una finestra pop-up per applicare la stessa maschera a tutte le volte di Fourier o alle successive volte di Fourier. Questo può anche essere applicato a singoli pixel in qualsiasi momento durante la riduzione dei dati. I pixel mascherati diventano verdi.
- Assicurarsi che i dati raccolti siano sotto forma di un segnale eco, cioè la funzione coseno in termini di corrente di fase, su ciascun pixel del rivelatore (vedere Figura 4A).
NOTA: La corrente di fase è proporzionale all'angolo di precessione dello spin neutronico; quindi, è comune rappresentare la corrente di fase come un angolo di fase come mostrato in Figura 4A. Per le misurazioni su sorgenti pulsata, ai dati vengono applicati calcoli aggiuntivi del tempo di volo per ottenere i segnali di eco in funzione della lunghezza d'onda del neutrone incidente all'interno di un impulso neutronico. - Iniziare adattando il file di risoluzione. Selezionare un file di risoluzione dall'elenco dei file caricati e fare clic con il pulsante destro del mouse sul file. Seleziona Adatta operazioni: Adatta echi (risoluzione) dal menu a comparsa.
- Assicurarsi che gli adattamenti dei segnali eco producano una serie di parametri di adattamento, incluso il parametro A, richiesto nel passaggio 4.8. Gli adattamenti vengono eseguiti automaticamente utilizzando la seguente espressione.

Qui, ζ è il periodo del segnale di eco (cioè la funzione coseno in Figura 4A), σ è la larghezza dell'involucro gaussiano determinato dalla lunghezza d'onda media e dalla diffusione della lunghezza d'onda del fascio di neutroni incidente, Φc è la corrente di fase e Φ0 è il punto di eco che dipende dal percorso di campo sperimentato dai neutroni50. Le informazioni fisiche sul campione sono codificate nell'ampiezza, A, della funzione coseno nell'equazione (1).
NOTA: La larghezza dell'inviluppo gaussiano si basa su valori predeterminati dallo scienziato dello strumento e non deve essere modificata. Gli altri parametri sono variabili che vengono adattate al segnale eco specifico su ciascun pixel. - Esaminate i risultati dell'adattamento facendo clic su ciascun pixel per visualizzare i parametri di adattamento risultanti, la qualità dell'adattamento e la deviazione quadrata media dell'adattamento. Per ispezionare l'errore associato a ciascun parametro di raccordo sull'intero rilevatore, selezionare Opzioni immagine e quindi selezionare il parametro di raccordo di interesse. Questo genererà una mappa con il valore del paramter di raccordo su ciascun pixel. Fare clic con il pulsante destro del mouse sull'immagine del rilevatore. Verrà visualizzata una finestra pop-up che mostra una mappa della barra di errore del parametro di raccordo selezionato.
- Se l'adattamento su un pixel specifico è insoddisfacente (ad esempio, adatta i parameeter con barre di errore di grandi dimensioni), riadatta il segnale su quel pixel specifico. Selezionare tale pixel, premere la scheda Raccordo e quindi premere Adatta pixel. Inserire nuovi parametri di partenza per la fase (Φ0) e il periodo (ζ) nella linguetta Raccordo per ottenere una vestibilità più soddisfacente.
NOTA: è utile tracciare la fase adattata in funzione del Tempo di Fourier. A tale scopo, passare alla finestra principale del plottaggio e selezionare Adatta fase v. Tempo di Fourier. Questa trama dovrebbe essere liscia e continua. Esaminate le discontinuità in questo plottaggio e rimontate i pixel a cui corrispondono.
- Ridurre il file di esempio o di cella selezionando il file corrispondente dall'elenco dei file caricati ed etichettati.
- Ispezionare tutti i pixel e mascherare quelli con statistiche errate come descritto nel passaggio 4.7.5.
- Fare clic con il pulsante destro del mouse sul file e selezionare Adatta operazioni: importa fasi (campione, cella). In questo modo vengono importate le fasi e la maschera applicata dal file di risoluzione.
- Adattare i segnali eco utilizzando la stessa procedura descritta in precedenza per il file di risoluzione (passaggi 4.7.8-4.7.10). Nel montaggio dei file di esempio e di cella, non modificare i valori del punto e del punto di fase dell'eco importati dagli adattamenti di risoluzione. Questi parametri dipendono dalle impostazioni strumentali e non dovrebbero variare con i campioni.
- Prima di procedere alla riduzione dei dati, inserire il beam center per tutti i file di dati. Selezionare il file di dati, andare alla scheda Generale e immettere i valori del centro del fascio X e Y. Questi valori vengono registrati durante l'esperimento.
- Una volta completati gli adattamenti ai file sample, cell e risoluzione, calcolare la funzione di scattering intermedio normalizzata da utilizzare in seguito nell'analisi e nell'interpretazione dei dati. Per fare ciò, fai clic con il pulsante destro del mouse sul file di esempio da ridurre dall'elenco dei file adattati e seleziona Calcola I (Q) dal menu a comparsa. Verrà visualizzata una finestra con le opzioni di immissione per i file di risoluzione e cella (ad esempio, buffer) e il numero di archi Q (vedere il passaggio 4.9). Dopo aver inserito tutte le informazioni richieste, premere il pulsante OK. I risultati appariranno in una nuova finestra.
NOTA: La riduzione dei dati viene eseguita secondo la seguente equazione per ottenere la funzione di scattering intermedia normalizzata69.
dove t è il tempo di Fourier, Nsu e Ngiù sono i conteggi dei neutroni nelle configurazioni non-spin-flip e spin-flip (misurati con le π/2-flipper spente e le π-flipper off e on, rispettivamente), e gli apici, BKG e R, corrispondono rispettivamente alle misure di sfondo e di risoluzione, come definito nei passaggi 4.4 e 4.6. Si noti che la polarizzazione del fascio , quindi i cambiamenti nello stato di spin a causa dello scambio di energia tra il neutrone e il campione viene rilevata come una goccia nella polarizzazione (dall'unità).
, quindi i cambiamenti nello stato di spin a causa dello scambio di energia tra il neutrone e il campione viene rilevata come una goccia nella polarizzazione (dall'unità).
- Infine, raggruppare i pixel del rivelatore in archi Q-come mostrato nella Figura 4B per ottenere la dipendenza Qdella funzione di scattering intermedia normalizzata, S(Q,t) / S(Q,0). Questo è tecnicamente indicato come data binning e dovrebbe essere fatto con giudizio, cioè tenendo conto delle statistiche di conteggio dal campione e della deviazione standard prevista dei dati sui pixel raggruppati.
- Per campioni fortemente scattering, dividere il rivelatore in più archi Q mantenendo barre di errore ragionevoli sulla funzione di scattering intermedio risultante, S(Q,t) / S(Q,0). Ciò produce più punti dati Q ed è importante per la procedura di analisi dei dati descritta di seguito. Tieni presente che per i campioni a dispersione debole, un binning eccessivo si traduce in scarsi segnali di decadimento, cioè grandi barre di errore su S(Q,t)/ S(Q,0),che potrebbero causare grandi incertezze.
5. Analisi e interpretazione dei dati
- Adatta le funzioni di scattering intermedie normalizzate, S(Q,t) / S(Q,0), ottenute dalla riduzione dei dati sopra a una funzione esponenziale allungata con un esponente di stretching di 2/370.

NOTA: un esempio di questi adattamenti è fornito nella Figura 5B. Gli adattamenti di S(Q,t) / S(Q,0) all'equazione (3) producono i tassi di rilassamento Q-dipendenti Γ (Q). - Tracciare Γ (Q) in funzione di Q e adattarsi a un modello adatto per estrarre i parametri di membrana rilevanti.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Gli studi NSE che accedono alle fluttuazioni di flessione vengono in genere eseguiti su un intervallo Q di ~ (0,04 - 0,2) Å-1. Questo intervallo Q corrisponde a scale di lunghezza intermedie tra lo spessore della membrana e il raggio liposomiale, dove dominano le dinamiche di flessione. La misurazione su un intervallo Q esteso può dare accesso a modalità dinamiche aggiuntive, tra cui la diffusione liposomiale e la dinamica intramembrana. Per maggiori dettagli sul cross-over nella dinamica delle membrane a cui ha avuto accesso NSE, consultare queste pubblicazioni pertinenti25,71. È importante sottolineare che i segnali NSE sono proporzionali a:  , dove Icoh e Iinc sono, rispettivamente, l'intensità di scattering coerente e incoerente del campione. Pertanto, è consigliabile preparare campioni liposomiali NSE in tamponi deuterati (cioè tamponi preparati con D2O invece di H2O) per ridurre al minimo il segnale di scattering incoerente, principalmente contribuito dal contenuto di idrogeno del campione. Tuttavia, in alcuni casi potrebbero essere necessari schemi di deuterazione intermedi (ad es. utilizzando miscele di D2O e H2O) per ottenere condizioni di contrasto ottimali. Tipicamente, le misurazioni NSE delle fluttuazioni di flessione della membrana vengono eseguite su liposomi completamente proziati in tampone deuterato, indicato come liposomi completamente contrastati nella Figura 5. Questo schema di deuterazione si traduce in una grande differenza di NSLD tra il nucleo della membrana (~0 ×10 -6 Å -2) e il suo ambiente fluido deuterato (~6,4 ×10 -6 Å-2), che migliora significativamente il segnale di scattering dalle membrane liposomiali e migliora le statistiche di misurazione della dinamica di flessione. Questo schema di contrasto(Figura 2A pannello di sinistra) è frequentemente utilizzato negli studi di rigidità di flessione delle membrane lipidiche con singoli componenti lipidici38,72 e multipli39,66 e negli studi di ammorbidimento/irrigidimento della membrana mediante inclusioni biologiche (ad esempio, colesterolo, molecole di farmaci, peptidi/proteine)36,37,73, 74,75e additivi sintetici (ad esempio, nanoparticelle)76,77.
, dove Icoh e Iinc sono, rispettivamente, l'intensità di scattering coerente e incoerente del campione. Pertanto, è consigliabile preparare campioni liposomiali NSE in tamponi deuterati (cioè tamponi preparati con D2O invece di H2O) per ridurre al minimo il segnale di scattering incoerente, principalmente contribuito dal contenuto di idrogeno del campione. Tuttavia, in alcuni casi potrebbero essere necessari schemi di deuterazione intermedi (ad es. utilizzando miscele di D2O e H2O) per ottenere condizioni di contrasto ottimali. Tipicamente, le misurazioni NSE delle fluttuazioni di flessione della membrana vengono eseguite su liposomi completamente proziati in tampone deuterato, indicato come liposomi completamente contrastati nella Figura 5. Questo schema di deuterazione si traduce in una grande differenza di NSLD tra il nucleo della membrana (~0 ×10 -6 Å -2) e il suo ambiente fluido deuterato (~6,4 ×10 -6 Å-2), che migliora significativamente il segnale di scattering dalle membrane liposomiali e migliora le statistiche di misurazione della dinamica di flessione. Questo schema di contrasto(Figura 2A pannello di sinistra) è frequentemente utilizzato negli studi di rigidità di flessione delle membrane lipidiche con singoli componenti lipidici38,72 e multipli39,66 e negli studi di ammorbidimento/irrigidimento della membrana mediante inclusioni biologiche (ad esempio, colesterolo, molecole di farmaci, peptidi/proteine)36,37,73, 74,75e additivi sintetici (ad esempio, nanoparticelle)76,77.
Le misurazioni delle fluttuazioni di flessione si traducono in tassi di rilassamento che seguono una dipendenza Q3, come previsto da Zilman e Granek per fogli sottili elastici termicamente ondulati70. Una forma raffinata di questa Q-dipendenzaè ottenuta dalle correzioni teoriche di Watson e Brown78, che tengono conto degli effetti dell'attrito intermonostrato proposti da Seifert e Langer79. Definendo inoltre il piano neutro all'interfaccia tra i gruppi di testa idrofili e le code idrofobiche della membrana, i tassi di rilassamento della flessione possono quindi essere adattati alla seguente espressione38.

dove ηbuff è la viscosità tampone, kBT è l'energia termica, κ e è la rigidità di flessione della membrana misurata (o della porzione contrastata della membrana in sistemi selettivamente deuterati). Questo tipo di misurazione consente il calcolo diretto delle proprietà elastiche della membrana sotto forma di modulo di rigidità di flessione. Si noti che κ viene estratto dalla pendenza dell'adattamento lineare di Γ rispetto a Q3, come mostrato nella Figura 5C.
D'altra parte, le misurazioni NSE delle fluttuazioni dello spessore della membrana mostrano deviazioni dalla dipendenza Q3in Γ(Q) intorno a valori Q che corrispondono allo spessore della membrana (vedi Figura 2 in rif.66). Per isolare il segnale di fluttuazione dello spessore, si può dividere Γ(Q) per Q3, come mostrato in Figura 5D. I dati risultanti mostrano che le dinamiche in eccesso dovute alle fluttuazioni di spessore seguono una funzione lorentziana in Q, come recentemente corroborato nelle simulazioni di dinamica molecolare a grana grossa (MD)67. Per adattarsi alle dinamiche in eccesso osservate, Nagao et al.38 hanno sviluppato un'espressione basata sul quadro teorico delle fluttuazioni di membrana di Bingham et al.80 come segue.

In questa espressione, Q0 è il valore Qdi picco corrispondente allo spessore della membrana (che può essere ottenuto indipendentemente dalle misurazioni SANS), μ è la viscosità della membrana nel piano, AL è l'area per lipide (misurata con SANS / SAXS) e KA è il modulo di comprimibilità dell'area. Supponendo che KA possa essere calcolato da κ utilizzando il modello di pennello polimerico, questa espressione si riduce a un parametro di adattamento, vale a dire, la viscosità della membrana μ, presentando un nuovo approccio per misurare la viscosità della membrana senza la necessità di etichettatura a fluorescenza o tethering / tracciamento delle particelle13. La premessa è che secondo i modelli di deformazione dei fogli sottili elastici81, κ e KA sono interdipendenti in modo tale che: ,  dove tm è lo spessore meccanico (o deformabile) della membrana e β è una costante che descrive l'accoppiamento interleaflet. L'ipotesi è che β = 12 per i foglietti completamente accoppiati, β = 48 per i foglietti completamente disaccoppiati e β = 24 per i foglietti accoppiati intermediamente. Quest'ultimo è indicato come il pennello polimerico modello81 e ha dimostrato di applicarsi in membrane lipidiche fluide monocomponenti e binarie39. Tuttavia, questo deve essere affrontato con cautela. Ad esempio, recenti simulazioni di Doktorova et al. 82 ha dimostrato che, affinché il modello di pennello polimerico contenga membrane lipidiche insature contenenti colesterolo, deve essere utilizzata un'espressione modificata dello spessore meccanico della membrana. Idealmente, se è possibile una misura indipendente di KA, ad esempio utilizzando l'aspirazione a micropipette83, allora combinare i risultati di KA con le misure di rigidità di flessione NSE presenterebbe un'opportunità unica per studiare l'accoppiamento interleaflet in membrane modello e biologiche - una questione di lunga data nella biofisica delle membrane e nella biologia strutturale. Una volta convalidati i valori di KA, possono essere utilizzati nell'equazione 5 per ottenere la viscosità mesoscopica della membrana.
dove tm è lo spessore meccanico (o deformabile) della membrana e β è una costante che descrive l'accoppiamento interleaflet. L'ipotesi è che β = 12 per i foglietti completamente accoppiati, β = 48 per i foglietti completamente disaccoppiati e β = 24 per i foglietti accoppiati intermediamente. Quest'ultimo è indicato come il pennello polimerico modello81 e ha dimostrato di applicarsi in membrane lipidiche fluide monocomponenti e binarie39. Tuttavia, questo deve essere affrontato con cautela. Ad esempio, recenti simulazioni di Doktorova et al. 82 ha dimostrato che, affinché il modello di pennello polimerico contenga membrane lipidiche insature contenenti colesterolo, deve essere utilizzata un'espressione modificata dello spessore meccanico della membrana. Idealmente, se è possibile una misura indipendente di KA, ad esempio utilizzando l'aspirazione a micropipette83, allora combinare i risultati di KA con le misure di rigidità di flessione NSE presenterebbe un'opportunità unica per studiare l'accoppiamento interleaflet in membrane modello e biologiche - una questione di lunga data nella biofisica delle membrane e nella biologia strutturale. Una volta convalidati i valori di KA, possono essere utilizzati nell'equazione 5 per ottenere la viscosità mesoscopica della membrana.

Figura 1: Progettazione dello strumento NSE e sovrapposizione sinergica con scale di lunghezza/tempi della dinamica mesoscopica della membrana. (A) Schema dei diversi elementi magnetici di uno strumento NSE, utilizzato per manipolare lo spin dei neutroni che attraversano lo strumento da sinistra a destra. Il neutrone evidenziato indica un cambiamento nell'orientamento dello spin (o perdita di polarizzazione) dovuto allo scambio di energia tra il neutrone e il campione, mentre il neutrone trasparente rappresenta lo spin-eco, cioè nessun cambiamento nello spin del neutrone a causa dello scambio di energia zero. La freccia grigia indica la possibilità di ruotare il secondo braccio dello spettrometro per accedere ad angoli di dispersione più grandi. (B) Rappresentazione pittorica della dinamica gerarchica nelle membrane lipidiche, mostrando vari modi dinamici che coprono più scale di lunghezza e tempo. L'area ombreggiata rappresenta le scale di lunghezza e tempo a cui si accede da NSE, che si sovrappongono alle mesoscale delle fluttuazioni collettive della membrana, vale a dire, flessione e fluttuazioni di spessore. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Esempi di possibili schemi di deuterazione in esperimenti NSE su membrane lipidiche. (A) Sinistra: Membrane completamente contrastate, ad esempio membrane protiate in tampone deuterato, che mostrano il profilo NSLD lungo la superficie normale della membrana. La differenza nella NSLD tra la regione della coda (~0 × 10-2 Å-2) e la regione del gruppo di testa (~4,5 × 10-6 Å-2) della membrana è dovuta all'idratazione del gruppo di testa con tampone deuterato. A destra: membrane abbinate al contrasto della coda in modo tale che la regione di coda dell'idrocarburo della membrana abbia lo stesso NSLD del tampone, come mostrato nel corrispondente profilo NSLD lungo la membrana normale. (B) Membrane che formano domini con due schemi di contrasto neutronico in cui i domini (al centro) o la matrice (a sinistra) sono abbinati al tampone, consentendo studi selettivi della dinamica della matrice o del dominio, rispettivamente. Questa cifra è stata modificata da Nickels et al., JACS 201541. (C) Membrane asimmetriche preparate mediante scambio di ciclodestrine tra vescicole lipidiche proziate e deuterate, con conseguente deuterazione di un foglietto di membrana mantenendo l'altro foglietto proziato. Ciò consente di studi sulla dinamica di flessione del foglietto proziato e fornisce informazioni sull'accoppiamento meccanico tra le foglioline opposte in membrane asimmetriche. Questa figura è stata modificata da Rickeard et al., Nanoscale 202040. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Illustrazione della configurazione per l'estrusione automatica dei liposomi. (A) Estrusore automatizzato su misura utilizzando una pompa a siringa, un mini set di estrusori e un telaio in alluminio/acciaio per consentire estrusi ciclici. (B) e (C) mostrano la differenza nell'aspetto visivo delle sospensioni lipidiche prima (bianco latte) e dopo (blu opale trasparente) e dopo l'estrusione (blu opale trasparente). Ciò è dovuto alla formazione iniziale di pile lipidiche di dimensioni micron o vescicole giganti che sono dell'ordine o più grandi della lunghezza d'onda della luce visibile. Dopo l'estrusione, la sospensione comprenderà vescicole nanoscopiche (~ 100 nm), che sono più piccole della lunghezza d'onda della luce visibile, producendo una sospensione trasparente. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Dati rappresentativi degli esperimenti NSE sulle sospensioni liposomiali. (A) Esempio di un eco-segnale su un singolo pixel del rivelatore (pixel marcato nel pannello B), che mostra gli adattamenti del segnale dell'eco usando l'equazione (1), con un'illustrazione dei diversi parametri richiesti nell'echo fit. Si noti che il segnale eco viene tracciato in funzione dell'angolo di fase anziché della corrente di fase come discusso nel passaggio 4.7 del protocollo. (B) Immagine del rivelatore NSE che mostra la variazione della conta neutronica per pixel. L'immagine mostra anche i pixel del rilevatore eliminati (verde) a causa di scarsi segnali di eco. Il binning dei pixel del rivelatore negli archi Q (noto anche come anelli di Debye-Scherrer) produce la dipendenza Q della funzione di scattering intermedio, necessaria per analizzare e interpretare i dati NSE. Questa cifra è stata modificata da Ashkar, J. Appl. Phys. 202050. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Risultati rappresentativi di esperimenti NSE su sospensioni liposomiali con diversi schemi di deuterazione. (A) Geometria di scattering di un neutrone che interagisce con un liposoma, mostrando l'angolo di scattering, 2θ, e il trasferimento del motovettore,  . (B) Funzioni di scattering intermedie, S(Q,t) / S(Q,0), mostrano decadimenti in funzione del tempo di Fourier. L'adattamento dei decadimenti misurati a una funzione esponenziale allungata data dall'equazione 3 produce i tassi di rilassamento, Γ(Q). (C) Per i liposomi completamente proziati in tampone deterrato, Γ(Q) segue una dipendenza Q3, tipica della dinamica di flessione. L'adattamento lineare dei dati ottenuti a un modello Zilman-Granek produce il modulo di rigidità di flessione della membrana. (D) Per i liposomi deuterati della coda, si osserva una dinamica in eccesso oltre alle fluttuazioni di flessione e sono più pronunciati a valori Q che corrispondono allo spessore della membrana. Adattare la dinamica in eccesso a una funzione lorentziana (equazione 5) consente l'estrazione della viscosità della membrana. I set di dati sono stati raccolti sullo spettrometro NSE del NIST. Fare clic qui per visualizzare una versione più grande di questa figura.
. (B) Funzioni di scattering intermedie, S(Q,t) / S(Q,0), mostrano decadimenti in funzione del tempo di Fourier. L'adattamento dei decadimenti misurati a una funzione esponenziale allungata data dall'equazione 3 produce i tassi di rilassamento, Γ(Q). (C) Per i liposomi completamente proziati in tampone deterrato, Γ(Q) segue una dipendenza Q3, tipica della dinamica di flessione. L'adattamento lineare dei dati ottenuti a un modello Zilman-Granek produce il modulo di rigidità di flessione della membrana. (D) Per i liposomi deuterati della coda, si osserva una dinamica in eccesso oltre alle fluttuazioni di flessione e sono più pronunciati a valori Q che corrispondono allo spessore della membrana. Adattare la dinamica in eccesso a una funzione lorentziana (equazione 5) consente l'estrazione della viscosità della membrana. I set di dati sono stati raccolti sullo spettrometro NSE del NIST. Fare clic qui per visualizzare una versione più grande di questa figura.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
NSE è una tecnica potente e unica nella misurazione della dinamica mesoscopica delle membrane lipidiche in varie condizioni. L'utilizzo efficace di NSE dipende dalla qualità del campione, dal contrasto neutronico e dalla gamma di dinamiche accessibili che possono essere sonde per un determinato campione. Pertanto, sono necessari diversi passaggi critici per eseguire esperimenti NSE di successo e raccogliere dati di alta qualità. Un passo fondamentale per garantire l'uso efficace del tempo del fascio di neutroni durante un esperimento NSE è quello di caratterizzare le sospensioni liposomiali con metodi basati su laboratorio prima dell'esperimento NSE. Per esempio, la distribuzione dimensionale (o costante di diffusione) dei liposomi estrusi può essere determinata mediante scattering dinamico della luce (DLS), prontamente disponibile nei singoli laboratori o in strutture condivise84. La microscopia crioeletletnica è un altro metodo di charcaterizzazione recentemente validato su campioni liposomiali, in cui immagini al microscopio ad alta risoluzione su sezioni criomicrotomizzate di sospensione liposomiale possono essere efficacemente utilizzate per esaminare l'unilamellarità liposomiale65,la formazione del dominio85,86,o l'incorporazione di additivi come le nanoparticelle76 e le proteine87. In alternativa, lo scattering a raggi X ad angolo piccolo (SAXS) può essere utilizzato per caratterizzare la struttura della membrana88,valutare la multilamellarità liposomiale65o valutare gli effetti degli additivi sulle proprietà strutturali della membrana89. Oltre a queste tecniche di laboratorio, è altamente consigliabile che le misurazioni NSE su campioni liposomiali siano abbinate a studi strutturali utilizzando lo scattering neutronico a piccolo angolo (SANS)54,90. SANS è un eccellente complemento a NSE, non solo per acquisire informazioni strutturali sulla membrana, ma anche per esaminare l'intensità del segnale di scattering neutronico dal campione, confermare lo schema di contrasto e fare una scelta informata sul Q-range su cui devono essere eseguite le misurazioni NSE. Pertanto, si consiglia agli utenti NSE di richiedere il beamtime SANS quando fanno domanda per gli esperimenti NSE.
Tuttavia, NSE soffre di limitazioni del campione negli studi sulle membrane biologiche. Uno dei principali fattori limitanti di tali esperimenti è la quantità standard di campione richiesta per le misurazioni NSE (2-4 ml) e le alte concentrazioni del campione pari a 100-200 mg di materiale di membrana (lipidi e proteine) per ottenere dati di alta qualità. In molti casi, la produzione di tali quantità di materiale biologico non è fattibile o è proibitiva in termini di costi. In tali scenari, è possibile ridurre la concentrazione a 20-25 mg/ml, ma ciò richiederebbe almeno un aumento di 4 volte del tempo di acquisizione al fine di ottenere statistiche comparabili ai campioni con concentrazioni di 50 mg/mL. Questi severi requisiti sul volume e sulla concentrazione del campione potrebbero essere alleviati con la prossima generazione di spettrometri NSE su sorgenti di neutroni a flusso più elevato, come la seconda stazione bersaglio presso l'Oak Ridge National Lab e la European Spallation Source. Un'altra limitazione critica nell'esecuzione di esperimenti NSE su membrane lipidiche che richiedono schemi di deuterazione selettiva è la mancanza di disponibilità commerciale di alcune varianti deuterate di molecole lipidiche o i loro prezzi esorbitanti, se disponibili. In alcuni casi, queste limitazioni possono essere aggirate richiedendo la sintesi di lipidi deuterati (o colesterolo, proteine) attraverso strutture di deuterazione degli utenti, come il laboratorio di bio-deuterazione presso l'Oak Ridge National Lab, l'impianto nazionale di deuterazione presso ANSTO o l'impianto di deuterazione presso l'ISIS Neutron and Muon Source. L'accesso a queste strutture e alle loro capacità di sintesi è disponibile attraverso proposte presentate dagli utenti che sono sottoposte a revisione paritaria in base al merito scientifico della sintesi del materiale proposto e al suo uso previsto negli studi sensibili agli isotopi.
Nonostante queste limitazioni, l'applicazione della spettroscopia NSE negli studi di meccanica delle membrane ha portato alla determinazione dei moduli di rigidità di flessione di membrane con vari gradi di complessità, dalle membrane lipidiche monocomponenti35,38 alle membrane biomimetiche multicomponenti41,66,91,che hanno fatto avanzare la nostra comprensione della natura dinamica delle membrane lipidiche. Ad esempio, le misure di rigidità di flessione NSE di membrane lipidiche con diverse unità molecolari, ad esempio lipidi di diverse lunghezze di catena acilica e saturazione della catena38,72,92, hanno fornito informazioni essenziali sul ruolo della chimica molecolare nella meccanica delle membrane. Se abbinate a informazioni strutturali, come lo spessore della membrana o l'imballaggio molecolare93,queste misurazioni iniziano a fornire nuove prospettive sull'interdipendenza tra struttura e dinamica della membrana e su come influenzano la funzione della membrana. Le scale mesoscopiche di NSE lo posizionano in modo univoco per tali indagini fondamentali delle relazioni struttura-proprietà, più rilevanti sulla scala di lunghezza degli assemblaggi molecolari. Questo argomento è stato recentemente esplorato in due studi NSE su membrane lipidiche ricche di colesterolo36 e in membrane lipidiche binarie con discrepanza idrofobica tra i due componenti lipidici39. Entrambi gli studi hanno trovato forti prove che la meccanica di membrana scala con l'area per lipide, corroborando le conclusioni di una recente simulazione MD all-atom di Doktorova et al.82. Questi risultati sottolineano la natura auto-assemblata delle membrane lipidiche e forniscono un quadro unificante dell'imballaggio molecolare come parametro chiave nella definizione delle proprietà dinamiche e funzionali della membrana.
Altre applicazioni di NSE coinvolgono studi sulla risposta meccanica delle membrane a piccoli additivi, tra cui molecole biologiche come colesterolo36,37, trealosio92e melittina73,94, o additivi inorganici come nanoparticelle per applicazioni di somministrazione di farmaci76. NSE è stato utilizzato anche per capire come la meccanica delle membrane risponde ai cambiamenti nel loro ambiente, tra cui la temperatura92,pH74e la presenza di macromolecole di affollamento96. Tali studi stanno contribuendo a una migliore comprensione dei fattori che influenzano l'ammorbidimento o l'irrigidimento delle membrane lipidiche, in condizioni biologiche legate alla salute e alla malattia e in contesti controllati per applicazioni terapeutiche. In particolare, le misurazioni NSE sono state utilizzate anche per sondare l'effetto dei peptidi antimicrobici sulla dinamica della membrana73,94,95. Ulteriori esempi di applicazioni NSE su biomembrane includono studi sulla dinamica di strutture di membrana appiattite, chiamate thylakoids, che ospitano il macchinario fotosintetico in cellule cianobatteriche97,98.
Si può anche utilizzare la deuterazione lipidica selettiva negli studi NSE per studiare la dinamica di specifiche caratteristiche della membrana che sono rilevanti per la funzione biologica. Ad esempio, Nickels et al. hanno utilizzato la deuterazione lipidica selettiva nelle membrane lipidiche che formano domini per generare un contrasto laterale all'interno della membrana, come precedentemente illustrato da Heberle et al.28. Questo schema di deuterazione ha permesso di misurare in modo indipendente la rigidità di flessione dei domini lipidici e della matrice lipidica ospite41 (vedi Figura 2B). I risultati hanno confermato che i due compartimenti di membrana hanno moduli di rigidità di flessione distinti, che potrebbero essere un meccanismo trainante per la formazione del dominio nelle membrane cellulari. In uno studio più recente, Rickeard et al. hanno utilizzato lo scambio di ciclodestrine tra liposomi proziati e deuterati per ottenere liposomi asimmetrici con foglietti isotopicamente etichettati40 (Figura 2C). I loro liposomi terminali avevano un foglietto proziato e un foglietto deuterato che è in contrasto con il tampone, consentendo studi sulla dinamica dei singoli foglietti e fornendo un primo resoconto sperimentale diretto dell'effetto dell'asimmetria e dell'accoppiamento del foglietto sulle fluttuazioni di flessione della membrana.
La deuterazione selettiva della membrana è stata utilizzata anche negli studi NSE sulle fluttuazioni dello spessore della membrana, una modalità dinamica a lungo prevista nelle membrane lipidiche99 che è stata osservata solo di recente con l'avvento della spettroscopia NSE35,100. Queste misurazioni utilizzano membrane deuterate di coda per amplificare il segnale proveniente dalle regioni del gruppo di testa della membrana e risolvere il segnale di fluttuazione dello spessore. Questo tipo di esperimenti NSE è relativamente recente, ma è stato efficacemente utilizzato per comprendere l'interdipendenza delle proprietà elastiche e viscose della membrana38, per esplorare il ridimensionamento della rigidità e della viscosità di flessione con l'impacchettamento molecolare nelle membrane lipidiche miste39e per sondare gli effetti locali del colesterolo sulla viscosità della membrana36. Un'altra area di significato biologico in cui questa modalità dinamica potrebbe avere implicazioni di vasta portata è l'interazione mesoscopica membrana-proteina95. È noto che la funzione delle proteine di membrana è strettamente legata alla corrispondenza idrofobica tra la proteina e la membrana ospite. Pertanto, le variazioni dello spessore della membrana, dovute alle fluttuazioni dello spessore, potrebbero fungere da meccanismo di regolazione per la funzione delle proteine di membrana. NSE è estremamente adatto per tali studi in quanto può sondare direttamente gli effetti del legame e dell'inserimento proteico sulle fluttuazioni dello spessore della membrana. Recenti misurazioni NSE del nostro gruppo (non pubblicate) suggeriscono che l'inserimento di proteine transmembrana potrebbe sopprimere significativamente le fluttuazioni dello spessore della membrana e potrebbe presentare un potenziale meccanismo per regolare gli eventi di segnalazione. Questa è un'area di ricerca pressante, ma sottosviluppata, in cui la NSE può avere un impatto significativo nella comprensione delle risposte dinamiche delle membrane al legame proteico e all'inserimento sulle scale temporali e temporali delle funzioni biologiche chiave impartite dalle interazioni delle proteine con le membrane cellulari.
In sintesi, nSE si è evoluto negli ultimi anni come un potente strumento per interrogare la dinamica della membrana su scale spaziali e temporali di funzioni biologiche vitali. La tecnica sta rapidamente guadagnando un interesse diffuso e il suo potenziale nel rispondere a domande chiave sulla funzione della membrana sta diventando ben riconosciuto. Le capacità di variazione del contrasto all'interno di NSE lo hanno posizionato come un approccio unico per misurare le proprietà mesoscopiche della membrana che altrimenti sarebbe difficile da ottenere. Un altro vantaggio significativo dell'NSE rispetto ai metodi di spettroscopia tradizionali negli studi di dinamica di membrana è la sua sovrapposizione con le scale di lunghezza e tempo accessibili con le simulazioni MD, consentendo studi sinergici sperimentali / di calcolo per ottenere una comprensione a livello molecolare dei diversi componenti molecolari che compongono le membrane. Nonostante la sua promessa, ci sono ancora alcune limitazioni nell'uso di NSE negli studi sulle membrane biologiche, tra cui il requisito di grandi volumi di campioni, la difficoltà nella deuterazione selettiva nei sistemi biologici e il flusso di neutroni relativamente basso sugli spettrometri NSE, che si traduce in tempi di misurazione più lunghi e disponibilità limitata di beamtime. Tuttavia, queste carenze potrebbero essere superate nel prossimo futuro con costanti sviluppi nelle sorgenti di neutroni e nella strumentazione insieme ai progressi negli impianti di deuterazione.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Gli autori non dichiarano conflitti di interesse e non hanno nulla da rivelare.
Acknowledgments
R. Ashkar ringrazia M. Nagao, L.-R. Stingaciu e P. Zolnierczuk per molte discussioni utili e per la loro frequente assistenza con esperimenti NSE sulle rispettive linee di fascio. Gli autori riconoscono l'uso di spettrometri eco-spin neutronici presso NIST e ORNL. Lo spettrometro NSE del NIST è supportato dal Center for High Resolution Neutron Scattering, una partnership tra il National Institute of Standards and Technology e la National Science Foundation sotto accordo n. DMR-1508249. Lo spettrometro NSE presso la sorgente di neutroni di spallazione dell'ORNL è supportato dalla Scientific User Facilities Division, Office of Basic Energy Sciences, US Department of Energy. Oak Ridge National Laboratory è gestito da UT-Battelle, LLC sotto il contratto DOE degli Stati Uniti n. DE-AC05-00OR22725.
Materials
| Name | Company | Catalog Number | Comments |
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. Ø, Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes--a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich's model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a "modeling manifesto". Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D. Neutrons in Soft Matter. Imae, T., Kanaya, T., Furusaka, M., Torikai, N. , Wiley. ch6 147-182 (2011).
- Pynn, R. Neutron Spin Echo. Mezei, F., Pappas, C., Gutberlet, T. , Springer. Berlin Heidelberg. 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , Oxford University Press. (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- Neutron activation and scattering calculator. , Available from: https://www.ncnr.nist.gov/resources/activation/ (2021).
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , Walter de Gruyter, Inc. 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles - as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O'Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -R., O'Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).
Tags
Biologia Numero 171 dinamica lipidica collettiva modulo di flessione comprimibilità dell'area fluttuazioni di spessore viscosità della membrana deuterazione selettiva preparazione dei liposomiErratum
Formal Correction: Erratum: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions
Posted by JoVE Editors on 08/06/2021.
Citeable Link.
An erratum was issued for: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions. The Introduction, Protocol, and Representative Results sections have been updated.
In the Introduction, the fith pargraph was updated from:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 2 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
to:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 0 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
In the Protocol, step 1.1 was updated from:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 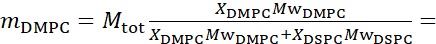 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
to:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 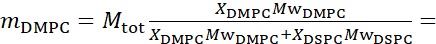 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
In the Representative Results, the fist pagargaph was updted from:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 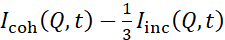 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
to:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 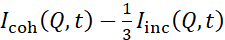 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
In the Representative Reults, Figure 2 was updated from:
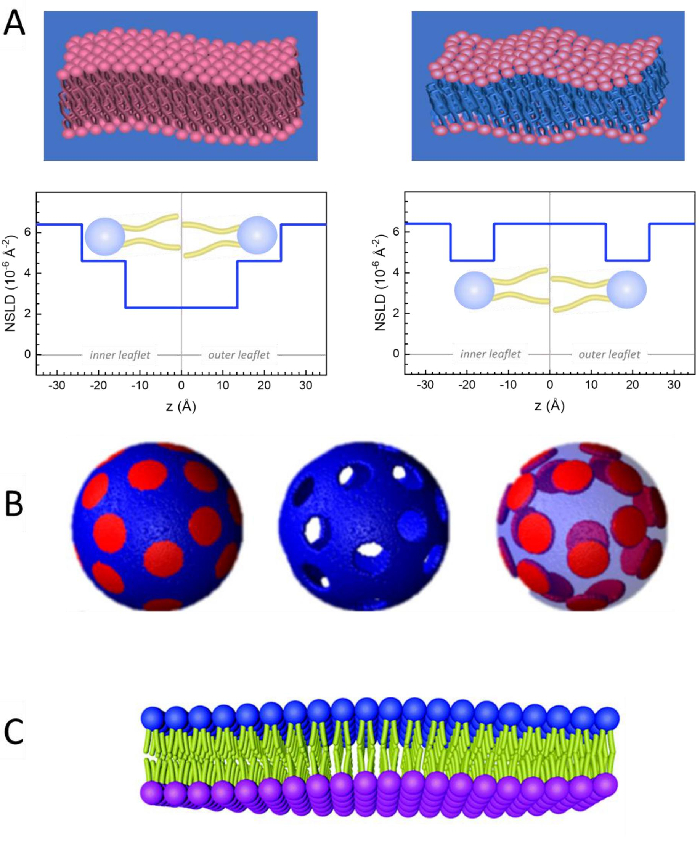
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the headgroup (~2 × 10-2 Å-2) and tail region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
to:
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the tail region (~0 × 10-2 Å-2) and headgroup region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.



 133,4 mg, dove XDMPC e XDSPC sono le frazioni molari di DMPC e DSPC, qui impostate rispettivamente a 0,7 e 0,3, e MwDMPC e MwDSPC sono i pesi molari dati rispettivamente da 677,9 g / mol e 790,1 g / mol. Allo stesso modo, mDSPC = 66,6 mg. Questo schema di deuterazione aumenta il contrasto di scattering tra la membrana (NSLD ~ 0 × 10-6 Å-2) e il tampone deuterato (NSLD ~ 6,4 × 10-6 Å -2) e amplifica il segnale dalle indulazioni di membrana (vedi Figura 2A pannello di sinistra).
133,4 mg, dove XDMPC e XDSPC sono le frazioni molari di DMPC e DSPC, qui impostate rispettivamente a 0,7 e 0,3, e MwDMPC e MwDSPC sono i pesi molari dati rispettivamente da 677,9 g / mol e 790,1 g / mol. Allo stesso modo, mDSPC = 66,6 mg. Questo schema di deuterazione aumenta il contrasto di scattering tra la membrana (NSLD ~ 0 × 10-6 Å-2) e il tampone deuterato (NSLD ~ 6,4 × 10-6 Å -2) e amplifica il segnale dalle indulazioni di membrana (vedi Figura 2A pannello di sinistra). Mtot è la massa totale, impostata qui a 200 mg. Vedi l'esempio sopra per i doppi strati lipidici DMPC-DSPC con diversi schemi di deuterazione.
Mtot è la massa totale, impostata qui a 200 mg. Vedi l'esempio sopra per i doppi strati lipidici DMPC-DSPC con diversi schemi di deuterazione.

 , quindi i cambiamenti nello stato di spin a causa dello scambio di energia tra il neutrone e il campione viene rilevata come una goccia nella polarizzazione (dall'unità).
, quindi i cambiamenti nello stato di spin a causa dello scambio di energia tra il neutrone e il campione viene rilevata come una goccia nella polarizzazione (dall'unità).
