ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Klonale expansie is een belangrijk kenmerk van antigeenspecifieke T-celrespons. De celcyclus van antigeen-reagerende T-cellen is echter slecht onderzocht, deels vanwege technische beperkingen. We beschrijven een flowcytometrische methode om klonaal uitdijende antigeenspecifieke CD8 T-cellen in milt- en lymfeklieren van gevaccineerde muizen te analyseren.
Abstract
De celcyclus van antigeenspecifieke T-cellen in vivo is onderzocht met behulp van een paar methoden, die allemaal enkele beperkingen hebben. Bromodeoxyuridine (BrdU) markeert cellen die zich in of onlangs voltooide S-fase bevinden, en carboxyfluoresceïne succinimidylester (CFSE) detecteert dochtercellen na deling. Deze kleurstoffen laten echter geen identificatie van de celcyclusfase toe op het moment van analyse. Een alternatieve benadering is om Ki67 te benutten, een marker die sterk tot expressie wordt gebracht door cellen in alle fasen van de celcyclus, behalve de rustige fase G0. Helaas staat Ki67 geen verdere differentiatie toe, omdat het geen cellen in de S-fase scheidt die zijn toegewijd aan mitose van die in G1 die in deze fase kunnenblijven, doorgaan met fietsen of naar G0gaan .
Hier beschrijven we een flowcytometrische methode voor het vastleggen van een "momentopname" van T-cellen in verschillende celcyclusfasen in secundaire lymfoïde organen van muizen. De methode combineert Ki67 en DNA-kleuring met major histocompatibiliteitscomplex (MHC)-peptide-multimerkleuring en een innovatieve gatingstrategie, waardoor we met succes onderscheid kunnen maken tussen antigeenspecifieke CD8 T-cellen in G0, in G1 en in S-G2/ M-fasen van de celcyclus in de milt en drainerende lymfeklieren van muizen na vaccinatie met virale vectoren die het modelantigeen gag van het humaan immunodeficiëntievirus (HIV) -1 dragen.
Kritische stappen van de methode waren de keuze van de DNA-kleurstof en de gating-strategie om de testgevoeligheid te verhogen en sterk geactiveerde / prolifererende antigeenspecifieke T-cellen op te nemen die volgens de huidige analysecriteria zouden zijn gemist. De DNA-kleurstof, Hoechst 33342, stelde ons in staat om een hoogwaardige discriminatie van de G0/ G 1 en G2 / M DNA-pieken te verkrijgen, met behoud vanmembraan-en intracellulaire kleuring. De methode heeft een groot potentieel om de kennis over T-celrespons in vivo te vergroten en de immunomonitoringanalyse te verbeteren.
Introduction
Naïeve T-cellen ondergaan klonale expansie en differentiatie op antigeen-priming. Gedifferentieerde T-cellen vertonen effectorfuncties die essentieel zijn voor antigeenklaring en voor het behoud van antigeenspecifiek geheugen, wat essentieel is voor langdurige bescherming. Tijdens de eerste stappen van de primaire respons is naïeve T-celinteractie met antigeen-presenterende cellen (APC's) binnen gespecialiseerde niches in lymfoïde organen van cruciaal belang om de enorme T-celproliferatie te induceren die kenmerkend is voor de klonale expansiefase1,2,3. T-cel-APC interactie wordt fijn gereguleerd door concentratie en persistentie van antigeen, co-stimulerende signalen en oplosbare factoren (cytokines en chemokines) die de kwantiteit en kwaliteit van het T-cel klonale nageslacht beïnvloeden4,5,6,7.
Ondanks intensieve studies van T-cel klonale expansie, is het nog steeds niet bekend of antigeen-geprimeerde T-cellen hun hele celcyclus voltooien op de plaats van antigeenherkenning, of dat ze migreren naar andere organen tijdens de progressie van de celcyclus. Dit gebrek aan kennis is te wijten aan de eigenschappen van beschikbare hulpmiddelen voor celcyclusanalyse. Deze omvatten monoklonale antilichamen (mAbs) specifiek voor de nucleaire marker, Ki67, en celkleurstoffen die ofwel cellen identificeren die de S-fase van de celcyclus hebben ondergaan (bijv. Bromodeoxyuridine (BrdU)) of discrimineren tussen dochtercellen en hun voorouders (bijv. Carboxyfluoresceïne succinimidylester (CFSE)).
Celetiketterende kleurstoffen, zoals CFSE en BrdU, laten echter niet toe om te bepalen of cellen in een bepaald orgaan lokaal woekeren of eerder naar deze site zijn gemigreerd na deling8,9. Bovendien is het intranucleaire eiwit, Ki67, alleen in staat om cellen in G0 (Ki67-negatieve cellen) te onderscheiden van die in een andere celcyclusfase (Ki67-positieve cellen). Ki67-analyse onderscheidt dus geen cellen in actieve proliferatie (d.w.z. in S, G2of M) van die in G1, die ofwel snel kunnen evolueren naar deling of gedurende lange perioden in G1 kunnen blijven of terugkeren naar rust10,11.
Hier beschrijven we een nieuwe flowcytometrische methode voor celcyclusanalyse van antigeenspecifieke CD8 T-cellen12 uit de milt- en lymfeklieren (LNs) van gevaccineerde muizen(figuur 1). De methode maakt gebruik van een combinatie van Ki67 en DNA-kleuring die eerder werd gebruikt om de celcyclus van hematopoëtische cellen van muizenbeenmerg (BM) te analyseren13,14. Hier hebben we met succes Ki67 plus DNA-kleuring toegepast, samen met de onlangs gepubliceerde innovatieve gating-strategie12, op de analyse van CD8 T-cel klonale expansie. We waren in staat om duidelijk onderscheid te maken tussen antigeenspecifieke CD8 T-cellen in G0,in G1,en in S-G2/ M-fasen in de milt en drainerende LNs van gevaccineerde muizen.
Protocol
Muizen werden gehuisvest in Plaisant Animal Facility en het werk werd uitgevoerd onder autorisatienummer 1065/2015-PR van het Italiaanse ministerie van Volksgezondheid. Het protocol volgde de richtlijnen voor dierverzorging volgens nationale en internationale wetten en beleid (UE-richtlijn 2010/63/UE; Italiaans wetsbesluit 26/2014).
1. Bereiding van medium en kleuroplossing
- Bereid volledig medium voor: Roswell Park Memorial Institute (RPMI) medium met 2 mM glutamine, 100 U / ml penicilline / streptomycine, 50 μM bèta-mercaptoethanol en 10% volume / volume (v / v) foetaal runderserum (FBS)
- Bereidingskleuringsbuffer: Fosfaat-gebufferde zoutoplossing zonder Ca2+/Mg2+ (PBS) met 1% gewicht/volume (w/v) runderserumalbumine (BSA) en 2 mM ethyleendiaminetetra-azijnzuur dinatriumzout (EDTA)
2. Muis behandeling
- Prime 7-8 weken oude, vrouwelijke Balb / c muizen door intramusculaire (i.m.) injectie in de quadriceps van humaan immunodeficiëntievirus (HIV)-1-gag-expressing-chimpansee adenovirale vector (ChAd3-gag) met een dosis van 107 virale deeltjes.
- Op 1-4 maanden na priming, boost eenmaal de muizen door i.m. injectie van HIV-1-gag-expressing gemodificeerd vaccinia Ankara virus (MVA-gag) met een dosis van 106 plaque-vormende eenheden.
- Offer op dag 3 na de boost de gebooste muizen op door cervicale dislocatie en analyseer ze parallel met onbehandelde muizen.
- Oogst de LNs drainerend de quadriceps (iliacale, popliteale en inguinale) en de milt van versterkte en onbehandelde muizen. Verzamel bovendien de BM van de twee achterpoten van onbehandelde muizen en gebruik deze BM voor flowcytometerinstellingen en als positieve controle voor celcyclusanalyse(figuur 2).
OPMERKING: Genereer ChAd3-gag en MVA-gag vectoren zoals eerder beschreven12,15,16,17.
3. Isolatie van drainerende LN-, milt- en BM-cellen
- Isolatie van milt- en LN-cellen
- Plaats 5 ml volledig medium in elk van de twee buizen van 15 ml en houd ze op ijs, klaar om organen te verzamelen.
- Offer een volwassen muis op door cervicale dislocatie.
- Plaats de muis op zijn rug en steriliseer het huidoppervlak met 70% v/v ethanol.
- Om inguinale LIAN's te verzamelen, maakt u een longitudinale incisie van ~ 1 cm op de buik met een schaar en strekt u de incisie uit met de tang.
- Visualiseer inguinale LAN's op het interne oppervlak van de huid en oogst ze met de tang. Plaats de inguinale LC's in een van de twee buizen van 15 ml die in stap 3.1.1 zijn voorbereid.
- Om de milt te verzamelen, maakt u een peritoneale incisie met een schaar en verwijdert u de milt. Nadat u het omliggende bindweefsel hebt doorgesneden, plaatst u de milt in de tweede buis van 15 ml die is voorbereid in stap 3.1.1.
- Om iliacale LIAN's te verzamelen, verplaatst u de darmen opzij en visualiseert u iliacale LC's dicht bij de inferieure vena cava en verzamelt u ze vervolgens met behulp van de tang. Plaats de iliacale LIAN's in dezelfde buis met de inguinale LC's.
OPMERKING: Om voldoende LN-cellen voor kleuring te verkrijgen (zie rubriek 4), is het vaak nodig om popliteale, inguinale en iliacale LC's van één muis te poolen. Deze LIAN's draineren allemaal de quadriceps (de plaats van i.m. vaccinatie). Dit protocol gebruikt slechts één buis van 15 ml gepoolde LN's. - Om popliteale LNs te verzamelen, pak je de huid van de achterpoten vast en trek je deze voorzichtig naar beneden om de spieren te blootleggen. Plaats vervolgens de tang tussen de spieren onder het kniegewricht en verzamel de popliteale RN's. Plaats de popliteale LAN's in dezelfde buis met inguinale en iliacale LAN's.
OPMERKING: Zie opmerking na 3.1.7. - Plaats de milt in een celzeef van 70 μm in een kweekschaal van 60 mm gevuld met 5 ml volledig medium. Gebruik een zuiger van een spuit van 5 ml en pureer het orgaan voorzichtig totdat het volledig is uitgesplitst.
- Verwijder de zeef en breng de celsuspensie over op een schone buis van 15 ml.
- Voeg 5 ml volledig medium toe aan de kweekschaal en was de schaal en de zeef voorzichtig om ervoor te zorgen dat alle cellen zijn teruggevonden. Zwembad met de rest van de miltcel suspensie in de buis van 15 ml.
- Bereid voor de gepoolde inguinale, iliacale en popliteale LIAN's een eencellige suspensie volgens een procedure die vergelijkbaar is met die in de stappen 3.1.9 tot en met 3.1.11 voor de milt.
- Centrifugeer cellen bij 400 × g gedurende 10 minuten bij 4 °C. Gooi het supernatant weg en resuspend de celkorrels in PBS.
- Tel de cellen met een Neubauer-kamer met behulp van rode bloedcellysisbuffer en 0,04% v / v trypan blauw in PBS.
- Isolatie van BM-cellen
- Plaats 5 ml compleet medium in een buis van 15 ml en houd het op ijs, klaar voor het verzamelen van achterpoten.
- Offer een volwassen muis op door cervicale dislocatie.
- Steriliseer het huidoppervlak met 70% v/v ethanol.
- Maak een transversale incisie van ~ 1 cm op de ventrale huid met een schaar, pak de huid aan beide zijden van de snede stevig vast en trek zachtjes naar beneden om de spieren van de achterpoten te ontdekken.
- Om de huid van de achterkant van de achterpoten te verwijderen, houdt u de muis in rugligging, plaatst u de klem onder de knie en trekt u omhoog om de spieren bloot te leggen.
- Snijd de botten aan de twee uiteinden van één achterbeen: het bekken/heupgewricht en de enkel.
- Breng beide achterpoten over op de buis van 15 ml die in stap 3.2.1 is voorbereid. Houd de buis op ijs.
- Neem de achterpoten uit de buis van 15 ml en breng ze over op tissuepapier. Snijd de achterpoten net onder het kniegewricht om het scheenbeen te verwijderen. Ontleed het dijbeen en scheenbeen van de omliggende spieren, verwijder overtollig weefsel met een schaar en maak het vloeipapier nat.
- Knip de botuiteinden met een schaar om de binnenste mergbacht bloot te leggen. Steek het scheenbeen en het dijbeen in de BM-extractiebuis (zie bereiding in 3.2.9.1-3.2.9.218), met het breedste uiteinde aan de onderkant.
- Snijd een pipetpunt van 200 μL op de lijn net boven het uiteinde van de punt en op de lijn van 100 μL.
- Plaats het middelste deel in het bovenste, grotere deel van de punt en plaats dit in een microfugebuis van 1,5 ml.
- Draai de BM-extractiebuis gedurende 1 minuut op 800 × g.
- Gooi het bot weg en oefen de pellet krachtig opnieuw op in 1 ml volledig medium om eventuele clusters te verwijderen. Filter de celsuspensie door een filter van 70 μm dat op de bovenkant van een buis van 15 ml is geplaatst.
- Was de BM-extractiebuis tweemaal met telkens 1 ml volledig medium. Filtreer door een filter van 70 μm en pool het volume met de rest van de celsuspensie verkregen in stap 3.2.11.
OPMERKING: Een enkele buis van 15 ml bevat cellen van beide achterpoten van een muis. - Centrifugeer cellen bij 400 × g gedurende 10 minuten bij 4 °C. Gooi het supernatant weg en resuspend de celkorrel in PBS.
- Tel de cellen met een Neubauer-kamer met behulp van rode bloedcellysisbuffer en 0,04% v / v trypan blauw in PBS.
4. Kleuring van milt-, LN- en BM-cellen
- Verdeel celmonsters die moeten worden gekleurd in 3 subgroepen: celmonsters ter compensatie, inclusief BM-cellen van onbehandelde muizen die alleen worden gekleurd met Hoechst 33342 (voortaan Hoechst genoemd) en miltcellen van onbehandelde muizen om te worden gebruikt om een dode / levende celmix te bereiden voor compensatie van dode celkleurstof; positieve controle voor celcyclusanalyse, bestaande uit een BM-monster van onbehandelde muizen; en experimentele monsters met milt- en LN-monsters van onbehandelde en gevaccineerde muizen.
OPMERKING: Zorg ervoor dat er voldoende milt- en LN-cellen zijn voor analyse van voldoende aantallen gag-specifieke CD8 T-cellen. Het is vaak nodig om gepoolde miltcellen en gepoolde LN-cellen van 3 gevaccineerde muizen te gebruiken en twee of meer identieke monsters van gepoolde cellen te kleuren, elk met 3 × 106 cellen. Voeg identieke monsters samen bij de stap van Hoechst-kleuring. Op dezelfde manier kleuren gepoolde miltcellen en LN-cellen van 3 onbehandelde muizen en voegen identieke monsters aan het einde samen. Zet een onbevlekt monster van miltcellen van een onbehandelde muis opzij om te worden gebruikt voor instrument- en compensatie-instellingen. - Bereid dode / levende celmix voor dode celkleurstofcompensatie (deze mix van cellen wordt alleen gekleurd met de dode celkleurstof).
- Verwarm een waterbad op 65 °C.
- Neem een aliquot van miltcellen (~ 3 × 106).
- Breng de celsuspensie over in een microfugebuis, plaats deze gedurende 5 minuten in het waterbad bij 65 °C en plaats hem vervolgens onmiddellijk gedurende 10 minuten op ijs.
- Meng de warmtedode cellen met levende miltcellen (~ 3 × 106) in een verhouding van 1: 1 en breng de helft van het mengsel over naar een 96 goed ronde bodemplaat (~ 3 × 106 cellen / goed voor de dode celkleuringscontrole).
- Dode celkleuring van experimentele monsters, positieve controle voor celcyclusanalyse en dode /levende celmix
- Breng milt, LN, BM-cellen (3 × 106 cellen/put) en het mengsel van dode/levende cellen (punt 4.2) over in een 96-put ronde bodemplaat, volgens het kleuringsschema (stap 4.1), en centrifugeer bij 400 × g gedurende 3 minuten bij 4 °C.
- Resuspend elke celkorrel in 50 μL dode celkleurstof verdund in PBS en resuspend door 3 keer onmiddellijk op en neer te pipetteren.
- Incubeer gedurende 30 minuten bij 4 °C, beschermd tegen licht.
- Was cellen 2 keer met kleuringsbuffer; de eerste keer met 200 μL en de tweede keer met 250 μL. Centrifugeer voor elke wasbeurt de plaat bij 400 × g gedurende 3 minuten bij 4 °C.
- Gooi het supernatant weg en resuspend de celkorrel in 20 μL PBS.
- Membraancelkleuring met major histocompatibiliteitscomplex (MHC)-peptidemultimeren en mAbs.
- Rekening houdend met de benodigde volumes volgens het kleuringsschema (instellingen van de flowcytometer, tabel 1),bereidt u de volgende reagentia voor:
- Verdun mAb 2.4G2 in de kleuringsbuffer volgens de juiste verdunning (zie Materiaaltabel); Gebruik voor elk te bevlekten monster 10 μL van deze verdunning.
OPMERKING: 2,4G2 mAb blokkeert niet-antigeenspecifieke binding van immunoglobulinen aan de FcγIII- en FcγII-receptoren. - Verdun het H-2k(d) AMQMLKETI allophycocyanine (APC)-gelabelde tetrameer (Tetr-gag) in de kleuringsbuffer om de juiste verdunning te verkrijgen (zie Materialentabel); Gebruik voor elk te bevlekken monster 20 μL van deze verdunning.
- Bereid het antilichaammengsel voor door mAbs in de kleuringsbuffer te verdunnen volgens de juiste verdunning (zie Tabel met materialen) die eerder is bepaald in titratie-experimenten; Gebruik voor elk te bevlekten monster 20 μL van dit antilichaammengsel.
OPMERKING: Hier werden anti-CD3e peridinine chlorofyl eiwit (PerCP-Cy5.5) (kloon 145-2C11), anti-CD8a briljant ultraviolet (BUV805) (kloon 53-6.7) en anti-CD62L fycoerythrin cyanine7 (PECy7) (kloon MEL-14) gebruikt.
- Verdun mAb 2.4G2 in de kleuringsbuffer volgens de juiste verdunning (zie Materiaaltabel); Gebruik voor elk te bevlekten monster 10 μL van deze verdunning.
- Voeg 10 μL van de eerder verdunde 2,4G2 mAb (stap 4.4.1.1) toe en incubeer gedurende 10 minuten bij 4 °C, beschermd tegen licht.
- Voeg 20 μL van de eerder verdunde Tetr-gag APC (stap 4.4.1.2) en 10 μL H-2k(d) AMQMLKETI fycoerythrin (PE) pentameer (pent-gag) toe. Incubeer gedurende 15 minuten bij 4 °C, beschermd tegen licht.
- Voeg 20 μL van het eerder bereide antilichaammengsel (stap 4.4.1.3) toe en incubeer 15 min bij 4 °C, beschermd tegen licht.
OPMERKING: Het uiteindelijke volume is dus 80 μL per put (stap 4.3.5, stap 4.4.2 tot en met 4.4.4). - Was cellen met 200 μL kleurbuffer. Centrifugeer bij 400 × g gedurende 5 minuten bij 4 °C.
- Resuspend de celkorrel in 250 μL kleuringsbuffer en breng de celsuspensie over naar buizen van 5 ml. Voeg 1 ml vlekbuffer toe aan de buis en centrifugeer bij 400 × g gedurende 5 minuten bij 4 °C.
- Neem het aliquot van BM-cellen (3 × 106 cellen) (zie lijst van celmonsters, rubriek 4.1) om het Hoechst-kanaal te compenseren (Hoechst 33342 wordt geëxciteerd door een ultraviolette laser (flowcytometerinstellingen (tabel 2)) en breng de celsuspensie over in een buis van 5 ml. Voeg 1 ml kleurbuffer toe aan de buis en centrifugeer 400 × g gedurende 5 minuten bij 4 °C.
- Rekening houdend met de benodigde volumes volgens het kleuringsschema (instellingen van de flowcytometer, tabel 1),bereidt u de volgende reagentia voor:
5. Fixatie/permeabilisatie
- Bereid verse fixatie/permeabilisatiebuffer voor door 1 deel fixatie/permeabilisatieconcentraat te verdunnen met 3 delen fixatie/permeabilisatieverdunningsmiddel, volgens de instructies van de fabrikant.
- Gooi het supernatant weg en puls vortex de monsters om de pellet volledig uit te splitsen.
- Voeg 1 ml van de vers bereide fixatie-/permeabilisatiebuffer toe aan elke buis, inclusief een buis met niet-gekleurde miltcellen (3 x 106, zie lijst met celmonsters, rubriek 4.1) en vortex.
- Incubeer gedurende 16 uur bij 4 °C.
OPMERKING: Het protocol kan hier worden gepauzeerd.
6. Intracellulaire kleuring
- Ki67 kleuring
- Bereid verse permeabilisatiebuffer 1x door permeabilisatiebuffer 10x te verdunnen met gedestilleerd water, volgens de instructies van de fabrikant. Voor gebruik moet de permeabilisatiebuffer 1x door een filter van 0,45 μm worden gefilterd om aggregaten te elimineren.
- Verdun mAb Ki67 fluoresceïne-isothiocyanaat (FITC) (kloon SolA15) in permeabilisatiebuffer 1x (zie Tabel met materialen), zoals eerder bepaald in titratie-experimenten (eindvolume van 100 μL per monster).
- Voeg 3 ml permeabilisatiebuffer 1x toe aan elke buis en centrifugeer bij 400 × g gedurende 5 minuten bij kamertemperatuur (RT).
- Gooi het supernatant weg en herhaal stap 6.1.3.
- Gooi het supernatant weg en resuspend de celkorrel in 100 μL eerder verdunde mAb Ki67 FITC (stap 6.1.2).
- Incubeer gedurende 30 minuten bij RT, beschermd tegen licht.
- Was cellen 2 keer met 4 ml permeabilisatiebuffer 1x. Voor elke wascentrifuge bij 400 × g gedurende 5 minuten bij RT.
- Resuspend de celkorrel in PBS rekening houdend met de volgende volumes: 350 μL PBS voor de monsters die rechtstreeks bij de flowcytometer moeten worden verkregen; 250 μL PBS voor de monsters die kort voor de flowcytometrie met Hoechst moeten worden geïncubeerd (rubriek 6.2).
- DNA-kleuring
- Voeg 250 μL van 4 μg/ml Hoechst in PBS toe aan elk monster (de uiteindelijke concentratie hoechst is 2 μg/ml).
OPMERKING: Indien twee of meer identieke monsters van 250 μL in PBS werden bereid, voeg ze dan in deze stap samen en voeg een gelijk volume van 4 μg/ml Hoechst-oplossing toe aan PBS (de uiteindelijke concentratie van Hoechst is 2 μg/ml). Het aantal cellen heeft een grote invloed op de DNA-kleuringsstap. Gebruik hetzelfde celnummer in elk monster. Houd er rekening mee dat zelfs een licht verminderd celgetal (bijvoorbeeld als gevolg van celverlies in eerdere wasstappen) resulteert in een hogere Hoechst-binding aan DNA en een hogere Hoechst-intensiteit. - Incubeer gedurende 15 minuten bij RT, beschermd tegen licht.
- Centrifugeer de monsters bij 400 × g gedurende 5 minuten bij RT.
- Resuspend de celkorrel in 350 μL PBS.
- Voeg 250 μL van 4 μg/ml Hoechst in PBS toe aan elk monster (de uiteindelijke concentratie hoechst is 2 μg/ml).
7. Bereiding van monsters van compensatieparels
- Bereid 5 μL van het antilichaam door mAb in de kleuringsbuffer op de juiste manier te verdunnen.
OPMERKING: Bereid voor elke fluorochroom-geconjugeerde mAb die in het experiment wordt gebruikt, het bijbehorende compensatieparelmonster voor. - Vortex Negatieve Controle en Anti-Rat/Hamster Ig,κ Comp Kralen voor gebruik.
- Breng voor elk monster één druppel (~ 20 μL) negatieve controlecompkralen en één druppel anti-rat / hamster Ig, k CompBeads in.
- Voeg 5 μL van het voorverwijde antilichaam (stap 7.1) toe aan de buis en pipetteer op en neer.
- Incubeer gedurende 15 minuten bij 4 °C, beschermd tegen licht.
- Was monsters met 2 ml vlekbuffer. Centrifugeer bij 400 × g gedurende 5 minuten bij 4 °C.
- Gooi het supernatant weg en resuspend de pellet door 500 μL PBS toe te voegen aan elke buis en vortex.
8. Instrument- en compensatie-instelling en experimentele monsterverwerving bij de flowcytometer
OPMERKING: Raadpleeg de instellingen van de flowcytometer(tabel 2)voor de configuratie van de cytometer.
- Algemene instrument- en compensatie-opstelling
- Open de software voor het verkrijgen van voorbeelden (zie Tabel met materialen) en maak een nieuw experiment door op Nieuw experiment te klikken in het lintgedeelte van de werkruimte en Nieuw leeg experimentte selecteren.
- Dubbelklik op het gemaakte experiment om het te openen.
- Klik in het venster Cytometerinstellingen op Parameters en selecteer alle kanalen (bijv. PE, APC, enz.) die in het kleuringspaneel worden gebruikt, inclusief forward scatter (FSC) en side scatter (SSC) parameters.
- Selecteer lineaire schaal als een Hoechst-parameter door de logschaal uit te schakelen en controleer de breedte (W) van de spanningspuls voor FCS, SSC en Hoechst.
OPMERKING: Alle parameters worden standaard weergegeven in logaritmische (log) schaal, behalve FSC en SSC die in lineaire schaal zijn. Alle parameters worden geanalyseerd door het gebied (A) en de hoogte (H) van de spanningspuls. - Maak op het globale werkbladeen puntplot met FSC-A op de x-as en SSC-A op de y-as.
- Voer het niet-gekleurde miltvoorbeeld uit door te klikken op Gegevens verkrijgen op het acquisitiedashboard.
- Stel de juiste FSC- en SSC-instellingen in om de cellen te visualiseren door de spanningswaarden in de sectie Parameters te wijzigen en maak een poort om alle cellen te selecteren die worden weergegeven in de FSC-A/SSC-A-puntenplot door op Polygon Gate te klikken op de werkruimtewerkbalk van het globale werkblad.
- Geef de gated cellen weer in histogrammen met elke fluorescentieparameter op de x-as.
- Voer onbevlekte en volledig gekleurde miltmonsters uit om de fluorescentiedetector (PMT) aan te passen om een duidelijke scheiding te hebben tussen negatieve en positieve signalen van de gekleurde cellen voor elke fluorescentieparameter.
- Als u compensatie-instellingen wilt uitvoeren, klikt u op Experimenteren op het lint van de werkruimte en selecteert u onder Het gedeelte Compensatie-instellingen de optie Compensatiebesturingselementen maken. Verwijder het vinkje bij Inclusief onbevlekte bedieningsbuis/put en klik op OK.
OPMERKING: Deze bewerking resulteert in het maken van een exemplaar met de naam Compensatiebesturingselementen en een normaal werkblad met verschillende bladen die overeenkomen met elke geselecteerde parameter. - Voer een voorbeeld uit van compensatiekralen (zie rubriek 7); stel de juiste FSC- en SSC-instellingen in om de kralen te visualiseren door de spanningswaarden en de acquisitiedrempel van 5.000 op FSC-parameters in het cytometervenster te wijzigen.
- Pas de P1-poort op de kralenpopulatie aan en controleer of de positieve en negatieve pieken beide zichtbaar zijn op de x-as. Herhaal deze bewerking voor elk monster van compensatiekralen en noteer ten slotte elk voorbeeldbestand door op Recordgegevens te klikken op het Acquisitiedashboard (noteer ten minste 5.000 gebeurtenissen voor elk monster).
- Stel voor elk geregistreerd kraalmonster de P2- en P3-poorten in op respectievelijk de positieve en negatieve pieken.
- Voer de celmonsters uit ter compensatie (zie stap 4.2 en 4.4.7 en rubrieken 5 en 6). Wijzig de FSC- en SSC-spanningen en de drempelwaarde om de cellen te visualiseren, pas de P1-poort aan en neem ten slotte elk monsterbestand op (registreer ten minste 10.000 gebeurtenissen). Stel de P2- en P3-poorten in op respectievelijk de positieve en negatieve pieken.
OPMERKING: Gebruik voor de compensatie van het Hoechst-kanaal de G0/G1 als de negatieve piek (P3) en de G2/M als de positieve (P2). - Klik op Experimenteren in het lintgedeelte van de werkruimte en selecteer in het gedeelte Compensatie instellen de optie Compensatie berekenen.
- Geef de gemaakte compensatie-instelling een naam, koppel deze en sla deze op in het huidige experiment.
- Experimentele monsterverwerving
- Open een exemplaar door op Nieuw exemplaar op de browserwerkbalk te klikken en maak de gatingstrategie in het algemene werkblad.
OPMERKING: De gatingstrategie van monsterverwerving is vergelijkbaar met die van steekproefanalyse, beschreven in figuur 3 en rubriek 9. - Alle gebeurtenispopulatie weergeven in een histogram met CD3-A op de x-as. Maak een intervalpoort om alleen deCD3 +-cellen te selecteren.
- Selecteer ophet acquisitiedashboard de optie opslaggate als Alle gebeurtenissen voor LN-voorbeelden en Alle gebeurtenissen of CD3+-cellen voor miltmonsters.
- Voer de experimentele monsters op lage snelheid uit en neem ten slotte alle bestanden op en zorg ervoor dat u ten minste 100-200 antigeenspecifieke CD8 T-cellen verzamelt voor elk monster van de gevaccineerde muizen.
OPMERKING: De bestandsgrootte van experimentele monsters is meestal groot (30-120 MB), vooral wanneer de frequentie van antigeenspecifieke CD8 T-cellen laag is. Daarom moeten grote aantallen gebeurtenissen (> 1 × 106) worden verzameld om ten minste 100-200 antigeenspecifieke CD8 T-cellen te registreren. Grote bestanden kunnen het daaropvolgende gegevensanalyseproces vertragen. De verwerving van alleen CD3+ cellen in miltmonsters (zie stap 8.2.2 hierboven) is nuttig om de bestandsgrootte kleiner te houden. - Voer de positieve controle uit en registreer deze voor celcyclusanalyse, d.w.z. BM-monster van onbehandelde muizen.
- Open een exemplaar door op Nieuw exemplaar op de browserwerkbalk te klikken en maak de gatingstrategie in het algemene werkblad.
9. Data-analyse
- Open de software (zie Tabel met materialen)en maak verschillende groepen die overeenkomen met de verschillende organen die moeten worden geanalyseerd door te klikken op Groep maken in het lintgedeelte van de werkruimte (d.w.z. groep "a-LAN's" maken; "b-milt"; "c-BM").
OPMERKING: Nieuw gemaakte groepen verschijnen in de groepslijst, terwijl de groep "Compensatie" automatisch wordt gegenereerd door de software. - Open het venster Groep wijzigen door te dubbelklikken op de groepsnaam en controleer of de nieuw gemaakte groepen zijn gesynchroniseerd. Als dit niet het geval is, plaatst u een vinkje op de functie Gesynchroniseerd.
- Sleep elk FCS-bestand naar de bijbehorende groep.
- Maak de gating-strategie die begint met de groep "a-LNs".
- Dubbelklik op het volledig gekleurde monster in de groep om het grafiekvenster te openen; x- en y-as zijn gelabeld zoals in de fcs-bestanden (zie de instellingen van de flowcytometer, tabel 2).
- Toon de totale gebeurtenissen die voor dit monster zijn verkregen in een dot plot met DNA-A op de x-as en DNA-W op de y-as.
- Selecteer alleen de populatie met één cel door op Rechthoek te klikken in het gedeelte gating tool van het grafiekvenster.
OPMERKING: Afzonderlijke cellen hebben DNA-A-waarden als volgt: 2N (laag): tussen 2N en 4N (intermediair), of gelijk aan 4N (hoog), terwijl DNA-W-waarden identiek zijn voor alle cellen (stap 1 van figuur 3). - Dubbelklik in het midden van de rechthoekige poort om enkele cellen weer te geven in een dot plot met FSC-A parameter op de x-as en dode cel kleurstof op de y-as.
- Selecteer alleen de levende celpopulatie door op Polygoon te klikken in het gedeelte gating tool van het grafiekvenster. Levende cellen zijn negatief voor de dode celkleurstof (stap 2 van figuur 3).
- Dubbelklik in het midden van de veelhoekige poort om de cellen weer te geven in een dot plot met FSC-A parameter op de x-as en SSC-A parameter op de y-as.
- Klik op Rechthoeken maak een "ontspannen" poort om alle afzonderlijke levende cellen in die grafiek12 op te nemen (stap 3 van figuur 3).
- Dubbelklik in het midden van de "ontspannen" poort om de cellen weer te geven in een dot plot met CD3 op de x-as en CD8 op de y-as.
- Selecteer de CD3+CD8+ cellen door te klikken op Polygoon (stap 4 van Figuur 3).
- Dubbelklik in het midden van de CD3+CD8+ gate om de cellen in een dot plot weer te geven met Tetr-gag op de x-as en Pent-gag op de y-as.
- Selecteer de antigeenspecifieke CD8 T-cellen (positief voor zowel Tetr-gag als Pent-gag) door op Polygon te klikken (stap 5 van figuur 3).
- Dubbelklik in het midden van de gag-specifieke poort om de cellen weer te geven in een dot plot met DNA-A op de x-as en Ki67 op de y-as (Figuur 4).
- Selecteer de cellen in de verschillende celcyclusfasen door op Quad te klikken in het gedeelte gating tool van het grafiekvenster.
OPMERKING: Cel in G0-fase zijn Ki67neg-DNA lage cellen (kwadrant linksonder); cellen in G1 zijn Ki67pos-DNA laag (kwadrant linksboven); cellen in S-G2/Mzijn Ki67pos-DNA intermediair/hoog (kwadrant rechtsboven) (Figuur 4). - Kopieer de gatingstrategie die in één voorbeeld is gemaakt naar de overeenkomstige groep om de poorten toe te passen op alle voorbeelden van de groep.
- Herhaal stap 9.5 tot 9.18 voor de "a-LN-groep".
- Controleer of alle poorten geschikt zijn voor elk monster van de groep "b-milt". Om de celcyclus tussen de BM-cellen (positieve controle) te analyseren, klikt u in het midden van de "ontspannen" poort om de cellen weer te geven in een dot plot met DNA-A op de x-as en Ki67 op de y-as.
- Controleer of alle poorten geschikt zijn voor elk monster van de 3 groepen (d.w.z. voor cellen uit milt, LN en BM).
OPMERKING: Single cell population gate (stap 9.7) en Quad gate for cell cycle (step 9.17) kunnen verschillende poortcoördinaten hebben in verschillende monsters, voornamelijk als gevolg van de mogelijke kleine verschillen van de Hoechst-kleurstofintensiteit tussen monsters (paragraaf 6.2). Om deze reden kan het nodig zijn om de Single cell population gate en de Quad gates voor celcyclus in elk monster te wijzigen. Dit wordt als volgt gedaan: dubbelklik op de groepsnaam en verwijder de synchronisatie uit de groepseigenschappen. Deze bewerking maakt het mogelijk om de poorten in één monster te wijzigen zonder dezelfde poorten in alle andere monsters van de groep te wijzigen. Pas na het verwijderen van de synchronisatie de poorten waar nodig aan. - Als u de resultaten van deze analyse wilt visualiseren, klikt u op Lay-outeditor in het lintgedeelte van de werkruimte om deze te openen. Sleep elke poort van de gatingstrategie in het voorbeeldvenster naar de lay-outeditor en plaats de plots volgens de volgorde van de gatingstrategie. Wijzig indien nodig het grafiektype door te dubbelklikken op de bijbehorende plot in de lay-out en het juiste type te selecteren in het venster Grafiekdefinitie.
- Klik op de groep en herhaal functies op het lay-outlint om de resultaten van elk orgel te visualiseren en verschillende monsters te vergelijken.
Representative Results
De celcyclusfasen van cellen uit milt, LNs en BM van Balb / c-muizen werden geanalyseerd met behulp van de fluorescerende DNA-kleurstof, Hoechst en een anti-Ki67 mAb, volgens het protocol samengevat in figuur 1. Deze kleuring maakte de differentiatie van cellen in de volgende fasen van de celcyclus mogelijk: G0 (Ki67neg, met 2N DNA gedefinieerd als DNAlow), G1 (Ki67pos, DNAlow) en S-G2/ M (Ki67pos, met een DNA-gehalte tussen 2N en 4N, of gelijk aan 4N DNA gedefinieerd als DNAintermediaat / hoog).
We voerden eerst celcyclusanalyse van BM-cellen uit om eerder gepubliceerde resultaten13,14 te reproduceren en analyseerden vervolgens de cellen van belang, d.w.z. CD8 T-cellen. Figuur 2 toont een typisch voorbeeld van celcyclusanalyse van BM-cellen (figuur 2A). Het protocol leverde een lage variatiecoëfficiënt (CV) op van G0/G1 en G2/M DNA-pieken, wat de uitstekende kwaliteit van de DNA-kleuring aangeeft (Figuur 2B, met een voorbeeld met CV < 2.5; CV werd altijd < 5 in alle experimenten).
Vervolgens pasten we hetzelfde protocol toe op antigeenspecifieke CD8 T-cellen van gevaccineerde muizen. BALB /c-muizen werden gevaccineerd tegen de antigeen gag van HIV-1 door Chad3-gag te gebruiken voor priming en MVA-gag voor boosting, beide ontworpen om HIV-1 gag te dragen. Op dag (d) 3 post-boost analyseerden we de frequentie van gag-specifieke CD8 T-cellen uit de milt en drainerende LNs. We hebben geprofiteerd van de nieuw gedefinieerde gating-strategie voor T-cellen in de vroege fase van immuunrespons, die in tegenstelling tot de conventionele strategie geschikt is voor het detecteren van sterk geactiveerde antigeen-reagerende CD8 T-cellen12. We hebben de nieuwe strategie in vijf opeenvolgende stappen uitgevoerd. In stap 1 sloten we doubletten of aggregaten uit door DNA-A / -W gate, en in stap 2 identificeerden we levende cellen door uitsluiting van dode celmarkers. In stap 3 identificeerden we de populatie van belang met behulp van een niet-conventionele "ontspannen" FSC-A / SSC-A-poort (Figuur 3A) in plaats van de canonieke smalle lymfocytenpoort12. Na gating op CD3+CD8+ cellen (stap 4 van figuur 3A),identificeerden we gag-specifieke CD8 T-cellen met behulp van twee verschillende MHC-multimeren, d.w.z. Pent-gag en Tetr-gag (stap 5 van figuur 3A). We gebruikten twee multimeren in plaats van één om de gevoeligheid van gag-specifieke CD8 T-celdetectie bij gevaccineerde muizen te verbeteren, zonder de kleurachtergrond bij onbehandelde muizen te vergroten(Figuur 3B en C,stap 5). Zo onderscheidden we met succes onbehandelde muizen (0,00% en 0,00% antigeenspecifieke CD8 T-cellen in respectievelijk LNs en milt) van gevaccineerde muizen (0,46% en 0,29% antigeenspecifieke CD8 T-cellen in respectievelijk LNs en milt, figuur 3B en C).
Met name het protocol stelde ons in staat om een extreem lage achtergrond te hebben in de antigeenspecifieke CD8 T-celpoort van LAN's en milt van onbehandelde muizen (meestal 0,00% en maximaal 0,02%). De vergelijking van gag-specifieke en niet gag-specifieke FSC-A / SSC-A-plots toonde aan dat de gag-specifieke cellen een hoge SSC-A en FSC-A(Figuur 3D)hadden, wat de noodzaak bevestigt om een "ontspannen" FSC-A / SSC-A-poort te gebruiken om deze cellen te vangen. Vervolgens evalueerden we de percentages gag-specifieke CD8 T-cellen in verschillende celcyclusfasen(figuur 4A). We ontdekten dat gag-specifieke CD8 T-cellen in de milt en zelfs meer in de drainerende LNs een hoog percentage cellen in S-G2/ M-fasen bevatten op dag 3 na de boost (respectievelijk 18,60% en 33,52%).
Bovendien vonden we dat gag-specifieke CD8 T-cellen in S-G2/ M-fasen een hoge FSC-A en SSC-A hadden, wanneer ze over de totale CD8 T-cellen van hetzelfde orgaan werden gelegd(figuur 4B). CD62L-expressie door gag-specifieke CD8 T-cellen was laag, zoals verwacht voor geactiveerde T-cellen, behalve een paar cellen in G0 in de LNs (Figuur 4C). Al met al bevestigden deze resultaten dat de "ontspannen" poort (stap 3 van figuur 3A, B en C)alle prolifererende antigeenspecifieke CD8 T-cellen12moest omvatten. Het protocol was uiterst waardevol voor een "snapshot" evaluatie van celcyclusfasen van antigeenspecifieke CD8 T-cellen op het moment van analyse en van CD62L-expressie door cellen in verschillende celcyclusfasen.

Figuur 1: Schema van het protocol voor celcyclusanalyse van antigeenspecifieke CD8 T-cellen. Klik hier om een grotere versie van deze figuur te bekijken.

Figuur 2: Celcyclusanalyse van BM-cellen. BM-cellen van onbehandelde Balb / c-muizen werden gekleurd en geanalyseerd door flowcytometrie. (A) Voorbeeld van gating strategie. We gated op enkele cellen in de DNA-A /-W plot (links) en vervolgens op levende cellen door dode celkleurstof uitsluiting (midden). Vervolgens werd voor alle BM-cellen een "ontspannen" FSC-A/SSC-A poort gebruikt (rechts). (B) Voorbeeld van celcyclusanalyse van BM-cellen (links). We gebruikten een combinatie van Ki67 en DNA-kleuring om cellen in de volgende fasen van de celcyclus te identificeren: G0 (kwadrant linksonder, Ki67neg-DNAlow-cellen), G1 (kwadrant linksboven, Ki67pos-DNAlow), S-G2/ M (kwadrant rechtsboven, Ki67pos-DNAintermediate / hoog). Fluorescentie Minus One (FMO) controle van Ki67 mAb (midden) en DNA histogram (rechts) worden getoond. In de DNA-histogramplot komen de linker- en rechterpoort overeen met respectievelijk de G0/ G1 en de G2/ M DNA-piek, en de getallen vertegenwoordigen de variatiecoëfficiënten (CV) van elke piek. In alle andere percelen vertegenwoordigen de getallen celpercentages in de aangegeven poorten. De figuur toont 1 representatief experiment op 5. In elk experiment analyseerden we gepoolde BM-cellen van 3 muizen. Klik hier om een grotere versie van deze figuur te bekijken.

Figuur 3: Analyse van antigeenspecifieke CD8 T-cellen uit LY's en milt. Balb/c-muizen werden intramusculair (i.m.) geprimed met Chad3-gag en geboost i.m. met MVA-gag. Op dag 3 na de boost werden drainerende LN- en miltcellen van gevaccineerde en onbehandelde controlemuizen gekleurd en geanalyseerd met behulp van flowcytometrie. (A) Schema van de gating-strategie in vijf stappen om afzonderlijke cellen te identificeren (stap 1); levende cellen (stap 2); lymfocyten (stap 3); CD8 T-cellen (stap 4); en gag-specifieke cellen (stap 5). (B-C) Voorbeeld van plots: analyse van cellen van (B) LY's en (C) milt van onbehandelde (boven) en gevaccineerde (onderste) muizen. We identificeerden enkele cellen op de DNA-A / -W plot in stap 1. Vervolgens selecteerden we in stap 2 levende cellen door uitsluiting van dode celkleurstof. In stap 3 gebruikten we een niet-canonieke "ontspannen" poort voor lymfocyten. In stap 4 identificeerden we CD8 T-cellen door hun dubbele expressie van CD3 en CD8. Vervolgens identificeerden we gag-specifieke cellen en niet gag-specifiek in stap 5, op basis van hun vermogen om respectievelijk fluorochroom-gelabeld H-2kd-gag-Pentamer (Pent-gag) en H-2kd-gag-Tetramer (Tetr-gag) te binden, of niet. (D) FSC-A/SSC-A-profielen van gag-specifieke (blauwe) en niet gag-specifieke (grijze) cellen na gating zoals hierboven beschreven. Getallen vertegenwoordigen celpercentages in de aangegeven poorten. De figuur toont 1 representatief experiment op 5. In elk experiment analyseerden we gepoolde milt- en gepoolde LN-cellen van 3 gevaccineerde muizen en 3 onbehandelde muizen. Klik hier om een grotere versie van deze figuur te bekijken.

Figuur 4: Celcyclusanalyse van antigeenspecifieke CD8 T-cellen. Muizen werden gevaccineerd zoals in figuur 3 en celcyclusanalyse van gag-specifieke cellen werd uitgevoerd op dag 3 na de boost, na gating in 5 stappen zoals in figuur 3. (A)Voorbeeld van celcyclusanalyse van gag-specifieke CD8 T-cellen van LNs (boven) en milt (onder) van gevaccineerde muizen. Celcyclusfasen werden geïdentificeerd zoals in figuur 2B. De panelen vertegenwoordigen cellen in G0,in G1en in S-G2/M (links) en Fluorescentie Minus One (FMO) controle van Ki67 mAb (rechts). Getallen vertegenwoordigen celpercentages in de aangegeven poorten. (B) FSC-A/SSC-A puntplots met gag-specifieke CD8 T-cellen in S-G2/M-fasen (in rood) bedekt met totale CD3+CD8+ T-cellen (in grijs) van LNs (boven) en milt (onder) van gevaccineerde muizen. (C) Offset histogrammen met CD62L-expressie door gag-specifieke CD8 T-cellen in G0 (groen), in G1 (blauw) en in S-G2/M (rood) van LNs (boven) en milt (onder) van gevaccineerde muizen. De y-assen geven een genormaliseerd aantal gebeurtenissen aan. De figuur toont 1 representatief voorbeeld van 5 onafhankelijke experimenten met in totaal 15 muizen. Klik hier om een grotere versie van deze figuur te bekijken.
Aanvullend materiaal: Instellingen van de flowcytometer. Klik hier om dit bestand te downloaden.
Discussion
Hoewel T-cel klonale expansie intensief is bestudeerd, blijven sommige aspecten onbekend, vooral omdat de beschikbare hulpmiddelen om het te onderzoeken weinig zijn en hun eigen nadelen hebben. Vanuit dit perspectief hebben we een zeer gevoelige flowcytometrische methode opgezet om de celcyclus van antigeenspecifieke CD8 T-cellen op vroege tijdstippen na vaccinatie in een muismodel te analyseren. Het protocol is gebaseerd op een combinatie van Ki67 en DNA-kleuring, die eerder werd gebruikt om de celcyclus van BM-hematopoëtische cellen bij muizen te analyseren13,14. Om het protocol aan te passen aan antigeenspecifieke CD8 T-cellen, moesten we rekening houden met een paar kritieke kwesties, waaronder de keuze van de DNA-kleurstof, de juiste omstandigheden om vergelijkbare DNA-kleuring over verschillende monsters te verkrijgen en de gating-strategie voor data-analyse.
Veel kleurstoffen zijn beschikbaar voor DNA-kleuring, waaronder propidiumjodide en 7-aminoactinomycine D; we kozen voor Hoechst omdat het compatibel was met membraankleuring en het milde fixatie / permeabilisatieprotocol dat vereist is voor Ki67-kleuring. Tegelijkertijd stelde het kleuren met Hoechst ons in staat om een DNA-histogram van uitstekende kwaliteit te verkrijgen, d.w.z. de G0/ G1 en G2/ M DNA-pieken hadden een veel lagere variatiecoëfficiënt (CV) dan DNA-pieken die gewoonlijk worden verkregen met andere DNA-kleurstoffen, bijvoorbeeld DRAQ519. Inderdaad, Hoechst kan DNA kleuren, zelfs in levende cellen20.
Sommige strategieën werden gebruikt om de fluctuatie in Hoechst-intensiteit in verschillende monsters van hetzelfde experiment te voorkomen. Hoechstkleuring werd uitgevoerd vlak voor de monsterverwerving bij de flowcytometer om de afname van de kleurstofintensiteit in de loop van de tijd te minimaliseren. Voor degenen die geïnteresseerd zijn in het reproduceren van het protocol in grote experimenten met talloze monsters, raden we aan om Hoechst-kleuring op een paar monsters tegelijk uit te voeren. Een ander nadeel is dat de intensiteit van Hoechst sterk kan worden beïnvloed door het celgetal tijdens de incubatie met de kleurstof. Om deze reden raden we ten zeerste aan om altijd hetzelfde aantal cellen en hetzelfde volume per monster te gebruiken voor DNA-kleuring. Als een groot aantal cellen nodig is voor acquisitie bij de flowcytometer, raden we aan om twee of meer identieke monsters te bereiden en ze vervolgens samen te voegen net voor de Hoechst-kleurstap.
Een belangrijk punt van het protocol is de gating-strategie voor data-analyse. We hebben onlangs een nieuwe strategie gepubliceerd voor T-celanalyse op vroege tijdstippen van de immuunrespons, waardoor we de gevoeligheid van detectie van antigeenspecifieke T-cellen konden verhogen12. We hebben deze strategie als volgt toegepast op de gegevens die hier worden weergegeven. Ten eerste hebben we celaggregaten uitgesloten in de DNA-A/W plot. Ten tweede hebben we, na het verwijderen van dode cellen, een vrij grote lymfocytenpoort gebruikt in het FSC / SSC-perceel ("ontspannen poort"). Door deze strategie waren we in staat om sterk geactiveerde antigeenspecifieke CD8 T-cellen op te nemen in S-G2/ M die meestal worden gemist door de huidige gating-strategieën, omdat deze cellen een hoge FSC-A en SSC-A hebben. Samenvattend vormt de data-analyse een cruciaal onderdeel van de methode, wat essentieel is om een gevoelige detectie van geactiveerde / prolifererende antigeenspecifieke T-cellen te verkrijgen.
De methode voorkomt de mogelijkheid van het missen van kritieke T-celgegevens in vroege fasen van immuunrespons en opent nieuwe perspectieven voor T-cel immunomonitoring. Een toekomstige verbetering zou kunnen zijn om kleuring voor fosfo-histon 3 op te nemen die differentiatie tussen G2 en M21mogelijk zou maken. Een huidige beperking is dat cellen moeten worden gefixeerd en gepermeabiliseerd om te kleuren voor de nucleaire marker, Ki67. Cellen kunnen dus niet worden gebruikt voor andere soorten analyses, zoals sorteren en daaropvolgende functionele analyse. Bovendien interfereren DNA-kleurstoffen, waaronder Hoechst, meestal met de genomische DNA-analyse en zijn ze niet geschikt voor dit type evaluatie. Identificatie van membraanmarkers die correleren met verschillende celcyclusfasen en die op levende cellen kunnen worden gekleurd, kan deze beperking overwinnen. Kortom, de methode heeft een groot potentieel voor de evaluatie van geactiveerde / prolifererende T-cellen in verschillende contexten, zoals vaccinatie, infectie, immunogemedieerde ziekten en immunotherapie.
Disclosures
A. Folgori en S. Capone zijn werknemers van Reithera Srl. A. Nicosia wordt genoemd uitvinder op patentaanvraag WO 2005071093 (A3) "Chimpansee adenovirus vaccindragers." De andere auteurs hebben niets te onthullen.
Acknowledgments
Dit werk werd ondersteund door Reithera, door MIUR-project 2017K55HLC_006 en door 5 × 1000 subsidie van Associazione Italiana Ricerca sul Cancro (AIRC). Het volgende tetrameer werd verkregen via de NIH Tetramer Facility: APC-geconjugeerde H-2K (d) HIV-gag 197-205 AMQMLKETI.
Materials
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
References
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- Mouse phenotype. , Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020).
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Tags
Immunologie en infectie antigeen-specifieke CD8 T-cellen celcyclus Ki67 DNA-kleurstof flowcytometrie milt lymfeklieren muisErratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021.
Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
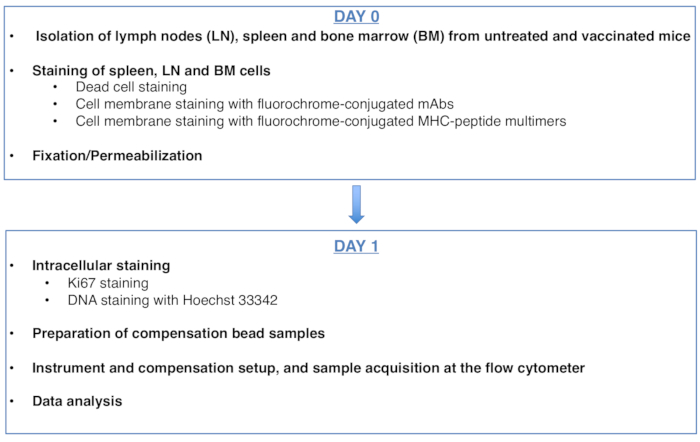
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:
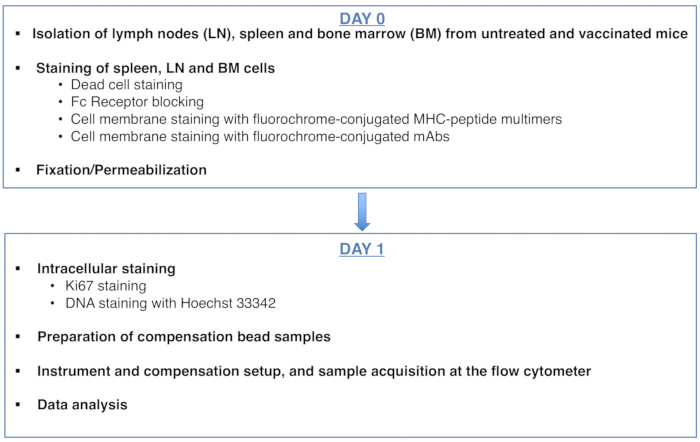
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.



