ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Kloningsudvidelse er et centralt element i antigenspecifik T-cellerespons. Imidlertid er cellecyklussen af antigenoptalende T-celler blevet dårligt undersøgt, dels på grund af tekniske begrænsninger. Vi beskriver en flow cytometrisk metode til at analysere clonally ekspanderende antigen-specifikke CD8 T-celler i milt og lymfeknuder af vaccinerede mus.
Abstract
Cellecyklussen af antigenspecifikke T-celler in vivo er blevet undersøgt ved hjælp af nogle få metoder, som alle har nogle begrænsninger. Bromodeoxyuridin (BrdU) markerer celler, der er i eller for nylig afsluttet S-fase, og carboxyfluorescein succinimidyl ester (CFSE) registrerer datterceller efter division. Disse farvestoffer tillader dog ikke identifikation af cellecyklusfasen på analysetidspunktet. En alternativ tilgang er at udnytte Ki67, en markør, der er stærkt udtrykt af celler i alle faser af cellecyklussen undtagen den hvilende fase G0. Desværre tillader Ki67 ikke yderligere differentiering, da det ikke adskiller celler i S-fase, der er forpligtet til mitose fra dem i G1, der kan forblive i denne fase, fortsætte ind i cykling eller flytte ind i G0.
Her beskriver vi en flowcytometrisk metode til at fange et "øjebliksbillede" af T-celler i forskellige cellecyklusfaser i mus sekundære lymfoidorganer. Metoden kombinerer Ki67 og DNA farvning med store histokompatibilitet kompleks (MHC)-peptid-multimer farvning og en innovativ gating strategi, så vi med succes kan skelne mellem antigen-specifikke CD8 T-celler i G0, i G1 og i S-G2/ M faser af cellecyklus i milten og dræne lymfeknuder af mus efter vaccination med virale vektorer transporterer modellen antigen gag af human immundefekt virus (HIV)-1.
Kritiske trin i metoden var valget af DNA-farvestof og gating strategi for at øge analysen følsomhed og til at omfatte stærkt aktiveret / udbrede antigen-specifikke T-celler, der ville have været savnet af de nuværende kriterier for analyse. DNA-farvestoffet, Hoechst 33342, gjorde det muligt for os at opnå en forskelsbehandling af G0/G1 og G2/M DNA-toppe af høj kvalitet, samtidig med at vi bevarede membran og intracellulære farvninger. Metoden har et stort potentiale til at øge kendskabet til T-cellerespons in vivo og til at forbedre immunovervågningsanalysen.
Introduction
Naive T-celler gennemgår kloniske ekspansion og differentiering ved antigen-priming. Differentierede T-celler viser effektorfunktioner, der er afgørende for antigenfrigang og til vedligeholdelse af antigenspecifik hukommelse, som er nøglen til langvarig beskyttelse. Under de første trin i den primære respons, naiv T celle interaktion med antigen-præsentere celler (APC'er) inden for specialiserede nicher i lymfoide organer er afgørende for at fremkalde den enorme T-celle spredning, der karakteriserer kloniske ekspansion fase1,2,3. T-celle-APC interaktion er fint reguleret af koncentration og vedholdenhed af antigen, co-stimulerende signaler, og opløselige faktorer (cytokiner og chemokiner), der påvirker mængden og kvaliteten af T-celle klon afkom4,5,6,7.
På trods af intensive undersøgelser af T-celle kloniske ekspansion, Det vides stadig ikke, om antigen-primet T-celler fuldføre hele deres celle cyklus på stedet for antigen anerkendelse, eller om de migrerer til andre organer under celle cyklus progression. Denne mangel på viden skyldes egenskaberne ved tilgængelige værktøjer til cellecyklusanalyse. Disse omfatter monoklonale antistoffer (mAbs), der er specifikke for atommarkøren, Ki67, og cellefarvestoffer, der enten identificerer celler, der har gennemgået S-fasen af cellecyklussen (f.eks. Bromodeoxyuridin (BrdU)) eller diskriminerer mellem datterceller og deres forfædre (f.eks. carboxyfluorescein kortnimidyl ester (CFSE)).
Cellemærkningsfarvestoffer, såsom CFSE og BrdU, tillader dog ikke at afgøre, om celler, der findes i et bestemt organ, spredes lokalt eller snarere migreret til dette sted efter8. Desuden er det intranukleare protein, Ki67, kun i stand til at skelne celler i G0 (Ki67-negative celler) fra dem i enhver anden cellecyklusfase (Ki67-positive celler). Ki67-analysen skelner således ikke celler i aktiv spredning (dvs. i S, G2eller M) fra cellerne i G1, som enten hurtigt kan udvikle sig til opdeling eller blive i lange perioder i G1 eller vende tilbage til quiescence10,11.
Her beskriver vi en ny flowcytometrisk metode til cellecyklusanalyse af antigenspecifikke CD8 T-celler12 fra milten og lymfeknuderne (LN'er) hos vaccinerede mus (Figur 1). Metoden udnytter en kombination af Ki67 og DNA farvning, der tidligere blev brugt til at analysere cellecyklus af musen knoglemarv (BM) hæmatoopoietic celler13,14. Her har vi med succes anvendt Ki67 plus DNA farvning, sammen med den nyligt offentliggjorte innovative gating strategi12, til analyse af CD8 T celle kloniske ekspansion. Vi var i stand til klart at skelne mellem antigenspecifikke CD8 T-celler i G0, i G1og i S-G2/M-faser i milten og drænende LN'er for vaccinerede mus.
Protocol
Mus blev anbragt på Plaisant Animal Facility, og arbejdet blev udført under det italienske sundhedsministeriums autorisationsnummer 1065/2015-PR. Protokollen fulgte retningslinjerne for dyrepleje i henhold til nationale og internationale love og politikker (UE-direktiv 2010/63/UE; italiensk lovdekret 26/2014).
1. Fremstilling af medium og farvningsopløsning
- Forbered Komplet Medium: Roswell Park Memorial Institute (RPMI) medium med 2 mM glutamin, 100 U/mL penicillin/streptomycin, 50 μM beta-mercaptoethanol og 10% volumen /volumen (v/v) af fosterkvæg serum (FBS)
- Forbered farvning Buffer: Fosfat-buffered saltvand uden Ca2 +/ Mg2 + (PBS) med 1% vægt / volumen (w / v) kvæg serum albumin (BSA) og 2 mM ethylendiamintetraacetic syre disodium salt (EDTA)
2. Musebehandling
- Prime 7-8-uger gamle, kvindelige Balb /c mus ved intramuskulær (i.m.) injektion i quadriceps af human immundefekt virus (HIV)-1-gag-udtrykke-chimpanse adenoviral vektor (ChAd3-gag) med en dosis på 107 viruspartikler.
- Ved 1-4 måneder efter priming, boost en gang musene ved i.m. injektion af HIV-1-gag-udtrykke modificeret vaccinia Ankara virus (MVA-gag) med en dosis på 106 plaque-dannende enheder.
- På dag 3 efter boost, ofre de boostede mus ved cervikal dislokation, og analysere dem parallelt med ubehandlede mus.
- Høst de LN'er dræne quadriceps (iliac, popliteal, og lyske) og milt fra boostet og ubehandlede mus. Desuden opsamle BM fra de to bagben fra ubehandlede mus, og bruge denne BM til flow cytometer indstillinger og som positiv kontrol for celle cyklus analyse (Figur 2).
BEMÆRK: Generer vektorer for ChAd3-gag og MVA-gag som beskrevet tidligere12,15,16,17.
3. Isolering af dræning LN, milt, og BM celler
- Isolering af milt- og LN-celler
- Placer 5 mL komplet medium i hver af to 15 ML rør, og holde dem på is, klar til organer, der skal indsamles.
- Ofre en voksen mus ved cervikal dislokation.
- Placer musen på ryggen, og steriliser hudoverfladen med 70% v/v ethanol.
- For at indsamle inguinal LNs, lav en ~ 1 cm langsgående snit på maven med en saks, og strække snittet med kraften.
- Visualiser inguinal LNs på den indre overflade af huden, og høst dem med sammentakkerne. Lyske-L'erne placeres i et af de to 15 ML-rør, der er fremstillet i trin 3.1.1.
- For at samle milten skal du lave et peritoneal snit med en saks og fjerne milten. Efter skæring af det omgivende bindevæv placeres milten i det andet 15 mL rør, der er fremstillet i trin 3.1.1.
- At indsamle iliac LP'er, flytte tarmene til side og visualisere iliac LP'er tæt på ringere vena cava, og derefter indsamle dem ved hjælp af sammentværingerne. Læg iliaca-LU'erne i det samme rør, der indeholder de lyske LN'er.
BEMÆRK: For at få nok LN-celler til farvning (se afsnit 4) er det ofte nødvendigt at samle popliteale, inguinale og iliac-L'er fra én mus. Disse LP'er dræner alle quadriceps (stedet for i.m. vaccination). Denne protokol bruger kun ét 15 ML-rør med puljede LN'er. - For at indsamle popliteale LNs skal du gribe bagbenenes hud og forsigtigt trække den nedad for at afdække musklerne. Indsæt derefter sammentakkerne mellem musklerne under knæleddet, og saml de popliteale LNs. Placer de popliteale LN'er i samme rør, der indeholder lyske og iliac-LP'er.
BEMÆRK: Se note efter 3.1.7. - Læg milten i en 70 μm cellesn si i en 60 mm kulturskål fyldt med 5 mL komplet medium. Brug et 5 mL sprøjtestemning, forsigtigt mose organet, indtil dets fuldstændige opdeling.
- Fjern si, og overfør celleaffjedringen til et rent 15 ML rør.
- Tilsæt 5 mL komplet medium til kulturskålen, og vask forsigtigt fadet og sien for at sikre, at alle celler er blevet genoprettet. Pool med resten af milten celle suspension i 15 mL rør.
- For de samlede inguinal-, ilia- og popliteale LN'er forberedes en enkelt celleaffjedring efter en procedure, der svarer til den, der anvendes i trin 3.1.9 til 3.1.11 for milten.
- Centrifugeceller ved 400 × g i 10 min ved 4 °C. Kassér supernatanten, og genbrug cellepillerne i PBS.
- Tæl cellerne med et Neubauer kammer ved hjælp af røde blodlegemer lysis buffer og 0,04% v / v trypan blå i PBS.
- Isolering af BM-celler
- Placer 5 mL komplet medium i en 15 ML rør, og holde det på is, klar til indsamling af bagben.
- Ofre en voksen mus ved cervikal dislokation.
- Steriliser hudoverfladen med 70% v/v ethanol.
- Lav en ~ 1 cm tværgående snit på ventral hud med en saks, fast greb huden på begge sider af snittet, og træk forsigtigt nedad for at afdække musklerne i bagbenene.
- For at fjerne huden fra bagsiden af bagbenene, holde musen i en liggende position, placere klemmen under knæet og trække opad for at udsætte musklerne.
- Skær knoglerne i de to ekstremiteter i det ene bagben: bækken / hofteled og anklen.
- Begge bagben overføres til det 15 ML-rør, der er fremstillet i trin 3.2.1. Hold røret på is.
- Tag bagbenene fra 15 ML-røret og overfør dem til silkepapir. Skær bagbenene lige under knæleddet for at fjerne skinnebenet. Dissekere lårbenet og skinnebenet fra de omkringliggende muskler, fjerne overskydende væv ved hjælp af en saks, og våd silkepapir.
- Skær knogleenderne med en saks for at eksponere den indvendige marvaksel. Skinnebenet og lårbenet i BM-ekstraktionsrøret (se præparatet i 3.2.9.1-3.2.9.218), med den bredeste ende i bunden.
- Skær en 200 μL pipettespids ved linjen lige over enden af spidsen og ved 100 μL-linjen.
- Placer den midterste del i den øverste, større del af spidsen, og læg den i et 1,5 mL mikrofugerør.
- Drej BM ekstraktionsrøret ved 800 × g i 1 min.
- Kassér knoglen, og gentag forsigtigt pelleten i 1 mL komplet medium for at fjerne eventuelle klynger. Celleaffjedringen filtreres gennem et 70 μm filter, der er placeret på toppen af et 15 mL rør.
- Vask BM-ekstraktionsrøret to gange med 1 mL komplet medium hver gang. Filtrer gennem et 70 μm filter, og volumenpuljes med resten af celleaffjedringen, der blev opnået i trin 3.2.11.
BEMÆRK: Et enkelt 15 ML rør vil indeholde celler fra begge bagben af en mus. - Centrifugeceller ved 400 × g i 10 min ved 4 °C. Kassér supernatanten, og brug cellepillen i PBS igen.
- Tæl cellerne med et Neubauer kammer ved hjælp af røde blodlegemer lysis buffer og 0,04% v / v trypan blå i PBS.
4. Farvning af milt-, LN- og BM-celler
- Del celleprøver, der skal farves, i 3 undergrupper: celleprøvertil kompensation , herunder BM-celler fra ubehandlede mus, der kun skal farves sammen med Hoechst 33342 (fremover benævnt Hoechst) og miltceller fra ubehandlede mus, der skal anvendes til at forberede en død/levende celleblanding til erstatning for døde cellefarvestoffer positiv kontrol med cellecyklusanalyse, bestående af en BM-prøve fra ubehandlede mus forsøgsprøver, der indeholder milt- og LN-prøver fra ubehandlede og vaccinerede mus.
BEMÆRK: Sørg for, at der er nok milt- og LN-celler til analyse af et tilstrækkeligt antal gag-specifikke CD8 T-celler. Det er ofte nødvendigt at bruge poolede miltceller og poolede LN-celler fra 3 vaccinerede mus og plette to eller flere identiske prøver af poolede celler, der hver indeholder 3 × 106 celler. Flet identiske prøver ved trinnet af Hoechst farvning. Tilsvarende plet pooled milt celler og LN celler fra 3 ubehandlede mus, og fusionere identiske prøver i slutningen. Sæt en uplettet prøve af miltceller fra en ubehandlet mus, der skal bruges til instrument- og kompensationsopsætning. - Forbered døde / levende celle mix for døde celle farvestof kompensation (denne blanding af celler vil blive farves kun med den døde celle farvestof).
- Opvarm et vandbad ved 65 °C.
- Tag en aliquot af miltceller (~ 3 × 106).
- Celleaffjedringen overføres til et mikrofugerør, læg den i vandbadet ved 65 °C i 5 min, og læg den derefter straks på is i 10 min.
- Bland de varmedraflivede celler med levende miltceller (~ 3 × 106) i et forhold på 1:1, og overfør halvdelen af blandingen til en 96 godt rund bundplade (~ 3 × 106 celler / brønd til den døde celle farvningskontrol).
- Døde cellefarvning af eksperimentelle prøver, positiv kontrol til cellecyklusanalyse og død/levende celleblanding
- Overfør milt, LN, BM-celler (3 × 106 celler/brønd) og den døde/levende celleblanding (punkt 4.2) til 96-brønd rundbundsplade i henhold til farvningsordningen (trin 4.1) og centrifuge ved 400 × g i 3 min ved 4 °C.
- Resuspend hver celle pellet i 50 μL af døde celle farvestof fortyndet i PBS, og genbruges ved pipetter op og ned 3 gange straks.
- Inkuberes i 30 min ved 4 °C, beskyttet mod lys.
- Vask celler 2 gange med farvningsbuffer; første gang med 200 μL og anden gang med 250 μL. For hver vask centrifuge pladen ved 400 × g i 3 min ved 4 °C.
- Kassér supernatanten, og brug cellepillen i 20 μL PBS igen.
- Membrancelle farvning med større histokompatibilitet kompleks (MHC)-peptid multimers og mAbs.
- Under hensyntagen til de nødvendige mængder i henhold til farvningsordningen (Flow cytometerindstillinger, tabel 1), skal følgende reagenser forberedes:
- Fortynd mAb 2,4 G2 i farvningsbufferen i henhold til den relevante fortynding (se materialetabel); for hver prøve, der skal farves, skal der anvendes 10 μL af denne fortynding.
BEMÆRK: 2,4 G2 mAb blokerer ikke-antigenspecifik binding af immunoglobuliner til FüIII- og FcγII-receptorerne. - Fortynd den H-2k(d) AMQMLKETI allophycocyanin (APC)-mærket tetramer (Tetr-gag) i farvningsbufferen for at opnå den relevante fortynding (se materialetabel); for hver prøve, der skal farves, skal der anvendes 20 μL af denne fortynding.
- Antistofblandingen forberedes ved at fortynde mAb'er i farvningsbufferen i henhold til den relevante fortynding (se materialetabel),der tidligere er blevet bestemt i titreringsforsøg. for hver prøve, der skal farves, skal du bruge 20 μL af denne antistofblanding.
BEMÆRK: Her blev anti-CD3e peridinin klorofylprotein (PerCP-Cy5.5) (klon 145-2C11), anti-CD8a strålende ultraviolet (BUV805) (klon 53-6.7) og anti-CD62L phycoerythrin cyanine7 (PECy7) (klon MEL-14) brugt.
- Fortynd mAb 2,4 G2 i farvningsbufferen i henhold til den relevante fortynding (se materialetabel); for hver prøve, der skal farves, skal der anvendes 10 μL af denne fortynding.
- Der tilsættes 10 μL af de tidligere fortyndede 2,4 G2 mAb (trin 4.4.1.1) og inkuberes i 10 min ved 4 °C, beskyttet mod lys.
- Der tilsættes 20 μL af den tidligere fortyndede Tetr-gag APC (trin 4.4.1.2) og 10 μL af H-2k(d) AMQMLKETI phycoerythrin (PE) pentamer (pent-gag). Inkuberes i 15 min ved 4 °C, beskyttet mod lys.
- Der tilsættes 20 μL af den tidligere forberedte antistofblanding (trin 4.4.1.3) og inkuberes 15 min ved 4 °C, beskyttet mod lys.
BEMÆRK: Derfor er det endelige volumen 80 μL pr. brønd (trin 4.3.5, trin 4.4.2 til 4.4.4). - Vask celler med 200 μL farvningsbuffer. Centrifuge ved 400 × g i 5 min ved 4 °C.
- Cellpillen genbruges i 250 μL farvningsbuffer, og celleaffjedringen overføres til 5 mL-rør. Røret tilsættes 1 minut og centrifuge ved 400 × g i 5 min ved 4 °C.
- Tag aliquot af BM-celler (3 × 106 celler) (se listen over celleprøver, punkt 4.1), der skal bruges til at kompensere Fort. Røret tilsættes 1 minut og centrifuge 400 × g i 5 min ved 4 °C.
- Under hensyntagen til de nødvendige mængder i henhold til farvningsordningen (Flow cytometerindstillinger, tabel 1), skal følgende reagenser forberedes:
5. Fiksering/permeabilisering
- Forbered ny fiksering /permeabilisering buffer ved fortynding 1 del af fiksering / permeabilisering koncentrat med 3 dele af fiksering / permeabilisering fortynding fortyndingsstof, i henhold til producentens anvisninger.
- Kassér supernatanten, og pulshvirvlen prøverne for helt at opdele pelleten.
- Der tilsættes 1 mL af den frisklavede fikserings-/permeabiliseringsbuffer til hvert rør, herunder et rør med ikke-farvede miltceller (3 x 106, se listen over celleprøver, afsnit 4.1) og vortex.
- Inkuberes i 16 timer ved 4 °C.
BEMÆRK: Protokollen kan sættes på pause her.
6. Intracellulær farvning
- Ki67 farvning
- Forbered frisk permeabilisering buffer 1x ved fortynding permeabilisering buffer 10x med destilleret vand, i henhold til producentens anvisninger. Før brug skal permeabiliseringsbufferen 1x filtreres gennem et 0,45 μm filter for at eliminere aggregater.
- Fortynd mAb Ki67 fluorescein isothiocyanat (FITC) (klon SolA15) i permeabiliseringsbuffer 1x (se Materialetabel), som tidligere fastlagt i titreringsforsøg (sidste volumen på 100 μL pr. prøve).
- Tilsæt 3 mL permeabiliseringsbuffer 1x til hvert rør og centrifuge ved 400 × g i 5 min ved stuetemperatur (RT).
- Supernatanten kasseres, og trin 6.1.3 gentages.
- Supernatanten kasseres, og cellepillen genudsluses i 100 μL af tidligere fortyndet mAb Ki67 FITC (trin 6.1.2).
- Inkuber i 30 minutter på RT, beskyttet mod lys.
- Vask cellerne 2 gange med 4 mL permeabiliseringsbuffer 1x. For hver vask centrifuge ved 400 × g i 5 min på RT.
- Brug cellepillen i PBS på en genbrug af cellepillen i PBS i betragtning af følgende mængder: 350 μL PBS for de prøver, der skal udtages direkte ved flowcytometeret; 250 μL PBS for de prøver, der skal inkuberes med Hoechst kort før flowcytometri (punkt 6.2).
- DNA-farvning
- Der tilsættes 250 μL på 4 μg/mL Hoechst i PBS til hver prøve (den endelige koncentration af Hoechst er 2 μg/mL).
BEMÆRK: Hvis der blev udtaget to eller flere identiske prøver på 250 μL i PBS, smeltes de ad ved dette trin, og der tilsættes lige store mængder 4 μg/mL Hoechst-opløsning i PBS (den endelige koncentration af Hoechst er 2 μg/mL). Antallet af celler påvirker i høj grad DNA-farvningstrinnet. Brug det samme cellenummer i hvert eksempel. Vær opmærksom på, at selv et lidt reduceret celletal (f.eks. på grund af celletab i tidligere vasketrin) resulterer i højere Hoechst-binding til DNA og højere Hoechst-intensitet. - Inkuber i 15 minutter på RT, beskyttet mod lys.
- Centrifuge prøverne ved 400 × g i 5 min på RT.
- Brug cellepillen igen i 350 μL AF PBS.
- Der tilsættes 250 μL på 4 μg/mL Hoechst i PBS til hver prøve (den endelige koncentration af Hoechst er 2 μg/mL).
7. Udarbejdelse af erstatning perleprøver
- 5 μL af antistoffet fremstilles ved at fortynde mAb i farvningsbufferen korrekt.
BEMÆRK: For hver fluorkrom-konjugeret mAb, der anvendes i forsøget, skal du forberede den tilsvarende kompensationsperleprøve. - Vortex Negativ Kontrol og Anti-Rat / Hamster Ig,κ Comp Perler før brug.
- For hver prøve skal der indføres en dråbe (~20 μL) af negative kontrolkompendier og en dråbe Anti-Rat/Hamster Ig,k CompBeads.
- 5 μL af det prædiluterede antistof (trin 7.1) til røret og pipet op og ned.
- Inkuberes i 15 min ved 4 °C, beskyttet mod lys.
- Vask prøver med 2 mL farvningsbuffer. Centrifuge ved 400 × g i 5 min ved 4 °C.
- Kassér supernatanten, og genbrug pelleten ved at tilsætte 500 μL PBS til hvert rør og vortex.
8. Instrument- og kompensationsopsætning og eksperimentel prøveopkøb ved flowcytometeret
BEMÆRK: Se flowcytometerindstillinger (Tabel 2) for cytometerkonfigurationen.
- Generel opsætning af instrument og kompensation
- Åbn softwaren til eksempelopsamling (se Materialetabel), og opret et nyt eksperiment ved at klikke på Nyt eksperiment i afsnittet arbejdsområdebånd og vælge Nyt tomt eksperiment.
- Dobbeltklik på det oprettede eksperiment for at åbne det.
- I vinduet Cytometerindstillinger skal du klikke på Parametre og vælge alle de kanaler (f.eks. PE, APC osv.), der bruges i farvningspanelet, herunder PARAMETRENE Forward Scatter (FSC) og Side Scatter (SSC).
- Vælg lineær skala som en Hoechst-parameter ved at fjerne markeringen af logskalaen, og kontroller bredden (W) af spændingspulsen for FCS, SSC og Hoechst.
BEMÆRK: Alle parametre vises som standard i logaritmisk (log) skala, bortset fra FSC og SSC, der er i lineær skala. Alle parametre analyseres af spændingspulsens område (A) og højde (H). - Opret et prikplot med FSC-A på x-aksen og SSC-A på y-aksen i det globale regneark.
- Kør det ikke-farvede milteksempel ved at klikke på Hent data på anskaffelsesdashboardet.
- Angiv de relevante FSC- og SSC-indstillinger for at visualisere cellerne ved at ændre spændingsværdierne i sektionen Parametre, og opret en port for at markere alle de celler, der vises i punktet FSC-A/SSC-A-A-punkt ved at klikke på Polygon Gate på arbejdsområdeværktøjslinjen i det globale regneark.
- Vis de indhegnede celler i histogrammer med hver fluorescensparameter på x-aksen.
- Kør ureblede og fuldt farvede miltprøver for at justere fluorescensdetektoren (PMT) for at have en klar adskillelse mellem negative og positive signaler fra de farvede celler for hver fluorescensparameter.
- Hvis du vil udføre kompensationsopsætning, skal du klikke på Eksperiment på båndet i arbejdsområdet og vælge Opret kompensationskontrolelementerunder Kompensationsopsætning . Fjern markeringen Medtag ikke-farvet kontrolrør /Brønd, og klik på OK.
BEMÆRK: Denne handling vil resultere i oprettelsen af en prøve med navnet Kompensationskontrolelementer og et normalt regneark, der indeholder flere ark svarende til hver valgt parameter. - Kør en prøve af kompensationsperler (se afsnit 7); indstille de relevante FSC- og SSC-indstillinger for at visualisere perlerne ved at ændre spændingsværdierne og anskaffelsestærsklen på 5.000 på FSC-parametre i Cytometer-vinduet.
- Juster P1-porten på perlepopulationen, og kontroller, at de positive og negative toppe begge er synlige på x-aksen. Gentag denne handling for hvert eksempel på kompensationsperle, og registrer til sidst hver eksempelfil ved at klikke på Postdata på anskaffelsesdashboardet (registrer mindst 5.000 hændelser for hvert eksempel).
- For hver registreret perleprøve skal du indstille henholdsvis P2- og P3-portene på de positive og negative toppe.
- Der køres celleprøverne for at få erstatning (se trin 4.2 og 4.4.7 og afsnit 5 og 6). Rediger FSC- og SSC-spændingerne og tærskelværdien for at visualisere cellerne, justere P1-porten og til sidst registrere hver eksempelfil (registrere mindst 10.000 hændelser). Indstil P2- og P3-portene på henholdsvis de positive og negative toppe.
BEMÆRK: For kompensation for Hoechst-kanalen skal du bruge G0/G1 som den negative top (P3) og G2/M som den positive (P2). - Klik på Eksperiment i områdets båndsektion, og vælg Beregn kompensationi sektionen Kompensationsopsætning .
- Navngiv den oprettede kompensationsindstilling, link og gem den i det aktuelle eksperiment.
- Eksperimentel stikprøve erhvervelse
- Åbn et eksempel ved at klikke på Ny prøve på browserens værktøjslinje, og opret gatingstrategien i det globale regneark.
BEMÆRK: Gating-strategien for erhvervelse af stikprøver svarer til den strategi for stikprøveanalyse, der er beskrevet i figur 3 og afsnit 9. - Vis alle hændelsespopulationer i et histogrammer med CD3-A på x-aksen. Opret en intervalport for kun at markere CD3+ -cellerne.
- Vælg lagerport som Alle hændelser for LN-eksempler på købsdashboardetog enten Alle hændelser eller CD3+ celler til milteksempler.
- Kør de eksperimentelle prøver ved lav hastighed, og til sidst optage alle de filer, der sørger for at indsamle mindst 100-200 antigen-specifikke CD8 T-celler for hver prøve fra de vaccinerede mus.
BEMÆRK: Filstørrelsen af eksperimentelle prøver er normalt stor (30-120 MB), især når hyppigheden af antigenspecifikke CD8 T-celler er lav. Derfor skal der indsamles et stort antal hændelser (> 1 × 106) for at optage mindst 100-200 antigenspecifikke CD8 T-celler. Store filer kan bremse den efterfølgende dataanalyseproces. Erhvervelsen af kun CD3+ celler i miltprøver (se trin 8.2.2 ovenfor) er nyttigt at holde filstørrelsen mindre. - Kør og registrer den positive kontrol til cellecyklusanalyse, dvs. BM-prøve fra ubehandlede mus.
- Åbn et eksempel ved at klikke på Ny prøve på browserens værktøjslinje, og opret gatingstrategien i det globale regneark.
9. Dataanalyse
- Åbn softwaren (se Materialetabel), og opret forskellige grupper, der svarer til de forskellige organer, der skal analyseres, ved at klikke på Opret gruppe i afsnittet arbejdsområdebånd (dvs. opret gruppen "a-LN'er"; "b-milt"; "c-BM").
BEMÆRK: Nyoprettede grupper vises på gruppelisten, mens gruppen "Kompensation" automatisk genereres af softwaren. - Åbn vinduet Rediger gruppe ved at dobbeltklikke på gruppenavnet, og kontroller, at de nyoprettede grupper synkroniseres. Hvis ikke, skal du markere funktionen Synkroniseret.
- Træk hver .fcs-fil i den tilsvarende gruppe.
- Opret gating-strategien, der starter med gruppen "a-LNs".
- Dobbeltklik på den fuldt farvede prøve i gruppen for at åbne grafvinduet. x-og y-aksen er mærket som i fcs-filerne (se flowcytometerindstillinger, tabel 2).
- Vis de samlede hændelser, der er opnået for denne prøve, i et prikplot med DNA-A på x-aksen og DNA-W på y-aksen.
- Marker kun enkeltcellepopulationen ved at klikke på Rektangel i gatingværktøjet i diagramvinduet.
BEMÆRK: Enkelte celler har DNA-A-værdier som følger: 2N (lav): mellem 2N og 4N (mellemliggende) eller lig med 4N (høj), mens DNA-W-værdier er identiske for dem alle (trin 1 i figur 3). - Dobbeltklik i midten af den rektangulære port for at få vist enkelte celler i et prikplot med FSC-A-parameter på x-aksen og døde cellefarve på y-aksen.
- Marker kun den levende cellepopulation ved at klikke på Polygon i gatingværktøjssektionen i grafvinduet. Levende celler er negative for det døde cellefarvestof (trin 2 i figur 3).
- Dobbeltklik i midten af den polygonale port for at få vist cellerne i et prikplot med FSC-A-parameter på x-aksen og SSC-A-parameteren på y-aksen.
- Klik på Rektangel, og opret en "afslappet" port for at medtage alle de enkelte dynamiske celler igrafen 12 (trin 3 i figur 3).
- Dobbeltklik i midten af den "afslappede" port for at få vist cellerne i et prikplot med CD3 på x-aksen og CD8 på y-aksen.
- Marker CD3+CD8+ cellerne ved at klikke på Polygon (trin 4 i figur 3).
- Dobbeltklik i midten af CD3+CD8+ porten for at få vist cellerne i et prikplot med Tetr-gag på x-aksen og Pent-gag på y-aksen.
- Vælg de antigenspecifikke CD8 T-celler (positiv for både Tetr-gag og Pent-gag) ved at klikke på Polygon (trin 5 i figur 3).
- Dobbeltklik i midten af den gag-specifikke port for at få vist cellerne i et prikplot med DNA-A på x-aksen og Ki67 på y-aksen (Figur 4).
- Marker cellerne i de forskellige cellecyklusfaser ved at klikke på Quad i gatingværktøjet i grafvinduet.
BEMÆRK: Celle i G0 fase er Ki67neg-DNA lavceller (nederst til venstre kvadrant); celler i G1 er Ki67pos-DNA lav (øvre venstre kvadrant); celler i S-G2/M er Ki67pos-DNA mellemliggende/høje (øverst til højre kvadrant) (Figur 4). - Kopier den gating strategi, der er oprettet i et eksempel til den tilsvarende gruppe for at anvende portene på alle gruppens prøver.
- Gentag trin 9.5 til 9.18 for "a-LN-gruppen".
- Kontroller, at alle porte er passende for hver prøve af "b-milten" gruppen. Hvis du vil analysere cellecyklussen mellem BM-cellerne (positiv kontrol), skal du klikke i midten af den "afslappede" port for at vise cellerne i et prikplot med DNA-A på x-aksen og Ki67 på y-aksen.
- Kontroller, at alle porte er passende for hver prøve af de 3 grupper (dvs. for celler fra milt, LN og BM).
BEMÆRK: Enkeltcellet population gate (trin 9.7) og Quad gate for cellecyklus (trin 9.17) kan have forskellige gate koordinater i forskellige prøver, hovedsagelig på grund af de mulige små forskelle i Hoechst farveintensiteten mellem prøverne (punkt 6.2). Af denne grund kan det være nødvendigt at ændre enkeltcellepopulationsporten og Quad-portene til cellecyklus i hver prøve. Dette gøres på følgende måde: Dobbeltklik på gruppenavnet, og fjern synkroniseringen fra gruppeegenskaberne. Denne operation gør det muligt at ændre portene i en prøve uden at ændre de samme porte i alle de andre prøver af gruppen. Når du har fjernet synkroniseringen, skal du ændre portene, hvor det er nødvendigt. - Hvis du vil visualisere de resultater, der opnås ved denne analyse, skal du klikke på Layouteditor i afsnittet på båndet i arbejdsområdet for at åbne den. Træk hver port i gatingstrategien i eksempelruden til layouteditoren, og placer plottet i overensstemmelse med rækkefølgen af gatingstrategien. Hvis det er nødvendigt, skal du ændre diagramtypen ved at dobbeltklikke på det tilsvarende afbildning i layoutet og vælge den relevante type i vinduet Grafdefinition.
- Klik på gruppen og iterate af funktioner på layoutbåndet for at visualisere de opnåede resultater i hvert organ, og sammenligne forskellige prøver.
Representative Results
Cellecyklusfaserne af celler fra milt,LN'er og BM af mus i Balb/c blev analyseret ved hjælp af det fluorescerende DNA-farvestof, Hoechst, og en anti-Ki67 mAb, ifølge protokollen opsummeret i figur 1. Dennefarvning gjorde det muligt at skelne celler i følgende faser af cellecyklussen: G 0 (Ki67neg, med 2N DNA defineret som DNAlow), G1 (Ki67pos, DNAlow) og S-G2/M (Ki67pos, med et DNA-indhold bestående mellem 2N og 4N, eller lig med 4N DNA defineret som DNAintermediate/høj).
Vi udførte først cellecyklusanalyse af BM-celler for at reproducere tidligere offentliggjorte resultater13,14 og analyserede derefter cellerne af interesse, dvs. CD8 T-celler. Figur 2 viser et typisk eksempel på cellecyklusanalyse af BM-celler (Figur 2A). Protokollen gav en lav variationskoefficient (CV) på G0/G1 og G2/M DNA-toppe, hvilket indikerer den fremragende kvalitet af DNA-farvningen (Figur 2B, der viser et eksempel med CV < 2,5; CV var altid < 5 i alle eksperimenter).
Vi anvendte derefter den samme protokol på antigenspecifikke CD8 T-celler fra vaccinerede mus. BALB/c mus blev vaccineret mod antigen gag af HIV-1 ved hjælp af Chad3-gag til priming og MVA-gag for at øge, begge manipuleret til at bære HIV-1 gag. På dag (d) 3 post-boost analyserede vi hyppigheden af gag-specifikke CD8 T-celler fra milten og dræning af LP'er. Vi benyttede os af den nyligt definerede gating strategi for T-celler i den tidlige fase af immunrespons, som i modsætning til den konventionelle strategi, er egnet til at opdage stærkt aktiveret antigen-reagere CD8 T-celler12. Vi udførte den nye strategi i fem efterfølgende trin. I trin 1 udelukkede vi doubler eller aggregater med DNA-A/ -W gate, og i trin 2 identificerede vi levende celler ved udelukkelse af døde celler. I trin 3 identificerede vi befolkningen af interesse ved hjælp af en ikke-konventionel "afslappet" FSC-A / SSC-A gate (Figur 3A) i stedet for den kanoniske smalle lymfocytport12. Efter gating på CD3+CD8+ celler (trin 4 i figur 3A),identificerede vi gag-specifikke CD8 T-celler ved hjælp af to forskellige MHC multimers, dvs Pent-gag og Tetr-gag (trin 5 i figur 3A). Vi brugte to multimers i stedet for en til at forbedre følsomheden af gag-specifikke CD8 T celle afsløring i vaccinerede mus, uden at øge farvning baggrund i ubehandlede mus(Figur 3B og C, trin 5). Således skelnede vi med succes ubehandlede mus (henholdsvis 0,00% og 0,00% antigenspecifikke CD8 T-celler i LN'er og milt) fra vaccinerede mus (henholdsvis 0,46% og 0,29% antigenspecifikke CD8 T-celler i LN'er og miltceller, henholdsvis figur 3B og C).
Især gav protokollen os mulighed for at have en ekstremt lav baggrund i den antigenspecifikke CD8 T-celleport af LN'er og milt af ubehandlede mus (normalt 0,00% og højst 0,02%). Sammenligningen af gag-specifikke og ikke gag-specifikke FSC-A / SSC-A parceller viste, at gag-specifikke celler havde høj SSC-A og FSC-A (Figur 3D), bekræfter behovet for at bruge en "afslappet" FSC-A / SSC-A gate til at fange disse celler. Vi evaluerede derefter procentdelene af gag-specifikke CD8 T-celler i forskellige cellecyklusfaser (Figur 4A). Vi fandt ud af, at gag-specifikke CD8 T-celler i milten og endnu mere i de drænende LN'er indeholdt en høj andel af celler i S-G2/ M faser på dag 3 post-boost (henholdsvis 18,60% og 33,52%).
Desuden fandt vi, at gag-specifikke CD8 T-celler i S-G2/ M faser havde høje FSC-A og SSC-A, når overlejret på den samlede CD8 T-celler fra samme organ (Figur 4B). CD62L-udtrykket med gag-specifikke CD8 T-celler var lavt, som forventet for aktiverede T-celler, bortset fra nogle få celler i G0 i LN'erne (Figur 4C). Alt i alt bekræftede disse resultater, at den "afslappede" port (trin 3 i figur 3A, B og C) var forpligtet til at omfatte alle de voksende antigenspecifikke CD8 T-celler12. Protokollen var yderst værdifuld for en "snapshot" evaluering af celle cyklus faser af antigen-specifikke CD8 T-celler på tidspunktet for analysen og CD62L udtryk af celler i forskellige celle cyklus faser.

Figur 1: Ordningen for protokollen til cellecyklusanalyse af antigenspecifikke CD8 T-celler. Klik her for at se en større version af dette tal.

Figur 2: Cellecyklusanalyse af BM-celler. BM-celler fra ubehandlede Balb/c mus blev farves og analyseret af flowcytometri. (A) Eksempel på gating strategi. Vi gated på enkelte celler i DNA-A /-W plot (venstre) og efterfølgende på levende celler ved døde celle farvestof udelukkelse (midten). Derefter blev en "afslappet" FSC-A / SSC-A-port brugt til alle BM-celler (højre). (B) Eksempel på cellecyklusanalyse af BM-celler (venstre). Vi brugte en kombination af Ki67 og DNA farvning til at identificere celler i følgende faser af cellecyklus: G0 (nederst til venstre kvadrant, Ki67neg-DNAlow celler), G1 (øverst til venstre kvadrant, Ki67pos-DNAlow), S-G2/ M (øverst til højre kvadrant, Ki67pos-DNAintermediate / høj). Fluorescens Minus One (FMO) kontrol af Ki67 mAb (midten) og DNA histogram (højre) er vist. I DNA-histogrammeplottet svarer venstre og højre porte til henholdsvis G0/G1 og G2/M DNA-toppen, og tallene repræsenterer variationskoefficienterne (CV) for hver top. I alle de andre plots repræsenterer tallene celleprocenter i de angivne porte. Figuren viser 1 repræsentativt eksperiment ud af 5. I hvert eksperiment analyserede vi poolede BM-celler fra 3 mus. Klik her for at se en større version af dette tal.

Figur 3: Analyse af antigenspecifikke CD8 T-celler fra LN'er og milt. Balb/c mus blev primet intramuskulært (i.m.) med Chad3-gag og boostet i.m. med MVA-gag. På dag 3 efter boost blev dræning af LN- og miltceller fra vaccinerede og ubehandlede kontrolmus farves og analyseret af flowcytometri. A) Ordningen for gatingstrategien i fem trin til identifikation af enkeltceller (trin 1) levende celler (trin 2); lymfocytter (trin 3); CD8 T-celler (trin 4); og gag-specifikke celler (trin 5). (B-C) Eksempel på observationsområder: analyse af celler fra (B) LN'er og (C) milt af ubehandlede (top) og vaccinerede (nederste) mus. Vi identificerede enkelte celler på DNA-A/ -W plottet i trin 1. I trin 2 valgte vi så levende celler ved at eksklude døde celler. I trin 3 brugte vi en ikke-kanonisk "afslappet" port til lymfocytter. I trin 4 identificerede vi CD8 T-celler ved deres dobbelte udtryk for CD3 og CD8. Vi identificerede derefter gag-specifikke celler og ikke gag-specifikke i trin 5, baseret på deres evne til at binde fluorokrom-mærket H-2kd-gag-Pentamer (Pent-gag) og H-2kd-gag-Tetramer (Tetr-gag), eller ej, henholdsvis. (D) FSC-A/SSC-A-profiler af gag-specifikke (blå) og ikke gag-specifikke (grå) celler efter gating som beskrevet ovenfor. Tal repræsenterer celleprocenter i de angivne porte. Figuren viser 1 repræsentativt eksperiment ud af 5. I hvert eksperiment analyserede vi poolede milt og samlede LN-celler fra 3 vaccinerede mus og 3 ubehandlede mus. Klik her for at se en større version af dette tal.

Figur 4: Cellecyklusanalyse af antigenspecifikke CD8 T-celler. Mus blev vaccineret som i figur 3, og cellecyklusanalyse af gag-specifikke celler blev udført på dag 3 efter boost efter gating i 5 trin som i figur 3. (A) Eksempel på cellecyklusanalyse af gag-specifikke CD8 T-celler fra LN'er (øverst) og milt (nederst) af vaccinerede mus. Cellecyklusfaser blev identificeret som i figur 2B. Panelerne repræsenterer celler i G0, i G1og i S-G2/M (venstre) og Fluorescens Minus One (FMO) kontrol af Ki67 mAb (højre). Tal repræsenterer celleprocenter i de angivne porte. (B) FSC-A/SSC-A prikplotter, der viser gag-specifikke CD8 T-celler i S-G2/M-faser(med rødt) overlejret på totale CD3+CD8+ T-celler (i grå) fra LN'er (øverst) og milt (nederst) af vaccinerede mus. (C) Forskydning af histogrammer, der viser CD62L-udtryk med gagspecifikke CD8 T-celler i G0 (grøn), i G1 (blå) og i S-G2/M (rød) fra LN'er (øverst) og milt (nederst) af vaccinerede mus. Y-akserne angiver det normaliserede antal hændelser. Figuren viser 1 repræsentativt eksempel ud af 5 uafhængige eksperimenter med i alt 15 mus. Klik her for at se en større version af dette tal.
Supplerende materiale: Flowcytometerindstillinger. Klik her for at downloade denne fil.
Discussion
Selvom T-celle klonisk ekspansion er blevet intensivt undersøgt, nogle aspekter forbliver ukendt, mest fordi de værktøjer til rådighed til at undersøge det er få og har deres egne ulemper. Fra dette perspektiv, vi oprette en meget følsom flow cytometrisk metode til at analysere cellecyklus af antigen-specifikke CD8 T-celler på tidlige tidspunkter efter vaccination i en musemodel. Protokollen er baseret på en kombination af Ki67 og DNA farvning, som tidligere blev brugt til at analysere cellecyklus af BM hæmatoopoietic celler i mus13,14. For at tilpasse protokollen til antigenspecifikke CD8 T-celler måtte vi overveje et par kritiske spørgsmål, herunder valget af DNA-farvestof, de passende betingelser for at opnå sammenlignelig DNA-farvning på tværs af forskellige prøver og gatingstrategien for dataanalyse.
Mange farvestoffer er tilgængelige for DNA farvning, herunder propidium jod og 7-aminoactinomycin D; vi valgte Hoechst, fordi det var kompatibelt med membranfarvning og den milde fiksering / permeabiliseringsprotokol, der kræves til Ki67 farvning. Samtidig gav farvning med Hoechst os mulighed for at opnå et DNA-histogram af fremragende kvalitet, dvs. at G0/G1 og G2/M DNA-toppe havde en meget lavere variationskoefficient (CV) end DNA-toppe, der normalt opnås med andre DNA-farvestoffer, f.eks. DRAQ519. Faktisk kan Hoechst plette DNA selv i levende celler20.
Nogle strategier blev brugt til at undgå udsving i Hoechst intensitet i forskellige prøver af det samme eksperiment. Hoechst farvning blev udført lige før prøve erhvervelse på flow cytometer for at minimere nedgangen i farvestof intensitet i tide. For dem, der er interesseret i at reproducere protokollen i store eksperimenter med mange prøver, anbefaler vi at udføre Hoechst farvning på et par prøver ad gangen. En anden ulempe er, at Hoechst intensitet kan være stærkt påvirket af cellenummer under inkubation med farvestoffet. Af denne grund anbefaler vi på det kraftigste altid at bruge det samme antal celler og det samme volumen pr. prøve til DNA-farvning. Hvis et stort antal celler er nødvendige for erhvervelse på flowcytometeret, anbefaler vi, at du forbereder to eller flere identiske prøver og derefter fletter dem lige før Hoechst-farvningstrinnet.
Et centralt punkt i protokollen er gating-strategien for dataanalyse. Vi har for nylig offentliggjort en ny strategi for T-celleanalyse på tidlige tidspunkter af immunresponset, som gjorde det muligt for os at øge følsomheden af påvisning af antigenspecifikke T-celler12. Vi anvendte denne strategi på de data, der er vist her som følger. For det første udelukkede vi celleaggregater i DNA-A/W-plottet. For det andet, efter gating ud døde celler, vi brugte en temmelig stor lymfocyt gate i FSC / SSC plot ("afslappet gate"). Med denne strategi var vi i stand til at inkludere stærkt aktiverede antigenspecifikke CD8 T-celler i S-G2/M, der normalt savnes af aktuelle gatingstrategier, da disse celler har høje FSC-A og SSC-A. Sammenfattende repræsenterer dataanalysen en kritisk del af metoden, som er afgørende for at opnå en følsom påvisning af aktiverede / spredte antigenspecifikke T-celler.
Metoden forhindrer muligheden for at gå glip af kritiske T-celledata i tidlige faser af immunrespons og åbner nye perspektiver for T-celle immunovervågning. En fremtidig forbedring kan være at omfatte farvning for fosfo-histone 3, der ville tillade differentiering mellem G2 og M21. En aktuel begrænsning er, at celler skal fastsættes og permeabilized at plette for den nukleare markør, Ki67. Celler kan således ikke bruges til andre typer analyser såsom sortering og efterfølgende funktionel analyse. Desuden forstyrrer DNA-farvestoffer, herunder Hoechst, normalt den genomiske DNA-analyse og er ikke egnede til denne type evaluering. Identifikation af membranmarkører, der korrelerer med forskellige cellecyklusfaser, og som kan farves på levende celler, kan overvinde denne begrænsning. Afslutningsvis har metoden et stort potentiale for evaluering af aktiverede/spredte T-celler i flere sammenhænge såsom vaccination, infektion, immunmedierede sygdomme og immunterapi.
Disclosures
A. Folgori og S. Capone er ansatte i Reithera Srl. A. Nicosia er opkaldt opfinder på patentansøgning WO 2005071093 (A3) "Chimpanse adenovirus vaccine luftfartsselskaber." De andre forfattere har intet at afsløre.
Acknowledgments
Dette arbejde blev støttet af Reithera, af MIUR-projektet 2017K55HLC_006 og med 5 × 1000 tilskud fra Associazione Italiana Ricerca sul Cancro (AIRC). Følgende tetramer blev opnået gennem NIH Tetramer-anlægget: APC-konjugeret H-2K (d) HIV gag 197-205 AMQMLKETI.
Materials
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
References
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- Mouse phenotype. , Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020).
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Tags
Immunologi og infektion Problem 167 antigen-specifikke CD8 T-celler cellecyklus Ki67 DNA-farvestof flowcytometri milt lymfeknuder musErratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021.
Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
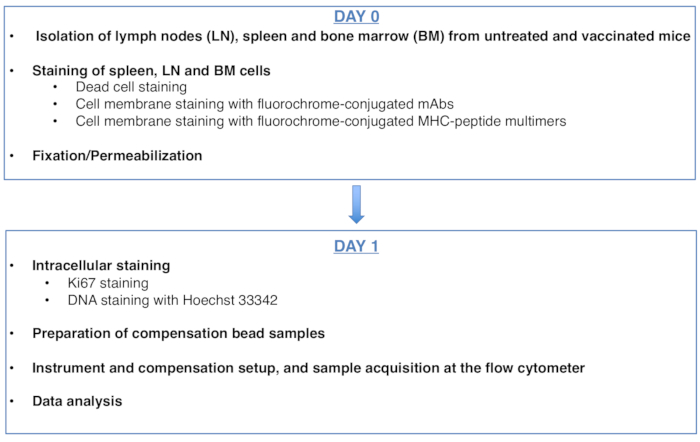
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:
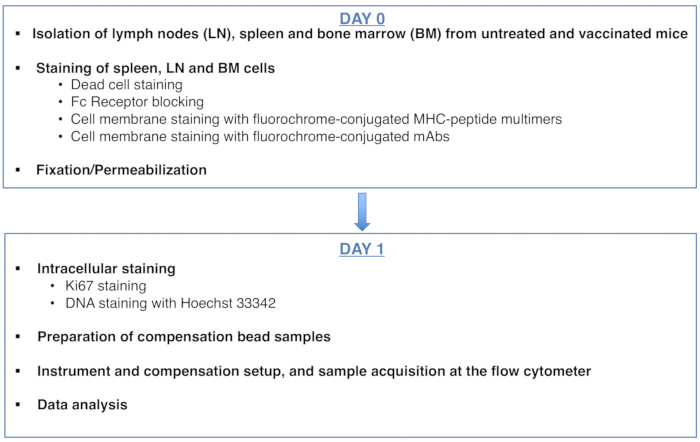
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.



