ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
クローン拡張は、抗原特異的T細胞応答の重要な特徴である。しかし、抗原応答性T細胞の細胞周期は、技術的な制限もあって、十分に調査されていない。ワクチン接種マウスの脾臓およびリンパ節におけるクローン膨張性抗原特異的CD8 T細胞を分析するフローサイトメトリック法について述べた。
Abstract
生体内の抗原特異的T細胞の細胞周期は、いくつかの方法を用いて調べられており、そのすべてがいくつかの制限を有する。ブロモデオキシウリジン(BrdU)は、S相中または最近完了した細胞をマークし、カルボキシフルオレセイン・スクシニミジルエステル(CFSE)は分裂後に娘細胞を検出する。しかし、これらの色素は分析時の細胞周期段階の同定を認めておらず。別のアプローチは、Ki67を利用することです, 静止期G0を除く細胞周期のすべての段階で細胞によって高度に発現しているマーカー.残念ながら、Ki67は、有糸分裂にコミットしているS相の細胞を、この段階に残ることができるG1の細胞から分離したり、サイクリングに進んだり、G0に移行したりしないため、さらなる分化は許されない。
ここでは、マウス二次リンパ器官における異なる細胞周期期におけるT細胞の「スナップショット」を捕捉するためのフローサイトメトリック法について述べた。この方法は、Ki67とDNA染色を主要組織適合性複合体(MHC)ペプチドマルチマー染色および革新的な格子戦略と組み合わせたものです。 ヒト免疫不全ウイルス(HIV)-1のモデル抗原ギャグを運ぶウイルスベクターを用いたワクチン接種後、マウスの脾臓およびリンパ節のG1およびS-G2/M相の抗原特異的CD8 T細胞の区別に成功した。
この方法の重要なステップは、DNA色素の選択と、アッセイ感度を高め、現在の分析基準で見逃されたであろう高度に活性化/増殖する抗原特異的T細胞を含む格言戦略であった。DNA染料であるHoechst 33342は、膜と細胞内染色を維持しながら、G0/G1およびG2/M DNAピークの高品質な差別を得ることを可能にしました。この方法は、生体内でのT細胞応答に関する知識を高め、免疫モニタリング分析を改善する大きな可能性を秘めています。
Introduction
ナイーブT細胞は、抗原プライミング時にクローン拡張と分化を受ける。分化されたT細胞は、抗原クリアランスや抗原特異的記憶の維持に不可欠なエフェクター機能を示し、これは長期的な保護の鍵となります。一次応答の最初のステップの間に、リンパ管内の特殊なニッチ内の抗原提示細胞(APC)とのナイーブT細胞相互作用は、クローン拡張フェーズ1、2、3を特徴付ける巨大なT細胞増殖を誘導するために重要である。T細胞-APC相互作用は、抗原の濃度および持続性によって細かく調節され、共刺激シグナル、および可溶性因子(サイトカインおよびケモカイン)がT細胞クローンの子孫4、5、6、7の量および質に影響を及ぼす。
T細胞クローナル拡張の鋭意研究にもかかわらず、抗原プライミングT細胞が抗原認識部位で細胞周期全体を完了するのか、細胞周期進行中に他の器官に移行するのかはまだ分かっていない。この知識の欠如は、細胞周期解析に利用できるツールの特性によるものです。これらには、核マーカーに特異的なモノクローナル抗体(mAbs)、Ki67、および細胞周期のS相を受けた細胞(例えばブロモデオキシウリジン(BrdU)))を識別するか、または娘細胞とその祖先(例えば、カルボキシフルオレセイン・スハクニミドリルエステル)を区別する細胞色素が含まれる。
しかし、CFSEやBrdUなどの細胞標識色素は、特定の器官に見つかった細胞が局所的に増殖するか、またはむしろ第8部、9の後にこの部位に移動したのかについて決定を許さない。また、核内タンパク質であるKi67は、G0(Ki67陰性細胞)の細胞を他の細胞周期期(Ki67陽性細胞)の細胞としか区別できない。したがって、Ki67解析では、活性増殖中の細胞(すなわち、S、G2、M)をG1内の細胞と区別せず、G1内で素早く分裂するか、またはG1に長期間滞在するか、静止10、11に戻る。
ここでは、ワクチン接種マウスの脾臓およびリンパ節(LNs)からの抗原特異的CD8 T細胞12の細胞周期解析のための新しいフローサイトメトリック法について説明する(図1)。この方法は、マウス骨髄(BM)造血細胞13,14の細胞周期を解析するために以前に使用されたKi67とDNA染色の組み合わせを利用する。ここでは、KI67とDNA染色を、最近発表された革新的な格言戦略12と共に、CD8 T細胞クローン拡張の分析にうまく適用しました。G0、G1、S-G2/M相では抗原特異的CD8 T細胞を明確に区別することができた。
Protocol
マウスはプレサント動物施設に収容され、作業はイタリア保健省の認可番号1065/2015-PRの下で行われました。このプロトコルは、国内および国際法およびポリシー(UE指令2010/63/UE;)に従って動物のケアガイドラインに従った。イタリアの立法令 26/2014).
1. 培地および染色液の調製
- 完全な媒体を準備する:2 mMグルタミン、100 U/mLペニシリン/ストレプトマイシン、50 μM β-メルカプトエタノール、ウシ胎児血清(FBS)の10%の体積/体積(v/v)を備えたロズウェルパーク記念研究所(RPMI)培地
- 染色バッファーの準備:リン酸緩衝塩分(Ca2+/Mg2+(PBS)なし、1%重量/体積(w/v)ウシ血清アルブミン(BSA)および2mMエチレンアミンテトラ酢酸二ナトリウム塩(EDTA)
2. マウス治療
- 7-8週齢の雌バルブ/cマウスは、ヒト免疫不全ウイルス(HIV)-1-gag発現チンパンジーアデノウイルスベクター(ChAd3-gag)の4頭筋への筋肉内(i.m.)注射による107ウイルス 粒子の用量を有する。
- プライミング後1〜4ヶ月後に、106 プラーク形成ユニットの用量でi.m.HIV-1ギャグ発現改変ワクシニアウイルス(MVA-gag)の注射によってマウスを1回後押しする。
- 3日目の後押しでは、子宮頸部脱臼によってブーストされたマウスを犠牲にし、未処理のマウスと並行して分析する。
- 四頭筋(腸骨、白癬、鼠)と脾臓を昇圧および未治療のマウスから排出するLNsを収穫します。さらに、未治療マウスから2本の後足からBMを収集し、フローサイトメーターの設定や細胞周期解析の陽性制御としてこのBMを使用する(図2)。
注意: 上記の説明に従って ChAd3-gag と MVA-gag ベクターを生成します12,15,16,17.
3. 排水LN、脾臓、およびBM細胞の分離
- 脾臓細胞とLN細胞の分離
- 2つの15 mLチューブのそれぞれに完全な培地の5 mLを入れ、それらを氷の上に置き、臓器を収集する準備ができています。
- 頸部脱臼で成虫マウスを犠牲にする。
- 背中にマウスを置き、70%v/vエタノールで皮膚表面を殺菌します。
- インギナルLNを収集するには、腹部に約1cmの縦切開をはさみで作り、鉗子で切開を伸ばします。
- 皮膚の内部表面にインジュナルのLNsを可視化し、鉗子で収穫します。ステップ3.1.1で作製した2本の15mLチューブの1つにインジュナルLを入れてください。
- 脾臓を採取するには、はさみで腹膜切開を行い、脾臓を取り除く。周囲の結合組織を切断した後、ステップ3.1.1で調製した第2の15mLチューブに脾臓を入れる。
- 腸骨の L を収集するには、腸を脇に移動し、下の大静脈の近くに腸骨の LUN を視覚化し、鉗子を使用して収集します。腸骨の LUN を含む同じチューブに腸骨の LUN を配置します。
注: 染色に十分な LN セルを取得するには (セクション 4 を参照)、多くの場合、1 つのマウスからポピタル、インジュナル、および iliac の LN をプールする必要があります。これらのRNはすべて四頭筋(i.m.ワクチン接種の部位)を排出している。このプロトコルは、プールされた LUN の 15 mL チューブのみを使用します。 - ポピタルのLNを収集するには、後ろ足の皮膚をつかみ、下にそっと引っ張って筋肉を明らかにする。次に、膝関節の下の筋肉の間に鉗子を挿入し、その後、その大台のLを集めます。
注: 3.1.7 以降の注を参照してください。 - 5 mLの培地を充填した60 mm培養皿内の70 μmの細胞ストレーナーに脾臓を入れます。5 mL シリンジ プランジャーを使用して、オルガンを完全に分解するまで軽くつぶします。
- ストレーナーを取り外し、細胞懸濁液を清潔な15 mLチューブに移します。
- 培養皿に5mLの培地を加え、皿とストレーナーを丁寧に洗って、すべての細胞が回収されるようにします。残りの脾臓細胞懸濁液を15 mLチューブにプールします。
- プールされたイングイナル、腸骨、およびポピタリのLNの場合、脾臓のステップ3.1.9から3.1.11で使用されるのと同様の手順に従って、単一の細胞懸濁液を準備します。
- 遠心分離細胞は400×gで10分間4°Cで10分間。 上清を捨て、PBS中の細胞ペレットを再懸濁する。
- PBSで赤血球溶菌バッファーと 0.04% v/v トリパンブルーを使用してノイバウアーチャンバーを持つ細胞を数えます。
- BM細胞の単離
- 5 mLコンプリートミディアムを15 mLチューブに入れ、氷の上に置いて、後ろ足のコレクションの準備をします。
- 頸部脱臼で成虫マウスを犠牲にする。
- 70%のv/vエタノールで皮膚表面を殺菌する。
- 腹側の皮膚に1cmの横切り切開をハサミで作り、切り口の両側の皮膚をしっかりとつかみ、後ろ足の筋肉を明らかにするために下向きにそっと引っ張ります。
- 後ろ足の後ろから皮膚を排除するには、マウスを膝の下に置き、クランプを膝の下に置き、筋肉を露出させるために上に引っ張ります。
- 片方の後ろ足の2つの四肢(骨盤/股関節と足首)で骨を切ります。
- ステップ3.2.1で準備した15 mLチューブに両後足を移します。チューブを氷の上に置いておきなさい。
- 15 mLチューブから後ろ足を取り、ティッシュペーパーに移します。膝関節のすぐ下の後ろ足を切って脛目を取り除きます。周囲の筋肉から大腿骨と脛骨を解剖し、はさみを使って余分な組織を取り除き、ティッシュペーパーを濡らします。
- 骨の端をはさみで切り、内側の骨髄シャフトを露出させます。脛骨と大腿骨をBM抽出チューブに挿入し(3.2.9.1-3.2.9.218の準備を参照)、最も広い端を底に入れてください。
- チップの端のすぐ上のラインと100 μLラインで200 μLのピペットチップを切ります。
- 先端の上部、より大きな部分に中央部を置き、これを1.5 mLマイクロフュージチューブに入れます。
- 800×gでBM抽出チューブを1分間回転させます。
- 骨を捨て、完全培地の1mLでペレットを激しく再懸濁し、任意のクラスターを除去する。15 mLチューブの上部に配置された70 μmのフィルターを通して、セルの懸濁液をフィルターします。
- BM抽出チューブを1 mLの完全な培地で2回洗浄します。70 μm フィルターを通してフィルターし、ステップ 3.2.11 で得られた残りのセル懸濁液とボリュームをプールします。
注:15 mLチューブ1本には、マウスの両後脚の細胞が含まれます。 - 遠心分離細胞は400×gで10分間4°Cで10分間。 上清を捨て、PBS中の細胞ペレットを再懸濁する。
- PBSで赤血球溶菌バッファーと 0.04% v/v トリパンブルーを使用してノイバウアーチャンバーを持つ細胞を数えます。
4. 脾臓、LN、およびBM細胞の染色
- 染色される細胞サンプルを3つのサブグループに分ける:治療されていないマウスからのBM細胞を含む 補償のための細胞サンプルは、Hoechst 33342(したがってHoechstと呼ばれる)でのみ染色され、未治療マウスの脾臓細胞は死細胞染色補償のための死死細胞/生細胞ミックスを調製するために使用される。 細胞周期分析のための陽性制御は、未治療マウスからのBMサンプルからなる;未治療およびワクチン接種マウスからの脾臓およびLNサンプルを含む 実験サンプル 。
注: 十分な数の gag 固有 CD8 T 細胞を分析するのに十分な脾臓および LN 細胞があることを確認してください。3つのワクチン接種マウスからプールされた脾臓細胞およびプールされたLN細胞を使用し、それぞれ3×106細胞を含むプールされた細胞の2つ以上の同一サンプルを染色することがしばしば必要である 。Hoechst染色のステップで同一のサンプルをマージします。同様に、3匹の未治療マウスから脾細胞とLN細胞を染色し、最後に同一のサンプルを結合する。無処置マウスから脾臓細胞の 染色されていないサンプル を取っておき、器具や補償のセットアップに使用します。 - 死んだ細胞染色補償のために死んだ/生きた細胞の混合物を準備する(細胞のこのミックスは死んだ細胞色素でのみ染色される)。
- 水浴を65°Cで熱します。
- 脾臓細胞のアリコートを取る (〜3 × 106).
- 細胞懸濁液をマイクロフュージチューブに移し、65°Cの水浴に5分間置き、すぐに10分間氷の上に置きます。
- 熱死滅した細胞を生きた脾臓細胞(約3×106)と1:1の比率で混合し、混合物の半分を96のウェルラウンドボトムプレートに移します(死細胞染色制御のために10個の細胞/ウェルを×。
- 実験サンプルの死細胞染色、細胞周期解析のための陽性制御、死細胞/生細胞混合
- 染色スキーム(ステップ4.1)に従って脾臓、LN、BM細胞(3×106細胞/ウェル)、死んだ/生きた細胞ミックス(セクション4.2)を96ウェルラウンドボトムプレートに移し、400×gで遠心分離機を4°Cで3分間移動させます。
- 各細胞ペレットをPBSで希釈した死細胞色素50μLで再懸濁し、直ちに3回上下にピペット処理して再懸濁する。
- 光から保護された4°Cで30分間インキュベートします。
- 細胞を染色バッファーで 2 回洗浄します。200 μL で初めて、2 回目は 250 μL です。各洗浄遠心分離機のプレートを400×gで4°Cで3分間洗浄する。
- 上清を捨て、細胞ペレットを20μLのPBSで再懸濁する。
- 膜細胞染色は主要組織適合性複合体(MHC)-ペプチド多数体およびmAbsを有する。
- 染色スキーム(フローサイトメーター設定、 表1)に従って必要な量を考慮して、次の試薬を準備します。
- 適切な希釈に従って染色バッファーにmAb 2.4G2を希釈する( 材料表を参照)。染色するサンプルごとに、この希釈液の10 μLを使用してください。
注: 2.4G2 mAb は、FcγIII および FcγII 受容体に対する免疫グロブリンの非抗原特異的結合をブロックします。 - 染色バッファーで H-2k(d) AMQMLKETI アロフィコシアニン (APC) ラベル付き四量体 (Tetr-gag) を希釈して、適切な希釈を得る ( 材料表を参照)。染色するサンプルごとに、この希釈液の20 μLを使用してください。
- 適切な希釈に従って、染色バッファー内の mAbs を希釈して抗体ミックスを準備します ( 材料表を参照) 滴定実験で以前に決定されています;染色するサンプルごとに、この抗体ミックスの20 μLを使用してください。
注:ここでは、抗CD3eペリジンクロロフィルタンパク質(PerCP-Cy5.5)(クローン145-2C11)、抗CD8aブリリアント紫外線(BUV805)(クローン53-6.7)、および抗CD62Lフィコエリスリンシアニン7(PECy7)(クローンMEL-14)を使用した。
- 適切な希釈に従って染色バッファーにmAb 2.4G2を希釈する( 材料表を参照)。染色するサンプルごとに、この希釈液の10 μLを使用してください。
- 以前希釈した2.4G2 mAb(ステップ4.4.1.1)の10μLを加え、光から保護された4°Cで10分間インキュベートします。
- 以前に希釈したテトルギャグAPC(ステップ4.4.1.2)の20 μLとH-2k(d)AMQMLKETIフィコエリスリン(PE)ペンタマー(ペントギャグ)の10 μLを加えます。光から保護された4°Cで15分間インキュベートします。
- 20 μLの前に調製した抗体ミックス(ステップ4.4.1.3)を加え、光から保護された4°Cで15分間インキュベートします。
注:したがって、最終体積はウェルあたり80 μLです(ステップ4.3.5、ステップ4.4.2から4.4.4)。 - 200 μLの染色バッファーで細胞を洗浄します。4°Cで5分間400×gで遠心分離機。
- 250 μLの染色バッファーで細胞ペレットを再懸濁し、細胞懸濁液を5 mLチューブに移します。チューブに1mLの染色バッファーを加え、遠心分離機を400×gで5分間4°Cで5分間加えます。
- ホーチストチャネルを補償するために使用するBM細胞(3×106細胞)(細胞サンプルのリストを参照してください4.1)のアリコートを取り、(Hoechst 33342は紫外線レーザー(フローサイトメーター設定(表2)によって励起される)、細胞懸濁液を5mLチューブに移します。 チューブに1mLの染色バッファーを加え、遠心分離機400×gを4°Cで5分間加えます。
- 染色スキーム(フローサイトメーター設定、 表1)に従って必要な量を考慮して、次の試薬を準備します。
5. 固定/透過
- メーカーの指示に従って、固定/透過濃縮濃縮物の1部を固定/透過希釈希釈液の3部で希釈して、新鮮な固定/透過バッファーを調製します。
- 上清とパルス渦を捨てて、ペレットを完全に分解します。
- 各チューブに、作りたての固定/透過性バッファーの1 mLを加え、未染色の脾臓細胞(3 x 106)を含むチューブ、細胞サンプルのリスト、セクション4.1、および渦を参照してください。
- 4°Cで16時間インキュベートする。
注: プロトコルはここで一時停止することができます。
6. 細胞内染色
- Ki67染色
- メーカーの指示に従って、パーメアビライゼーションバッファー10xを蒸留水で希釈して新鮮な透過バッファー1xを調製します。使用前に、パーメアビライゼーションバッファー1xは、0.45 μmフィルタを通してフィルタ処理して、凝集を除去する必要があります。
- 希釈mAb Ki67フルオレセインイソチオシアネート(FITC)(クローンSolA15)は、滴定実験(サンプル当たり100μLの最終体積)で以前に決定されたパーメアビライズバッファー1x( 材料表を参照)に含まれる。
- 各チューブに透過バッファー1xの3mLを加え、400×gで遠心分離機を室温(RT)で5分間加えます。
- 上清を破棄し、ステップ6.1.3を繰り返します。
- 上清を捨て、以前に希釈したmAb Ki67 FITCの100μLで細胞ペレットを再懸濁します(ステップ6.1.2)。
- RTで30分間インキュベートし、光から保護します。
- 4 mLのパーメビライゼーションバッファー1xで細胞を2回洗浄します。各洗浄用遠心分離機を400×gでRTで5分間洗浄します。
- 以下の体積を考慮してPBSの細胞ペレットを再懸濁します:350 μLのサンプルをフローサイトメーターで直接取得します。250 μLのPBSをフローサイトメトリーの直前にHoechstでインキュベートするサンプル用(セクション6.2)。
- DNA染色
- 各サンプルに4 μg/mLのHoechstを250 μLずつ加えます(最終濃度のHoechstは2 μg/mL)。
注:PBSで250μLの2つ以上の同一サンプルを調製した場合、このステップでそれらをマージし、PBSで4 μg/ mLのHoechst溶液の等容量を追加します(Hoechstの最終濃度は2μg/mLです)。細胞の数はDNA染色工程に大きく影響します。各サンプルで同じセル番号を使用します。細胞数が少し減少しても(例えば、以前の洗浄工程での細胞喪失による)結果として、DNAに対するHoechst結合が高くなり、Hoechst強度が高くなることに注意してください。 - RTで15分間インキュベートし、光から保護します。
- サンプルを400×gで5分間RTで遠心分離します。
- 細胞ペレットを350μLのPBSで再懸濁します。
- 各サンプルに4 μg/mLのHoechstを250 μLずつ加えます(最終濃度のHoechstは2 μg/mL)。
7. 報酬ビーズサンプルの調製
- 5μLの抗体を、適宜染色バッファーに希釈してmAbを調製する。
注: 実験で使用したフルオロクロム共役 mAb ごとに、対応する補正ビード サンプルを準備します。 - ボルテックスネガティブコントロールと抗ラット/ハムスターIg、κコンプビーズ使用前に.
- 各サンプルに対して、ネガティブコントロールコンプビーズの1滴(約20μL)とアンチラット/ハムスターIg,kコンビーズを1滴導入します。
- 5 μLの希釈済み抗体(ステップ7.1)をチューブに加え、上下にピペットします。
- 光から保護された4°Cで15分間インキュベートします。
- 2 mLの染色バッファーでサンプルを洗浄します。4°Cで5分間400×gで遠心分離機。
- 上清を捨て、各チューブと渦に500μLのPBSを加えてペレットを再懸濁します。
8. フローサイトメーターでの計測器と補償のセットアップと実験サンプルの取得
メモ:サイトメーターの構成については、フローサイトメーターの設定(表2)を参照してください。
- 一般的な計器と補償の設定
- サンプル取得用のソフトウェアを開き(「 材料表」を参照)、ワークスペースリボンセクションの [新規実験 ]をクリックし、[ 新規空白実験]を選択して新しい実験を作成します。
- 作成した実験をダブルクリックして開きます。
- [サイトメーター設定]ウィンドウで、[パラメータ]をクリックし、前方散乱(FSC)およびサイドスキャッタ(SSC)パラメータを含む染色パネルで使用されるすべてのチャンネル(例えば、PE、APCなど)を選択します。
- ログスケールのチェックを外して、Hoechstパラメータとして線形スケールを選択し、FCS、SSC、およびHoechstの電圧パルスの幅(W)をチェックします。
注: すべてのパラメータは、線形スケールの FSC および SSC を除き、対数 (対数) スケールで既定で表示されます。すべてのパラメータは、電圧パルスの面積(A)と高さ(H)で分析されます。 - グローバル ワークシートで、x軸にFSC-A、Y軸にSSC-Aを持つドットプロットを作成します。
- 取得ダッシュボードの [データの取得] をクリックして、染色されていない脾臓サンプルを実行します。
- [パラメータ]セクションの電圧値を変更してセルを視覚化し、グローバルワークシートのワークスペースツールバーのポリゴンゲートをクリックして、FSC-A/SSC-Aドットプロットに表示されるすべてのセルを選択するゲートを作成するには、適切なFSCおよびSSC設定を設定します。
- X軸上の各蛍光パラメータを持つヒストグラムでゲートされた細胞を表示します。
- 染色されていない完全に染色された脾臓サンプルを実行して、蛍光検出器(PMT)を調整し、各蛍光パラメータに対して染色された細胞の陰性シグナルと陽性シグナルの間で明確な分離を行います。
- 報酬の設定を実行するには、ワークスペース リボンの [実験 ] をクリックし、[ 補正の設定] セクションで [ 報酬コントロールの作成] を選択します。[ 未染色コントロール チューブ/ウェルを含める ] チェック ボックスをオフにし 、[OK]をクリックします。
注: この操作により、 補正コントロール という名前の検体と、選択した各パラメータに対応する複数のシートを含む 標準ワークシート が作成されます。 - 補正ビードのサンプルを実行します(セクション7を参照)。 Cytometer ウィンドウの FSC パラメータの電圧値と取得しきい値 5,000 を変更してビーズを視覚化するために適切な FSC および SSC 設定を設定します。
- ビードの母集団の P1 ゲートを調整し、正と負のピークが両方とも x 軸に表示されることを確認します。各補正ビードサンプルに対してこの操作を繰り返し、最後に取得ダッシュボードの[データを記録]をクリックして各サンプルファイルを記録します(各サンプルに対して少なくとも5,000件のイベントを記録します)。
- 各記録ビードサンプルに対して、P2とP3のゲートを正と負のピークにそれぞれ設定します。
- 補正のためにセルサンプルを実行します(ステップ 4.2 および 4.4.7、およびセクション 5 と 6 を参照)。FSC および SSC 電圧としきい値を変更してセルを視覚化し、P1 ゲートを調整し、最後に各サンプル ファイルを記録します (10,000 以上のイベントを記録します)。P2 と P3 のゲートをそれぞれ正のピークと負のピークに設定します。
注: Hoechst チャネルの補正のために、負のピーク(P3)として G0/G1を、正の 1 つとして G2/Mを使用します(P2)。 - ワークスペース リボン セクションで [実験 ] をクリックし、[ 補正の設定] セクションで [ 報酬の計算] を選択します。
- 作成した補正設定に名前を付け、リンクし、現在の実験に保存します。
- 実験サンプル取得
- ブラウザのツールバーの [ 新規標本 ] をクリックして標本を開き、 グローバルワークシートでギャティング戦略を作成します。
注: サンプル取得の格言戦略は、 図 3 およびセクション 9 で説明したサンプル分析の場合と同様です。 - X 軸上に CD3-A を使用して、 すべてのイベント の母集団をヒストグラムで表示します。 間隔ゲート を作成して、CD3+ セルのみを選択します。
- 取得ダッシュボードで、ストレージ ゲートを LN サンプルのすべてのイベントとして、脾臓サンプルの場合は [すべてのイベント]または [CD3+ セル] を選択します。
- 実験サンプルを低速で実行し、最後に、ワクチン接種されたマウスから各サンプルに対して少なくとも100〜200個の抗原特異的CD8 T細胞を収集することを確認するすべてのファイルを記録します。
注:実験サンプルのファイルサイズは、特に抗原特異的CD8 T細胞の周波数が低い場合は、通常、大きい(30〜120 MB)です。したがって、100~200個以上の抗原特異的CD8 T細胞を記録するために、多数のイベント(>1×106)を収集する必要があります。大きなファイルは、後続のデータ分析プロセスの速度を低下させる可能性があります。脾臓サンプル内の CD3+セルのみの取得 (上記のステップ 8.2.2 を参照) は、ファイルサイズを小さくしておくと便利です。 - 細胞周期分析のための陽性制御、すなわち、未処理マウスからのBMサンプルを実行して記録する。
- ブラウザのツールバーの [ 新規標本 ] をクリックして標本を開き、 グローバルワークシートでギャティング戦略を作成します。
9. データ分析
- ソフトウェアを開き ( 材料表を参照)、ワークスペース リボン セクションの [グループの作成 ] をクリックして、分析するさまざまな臓器に対応するさまざまなグループを作成します (つまり、グループ "a-LNs" を作成します)。「b-脾臓」;"c-BM")。
注: 新しく作成されたグループはグループリストに表示され、「補正」グループはソフトウェアによって自動的に生成されます。 - グループ名をダブルクリックして「 グループの変更 」ウィンドウを開き、新しく作成したグループが同期されていることを確認します。ない場合は、関数 の同期にチェックマークを挿入します。
- 各 .fcs ファイルを対応するグループにドラッグします。
- 「a-LNs」グループから始まる格子戦略を作成します。
- グループ内の完全に染色されたサンプルをダブルクリックしてグラフウィンドウを開きます。x 軸と y 軸は、fcs ファイルと同様にラベル付けされます (フローサイトメーターの設定、 表 2を参照)。
- このサンプルで獲得したイベントの総数を、x 軸に DNA-A、y 軸に DNA-W を含むドット プロットで表示します。
- グラフ ウィンドウの格言ツール セクションで [四角形 ] をクリックして、単一セルの母集団のみを選択します。
注: 単一細胞の DNA-A 値は次のとおりです: 2N (低): 2N から 4N (中間) の間、または 4N (高) に等しいのに対し、DNA-W 値は全て同じです ( 図 3のステップ 1)。 - 矩形ゲートの中央をダブルクリックすると、X 軸に FSC-A パラメータを持ち、y 軸に死細胞色素を使用して、単一のセルをドット プロットに表示します。
- グラフ ウィンドウの格言ツール セクションで [ポリゴン ] をクリックして、ライブ セルの母集団のみを選択します。生細胞は死細胞色素に対して負である( 図3のステップ2)。
- ポリゴン ゲートの中央をダブルクリックすると、X 軸に FSC-A パラメータ、Y 軸に SSC-A パラメータが付いたドット プロットのセルが表示されます。
- [Rectangle]をクリックし、そのグラフ12に 1 つのライブ セルをすべて含める "リラックスした" ゲートを作成します (図 3の手順 3)。
- 「リラックスした」ゲートの中央をダブルクリックすると、X 軸に CD3、y 軸に CD8 を使用したドット プロットのセルが表示されます。
- [ポリゴン] をクリックして CD3+CD8+セルを選択します (図 3のステップ 4)。
- CD3+CD8+ ゲートの中央をダブルクリックすると、X軸に Tetr-gag、Y軸にペントギャグを使用して、ドットプロットのセルが表示されます。
- 多角形をクリックして抗原特異的CD8 T細胞(テトルギャグとペントギャグの両方に陽性)を選択します(図3のステップ5)。
- gag 固有ゲートの中央をダブルクリックすると、X 軸に DNA-A、Y 軸に Ki67 を持つドットプロットのセルが表示されます (図 4)。
- グラフ ウィンドウの格言ツール セクションで [クワッド ]をクリックして、異なるセルサイクルフェーズのセルを選択します。
注:G0相の細胞はKi67neg-DNA低細胞(左下象限)です。G1の細胞は Ki67pos-DNA 低 (左上象限) です。S-G2/Mの細胞は、Ki67pos-DNA中間/高(右上象限)である(図4)。 - 1 つのサンプルで作成したゲーティングストラテジーを対応するグループにコピーして、グループのすべてのサンプルにゲートを適用します。
- 「a-LN グループ」について、手順 9.5 ~ 9.18 を繰り返します。
- すべてのゲートが「b-脾臓」グループの各サンプルに適していることを確認します。BM細胞間の細胞周期を解析するには(正のコントロール)、「リラックスした」ゲートの中央をクリックして、X軸にDNA-A、Y軸上にKi67を持つドットプロットの細胞を表示します。
- すべてのゲートが3つのグループの各サンプル(すなわち、脾臓、LN、およびBMからの細胞に対して)に適していることを確認してください。
注: 単一細胞母集団ゲート(ステップ 9.7)とセル周期用クワッドゲート(ステップ9.17)は、主にサンプル間のHoechst染料強度のわずかな差が考えられるため、異なるサンプルで異なるゲート座標を有する可能性があります(セクション6.2)。このため、各サンプルの単一セル母集団ゲートと、セル周期のクアッド ゲートを修正する必要がある場合があります。これは、グループ名をダブルクリックし、グループのプロパティから同期を削除します。この操作により、グループの他のすべてのサンプルで同じゲートを修正することなく、1つのサンプル内のゲートを変更できます。同期の削除後、必要に応じてゲートを変更します。 - この解析で得られた結果を視覚化するには、ワークスペース リボン セクションの [レイアウト エディター ] をクリックして開きます。サンプルペインのゲーティングストラテジーの各ゲートをレイアウトエディタにドラッグし、ゲーティング戦略の順序に従ってプロットを配置します。必要に応じて、レイアウト内の対応するプロットをダブルクリックし、[グラフ定義]ウィンドウで適切な タイプを選択して 、グラフ タイプを変更します。
- [グループ]をクリックし、レイアウトリボンの関数を反復して、各臓器で得られた結果を視覚化し、異なるサンプルを比較します。
Representative Results
図1に要約したプロトコルによれば、バルブ/cマウスの脾臓、RN、およびBM由来の細胞の細胞周期相を、蛍光DNA色素、Hoechst、および抗Ki67 mAbを用いて分析した。この染色により、細胞周期の次の段階で細胞の分化が可能となった:G 0(Ki67neg、DNAlowと定義されたDNAの2N)、G 1(Ki67pos、DNAlow)、およびS-G2/M(Ki67pos、2Nと4Nの間で構成されるDNA含有量、またはDNA中間/高と定義されるDNAの4Nに等しい)。
まず、BM細胞の細胞周期解析を行い、以前に公表された結果13,14を再現し、次に目的の細胞、すなわちCD8 T細胞を解析した。図2は、BM細胞の細胞周期解析の代表的な例を示す(図2A)。このプロトコルは、G0/G1およびG2/MDNAピークの低変動係数(CV)をもたらし、DNA染色の優れた品質を示す(図2B、CV<2.5との例を示す;CVは、すべての実験において常に5)<5)
次に、ワクチン接種を受けたマウスから抗原特異的CD8 T細胞に同じプロトコルを適用しました。BALB/cマウスは、プライミングにChad3-gagを使用し、HIV-1ギャグを運ぶように設計されたMVAギャグを増強することによって、HIV-1の抗原ギャグに対してワクチン接種を行った。日目(d)3ポストブーストでは、脾臓と排出LNからのギャグ特異的CD8 T細胞の頻度を分析した。我々は、免疫応答の初期段階におけるT細胞に対する新たに定義された格子戦略を利用したが、これは従来の戦略とは対照的に、高度に活性化された抗原応答性CD8 T細胞12を検出するのに適している。その後5つのステップで新しい戦略を実行しました。ステップ1では、DNA-A/-Wゲートでダブレットまたは凝集体を除外し、ステップ2では死細胞マーカー排除によって生細胞を同定した。ステップ3では、正規狭いリンパ球ゲート12の代わりに非従来型の「リラックスした」FSC-A/SSC-Aゲート(図3A)を用いて対象集団を同定した。CD3+CD8+細胞をギガティングした後 (図 3Aのステップ 4)、2 つの異なる MHC マルチマー、すなわち、ペントギャグと Tetr-gag (図 3Aのステップ 5) を使用して、ギャグ特異的 CD8 T 細胞を同定しました。ワクチン接種マウスにおけるギャグ特異的CD8 T細胞検出の感度を向上させるために、1個ではなく2つのマルチマーを用いたが、未治療マウスにおける染色の背景を増加させることなく(図3B及びC、ステップ5)。したがって、ワクチン接種を受けたマウス(それぞれ0.46%および0.29%の抗原特異的CD8 T細胞、図3BおよびC)から、未治療マウス(それぞれ0.00%および0.00%の抗原特異的CD8 T細胞)を同定した。
特に、このプロトコルは、LNの抗原特異的CD8 T細胞ゲートと未治療マウスの脾臓(通常0.00%および最大0.02%)のバックグラウンドを非常に低くすることができました。ギャグ特異的とギャグ特異的ではないFSC-A/SSC-Aプロットの比較は、ギャグ特異的細胞が高いSSC-AおよびFSC-A(図3D)を持っていることを示し、これらの細胞を捕捉するために「リラックスした」FSC-A/SSC-Aゲートを使用する必要性を確認した。次に、異なる細胞周期期におけるギャグ特異的CD8 T細胞の割合を評価した(図4A)。脾臓内のギャグ特異的CD8 T細胞および排水中のLNのさらに多くの細胞は、3日目のポストブースト(それぞれ18.60%と33.52%)でS-G2/M相の細胞の高い割合を含むことがわかりました。
さらに、S-G2/M相のギャグ特異的CD8 T細胞は、同じ器官から全CD8T細胞に重ね合わされた場合、FSC-AおよびSSC-Aが高いことがわかりました(図4B)。ギャグ特異的CD8T細胞によるCD62L発現は低く、活性化T細胞に対して期待通り、LNs中のG0内の数個の細胞を除く(図4C)。全体として、これらの結果は、「緩和された」ゲート(図3A、B、およびCのステップ3)が、増殖抗原特異的CD8T細胞12のすべてを含む必要があることを確認した。このプロトコルは、分析時の抗原特異的CD8 T細胞の細胞周期期の「スナップショット」評価と、異なる細胞周期期の細胞によるCD62L発現の「スナップショット」評価にとって極めて有用であった。

図1:抗原特異的CD8T細胞の細胞周期解析用プロトコルのスキームこの図の大きなバージョンを表示するには、ここをクリックしてください。

図2:BM細胞の細胞周期解析未治療のBalb/cマウスからのBM細胞を染色し、フローサイトメトリーによって分析した。(A)格言戦略の例DNA-A/-Wプロット(左)の単一細胞にゲートを付け、その後死細胞色素排除(中央)によって生細胞にゲートを付けた。その後、すべてのBM細胞に「リラックスした」FSC-A/SSC-Aゲートを使用しました(右)。(B)BM細胞の細胞周期解析例(左)。Ki67とDNA染色の組み合わせを使用して、細胞周期の次の段階で細胞を同定しました:G0(左下象限、Ki67neg-DNAlow細胞)、G1(左上象限、Ki67pos-DNAlow)、S-G2/M(右上象限、Ki67pos-DNA中間/高)。Ki67 mAb(中央)およびDNAヒストグラム(右)の蛍光マイナス1(FMO)制御が示されている。DNAヒストグラムプロットでは、左右のゲートはそれぞれG0/G1とG2/MDNAピークに対応し、その数は各ピークの変動係数(CV)を表します。他のすべてのプロットでは、数字は示されたゲートのセルパーセンテージを表します。図は、5の中の1つの代表的な実験を示しています。各実験では、3匹のマウスからプールされたBM細胞を分析した。この図の大きなバージョンを表示するには、ここをクリックしてください。

図3:LNsおよび脾臓からの抗原特異的CD8 T細胞の解析 Balb/cマウスは、チャド3ギャグで筋肉内(i.m.)をプライミングし、MVAギャグでi.mを後押しした。3日目のブースト後、ワクチン接種および未治療の対照マウスからLNおよび脾臓細胞を排出し、フローサイトメトリーによって染色および分析した。 (A) 単一細胞を同定するための5つのステップにおける格言戦略のスキーム(ステップ1)。生細胞(ステップ2);リンパ球(ステップ3);CD8 T細胞(ステップ4);そしてギャグ特異的細胞(ステップ5)。 (B-C) プロットの例 :(B)RN および (C) 未治療(上)およびワクチン接種(下)マウスの脾臓からの細胞の分析。ステップ1でDNA-A/-Wプロット上の単一細胞を同定した。そして、ステップ2では、死細胞染色除外により生細胞を選択した。ステップ3では、リンパ球に非正規の「リラックスした」ゲートを使用しました。ステップ4では、CD3とCD8の二重発現によりCD8 T細胞を同定した。次に、フルオロクロムラベル付きH-2kd-gag-Pentamer(Pent-gag)とH-2kd-gag-テトラマー(Tetr-gag)をそれぞれ結合する能力に基づいて、ステップ5でギャグ特異的細胞とギャグ特異的でない細胞を同定しました。 (D) 上記のようにガッティングした後のギャグ特異的(青)およびギャグ特異的(灰色)細胞のFSC-A/SSC-Aプロファイル。数値は、示されたゲートのセルのパーセンテージを表します。図は、5の中の1つの代表的な実験を示しています。各実験では、3つのワクチン接種マウスおよび3つの未治療マウスからプールされた脾臓およびプールLN細胞を分析した。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図4:抗原特異的CD8T細胞の細胞周期解析マウスは、図3のようにワクチンを接種し、細胞周期分析のギャグ特異的細胞を3日目の後昇圧で行い、図3のように5段階で食い込んだ後に行った。(A)ワクチン接種マウスのLNs(上)および脾臓(下)からのギャグ特異的CD8 T細胞の細胞周期解析の例細胞周期相は図2Bに示すように同定された。パネルは、G0、G1、および S-G2/M (左)および蛍光マイナス 1 (FMO) の Ki67 mAb (右) のセルを表します。数値は、示されたゲートのセルのパーセンテージを表します。(B) S-G2/M相(赤)のギャグ特異的CD8 T細胞(赤)を示すSC-A/SSC-Aドットプロットは、リンから合計CD3+CD8+ T細胞(灰色)に重ね合せ、ワクチン接種されたマウスの脾臓(下)に重ね合った。(C)G0(緑色)においてギャグ特異的CD8T細胞によるCD62L発現を示すオフセットヒストグラムを、G1(青)において、S-G2/M(赤)で、Nから(上)および脾臓(下)ワクチン接種マウスの。y 軸は、正規化されたイベント数を示します。図は、合計15匹のマウスを用いた5つの独立した実験のうち1つの代表的な例を示している。この図の大きなバージョンを表示するには、ここをクリックしてください。
補足材料: フローサイトメーターの設定。 このファイルをダウンロードするには、ここをクリックしてください。
Discussion
T細胞クローン拡張は集中的に研究されているが、いくつかの側面は未知のままであるが、主にそれを調査するために利用可能なツールは少なく、独自の欠点を有する。この観点から、マウスモデルでワクチン接種後早起き早期に抗原特異的CD8T細胞の細胞周期を解析する高感度フローサイトメトリック法を設定した。このプロトコルは、Ki67とDNA染色の組み合わせに基づいており、以前はマウス13,14におけるBM造血細胞の細胞周期を解析するために使用された。このプロトコルを抗原特異的CD8 T細胞に適応させるためには、DNA染料の選択、異なるサンプル間で同等のDNA染色を得るための適切な条件、データ分析のための格言戦略など、いくつかの重要な問題を考慮する必要がありました。
多くの色素は、DNA染色のために利用可能です, ヨウ化プロピジウムと7-アミノアクチノマイシンDを含む;Hoechstを選んだのは、膜染色とKi67染色に必要な軽度の固定/透過性プロトコルと互換性があったからです。同時に、Hoechstによる染色は、優れた品質のDNAヒストグラムを得ることを可能にし、すなわち、G0/G1およびG2/MDNAピークは、通常他のDNA色素で得られるDNAピークよりも変動係数(CV)がはるかに低かった、例えばDRAQ519。実際、Hoechstは生細胞20でもDNAを染色することができる。
いくつかの戦略は、同じ実験の異なるサンプルでHoechst強度の変動を避けるために使用されました。フローサイトメーターでサンプル集録の直前に、経時の色素強度の低下を最小限に抑えるために、Hoechst染色を行った。多数のサンプルを用いた大きな実験でプロトコルを再現することに興味がある方には、一度に数サンプルのHoechst染色を行うことをお勧めします。もう一つの欠点は、色素とのインキュベーション中に、Hoechstの強度が細胞数によって大きく影響されることです。そのため、DNA染色には、常に同じ数の細胞とサンプル当たりの体積を使用することを強くお勧めします。フローサイトメーターでの取得に多数の細胞が必要な場合は、2つ以上の同一サンプルを調製し、Hoechst染色工程の直前に結合することをお勧めします。
プロトコルの重要なポイントは、データ分析のための格言戦略です。我々は最近、免疫応答の早期にT細胞分析のための新しい戦略を発表し、抗原特異的T細胞12の検出の感度を高めさせた。この戦略を、以下のようにして、ここに示すデータに適用しました。まず、DNA-A/Wプロットで細胞凝集体を除外しました。第二に、死んだ細胞をゲートアウトした後、FSC/SSCプロット(「リラックスゲート」)でかなり大きなリンパ球ゲートを使用しました。この戦略により、高いFSC-AおよびSSC-Aを有するため、現在の格言戦略では通常見逃される高活性抗原特異的CD8T細胞をS-G2/Mに含めることができた。要約すると、データ分析は、活性化/増殖抗原特異的T細胞の感受性検出を得るために不可欠である方法の重要な部分を表す。
この方法は、免疫応答の初期段階で重要なT細胞データが欠落する可能性を防ぎ、T細胞免疫モニタリングのための新しい視点を開きます。将来の改善は、G2とM21の間で分化を可能にするホスホヒストン3の染色を含むかもしれません。現在の制限は、核マーカーKi67の染色には細胞を固定し、透過させなければならないということです。したがって、並べ替えやそれに続く機能解析など、他のタイプの解析にはセルを使用できません。また、Hoechstを含むDNA染料は、通常ゲノムDNA解析を妨げ、この種の評価には適さない。異なる細胞周期相と相関し、生きた細胞に染色できる膜マーカーの同定は、この限界を克服できる。結論として、この方法は、ワクチン接種、感染、免疫媒介性疾患、免疫療法などのいくつかの文脈で活性化/増殖T細胞の評価に大きな可能性を秘めています。
Disclosures
A. フォルゴリとS.カポネは、特許出願WO 2005071093(A3)「チンパンジーアデノウイルスワクチンキャリア」の発明者に指名されているRona Srl.A.ニコシアの従業員です。他の著者は開示するものは何もない。
Acknowledgments
この作品は、Ronea、MIURプロジェクト2017K55HLC_006、アソシアツィオーネ・イタリアーノ・リセルカ・スル・カンクロ(AIRC)から5×1000助成金によって支援されました。以下の四トラマーはNIHテトラマー施設を通じて得られた:APC共役H-2K(d)HIVギャグ197-205 AMQMLKETI。
Materials
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
References
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- Mouse phenotype. , Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020).
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Tags
免疫学と感染症,問題167,抗原特異的CD8T細胞,細胞周期,Ki67,DNA色素,フローサイトメトリー,脾臓,リンパ節,マウスErratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021.
Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
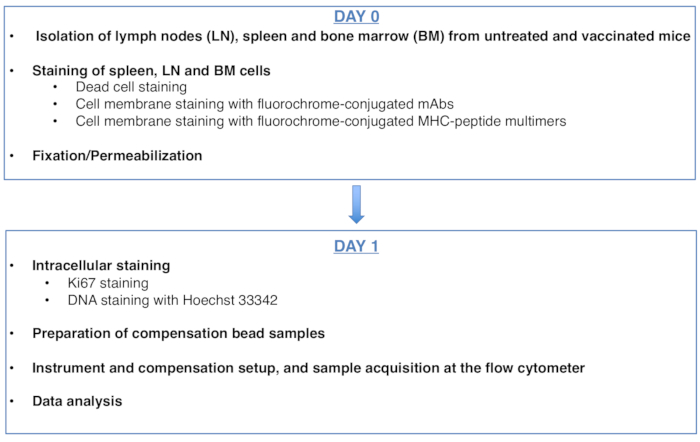
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:
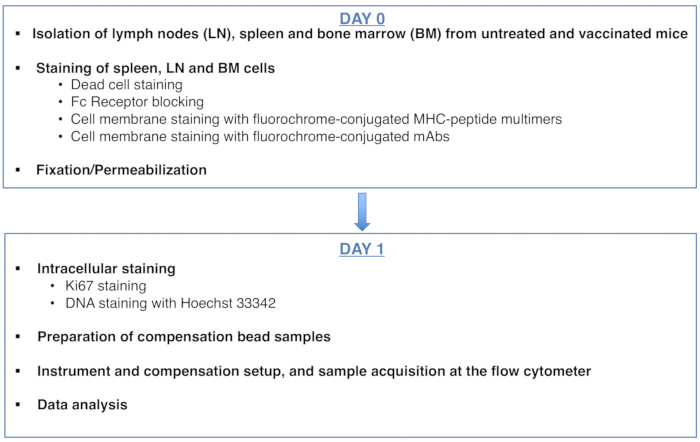
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.



