ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Klonal genleşme antijene özgü T hücre yanıtının önemli bir özelliğidir. Bununla birlikte, antijen yanıt veren T hücrelerinin hücre döngüsü, kısmen teknik sınırlamalar nedeniyle kötü bir şekilde araştırılmıştır. Aşılanmış farelerin dalak ve lenf düğümlerinde klonal olarak genişleyen antijene özgü CD8 T hücrelerini analiz etmek için bir akış sitometrik yöntemi açıklıyoruz.
Abstract
Vivo antijene özgü T hücrelerinin hücre döngüsü, hepsi bazı sınırlamalara sahip olan birkaç yöntem kullanılarak incelenmiştir. Bromodeoxyuridine (BrdU), S fazında veya yakın zamanda tamamlanan hücreleri işaretler ve karboksifluorescein succinimidyl ester (CFSE) bölünmeden sonra kız hücrelerini algılar. Bununla birlikte, bu boyalar analiz sırasında hücre döngüsü fazının tanımlanmasına izin vermez. Alternatif bir yaklaşım, hücre döngüsünün tüm aşamalarında hücreler tarafından yüksek oranda ifade edilen bir işaretleyici olan Ki67'denyararlanmaktır. Ne yazık ki, Ki67 daha fazla farklılaşmaya izin vermez, çünkü S fazında mitoz için taahhüt edilen hücreleri G1'dekilerden ayırmaz bu aşamada kalabilir, bisiklete binin veya G0'a geçin .
Burada, fare ikincil lenfoid organlarda farklı hücre döngüsü aşamalarında T hücrelerinin "anlık görüntüsünü" yakalamak için bir akış sitometrik yöntemini açıklıyoruz. Yöntem, Ki67 ve DNA boyamayı büyük histocompatibility complex (MHC)-peptit-multimer boyama ve yenilikçi bir gating stratejisi ile birleştirir, G0'dakiantijene özgü CD8 T hücreleri ile dalaktaki hücre döngüsünün S-G2 /M fazlarında başarılı bir şekilde ayırt etmemizi ve insan immün yetmezlik virüsünün (HIV)-1 model antijen gagını taşıyan viral vektörlerle aşılamadan sonra farelerin lenf düğümlerini boşaltmamızı sağlar.
Yöntemin kritik adımları, DNA boyasının seçimi ve test duyarlılığını artırmak ve mevcut analiz kriterleri tarafından kaçırılacak yüksek oranda aktive/çoğalan antijene özgü T hücrelerini dahil etmek için gating stratejisiydi. Hoechst 33342 DNA boyası, membran ve hücre içi lekelenmeyi korurken G0/G1 ve G2/M DNA zirvelerinde yüksek kaliteli bir ayrımcılık elde etmemizi sağladı. Yöntem, in vivo T hücre yanıtı hakkında bilgiyi artırmak ve immün izleme analizini geliştirmek için büyük bir potansiyele sahiptir.
Introduction
Naif T hücreleri antijen astarlama üzerine klonal genişleme ve farklılaşmaya uğrar. Farklılaştırılmış T hücreleri, antijen boşluğu ve uzun süreli koruma için anahtar olan antijene özgü belleğin bakımı için gerekli olan efektör işlevlerini görüntüler. Birincil yanıtın ilk adımları sırasında, lenfoid organlarda özel nişler içinde antijen sunan hücrelerle (APC' ler) naif T hücre etkileşimi, klonal genişlemefazı 1,2,3'ü karakterize eden büyük T hücre çoğalmasını teşvik etmek için kritik öneme sahiptir. T hücre-APC etkileşimi, T hücre klonal soyunun miktarını ve kalitesini etkileyen antijen, ko-uyarıcı sinyaller ve çözünür faktörlerin (sitokinler ve kemokinler) konsantrasyonu ve kalıcılığı ile ince bir şekilde düzenlenir4,5,6,7.
T hücre klonal genişlemesinin yoğun çalışmalarına rağmen, antijen astarlı T hücrelerinin tüm hücre döngülerini antijen tanıma alanında tamamlayıp tamamlamadıkları veya hücre döngüsü ilerlemesi sırasında diğer organlara göç edip etmedikleri hala bilinmemektedir. Bu bilgi eksikliği, hücre döngüsü analizi için mevcut araçların özelliklerinden kaynaklanmaktadır. Bunlar arasında nükleer işaretleyiciye özgü monoklonal antikorlar (mAbs), Ki67 ve hücre döngüsünün S fazını geçiren hücreleri tanımlayan hücre boyaları (örneğin, Bromodeoxyuridine (BrdU)) veya kız hücreleri ve ataları (örneğin, karboksifluorescein succinimidyl ester (CFSE)) arasında ayrım yapmak sayabilirsiniz.
Bununla birlikte, CFSE ve BrdU gibi hücre etiketleme boyaları, belirli bir organda bulunan hücrelerin yerel olarak çoğalıp çoğalmadığı veya bölüm8,9'dansonra bu bölgeye geçirilip geçirilmediği konusunda tespite izin vermez. Ayrıca, intranükleer protein, Ki67, sadece G0'daki hücreleri (Ki67 negatif hücreler) diğer hücre döngüsü fazlarındakilerden (Ki67-pozitif hücreler) ayırt edebilir. Bu nedenle, Ki67 analizi aktif çoğalmadaki hücreleri (yani S, G2veya M'de) G1'dekilerden ayırt etmez , bu da hızla bölünmeye ilerleyebilir veya G1'de uzun süre kalabilir veya sessize alma10,11'e geri dönebilir.
Burada, aşılanmış farelerin dalak ve lenf düğümlerinden (LN) antijene özgü CD8 T hücrelerinin12 hücrelerinin hücre döngüsü analizi için yeni bir akış sitometrik yöntemi açıklıyoruz (Şekil 1). Yöntem, daha önce fare kemik iliği (BM) hematopoetik hücrelerinin hücre döngüsünü analiz etmek için kullanılan Ki67 ve DNA boyama kombinasyonundan yararlanır13,14. Burada, cd8 T hücre klonal genişlemesi analizine son yayınlanan yenilikçi gating stratejisi12ile birlikte Ki67 artı DNA boyamayı başarıyla uyguladık. G0,G1'deantijene özgü CD8 T hücreleri ile dalaktaki S-G 2 /M evrelerinde ve aşılanmış farelerin LN'lerini boşaltmada açıkça ayrım yapabildik.
Protocol
Fareler Plaisant Hayvan Tesisi'nde barındırıldı ve çalışma İtalya Sağlık Bakanlığı'nın 1065/2015-PR yetki numarası altında gerçekleştirildi. Protokol, ulusal ve uluslararası yasa ve politikalara göre hayvan bakım yönergelerine uydu (UE Direktifi 2010/63/UE; İtalya Yasama Kararnamesi 26/2014).
1. Orta ve boyama çözeltisinin hazırlanması
- Komple Orta Hazırlanın: Roswell Park Memorial Institute (RPMI) 2 mM glutamin, 100 U/mL penisilin/streptomisin, 50 μM beta-mercaptoethanol ve % 10 hacim/hacim (v/v) fetal sığır serumu (FBS) ile orta
- Boyama Tamponu Hazırlayın:%1ağırlık/hacimli (w/v) sığır serum albümini (BSA) ve2 mM etileniaminetetraasetik asit disodium tuzu (EDTA) ile Ca 2+ /Mg 2+ (PBS) olmadan fosfat tamponlu salin
2. Fare tedavisi
- Prime 7-8 haftalık, dişi Balb/c fareler intramüsküler (i.m.) enjeksiyonu ile insan immün yetmezlik virüsünün (HIV)-1-gag-ifade-şempanze adenoviral vektör (ChAd3-gag) 107 viral parçacıklar doz ile kuadriseps.
- Astarlamadan 1-4 ay sonra, fareleri i.m. hiv-1-gag-ekspresyon modifiye vaksinia Ankara virüsü (MVA-gag) enjeksiyonu ile 106 plak oluşturan üniteler ile güçlendirin.
- 3. günde, güçlendirilmiş fareleri servikal çıkık ile kurban edin ve tedavi edilmemiş farelere paralel olarak analiz edin.
- Kuadriseps (iliak, popliteal ve kasık) ve dalakları güçlendirilmiş ve tedavi edilmemiş farelerden boşaltan LN'leri hasat edin. Ayrıca, BM'yi iki arka bacaktan tedavi edilmemiş farelerden toplayın ve bu BM'yi akış sitometresi ayarları ve hücre döngüsü analizi için pozitif kontrol olarak kullanın (Şekil 2).
NOT: Daha önce açıklandığı gibi ChAd3-gag ve MVA-gag vektörleri oluşturun12,15,16,17.
3. LN, dalak ve BM hücrelerinin boşaltılmasının izolasyonu
- Dalak ve LN hücrelerinin izolasyonu
- İki 15 mL tüpün her birine 5 mL tam orta yerleştirin ve organların toplanması için hazır olarak buzda tutun.
- Servikal çıkık ile yetişkin bir fare kurban.
- Fareyi sırtına yerleştirin ve cilt yüzeyini% 70 v / v etanol ile sterilize edin.
- Kasık LN'lerini toplamak için, makasla karın üzerinde ~1 cm uzunlamasına bir kesi yapın ve kesiği uzunlamasına ile uzatın.
- Cildin iç yüzeyinde kasık LN'lerini görselleştirin ve forsepslerle hasat edin. Kasık LN'lerini adım 3.1.1'de hazırlanan iki 15 mL tüplerden birine yerleştirin.
- Dalağını toplamak için makasla periton kesiği yapın ve dalağını çıkarın. Çevredeki bağ dokusunu kestikten sonra, dalağını 3.1.1 adımda hazırlanan ikinci 15 mL tüpe yerleştirin.
- İlyak LN'leri toplamak için bağırsakları bir kenara hareket ettirın ve iliak LN'leri alt vena kavaya yakın görselleştirin ve ardından forsepsleri kullanarak toplayın. Iliak LN'leri kasık LN'lerini içeren aynı tüpe yerleştirin.
NOT: Boyama için yeterli LN hücresi elde etmek için (bkz. bölüm 4), genellikle popliteal, kasık ve iliyak LN'leri bir fareden havuza almak gerekir. Bu LN'lerin hepsi kuadrisepsleri boşaltıyor (i.m. aşılama alanı). Bu protokol yalnızca bir adet 15 mL havuza sahip LN tüpü kullanır. - Popliteal LN'leri toplamak için, arka bacakların derisini kavrayın ve kasları ortaya çıkarmak için hafifçe aşağı doğru çekin. Ardından, diz ekleminin altındaki kasların arasına toparlamayı yerleştirin ve popliteal LN'leri toplayın.
NOT: 3.1.7'den sonraki nota bakın. - Dalağını 5 mL tam orta ile dolu 60 mm'lik bir kültür kabı içinde 70 μm hücre süzgecine yerleştirin. 5 mL şırınna pistonu kullanarak, organı tamamen dağılana kadar hafifçe püre haline getirin.
- Süzgeci çıkarın ve hücre süspansiyonunu temiz bir 15 mL tüpe aktarın.
- Kültür kabına 5 mL tam orta ekleyin ve tüm hücrelerin kurtarıldığından emin olmak için kabı ve süzgeci dikkatlice yıkayın. Dalak hücresi süspansiyonunun geri kalanı ile 15 mL tüpe havuz.
- Havuza alınan kasık, iliiac ve popliteal LN'ler için, dalak için 3.1.9 ila 3.1.11 adımlarında kullanılana benzer bir prosedürü izleyerek tek bir hücre süspansiyonu hazırlayın.
- 4 °C'de 10 dakika boyunca 400 × g'da santrifüj hücreleri. Üstnatant atın ve HÜCRE peletlerini PBS'de yeniden süzülür.
- PBS'de kırmızı kan hücresi liziz tamponu ve %0,04 v/v trippan mavisi kullanarak Neubauer odası olan hücreleri sayın.
- BM hücrelerinin izolasyonu
- 5 mL komple ortayı 15 mL'lik bir tüpe yerleştirin ve arka ayakların toplanması için hazır olarak buzda tutun.
- Servikal çıkık ile yetişkin bir fare kurban.
- Cilt yüzeyini %70 v/v etanol ile sterilize edin.
- Makasla ventral ciltte ~1 cm enine bir kesi yapın, kesimin her iki tarafındaki cildi sıkıca kavrayın ve arka bacakların kaslarını ortaya çıkarmak için hafifçe aşağı doğru çekin.
- Arka bacakların arkasından cildi ortadan kaldırmak için, fareyi sırtüstü pozisyonda tutmak için kelepçeyi dizinin altına yerleştirin ve kasları açığa çıkarmak için yukarı doğru çekin.
- Kemikleri bir arka bacağın iki ekstremitesinde kesin: pelvik/ kalça eklemi ve ayak bileği.
- Her iki arka ayağı da adım 3.2.1'de hazırlanan 15 mL'lik tüpe aktarın. Tüpü buzda tut.
- Arka ayakları 15 mL tüpten alın ve kağıt mendile aktarın. Kaval kemiğini çıkarmak için arka bacakları diz ekleminin hemen altında kesin. Uyluk kemiğini ve kaval kemiğini çevredeki kaslardan parçalara çıkarın, makas kullanarak fazla dokuyu çıkarın ve kağıt mendili ıslatın.
- İç ilik şaftını açığa çıkarmak için kemik uçlarını makasla kesin. Kaval kemiğini ve uyluk kemiğini BM ekstraksiyon tüpüne yerleştirin (bkz. hazırlık 3.2.9.1-3.2.9.218),en geniş ucu altta olacak şekilde.
- Ucun hemen üstündeki hatta ve 100 μL çizgisinde 200 μL pipet ucu kesin.
- Orta kısmı ucun üst, daha büyük bölümüne yerleştirin ve bunu 1,5 mL'lik bir mikroyakıt tüpüne yerleştirin.
- BM ekstraksiyon tüpünü 1 dakika boyunca 800 × g'da döndürün.
- Kemiği atın ve herhangi bir kümeyi çıkarmak için peletin 1 mL tam ortamda kuvvetlice yeniden süzülür. Hücre süspansiyonu, 15 mL tüpün üstüne yerleştirilmiş 70 μm'lik bir filtreden filtreleyin.
- BM ekstraksiyon tüpünü her seferinde 1 mL tam ortam ile iki kez yıkayın. 70 μm filtreden filtreleyin ve hacmi 3.2.11 adımında elde edilen hücre süspansiyonunun geri kalanıyla bir araya alın.
NOT: Tek bir 15 mL tüp, bir farenin her iki arka ayağından hücreler içerecektir. - 4 °C'de 10 dakika boyunca 400 × g'da santrifüj hücreleri. Üstnatant atın ve HÜCRE peletINI PBS'de yeniden süzülür.
- PBS'de kırmızı kan hücresi liziz tamponu ve %0,04 v/v trippan mavisi kullanarak Neubauer odası olan hücreleri sayın.
4. Dalak, LN ve BM hücrelerinin lekesi
- Lekelenecek hücre örneklerini 3 alt gruba ayırın: tedavi edilmemiş farelerden alınan BM hücrelerinin sadece Hoechst 33342 (bundan böyle Hoechst olarak adlandırılır) ile lekelenecek BM hücreleri ve ölü hücre boyası telafisi için ölü/canlı hücre karışımı hazırlamak için kullanılacak tedavi edilmemiş farelerden dalak hücreleri dahil olmak üzere tazminat için hücre örnekleri; hücre döngüsü analizi için pozitif kontrol, tedavi edilmemiş farelerden bir BM örneğinden oluşan; ve tedavi edilmemiş ve aşılanmamış farelerden dalak ve LN örnekleri içeren deneysel örnekler.
NOT: Gaga özgü CD8 T hücrelerinin yeterli sayıda analizi için yeterli dalak ve LN hücresi olduğundan emin olun. Genellikle aşılanmış 3 fareden havuzlanmış dalak hücreleri ve havuza alınan LN hücrelerini kullanmak ve her biri 3 × 106 hücre içeren iki veya daha fazla özdeş havuza alınan hücre örneğini lekelamak gerekir. Hoechst boyama adımında aynı örnekleri birleştirin. Benzer şekilde, tedavi edilmemiş 3 fareden leke havuzlu dalak hücreleri ve LN hücreleri ve sonunda aynı örnekleri birleştirir. Alet ve kompanzasyon kurulumu için kullanılmak üzere işlenmemiş bir fareden dalak hücrelerinin tespit edilmemiş bir örneğini bir kenara koyun. - Ölü hücre boyası telafisi için ölü/canlı hücre karışımı hazırlayın (bu hücre karışımı sadece ölü hücre boyası ile lekelenecektir).
- Bir su banyosuza 65 °C'de ısıtın.
- Dalak hücrelerinin bir aliquot alın (~3 × 106).
- Hücre süspansiyonu bir mikrofuge tüpüne aktarın, 5 dakika boyunca 65 ° C'de su banyosuna yerleştirin ve hemen 10 dakika boyunca buza yerleştirin.
- Isıdan etkilenen hücreleri canlı dalak hücreleriyle (~3 ×10 6)1:1 oranında karıştırın ve karışımın yarısını 96 iyi yuvarlak alt plakaya aktarın (~3 × ölü hücre boyama kontrolü için10 6 hücre/kuyu).
- Deneysel örneklerin ölü hücre lekesi, hücre döngüsü analizi için pozitif kontrol ve ölü/canlı hücre karışımı
- Dalak, LN, BM hücrelerini (3 ×10 6 hücre/kuyu) ve ölü/canlı hücre karışımını (bölüm 4.2) boyama şemasına (adım 4.1) göre 96 kuyulu yuvarlak alt plakaya aktarın ve 4 °C'de 3 dakika boyunca 400 × g'da santrifüj.
- Her hücre peletini PBS'de seyreltilmiş 50 μL ölü hücre boyasında yeniden ıslatın ve hemen 3 kez yukarı ve aşağı pipetleme ile yeniden hissedin.
- Işıktan korunan 4 °C'de 30 dakika kuluçkaya yat.
- Hücreleri boyama tamponu ile 2 kez yıkayın; ilk kez 200 μL ve ikinci kez 250 μL ile. Her yıkama için plakayı 4 °C'de 3 dakika boyunca 400 × g'da santrifüj edin.
- Üstnatant atın ve hücre peletini 20 μL PBS'de yeniden atın.
- Ana histocompatibility complex (MHC)-peptit multimers ve mAbs ile membran hücre boyama.
- Boyama şemasına göre gerekli hacimleri dikkate alarak (Akış sitometresi ayarları, Tablo 1), aşağıdaki reaktifleri hazırlayın:
- Lekeleme tamponunda mAb 2.4G2'yi uygun seyreltmeye göre seyreltin (bkz. Malzeme Tablosu); her numunenin lekeli olması için bu seyreltmenin 10 μL'sini kullanın.
NOT: 2.4G2 mAb, immünoglobulinlerin FcφIII ve FcφII reseptörlerine antijene özgü olmayan bağlanmasını engeller. - Uygun seyreltmeyi elde etmek için boyama tamponunda H-2k(d) AMQMLKETI allophycocyanin (APC) etiketli tetramer (Tetr-gag) seyreltin (bkz. Malzeme Tablosu); her numunenin lekeli olması için bu seyreltmenin 20 μL'sini kullanın.
- Daha önce titrasyon deneylerinde belirlenen uygun seyreltmeye göre boyama tamponundaki mAb'leri seyrelterek antikor karışımını hazırlayın ( bkz. Malzeme Tablosu); her numunenin lekeli olması için bu antikor karışımının 20 μL'sini kullanın.
NOT: Burada anti-CD3e peridinin klorofil proteini (PerCP-Cy5.5) (klon 145-2C11), anti-CD8a parlak ultraviyole (BUV805) (klon 53-6.7) ve anti-CD62L fikoerythrin siyanin7 (PECy7) (klon MEL-14) kullanılmıştır.
- Lekeleme tamponunda mAb 2.4G2'yi uygun seyreltmeye göre seyreltin (bkz. Malzeme Tablosu); her numunenin lekeli olması için bu seyreltmenin 10 μL'sini kullanın.
- Önceden seyreltilmiş 2.4G2 mAb'nin 10 μL'sini ekleyin (adım 4.4.1.1) ve ışıktan korunarak 4 °C'de 10 dakika kuluçkaya yatın.
- Daha önce seyreltilmiş Tetr-gag APC'nin 20 μL'si (adım 4.4.1.2) ve 10 μL H-2k(d) AMQMLKETI fizikerythrin (PE) pentamerini (pent-gag) ekleyin. Işıktan korunan 4 °C'de 15 dakika kuluçkaya yat.
- Önceden hazırlanmış antikor karışımının 20 μL'sini ekleyin (adım 4.4.1.3) ve ışıktan korunan 4 °C'de 15 dakika kuluçkaya yatın.
NOT: Bu nedenle, son hacim kuyu başına 80 μL'dir (adım 4.3.5, adım 4.4.2 ila 4.4.4). - Hücreleri 200 μL boyama tamponu ile yıkayın. 4 °C'de 5 dakika boyunca 400 × g'da santrifüj.
- Hücre peletini 250 μL boyama tamponunda yeniden süzülür ve hücre süspansiyonu 5 mL tüplere aktarın. Tüpe 1 mL boyama tamponu ekleyin ve 4 °C'de 5 dakika boyunca 400 × g'da santrifüj ekleyin.
- Hoechst kanalını telafi etmek için kullanılacak BM hücrelerinin aliquot'ını (3 ×10 6 hücre) alın (hücre örneklerinin listesine bakın, bölüm 4.1) (Hoechst 33342 ultraviyole lazer (akış sitometre ayarları (Tablo 2) ile heyecanlanır) ve hücre süspansiyonu 5 mL tüpe aktarın. Tüpe 1 mL boyama tamponu ekleyin ve 4 °C'de 5 dakika boyunca 400 × g santrifüjleyin.
- Boyama şemasına göre gerekli hacimleri dikkate alarak (Akış sitometresi ayarları, Tablo 1), aşağıdaki reaktifleri hazırlayın:
5. Fiksasyon/permeabilizasyon
- Üreticinin talimatlarına göre, fiksasyon/permeabilizasyon konsantresinin 1 kısmını 3 parça fiksasyon/permeabilizasyon seyreltici ile seyrelterek taze fiksasyon/permeabilizasyon tamponu hazırlayın.
- Peleti tamamen parçalamak için örneklerin süpernatant ve darbe girdabını atın.
- Her tüpe, lekesiz dalak hücrelerine sahip bir tüp (3 x 106,hücre örneklerinin listesine bakın, bölüm 4.1) ve girdap dahil olmak üzere her tüpe 1 mL taze hazırlanmış fiksasyon / permeabilizasyon tamponu ekleyin.
- 4 °C'de 16 saat kuluçkaya yaslanın.
NOT: Protokol burada duraklatılabilir.
6. Hücre içi boyama
- Ki67 boyama
- Üreticinin talimatlarına göre permeabilizasyon tamponunu damıtılmış su ile 10x seyrelterek taze permeabilizasyon tamponu 1x hazırlayın. Kullanmadan önce, permeabilizasyon tamponu 1x, agregaları ortadan kaldırmak için 0,45 μm filtreden geçirilmelidir.
- Titrasyon deneylerinde daha önce belirlendiği gibi permeabilizasyon tamponu 1x'te (bkz. Malzeme Tablosu)mAb Ki67 floresan izotiyosiyanat (FITC) (klon SolA15) seyreltin (numune başına 100 μL'lik son hacim).
- Her tüpe 3 mL permeabilizasyon tamponu 1x ekleyin ve oda sıcaklığında (RT) 5 dakika boyunca 400 × g'da santrifüjleyin.
- Üstnatant atın ve 6.1.3 adımını yineleyin.
- Süpernatantı atın ve hücre peletini daha önce seyreltilmiş 100 μL'lik mAb Ki67 FITC'de (adım 6.1.2) yeniden atın.
- RT'de 30 dakika kuluçkaya yat, ışıktan korun.
- Hücreleri 4 mL permeabilizasyon tamponu 1x ile 2 kez yıkayın. Her yıkama santrifüjü için RT'de 5 dakika boyunca 400 × g'da.
- Aşağıdaki hacimleri göz önünde bulundurarak hücre peletini PBS'de yeniden diriltin: Doğrudan akış sitometresinde elde edilecek numuneler için 350 μL PBS; Akış sitometrisinden kısa bir süre önce Hoechst ile inkübe edilecek numuneler için 250 μL PBS (bölüm 6.2).
- DNA boyama
- Her numuneye PBS'de 250 μL 4 μg/mL Hoechst ekleyin (Hoechst'in son konsantrasyonu 2 μg/mL'dir).
NOT: PBS'de 250 μL'lik iki veya daha fazla özdeş numunenin hazırlanması durumunda, bu adımda birleştirin ve PBS'de eşit hacimde 4 μg/mL Hoechst çözeltisi ekleyin (Hoechst'in son konsantrasyonu 2 μg/mL'dir). Hücre sayısı DNA boyama adımını büyük ölçüde etkiler. Her örnekte aynı hücre numarasını kullanın. Biraz daha az bir hücre sayısının bile (örneğin, önceki yıkama adımlarındaki hücre kaybı nedeniyle) DNA'ya daha yüksek Hoechst bağlanmasına ve daha yüksek Hoechst yoğunluğuna neden olduğunu unutmayın. - RT'de 15 dakika kuluçkaya yat, ışıktan korun.
- Rt'de 5 dakika boyunca numuneleri 400 × g'da santrifüj edin.
- Hücre peletini 350 μL PBS'de yeniden satın.
- Her numuneye PBS'de 250 μL 4 μg/mL Hoechst ekleyin (Hoechst'in son konsantrasyonu 2 μg/mL'dir).
7. Kompanzasyon boncuk örneklerinin hazırlanması
- Lekeleme tamponunda mAb'yi uygun şekilde seyrelterek antikorun 5 μL'sini hazırlayın.
NOT: Deneyde kullanılan her florokrom konjuge mAb için, karşılık gelen kompanzasyon boncuk örneğini hazırlayın. - Vortex Negatif Kontrol ve Anti-Sıçan / Hamster Ig, φ Comp Boncuk kullanmadan önce.
- Her örnek için, negatif kontrol compbeads bir damla (~20 μL) ve Anti-Rat / Hamster Ig bir damla tanıtmak, k CompBeads.
- Tüpe önceden seyreltilmiş antikorun 5 μL'sini (adım 7.1) ekleyin ve yukarı ve aşağı boruleyin.
- Işıktan korunan 4 °C'de 15 dakika kuluçkaya yat.
- Numuneleri 2 mL boyama tamponu ile yıkayın. 4 °C'de 5 dakika boyunca 400 × g'da santrifüj.
- Süpernatant atın ve her tüp ve girdap içine 500 μL PBS ekleyerek peleti yeniden depola.
8. Akış sitometresinde cihaz ve kompanzasyon kurulumu ve deneysel numune alımı
NOT: Sitometre yapılandırması için akış sitometresi ayarlarına (Tablo 2) bakın.
- Genel enstrüman ve kompanzasyon kurulumu
- Örnek alma için yazılımı açın (bkz. Malzeme Tablosu)ve çalışma alanı şeridi bölümünde Yeni Deneme 'yi tıklatıp Yeni Boş Deneme 'yi seçerek yeni bir denemeoluşturun.
- Oluşturulan denemeyi açmak için çift tıklatın.
- Cytometer Ayarları penceresinde Parametreler'e tıklayın ve boyama panelinde kullanılan İleri Saçılma (FSC) ve Yan Dağılım (SSC) parametreleri de dahil olmak üzere tüm kanalları (örneğin, PE, APC vb.) seçin.
- Günlük ölçeğinin işaretini kaldırarak Hoechst parametresi olarak doğrusal ölçeği seçin ve FCS, SSC ve Hoechst için voltaj darbesinin Genişliğini (W) kontrol edin.
NOT: Doğrusal ölçekteki FSC ve SSC dışında, tüm parametreler varsayılan olarak logaritmik (günlük) ölçeğinde gösterilir. Tüm parametreler, gerilim darbesinin Alanı (A) ve Yüksekliği (H) tarafından analiz edilir. - Genel Çalışma Sayfasında,x ekseninde FSC-A ve y ekseninde SSC-A içeren bir nokta çizimi oluşturun.
- Edinme Panosu'nda Veri Al 'ı tıklatarak, lekelenmemiş dalak örneğini çalıştırın.
- Parametreler bölümündeki voltaj değerlerini değiştirerek hücreleri görselleştirmek için uygun FSC ve SSC ayarlarını ayarlayınve Genel Çalışma Sayfası'nın çalışma alanı araç çubuğundaki Çokgen Kapısı'nı tıklatarak FSC-A/SSC-A nokta çiziminde görüntülenen tüm hücreleri seçmek için bir kapı oluşturun.
- Kapılı hücreleri histogramlarda x eksenindeki her floresan parametresiyle görüntüleyin.
- Floresan dedektörünü (PMT) her floresan parametresi için lekeli hücrelerin negatif ve pozitif sinyalleri arasında net bir ayrım yapacak şekilde ayarlamak için lekesiz ve tamamen lekeli dalak örneklerini çalıştırın.
- Ücret kurulumu gerçekleştirmek için, çalışma alanı şeridinde Deneme'yi tıklatın ve Ücret Kurulumu bölümünün altında Ücret Denetimleri Oluştur 'useçin. Unstained Control Tube/Well'i Ekle seçeneğinin işaretini kaldırın ve Tamam'ı tıklatın.
NOT: Bu işlem, ücret denetimleri adlı bir örnek ve seçilen her parametreye karşılık gelen birkaç sayfa içeren normal bir çalışma sayfası oluşturulmasına neden olur. - Bir ücret boncuk örneği çalıştırın (bkz. bölüm 7); Cytometer penceresinde FSC parametrelerinde voltaj değerlerini ve 5.000 alım eşiğini değiştirerek boncukları görselleştirmek için uygun FSC ve SSC ayarlarını ayarlayın.
- Boncuk popülasyonu üzerindeki P1 kapısını ayarlayın ve pozitif ve negatif tepelerin x ekseninde görünür olup olmadığını kontrol edin. Her ücret boncuk örneği için bu işlemi yineleyin ve son olarak Edinme Panosunda Verileri Kaydet'i tıklatarak her örnek dosyayı kaydedin (her örnek için en az 5.000 olay kaydedin).
- Kaydedilen her boncuk örneği için P2 ve P3 kapılarını sırasıyla pozitif ve negatif tepelere ayarlayın.
- Hücre örneklerini tazminat için çalıştırın (bkz. adımlar 4.2 ve 4.4.7 ve bölüm 5 ve 6). Hücreleri görselleştirmek, P1 kapısını ayarlamak ve son olarak her örnek dosyayı kaydetmek için FSC ve SSC gerilimlerini ve eşik değerini değiştirin (en az 10.000 olay kaydedin). P2 ve P3 kapılarını sırasıyla pozitif ve negatif zirvelere ayarlayın.
NOT: Hoechst kanalının telafisi için negatif tepe (P3) olarak G0/G1'i ve pozitif kanal olarak G2/M'yi (P2) kullanın. - Çalışma alanı şeridi bölümünde Deneme'yi tıklatın ve Ücret Kurulumu bölümünde Tazminatı Hesapla 'yıseçin.
- Oluşturulan ücret ayarını adlandırın, bağlayın ve geçerli denemeye kaydedin.
- Deneysel örnek alımı
- Tarayıcı araç çubuğunda Yeni Örnek 'i tıklatarak bir örnek açın ve Genel Çalışma Sayfası'nda gating stratejisini oluşturun.
NOT: Örnek alımının gating stratejisi, Şekil 3 ve bölüm 9'da açıklanan örnek analizine benzer. - X ekseninde CD3-A bulunan bir histogramda Tüm Olay popülasyonunu görüntüleyin. Yalnızca CD3+ hücrelerini seçmek için bir Aralık Kapısı oluşturun.
- Edinme Panosu'nda, LN örnekleri için Tüm Olaylar olarak depolama kapısını ve dalak örnekleri için Tüm Olaylar veya CD3+ hücreleri'ni seçin.
- Deneysel örnekleri düşük hızda çalıştırın ve son olarak aşılanmış farelerden her örnek için en az 100-200 antijene özgü CD8 T hücresi topladığınızdan emin olan tüm dosyaları kaydedin.
NOT: Deneysel örneklerin dosya boyutu genellikle büyüktür (30-120 MB), özellikle antijene özgü CD8 T hücrelerinin sıklığı düşük olduğunda. Bu nedenle, en az 100-200 antijene özgü CD8 T hücresini kaydetmek için yüksek sayıda olay (1 × 106>) toplanması gerekir. Büyük dosyalar sonraki veri analizi işlemini yavaşlatabilir. Dalak örneklerinde yalnızca CD3+ hücrelerin alınması (yukarıdaki adım 8.2.2'ye bakın) dosya boyutunu daha küçük tutmak için yararlıdır. - Hücre döngüsü analizi için pozitif kontrolü çalıştırın ve kaydedin, yani tedavi edilmemiş farelerden BM örneği.
- Tarayıcı araç çubuğunda Yeni Örnek 'i tıklatarak bir örnek açın ve Genel Çalışma Sayfası'nda gating stratejisini oluşturun.
9. Veri Analizi
- Yazılımı açın (bkz. Malzeme Tablosu)ve çalışma alanı şeridi bölümünde Grup Oluştur'u tıklatarak analiz edilecek farklı organlara karşılık gelen farklı gruplar oluşturun (örneğin, "a-LN" grubu oluşturun; "b-dalak"; "c-BM").
NOT: Yeni oluşturulan gruplar grup listesinde görünürken, "Ücretlendirme" grubu yazılım tarafından otomatik olarak oluşturulur. - Grup adını çift tıklatarak Grubu Değiştir penceresini açın ve yeni oluşturulan grupların eşitlendiğinden olup olmadığını denetleyin. Değilse, Eşitlendiişlevine onay işareti ekleyin.
- Her .fcs dosyasını karşılık gelen grubunda sürükleyin.
- "a-LNs" grubundan başlayarak gating stratejisini oluşturun.
- Grafik penceresini açmak için gruptaki tamamen lekeli örneğe çift tıklayın; x-ve y ekseni FCS dosyalarında olduğu gibi etiketlenir (bkz. akış sitometresi ayarları, Tablo 2).
- Bu örnek için elde edilen toplam olayları x ekseninde DNA-A ve y ekseninde DNA-W bulunan bir nokta grafiğinde görüntüleyin.
- Grafik penceresinin geçiş aracı bölümünde dikdörtgen'i tıklatarak yalnızca tek hücre popülasyonu seçin.
NOT: Tek hücrelerin DNA-A değerleri aşağıdaki gibidir: 2N (düşük): 2N ile 4N (orta) arasında veya 4N'ye (yüksek) eşitken, DNA-W değerleri hepsi için aynıdır (Şekil 3'ün 1.adımı). - X ekseninde FSC-A parametresi ve y ekseninde ölü hücre boyası bulunan bir nokta çiziminde tek hücreleri görüntülemek için dikdörtgen kapının ortasına çift tıklayın.
- Grafik penceresinin gating aracı bölümündeKi Çokgen'i tıklatarak yalnızca canlı hücre popülasyonu seçin. Canlı hücreler ölü hücre boyası için negatiftir (Şekil 3'ün 2.adımı).
- Hücreleri x ekseninde FSC-A parametresi ve y ekseninde SSC-A parametresi bulunan bir nokta çiziminde görüntülemek için çokgen kapının ortasına çift tıklayın.
- Rectangle'a tıklayın ve bu grafik12'deki tüm tek canlı hücreleri dahil etmek için "rahat" bir kapı oluşturun (Şekil 3'ün 3.adımı).
- Hücreleri x ekseninde CD3 ve y ekseninde CD8 ile nokta çiziminde görüntülemek için "rahat" kapının ortasına çift tıklayın.
- Çokgen'e tıklayarak CD3+CD8+ hücreleri seçin (Şekil 3'ün 4.adımı).
- Hücreleri x ekseninde Tetr-gag ve y ekseninde Pent-gag ile nokta çiziminde görüntülemek için CD3+CD8+ kapının ortasına çift tıklayın.
- Poligon'a tıklayarak antijene özgü CD8 T hücrelerini (hem Tetr-gag hem de Pent-gag için pozitif) seçin (Şekil 3'ün 5.adımı).
- Hücreleri x ekseninde DNA-A ve y ekseninde Ki67 ile noktasal bir çizimde görüntülemek için gaga özgü kapının ortasına çift tıklayın (Şekil 4).
- Grafik penceresinin geçiş aracı bölümünde Quad'ı tıklatarak farklı hücre döngüsü aşamalarındaki hücreleri seçin.
NOT: G0 fazındaki hücre Ki67neg-DNA düşük hücreleridir (sol alt kadran); G1'deki hücreler Ki67pos-DNA düşüklüğündedir (sol üst kadran); S-G2/M'deki hücreler Ki67pos-DNA orta/yüksek (sağ üst kadran)(Şekil 4). - Kapıları grubun tüm örneklerine uygulamak için tek bir örnekte oluşturulan gating stratejisini ilgili gruba kopyalayın.
- "A-LN grubu" için 9,5 ile 9,18 arası adımları yineleyin.
- Tüm kapıların "b-dalak" grubunun her örneği için uygun olup olmadığını kontrol edin. BM hücreleri arasındaki hücre döngüsünü (pozitif kontrol) analiz etmek için, hücreleri x ekseninde DNA-A ve y ekseninde Ki67 ile bir nokta grafiğinde görüntülemek için "rahat" kapının ortasına tıklayın.
- Tüm kapıların 3 grubun her örneği için uygun olup olmadığını kontrol edin (yani dalak, LN ve BM'den hücreler için).
NOT: Hücre döngüsü için tek hücreli popülasyon kapısı (adım 9.7) ve Dörtlü kapı (adım 9.17), esas olarak numuneler arasındaki Hoechst boya yoğunluğunun olası küçük farkları nedeniyle farklı örneklerde farklı kapı koordinatlarına sahip olabilir (bölüm 6.2). Bu nedenle, her örnekteki hücre döngüsü için Tek hücre popülasyon kapısını ve Dört kapıyı değiştirmek gerekebilir. Bu aşağıdaki gibi yapılacaktır: grup adına çift tıklayın ve eşitlemeyi grup özelliklerinden kaldırın. Bu işlem, grubun diğer tüm örneklerinde aynı kapıları değiştirmeden bir numunedeki kapıların değiştirilmesine izin verir. Eşitlemeyi kaldırma işleminden sonra, gerektiğinde kapıları değiştirin. - Bu çözümlemeyle elde edilen sonuçları görselleştirmek için, çalışma alanı şeridi bölümündeki Düzen Düzenleyicisi'ni tıklatarak açın. Örnek bölmedeki gating stratejisinin her kapısını düzen düzenleyicisine sürükleyin ve çizimleri gating stratejisinin sırasına göre yerleştirin. Gerekirse, düzende karşılık gelen çizimi çift tıklatıp Grafik Tanımı penceresinde uygun Türü seçerek grafik türünü değiştirin.
- Her organda elde edilen sonuçları görselleştirmek ve farklı örnekleri karşılaştırmak için Grup'a tıklayın ve düzen şeridindeki işlevlere göre yinele.
Representative Results
Balb/c farelerinin dalak, LN ve BM hücre döngüsü aşamaları, Şekil 1'de özetlenen protokole göre floresan DNA boyası, Hoechst ve bir Anti-Ki67 mAb kullanılarak analiz edilmiştir. Bu boyama, hücre döngüsünün aşağıdaki aşamalarında hücrelerin farklılaşmasına izin verdi: G0 (Ki67neg, DNAlow olarak tanımlanan 2N DNA ile), G1 (Ki67pos, DNAlow) ve S-G2/M (Ki67pos, 2N ile 4N arasında bir DNA içeriğine sahip veya DNAintermediate/high olarak tanımlanan 4N DNA'ya eşit).
Daha önce yayınlanan sonuçlar13,14'ü çoğaltmak için önce BM hücrelerinin hücre döngüsü analizini yaptık ve daha sonra ilgi çekici hücreleri, yani CD8 T hücrelerini analiz ettik. Şekil 2, BM hücrelerinin hücre döngüsü analizinin tipik bir örneğini göstermektedir (Şekil 2A). Protokol, DNA boyamanın mükemmel kalitesini gösteren düşük bir G0/ G1 ve G2/ M DNA zirveleri değişim katsayısı (CV) sağladı (Şekil 2B, CV < 2.5 ile bir örnek gösterdi; CV her zaman tüm deneylerde 5 <).
Daha sonra aşılanmış farelerden antijene özgü CD8 T hücrelerine aynı protokolü uyguladık. BALB/c fareleri, Astarlama için Chad3-gag ve takviye için MVA-gag kullanılarak HIV-1'in antijen gag'ına karşı aşılandı, her ikisi de HIV-1 gag taşımak için tasarlandı. Gün (d) 3 post-boost'ta, dalaktan gag spesifik CD8 T hücrelerinin sıklığını analiz ettik ve LN'leri boşalttık. Bağışıklık yanıtının erken evresinde T hücreleri için yeni tanımlanan gating stratejisinden yararlandık, bu da geleneksel stratejinin aksine, yüksek oranda aktive edilen antijen yanıt veren CD8 T hücrelerini tespit etmek için uygundur12. Yeni stratejiyi sonraki beş adımda uyguladık. 1. adımda, DNA-A/ -W kapısı ile çiftleri veya agregaları hariç tuttuk ve 2. 3. adımda, kurallı dar lenfosit kapısı12yerine geleneksel olmayan "rahat" bir FSC-A / SSC-A kapısı (Şekil 3A) kullanarak ilgi nüfusunu belirledik. CD3+CD8+ hücrelerde (Şekil 3A'nın 4.adımı) gating yaptıktan sonra, pent-gag ve Tetr-gag (Şekil 3A'nın5. adımı) olmak üzere iki farklı MHC multimer kullanarak gag'a özgü CD8 T hücrelerini tanımladık. Tedavi edilmemiş farelerde boyama arka planını artırmadan, aşılanmış farelerde gag spesifik CD8 T hücre algılama hassasiyetini artırmak için bir yerine iki multimer kullandık(Şekil 3B ve C, adım 5). Bu nedenle, tedavi edilmemiş fareleri (LN'lerde ve dalaklarda sırasıyla% 0.00 ve% 0.00 antijene özgü CD8 T hücreleri) aşılanmış farelerden (sırasıyla LN'lerde ve dalaklarda% 0.46 ve% 0.29 antijene özgü CD8 T hücreleri, Şekil 3B ve C)başarıyla ayırt ettik.
Özellikle, protokol, LN'lerin antijene özgü CD8 T hücre kapısında ve tedavi edilmemiş farelerin dalaklarında (genellikle% 0.00 ve maksimum% 0.02) son derece düşük bir arka plana sahip olmamızı sağladı. Gag-spesifik ve gag-spesifik olmayan FSC-A / SSC-A arazilerinin karşılaştırılması, gag'a özgü hücrelerin yüksek SSC-A ve FSC-A'ya sahip olduğunu göstermiştir (Şekil 3D), bu hücreleri yakalamak için "rahat" bir FSC-A / SSC-A kapısı kullanma ihtiyacını doğrulamamıştır. Daha sonra farklı hücre döngüsü aşamalarında gag spesifik CD8 T hücrelerinin yüzdelerini değerlendirdik (Şekil 4A). Dalaktaki gag spesifik CD8 T hücrelerinin ve hatta boşaltılan LN'lerdeki daha fazlasının, 3.
Ayrıca, S-G2/M fazlarındaki gag spesifik CD8 T hücrelerinin, aynı organdan toplam CD8 T hücrelerine bindirildiğinde yüksek FSC-A ve SSC-A'ya sahip olduğunu bulduk (Şekil 4B). GAG'a özgü CD8 T hücreleri tarafından CD62L ifadesi, LN'lerdeki G0'daki birkaç hücre dışında, aktif T hücreleri için beklendiği gibi düşüktü (Şekil 4C). Tamamen, bu sonuçlar "rahat" kapının (Şekil 3A, B ve C'nin 3.adımı) çoğalan antijene özgü CD8 T hücrelerinin12'siniiçermesi gerektiğini doğruladı. Protokol, analiz anında antijene özgü CD8 T hücrelerinin hücre döngüsü aşamalarının ve farklı hücre döngüsü aşamalarındaki hücreler tarafından CD62L ekspresyonunun "anlık görüntü" değerlendirmesi için son derece değerliydi.

Şekil 1: Antijene özgü CD8 T hücrelerinin hücre döngüsü analizi için protokolün şeması. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: BM hücrelerinin hücre döngüsü analizi. Tedavi edilmeyen Balb/c farelerinden alınan BM hücreleri akış sitometrisi ile boyandı ve analiz edildi. (A) Gating stratejisi örneği. DNA-A/W grafiğindeki (solda) tek hücrelere ve daha sonra ölü hücre boyası dışlama (ortada) ile canlı hücrelere geçiş yaptık. Daha sonra tüm BM hücreleri için "rahat" bir FSC-A/SSC-A kapısı kullanıldı (sağda). (B) BM hücrelerinin hücre döngüsü analizi örneği (solda). Hücre döngüsünün aşağıdaki aşamalarında hücreleri tanımlamak için Ki67 ve DNA boyama kombinasyonu kullandık: G0 (sol alt kadran, Ki67neg-DNAlow hücreleri), G1 (sol üst kadran, Ki67pos-DNAlow), S-G2/M (sağ üst kadran, Ki67pos-DNAintermediate/yüksek). Ki67 mAb (ortada) ve DNA histogramının (sağda) floresan Eksi Bir (FMO) kontrolü gösterilmiştir. DNA histogramı grafiğinde, sol ve sağ kapılar sırasıyla G0/G1 ve G2/M DNA zirvesine karşılık gelir ve sayılar her tepenin değişim katsayılarını (CV) temsil eder. Diğer tüm çizimlerde, sayılar belirtilen kapılardaki hücre yüzdelerini temsil eder. Rakam, 5 üzerinden 1 temsili deney göstermektedir. Her deneyde, 3 fareden havuzlanmış BM hücrelerini analiz ettik. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3: LN'lerden ve dalaklardan antijene özgü CD8 T hücrelerinin analizi. Balb/c fareleri Chad3-gag ile intramüsküler (i.m.) astarlandı ve I.m. 3. günde, LN ve dalak hücrelerinin aşılanmış ve tedavi edilmemiş kontrol farelerinden boşaltılması akış sitometrisi ile lekelendi ve analiz edildi. (A) Tek hücreleri tanımlamak için beş adımda gating stratejisinin şeması (Adım 1); canlı hücreler (Adım 2); lenfositler (Adım 3); CD8 T hücreleri (Adım 4); ve gag'a özgü hücreler (Adım 5). (B-C) Çizimlere örnek: (B) LN'lerden ve (C) tedavi edilmemiş (üstten) ve aşılanmış (altta) farelerden hücrelerin analizi. Adım 1'deki DNA-A/ -W arsasında tek bir hücre tespit ettik. Daha sonra, Adım 2'de, ölü hücre boyası dışlama ile canlı hücreler seçtik. Adım 3'te lenfositler için kanonik olmayan "rahat" bir kapı kullandık. Adım 4'te, CD8 T hücrelerini CD3 ve CD8'in çift ekspresyyleriyle tanımladık. Daha sonra, sırasıyla florokrom etiketli H-2kd-gag-Pentamer (Pent-gag) ve H-2kd-gag-Tetramer (Tetr-gag) bağlama kapasitelerine dayanarak, Adım 5'te gag'a özgü olmayan ve gag spesifik olmayan hücreler belirledik. (D) FSC-A/SSC-A profilleri gag-spesifik (mavi) ve yukarıda açıklandığı gibi gating sonra gag-spesifik (gri) hücreleri değil. Sayılar belirtilen kapılardaki hücre yüzdelerini temsil eder. Rakam, 5 üzerinden 1 temsili deney göstermektedir. Her deneyde, aşılanmış 3 fare ve 3 tedavi edilmemiş fareden havuzlanmış dalak ve havuza alınan LN hücrelerini analiz ettik. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 4: Antijene özgü CD8 T hücrelerinin hücre döngüsü analizi. Fareler Şekil 3'te olduğu gibi aşılandı ve gag'a özgü hücrelerin hücre döngüsü analizi, Şekil 3'te olduğu gibi 5 adımda gating yapıldıktan sonra, 3. (A)Aşılanmış farelerin LN'lerinden (üstte) ve dalaklardan (altta) gag spesifik CD8 T hücrelerinin hücre döngüsü analizi örneği. Hücre döngüsü aşamaları Şekil 2B'deolduğu gibi tanımlanmıştır. Paneller G0, G1ve Ki67 mAb (sağda) S-G2/M (solda) ve Floresan Eksi Bir (FMO) kontrolündeki hücreleri temsil eder. Sayılar belirtilen kapılardaki hücre yüzdelerini temsil eder. (B) Aşılanmış farelerin LN'lerinden (üstte) ve dalaktan (altta) toplam CD3+CD8+ T hücrelerine (gri) üst üste bindirilmiş S-G2/ M fazlarındaki (kırmızı) gag'a özgü CD8 T hücrelerini gösteren FSC-A/SSC-A nokta çizimleri. (C) CD62L ekspresyonunu gösteren histogramları, aşılanmış farelerin G0 (yeşil), G1 (mavi) ve S-G 2 /M (kırmızı) ile LN'lerden (üst) ve dalak (altta) gag spesifik CD8 T hücreleriyle dengeleyin. y eksenleri normalleştirilmiş olay sayısını gösterir. Rakam, toplam 15 fare ile yapılan 5 bağımsız deneyden 1 temsili örnek göstermektedir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tamamlayıcı Malzeme: Akış sitometresi ayarları. Bu dosyayı indirmek için lütfen tıklayınız.
Discussion
T hücre klonal genişlemesi yoğun bir şekilde çalışılmış olsa da, bazı yönler bilinmemektedir, çünkü araştırma için mevcut araçlar azdır ve kendi dezavantajları vardır. Bu açıdan bakıldığında, bir fare modelinde aşılamadan sonra erken zamanlarda antijene özgü CD8 T hücrelerinin hücre döngüsünü analiz etmek için son derece hassas bir akış sitometrik yöntemi kurduk. Protokol, daha önce farelerde BM hematopoetik hücrelerinin hücre döngüsünü analiz etmek için kullanılan Ki67 ve DNA boyama kombinasyonuna dayanmaktadır13,14. Protokolü antijene özgü CD8 T hücrelerine uyarlamak için, DNA boyasının seçimi, farklı örnekler arasında karşılaştırılabilir DNA boyama elde etmek için uygun koşullar ve veri analizi için gating stratejisi de dahil olmak üzere birkaç kritik konuyu göz önünde bulundurmamız gerekiyordu.
Propidium İyodür ve 7-aminoaktinomisin D dahil olmak üzere DNA boyama için birçok boya mevcuttur; Membran lekelenmesi ve Ki67 boyama için gerekli olan hafif fiksasyon / permeabilizasyon protokolü ile uyumlu olduğu için Hoechst'i seçtik. Aynı zamanda, Hoechst ile lekeleme, mükemmel kalitede bir DNA histogramı elde etmemizi sağladı, yani G0/ G1 ve G2/ M DNA zirveleri, genellikle diğer DNA boyalarıyla elde edilen DNA tepelerinden çok daha düşük bir varyasyon katsayısına (CV) sahipti, örneğin DRAQ519. Gerçekten de, Hoechst canlı hücrelerde bile DNA lekeleyebilir20.
Aynı deneyin farklı örneklerinde Hoechst yoğunluğundaki dalgalanmayı önlemek için bazı stratejiler kullanılmıştır. Hoechst lekeleme, zaman içinde boya yoğunluğunun azalmasını en aza indirmek için akış sitometresinde numune alımından hemen önce gerçekleştirildi. Protokolü çok sayıda örnekle yapılan büyük deneylerde yeniden üretmek isteyenler için hoechst boyamayı aynı anda birkaç örnek üzerinde yapmanızı öneririz. Diğer bir dezavantajı, Hoechst yoğunluğunun boya ile inkübasyon sırasında hücre sayısından yoğun bir şekilde etkilenebilmesidir. Bu nedenle, DNA boyama için her zaman aynı sayıda hücre ve örnek başına aynı hacim kullanılmasını şiddetle öneririz. Akış sitometresinde elde etmek için yüksek sayıda hücre gerekiyorsa, iki veya daha fazla özdeş numune hazırlamanızı ve ardından Hoechst boyama adımından hemen önce birleştirmenizi öneririz.
Protokolün önemli bir noktası, veri analizi için gating stratejisidir. Yakın zamanda bağışıklık yanıtının erken zamanlarında T hücre analizi için yeni bir strateji yayınladık, bu da antijene özgü T hücrelerinin tespit hassasiyetini artırmamızı sağladı12. Bu stratejiyi burada gösterilen verilere aşağıdaki gibi uyguladık. İlk olarak, DNA-A/W çiziminde hücre agregalarını hariç tutmuşuz. İkincisi, ölü hücrelerin gating sonra, FSC / SSC arsa oldukça büyük bir lenfosit kapısı kullandık ("rahat kapı"). Bu strateji ile, S-G2/ M'de genellikle mevcut gating stratejileri tarafından kaçırılan yüksek aktif antijene özgü CD8 T hücrelerini dahil edebildik, çünkü bu hücreler yüksek FSC-A ve SSC-A'ya sahip. Özetle, veri analizi, aktif /çoğalan antijene özgü T hücrelerinin hassas bir şekilde algılanmasını sağlamak için gerekli olan yöntemin kritik bir bölümünü temsil eder.
Yöntem, immün yanıtın erken aşamalarında kritik T hücre verilerinin eksik olma olasılığını önler ve T hücre immün izleme için yeni bakış açıları açar. Gelecekteki bir gelişme, G2 ve M21arasında farklılaşmaya izin verecek fosfo-histon 3 için boyama içerebilir. Mevcut bir sınırlama, hücrelerin nükleer işaretleyici Ki67 için lekelenmek için sabit ve geçirgen olması gerektiğidir. Bu nedenle, hücreler sıralama ve sonraki fonksiyonel analiz gibi diğer analiz türleri için kullanılamaz. Ayrıca, Hoechst de dahil olmak üzere DNA boyaları genellikle genomik DNA analizine müdahale eder ve bu tür değerlendirmeler için uygun değildir. Farklı hücre döngüsü evreleriyle ilişkili ve canlı hücrelerde lekelenebilen membran belirteçlerinin tanımlanması bu sınırlamanın üstesinden gelebilir. Sonuç olarak, yöntem aşı, enfeksiyon, immün aracılı hastalıklar ve immün tedavi gibi çeşitli bağlamlarda aktive /çoğalan T hücrelerinin değerlendirilmesi için büyük bir potansiyele sahiptir.
Disclosures
A. Folgori ve S. Capone Reithera Srl. A. Lefkoşa'nın çalışanlarıdır ve WO 2005071093 (A3) "Şempanze adenovirüs aşı taşıyıcıları" patent başvurusunda mucit olarak adlandırılmıştır. Diğer yazarların açıklayacak bir şeyi yok.
Acknowledgments
Bu çalışma Reithera, MIUR proje 2017K55HLC_006 ve Associazione Italiana Ricerca sul Cancro (AIRC) tarafından 5 × 1000 hibe ile desteklendi. Nih Tetramer Tesisi aracılığıyla aşağıdaki tetramer elde edildi: APC-konjuge H-2K (d) HIV gag 197-205 AMQMLKETI.
Materials
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
References
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- Mouse phenotype. , Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020).
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Tags
İmmünoloji ve Enfeksiyon Sayı 167 antijene özgü CD8 T hücreleri hücre döngüsü Ki67 DNA boyası akış sitometrisi dalak lenf düğümleri fareErratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021.
Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
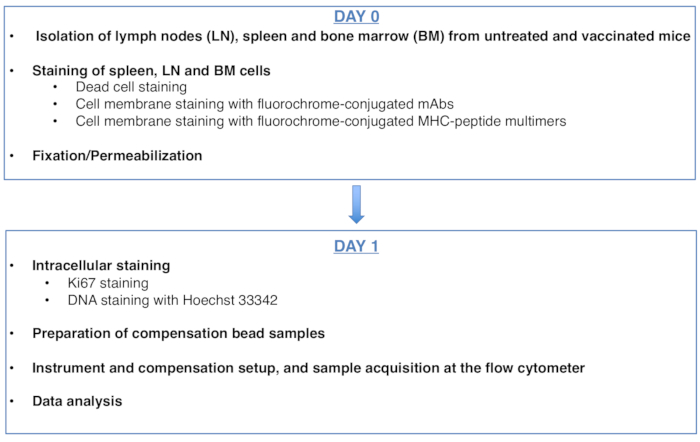
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:
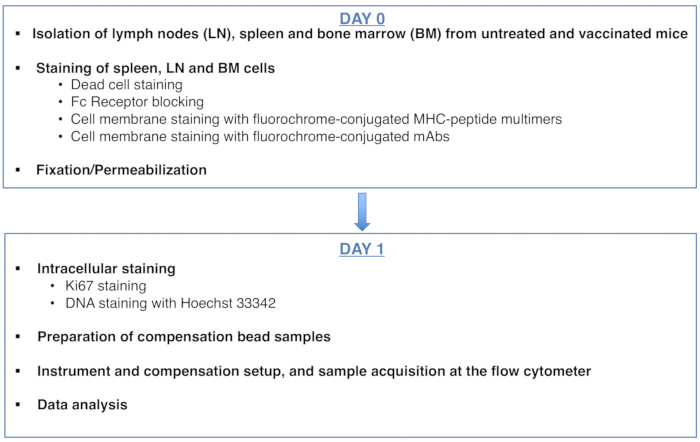
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.



