ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
克隆扩增是抗原特异性T细胞反应的关键特征。然而,抗原反应T细胞的细胞周期研究不佳,部分原因是技术限制。我们描述了一种流式细胞术方法,用于分析接种疫苗小鼠的脾脏和淋巴结中的克隆扩增抗原特异性CD8 T细胞。
Abstract
抗原特异性T细胞在体内的细胞周期已经通过使用几种方法进行了检查,所有这些方法都具有一定的局限性。溴脱氧尿苷(BrdU)标记处于或最近完成的S期细胞,羧基荧光素琥珀酰亚胺酯(CFSE)在分裂后检测子细胞。然而,这些染料不允许在分析时鉴定细胞周期相。另一种方法是利用Ki67,这是一种标记物,在细胞周期的所有阶段(静态阶段G0)中,细胞都高度表达。不幸的是,Ki67不允许进一步分化,因为它不会将S期致力于有丝分裂的细胞与G1 中可以保持该阶段的细胞分开, 进入循环或进入G0。
在这里,我们描述了一种流式细胞术方法,用于捕获小鼠次级淋巴器官中不同细胞周期阶段的T细胞的"快照"。该方法将Ki67和DNA染色与主要组织相容性复合物(MHC)肽多聚体染色和创新门控策略相结合,使我们能够成功地区分G0,G 1和脾脏中S-G2 / M阶段的抗原特异性CD8T细胞,并在接种携带人类免疫缺陷病毒(HIV)-1模型抗原凝集的病毒载体后引流小鼠淋巴结。
该方法的关键步骤是选择DNA染料和门控策略,以提高测定灵敏度,并包括当前分析标准可能遗漏的高度活化/增殖的抗原特异性T细胞。DNA染料Hoechst 33342使我们能够获得G0/ G1 和G2/ M DNA峰的高质量鉴别,同时保留膜和细胞内染色。该方法具有增加体内T细胞反应知识和改善免疫监测分析的巨大潜力。
Introduction
朴素T细胞在抗原启动时经历克隆扩增和分化。分化的T细胞显示出对抗原清除和维持抗原特异性记忆至关重要的效应器功能,这是持久保护的关键。在初级反应的第一步中,幼稚的T细胞与淋巴器官中特殊生态位内的抗原呈递细胞(APC)的相互作用对于诱导表征克隆扩增期1,2,3的巨大T细胞增殖至关重要。T细胞 - APC相互作用通过抗原,共刺激信号和可溶性因子(细胞因子和趋化因子)的浓度和持久性进行精细调节,这些因子影响T细胞克隆后代的数量和质量4,5,6,7。
尽管对T细胞克隆扩增进行了深入的研究,但仍然不知道抗原引发的T细胞是否在抗原识别的位点完成其整个细胞周期,或者它们是否在细胞周期进展过程中迁移到其他器官。这种知识的缺乏是由于可用于细胞周期分析的工具的特性。这些包括对核标志物,Ki67和细胞染料特异性的单克隆抗体(mAbs)和细胞染料,这些染料要么识别经历过细胞周期S期的细胞(例如,溴脱氧尿苷(BrdU))或区分子细胞及其祖先(例如,羧基荧光素琥珀酰亚胺酯(CFSE))。
然而,细胞标记染料,如CFSE和BrdU,不允许确定在特定器官中发现的细胞是否局部增殖,或者更确切地说是在分裂后迁移到该位点8,9。此外,核内蛋白Ki67只能区分G 0(Ki67阴性细胞)和任何其他细胞周期期(Ki67阳性细胞)中的细胞。因此,Ki67分析不能区分活跃增殖(即S,G2或M)中的细胞与G1中的细胞,G1中的细胞可能迅速进展到分裂或长时间停留在G1中或恢复到静止10,11。
在这里,我们描述了一种新的流式细胞术方法,用于从接种疫苗的小鼠的脾脏和淋巴结(LNs)中分析抗原特异性CD8 T细胞12的细胞周期(图1)。该方法利用了Ki67和DNA染色的组合,该染色以前用于分析小鼠骨髓(BM)造血细胞的细胞周期13,14。在这里,我们成功地将Ki67加DNA染色与最近发表的创新门控策略12一起应用于CD8 T细胞克隆扩增的分析。我们能够清楚地区分G0、G1和S-G2/M期的抗原特异性CD8 T细胞,以及接种疫苗的小鼠的脾脏和引流LNs。
Protocol
将小鼠安置在普莱桑特动物设施,并根据意大利卫生部授权号1065 / 2015-PR进行这项工作。该协议遵循国家和国际法律和政策(UE指令2010/63 / UE;意大利第26/2014号法令)。
1. 培养基和染色液的制备
- 制备完整培养基:罗斯威尔公园纪念研究所(RPMI)培养基,含2 mM谷氨酰胺,100 U / mL青霉素/链霉素,50μMβ-巯基乙醇和10%体积/体积(v / v)的胎牛血清(FBS)
- 制备染色缓冲液:磷酸盐缓冲盐水,不含Ca2 +/ Mg2 +(PBS),具有1%重量/体积(w / v)牛血清白蛋白(BSA)和2mM乙二胺四乙酸二钠盐(EDTA)
2. 小鼠治疗
- 通过肌内注射(i.m.)在人类免疫缺陷病毒(HIV)的股四头肌中注射7-8周龄的雌性Balb / c小鼠-1-gag-表达-黑猩猩腺病毒载体(ChAd3-gag),剂量为107 个病毒颗粒。
- 在启动后1-4个月,通过i.m.注射表达HIV-1-gag的改良牛痘安卡拉病毒(MVA-gag)以106 个斑块形成单位的剂量对小鼠进行增强。
- 在增强后的第3天,通过宫颈脱位处死增强的小鼠,并与未处理的小鼠并行分析它们。
- 从增强和未治疗的小鼠中收获排出股四头肌(髂头,腘窝和腹股沟)和脾脏的LNs。此外,从未处理的小鼠的两条后腿收集BM,并将该BM用于流式细胞仪设置并作为细胞周期分析的阳性对照(图2)。
注意:生成 ChAd3-gag 和 MVA-gag 向量,如前所述12、15、16、17。
3. 分离引流LN、脾脏和BM细胞
- 脾脏和LN细胞的分离
- 将5 mL完整培养基放入两个15 mL管中的每一个中,并将它们放在冰上,准备收集器官。
- 通过颈椎脱位处死一只成年小鼠。
- 将鼠标放在其背部,并用70%v / v乙醇对皮肤表面进行消毒。
- 要收集腹股沟LN,请用剪刀在腹部做一个~1厘米的纵向切口,并用镊子拉伸切口。
- 在皮肤内表面可视化腹股沟LNs,并用镊子收获它们。将腹股沟LNs放入步骤3.1.1中制备的两个15 mL管之一中。
- 要收集脾脏,请用剪刀做腹膜切口并切除脾脏。切割周围结缔组织后,将脾脏放入步骤3.1.1中制备的第二个15mL管中。
- 要收集髂骨LNs,请将肠道移到一边,并观察靠近下腔静脉的髂骨LNs,然后使用镊子收集它们。将髂骨LNs放在含有腹股沟LNs的同一管中。
注意:为了获得足够的LN细胞用于染色(见第4节),通常需要从一只小鼠中汇集腘窝,腹股沟和髂膜LNs。这些LN都排出股四头肌(i.m疫苗接种的部位)。该协议仅使用一个15 mL的合并LN管。 - 要收集腘窝LN,抓住后腿的皮肤并轻轻向下拉以露出肌肉。然后,将镊子插入膝关节下方的肌肉之间,并收集腘窝LNs。将腘窝LNs放在含有腹股沟和髂静脉LNs的同一管中。
注意:请参阅 3.1.7 之后的注释。 - 将脾脏放入装有5mL完整培养基的60mm培养皿中的70μm细胞过滤器中。使用5 mL注射器柱塞,轻轻捣碎器官,直到其完全分解。
- 取出过滤器,并将细胞悬浮液转移到干净的15 mL管中。
- 向培养皿中加入5mL完整培养基,并仔细清洗培养皿和过滤器,以确保所有细胞都已回收。与剩余的脾细胞悬浮液一起池入15 mL管中。
- 对于合并的腹股沟、髂窝和腘窝LNs,按照与步骤3.1.9至3.1.11中使用的脾脏相似的程序制备单细胞悬浮液。
- 在4°C下以400×g离心细胞10分钟。 弃去上清液,并将细胞沉淀重悬于PBS中。
- 使用PBS中的红细胞裂解缓冲液和0.04%v v /v台盼蓝用Neubauer室计数细胞。
- 脱脂细胞的分离
- 将 5 mL 完整培养基放入 15 mL 管中,并将其放在冰上,准备收集后腿。
- 通过颈椎脱位处死一只成年小鼠。
- 用70%v / v乙醇对皮肤表面进行消毒。
- 用剪刀在腹侧皮肤上做一个~1厘米的横向切口,牢牢抓住切口两侧的皮肤,然后轻轻向下拉,露出后腿的肌肉。
- 为了消除后腿后部的皮肤,保持鼠标处于仰卧位,将夹子放在膝盖下方,然后向上拉以暴露肌肉。
- 切开一条后腿两端的骨头:骨盆/髋关节和脚踝。
- 将双后腿转移到步骤3.2.1中制备的15 mL管中。将管子放在冰上。
- 从15 mL管中取出后腿,并将其转移到薄纸上。切开膝关节正下方的后腿以去除胫骨。从周围的肌肉中解剖股骨和胫骨,用剪刀去除多余的组织,并弄湿纸巾。
- 用剪刀剪断骨头末端,露出内侧髓轴。将胫骨和股骨插入BM抽出管(参见3.2.9.1-3.2.9.218中的制备),最宽的一端在底部。
- 在吸头末端正上方的管路和100 μL管路处切割200 μL移液器吸头。
- 将中间部分放入尖端的上部,较大的部分,并将其放入1.5 mL微量离心管中。
- 将BM提取管以800×g旋转1分钟。
- 丢弃骨头,并将沉淀在1mL完整培养基中剧烈重悬以除去任何簇。通过放置在15 mL管顶部的70μm过滤器过滤细胞悬浮液。
- 每次用1 mL完整培养基洗涤BM提取管两次。通过70μm过滤器过滤,并将体积与步骤3.2.11中获得的其余细胞悬浮液混合。
注意:单个15 mL管将包含来自小鼠后腿的细胞。 - 在4°C下以400×g离心细胞10分钟。 弃去上清液,并将细胞沉淀重悬于PBS中。
- 使用PBS中的红细胞裂解缓冲液和0.04%v v /v台盼蓝用Neubauer室计数细胞。
4. 脾脏、LN 和 BM 细胞染色
- 将要染色的细胞样品分为3个亚组: 用于补偿的细胞样品,包括仅用Hoechst 33342(以下简称Hoechst)染色的未经处理小鼠的BM细胞和来自未处理小鼠的脾细胞,用于制备用于死细胞染料补偿的死细胞/活细胞混合物; 细胞周期分析的阳性对照,包括来自未处理小鼠的BM样品;以及含有来自未经处理和接种疫苗的小鼠的脾脏和LN样本的 实验 样本。
注意:确保有足够的脾脏和LN细胞来分析足够数量的呕吐特异性CD8 T细胞。通常需要使用来自3只接种疫苗的小鼠的混合脾细胞和混合的LN细胞,并对两个或多个相同的混合细胞样品进行染色,每个样品含有3×106 个细胞。在Hoechst染色的步骤中合并相同的样品。同样,染色来自3只未处理小鼠的脾细胞和LN细胞,并在最后合并相同的样品。从未经处理的小鼠中留出未染色的脾细胞 样本 ,用于仪器和补偿设置。 - 准备死细胞/活细胞混合物以进行死细胞染料补偿(这种细胞混合物将仅用死细胞染料染色)。
- 在65°C下加热水浴。
- 取一等分试样的脾脏细胞(〜3×106)。
- 将细胞悬浮液转移到微量离心管中,将其置于65°C的水浴中5分钟,然后立即将其置于冰上10分钟。
- 以1:1的比例将热杀伤细胞与活脾细胞(〜3×106)混合,并将混合物的一半转移到96孔圆底板(〜3×106 个细胞/孔用于死细胞染色控制)。
- 实验样品的死细胞染色,细胞周期分析和死细胞/活细胞混合物的阳性对照
- 根据染色方案(步骤4.1),将脾脏,LN,BM细胞(3×106个细胞/孔)和死细胞/活细胞混合物(第4.2节)转移到96孔圆底板中,并在4°C下以400×g离心3分钟。
- 将每个细胞沉淀重悬于50μL在PBS中稀释的死细胞染料中,并立即通过上下移液3次来重悬。
- 在4°C下孵育30分钟,避光。
- 用染色缓冲液洗涤细胞2次;第一次用200μL,第二次用250μL。对于每个洗涤离心机,在4°C下以400×g离心3分钟。
- 弃去上清液,并将细胞沉淀重悬于20μLPBS中。
- 使用主要组织相容性复合物(MHC)肽多聚体和单克隆抗体进行膜细胞染色。
- 根据染色方案(流式细胞仪设置, 表1)考虑必要的体积,准备以下试剂:
- 根据适当的稀释度在染色缓冲液中稀释mAb 2.4G2(见 材料表);对于每个要染色的样品,使用10μL该稀释液。
注:2.4G2单克隆抗体阻断免疫球蛋白与FcγIII和FcγII受体的非抗原特异性结合。 - 在染色缓冲液中稀释H-2k(d)AMQMLKETI别癣菌蓝蛋白(APC)标记的四聚体(Tetr-gag)以获得适当的稀释度(见 材料表);对于每个要染色的样品,使用20μL该稀释液。
- 根据先前在滴定实验中确定的适当稀释度(见 材料表),通过稀释染色缓冲液中的mAbs来制备抗体混合物;对于每个要染色的样品,使用20μL该抗体混合物。
注:这里使用抗CD3e叶黄素叶绿素蛋白(PerCP-Cy5.5)(克隆145-2C11),抗CD8a亮紫外(BUV805)(克隆53-6.7)和抗CD62L藻红素菁7(PECy7)(克隆MEL-14)。
- 根据适当的稀释度在染色缓冲液中稀释mAb 2.4G2(见 材料表);对于每个要染色的样品,使用10μL该稀释液。
- 加入10μL先前稀释的2.4G2 mAb(步骤4.4.1.1),并在4°C下孵育10分钟,避光。
- 加入20μL先前稀释的Tetr-gag APC(步骤4.4.1.2)和10μL的H-2k(d)AMQMLKETI藻红素(PE)五聚体(pent-gag)。在4°C下孵育15分钟,避光。
- 加入20μL先前制备的抗体混合物(步骤4.4.1.3),并在4°C下孵育15分钟,避光。
注:因此,最终体积为每孔80μL(步骤4.3.5,步骤4.4.2至4.4.4)。 - 用200μL染色缓冲液洗涤细胞。在4°C下以400×g离心5分钟。
- 将细胞沉淀重悬于250μL染色缓冲液中,并将细胞悬浮液转移到5mL管中。向管中加入1mL染色缓冲液,并在4°C下以400×g离心5分钟。
- 取等分试样BM细胞(3×106个细胞)(参见细胞样品列表,第4.1节)以补偿Hoechst通道(Hoechst 33342被紫外激光激发(流式细胞仪设置(表2)),并将细胞悬浮液转移到5 mL管中。向管中加入1mL染色缓冲液,并在4°C下离心400×g5分钟。
- 根据染色方案(流式细胞仪设置, 表1)考虑必要的体积,准备以下试剂:
5. 固定/透化
- 根据制造商的说明,用3份固定/透化稀释剂稀释1份固定/透化浓缩液,制备新鲜的固定/透化缓冲液。
- 弃去上清液并对样品进行脉冲涡旋以完全分解沉淀。
- 向每个试管中加入1mL新鲜制备的固定/透化缓冲液,包括具有未染色脾脏细胞的试管(3×106,参见细胞样品列表,第4.1节)和涡旋。
- 在4°C下孵育16小时。
注意:协议可以在此处暂停。
6. 细胞内染色
- Ki67 染色
- 根据制造商的说明,通过用蒸馏水稀释10x的透化缓冲液来制备新鲜的透化缓冲液1x。使用前,必须通过0.45μm过滤器过滤透化缓冲液1x以消除聚集体。
- 将稀释的mAb Ki67荧光素异硫氰酸酯(FITC)(克隆SolA15)在透化缓冲液1x(见 材料表)中,如前所述在滴定实验中确定(每个样品的最终体积为100μL)。
- 向每管中加入3mL透化缓冲液1x,并在室温(RT)下以400×g离心5分钟。
- 弃去上清液并重复步骤6.1.3。
- 弃去上清液,并将细胞沉淀重悬于100μL先前稀释的mAb Ki67 FITC中(步骤6.1.2)。
- 在室温下孵育30分钟,避光。
- 用4mL渗透缓冲液1x洗涤细胞2次。对于每个洗涤离心机,在室温下以400×g离心5分钟。
- 考虑以下体积,将细胞沉淀重悬于PBS中:350μLPBS,用于直接在流式细胞仪上获取样品;250 μL PBS,用于在流式细胞术前不久与Hoechst一起孵育的样品(第6.2节)。
- 基因染色
- 向每个样品中加入250μL4μg/ mL PBS中的Hoechst(Hoechst的最终浓度为2μg/ mL)。
注意:如果制备两个或多个相同的250μLPBS样品,请在此步骤中将它们合并,并在PBS中加入等体积的4μg/ mLHoechst溶液(Hoechst的最终浓度为2μg/ mL)。细胞的数量极大地影响了DNA染色步骤。在每个样品中使用相同的细胞编号。请注意,即使细胞数量略有减少(例如,由于先前洗涤步骤中的细胞丢失)也会导致更高的Hoechst与DNA的结合和更高的Hoechst强度。 - 在室温下孵育15分钟,避光。
- 在室温下以400×g离心样品5分钟。
- 将细胞沉淀重悬于350μLPBS中。
- 向每个样品中加入250μL4μg/ mL PBS中的Hoechst(Hoechst的最终浓度为2μg/ mL)。
7. 补偿珠样品的制备
- 通过在染色缓冲液中适当稀释mAb来制备5μL抗体。
注意:对于实验中使用的每种荧光染料偶联单克隆抗体,准备其相应的补偿微珠样品。 - 涡旋阴性对照和抗大鼠/仓鼠Ig,κ Comp珠在使用前。
- 对于每个样品,引入一滴(约20μL)阴性对照CompBeads和一滴抗大鼠/仓鼠Ig,k CompBeads。
- 将5μL预稀释的抗体(步骤7.1)加入试管中,并上下移液。
- 在4°C下孵育15分钟,避光。
- 用2 mL染色缓冲液洗涤样品。在4°C下以400×g离心5分钟。
- 弃去上清液,并通过向每个管和涡旋中加入500μLPBS来重悬沉淀。
8. 流式细胞仪的仪器和补偿设置以及实验样品采集
注:有关细胞仪配置,请参阅流式细胞仪设置(表2)。
- 通用仪器和补偿设置
- 打开软件进行样品采集(请参见 材料表),然后单击工作区功能区中的" 新建 实验"部分并选择" 新建空白实验"来创建新实验。
- 双击创建的实验将其打开。
- 在 "细胞计数器设置" 窗口中,单击" 参数 ",然后选择染色面板中使用的所有通道(例如 PE、APC 等),包括前向散射 (FSC) 和侧散射 (SSC) 参数。
- 通过取消选中对数刻度,选择线性刻度作为Hoechst参数,并检查FCS,SSC和Hoechst的电压脉冲的宽度(W)。
注意:默认情况下,所有参数都以对数(对数)刻度显示,但 FSC 和 SSC 除外,它们以线性刻度显示。所有参数均由电压脉冲的面积 (A) 和高度 (H) 进行分析。 - 在 全局工作表上,创建一个点阵图,其中 FSC-A 在 x 轴上,SSC-A 在 y 轴上。
- 通过单击采集仪表板上的"获取数据"来运行未染色的脾脏样本。
- 设置适当的 FSC 和 SSC 设置,通过修改 "参数 "部分中的电压值来可视化像元,并创建一个门,通过单击"全局工作表"工作区工具栏上的" 多边形门 "来选择 FSC-A/SSC-A 点图中显示的所有 像元。
- 在直方图中显示门控细胞,每个荧光参数都位于x轴上。
- 运行未染色和完全染色的脾脏样品以调整荧光检测器(PMT),以在每个荧光参数下清晰地分离染色细胞的阴性和阳性信号。
- 若要执行补偿设置,请单击工作区功能区中的" 试验 ",然后在 "补偿设置" 部分下,选择" 创建补偿控件"。取消选中 包括未染色的控制管/孔, 然后单击 确定。
注意:此操作将导致创建一个名为" 补偿控制" 的试样和一个包含与每个选定参数对应的多个工作表的 "普通工作表 "。 - 运行补偿珠样品(见第7节);设置适当的FSC和SSC设置,通过在 "细胞计数器 "窗口中修改FSC参数上的电压值和采集阈值5,000来可视化磁珠。
- 调整磁珠群上的 P1 栅极,并检查正峰和负峰在 x 轴上是否都可见。对每个补偿微珠样品重复此操作,最后通过单击采集仪表板上的"记录数据"来记录每个样本文件(为每个样本记录至少 5,000 个事件)。
- 对于每个记录的磁珠样品,分别在正峰和负峰上设置 P2 和 P3 门。
- 运行细胞样品进行补偿(参见步骤4.2和4.4.7以及第5和第6节)。修改FSC和SSC电压以及阈值以可视化电池,调整P1门,最后记录每个样本文件(记录至少10,000个事件)。将 P2 和 P3 门分别设置为正峰和负峰。
注意:对于Hoechst通道的补偿,请使用G0/G1 作为负峰(P3),使用G2/ M作为正峰(P2)。 - 单击工作区功能区部分中的"试验",然后在"补偿设置"部分,选择"计算补偿"。
- 命名创建的补偿设置,链接并将其保存到当前实验。
- 实验样品采集
- 通过单击浏览器工具栏上的" 新建标本 "打开样本,然后在 "全局工作表"中创建门控策略。
注意:样品采集的门控策略类似于样品分析, 如图3 和第9节所述。 - 在直方图中显示 所有事件 总体,CD3-A 位于 x 轴上。创建 间隔门 以仅选择CD3+ 单元格。
- 在 采集仪表板上,选择存储门作为LN样本 的所有事件 ,选择 所有事件 或CD3+ 细胞作为脾脏样本。
- 以低速运行实验样品,最后记录所有文件,确保从接种疫苗的小鼠的每个样品中收集至少100-200个抗原特异性CD8 T细胞。
注意:实验样品的文件大小通常很大(30-120 MB),特别是当抗原特异性CD8 T细胞的频率较低时。因此,必须收集大量事件(>1×106)来记录至少100-200个抗原特异性CD8 T细胞。大文件可能会减慢后续数据分析过程的速度。在脾脏样本中仅采集CD3+ 细胞(参见上面的步骤8.2.2)有助于保持较小的文件大小。 - 运行并记录用于细胞周期分析的阳性对照,即来自未处理小鼠的BM样品。
- 通过单击浏览器工具栏上的" 新建标本 "打开样本,然后在 "全局工作表"中创建门控策略。
9. 数据分析
- 打开软件(参见 材料表),点击工作区功能区部分的 创建 组,创建与要分析的不同器官对应的不同组(即创建组"a-LNs";"b-脾脏";"c-BM")。
注意:新创建的组将显示在组列表中,而"补偿"组由软件自动生成。 - 通过双击 组 名称打开"修改组"窗口,并检查新创建的组是否已同步。如果不是,请在函数 "已同步"上插入复选标记。
- 拖动其相应组中的每个 .fcs 文件。
- 创建从"a-LN"组开始的门控策略。
- 双击组中完全染色的样品以打开图形窗口;x轴和y轴的标记与fcs文件中相同(参见流式细胞仪设置,表2)。
- 在点阵图中显示为此样本获取的总事件,其中 DNA-A 在 x 轴上,DNA-W 在 y 轴上。
- 通过单击图形窗口的门控工具部分中的 矩形 ,仅选择单个像元群。
注意:单个细胞的DNA-A值如下:2N(低):在2N和4N(中间)之间,或等于4N(高),而所有细胞的DNA-W值都是相同的(图3的步骤1)。 - 双击矩形门的中心,以点图显示单个细胞,X 轴上为 FSC-A 参数,y 轴上为死细胞染料。
- 通过单击图形窗口的门控工具部分中的 面 ,仅选择活细胞群。活细胞对死细胞染料呈阴性( 图3的步骤2)。
- 双击多边形门的中心以点图显示点图中的像元,其中 FSC-A 参数位于 x 轴上,SSC-A 参数位于 y 轴上。
- 单击"矩形",然后创建一个"放松"门,以包括该图12中的所有单个活细胞(图3的步骤3)。
- 双击"放松"门的中心,以点图显示单元格,其中 CD3 在 x 轴上,CD8 在 y 轴上。
- 通过单击Polygon选择CD3+CD8+单元格(图3的步骤4)。
- 双击 CD3+CD8+ 门的中心,以点阵图显示单元格,其中 Tetr-gag 在 x 轴上,Pent-gag 在 y 轴上。
- 通过单击 Polygon 选择抗原特异性CD8 T细胞(Tetr-gag和Pent-gag均呈阳性)( 图3的步骤5)。
- 双击特定嘎嘎门的中心,以点图显示细胞,其中DNA-A在x轴上,Ki67在y轴上(图4)。
- 通过单击图形窗口的门控工具部分中的 Quad 来选择不同细胞周期阶段中的单元格。
注意:G0期细胞是Ki67neg-DNA低细胞(左下象限);G1中的细胞是Ki67pos-DNA低(左上象限);S-G2/ M中的细胞是Ki67pos-DNA中级/高(右上象限)(图4)。 - 在一个样本中创建的门控策略复制到相应的组,以将门应用于该组的所有样本。
- 对"a-LN 组"重复步骤 9.5 到 9.18。
- 检查所有门是否适合"b-脾"组的每个样本。要分析BM细胞(阳性对照)之间的细胞周期,请单击"放松"门的中心以点阵图显示细胞,其中DNA-A在x轴上,Ki67在y轴上。
- 检查所有门是否适合3组的每个样本(即,来自脾脏,LN和BM的细胞)。
注:单细胞群门(步骤9.7)和细胞周期的四门(步骤9.17)在不同样品中可能具有不同的门坐标,主要是由于样品之间Hoechst染料强度可能存在细微差异(第6.2节)。因此,可能需要修改每个样品中细胞周期的单细胞群门和四元门。这将按如下方式完成:双击组名称,然后从组属性中删除同步。此操作允许修改一个样本中的门,而无需修改组中所有其他样本中的相同门。删除同步后,根据需要修改门。 - 要可视化此分析获得的结果,请单击工作区功能区部分中的"布局编辑器"将其打开。将示例窗格中门控策略的每个门拖到布局编辑器中,并根据门控策略的顺序放置图。如有必要,请通过双击布局中的相应绘图并在"图形定义"窗口中选择适当的类型来更改图形类型。
- 单击" 组 "并按布局功能区上的函数 进行迭代 ,以可视化在每个器官中获得的结果,并比较不同的样本。
Representative Results
根据图1中总结的方案,使用荧光DNA染料Hoechst和抗Ki67 mAb分析了Balb / c小鼠脾脏,LNs和BM细胞的细胞周期阶段。这种染色允许细胞在细胞周期的以下阶段分化:G 0(Ki67neg,2N的DNA定义为DNAlow),G1(Ki67pos,DNAlow)和S-G 2/ M(Ki67pos,DNA含量在2N和4N之间,或等于4N的DNA定义为DNA中介体/高)。
我们首先对BM细胞进行细胞周期分析,以重现先前发表的结果13,14,然后分析感兴趣的细胞,即CD8 T细胞。图2显示了BM细胞细胞周期分析的典型示例(图2A)。该方案产生了G0/ G1和G2/ M DNA峰的低变异系数(CV),表明DNA染色的质量优异(图2B,显示了CV<2.5的示例;CV总是<所有实验中的5)。
然后,我们将相同的方案应用于来自接种疫苗小鼠的抗原特异性CD8 T细胞。BALB / c小鼠通过使用Chad3-gag进行启动和MVA-gag用于增强来接种HIV-1的抗原gag,两者都被设计为携带HIV-1 gag。在增强后(d)第3天,我们分析了来自脾脏和引流LNs的呕吐特异性CD8 T细胞的频率。我们在免疫应答的早期阶段利用了新定义的T细胞门控策略,与传统策略相比,该策略适用于检测高度激活的抗原应答CD8 T细胞12。我们在随后的五个步骤中执行了新颖的策略。在步骤1中,我们通过DNA-A / -W门排除了二倍体或聚集体,在步骤2中,我们通过死细胞标记物排除鉴定活细胞。在步骤3中,我们使用非常规的"松弛"FSC-A / SSC-A门(图3A)而不是规范的狭窄淋巴细胞门12来识别感兴趣的人群。在CD3+CD8+细胞上进行门控后(图3A的步骤4),我们通过使用两种不同的MHC多聚体(即Pent-gag和Tetr-gag)鉴定了gag特异性CD8 T细胞(图3A的步骤5)。我们使用两个多聚体而不是一个来提高接种疫苗的小鼠中gag特异性CD8 T细胞检测的灵敏度,而不增加未处理小鼠的染色背景(图3B和C,步骤5)。因此,我们成功地区分了未处理的小鼠(LNs和脾脏中分别为0.00%和0.00%抗原特异性CD8 T细胞)与接种疫苗的小鼠(分别为LNs和脾脏中的0.46%和0.29%抗原特异性CD8 T细胞,图3B和C)。
值得注意的是,该协议允许我们在未经治疗的小鼠的LNs和脾脏的抗原特异性CD8 T细胞门中具有极低的背景(通常为0.00%,最大为0.02%)。对噗声特异性和非噘声特异性FSC-A / SSC-A图的比较表明,噗声特异性细胞具有高SSC-A和FSC-A(图3D),证实了需要使用"松弛"的FSC-A / SSC-A门来捕获这些细胞。然后,我们评估了不同细胞周期阶段中gag特异性CD8 T细胞的百分比(图4A)。我们发现,在扩增后的第3天,脾脏中甚至更多,脾脏中的gag特异性CD8 T细胞含有高比例的S-G2/ M期细胞(分别为18.60%和33.52%)。
此外,我们发现S-G2/ M相中的gappa特异性CD8 T细胞在覆盖到来自同一器官的总CD8 T细胞上时具有较高的FSC-A和SSC-A(图4B)。gag特异性CD8 T细胞的CD62L表达较低,正如激活的T细胞所预期的那样,除了LNs中G0 中的少数细胞(图4C)。总之,这些结果证实了"松弛"门( 图3A,B和C的步骤3)需要包括所有增殖抗原特异性CD8 T细胞12。该协议对于分析时抗原特异性CD8 T细胞的细胞周期阶段的"快照"评估以及不同细胞周期阶段细胞的CD62L表达非常有价值。

图1:抗原特异性CD8 T细胞的细胞周期分析方案。请点击此处查看此图的放大版本。

图2:BM细胞的细胞周期分析。 对来自未经处理的Balb / c小鼠的BM细胞进行染色并通过流式细胞术进行分析。 (A) 门控策略示例。我们在DNA-A/-W图(左)中对单个细胞进行门控,随后通过死细胞染料排除(中间)对活细胞进行门控。然后,所有BM细胞都使用"放松"的FSC-A / SSC-A门(右)。 (B)BM 细胞的细胞周期分析示例(左)。我们使用Ki67和DNA染色的组合来鉴定细胞周期以下阶段的细胞:G0( 左下象限,Ki67neg-DNAlow细胞),G1( 左上象限,Ki67pos-DNAlow),S-G2/ M(右上象限,Ki67pos-DNA中介体/高)。显示Ki67 mAb(中)和DNA直方图(右)的荧光减一(FMO)对照。在DNA直方图中,左门和右门分别对应于G0/G1 和G2/M DNA峰,数字表示每个峰的变异系数(CV)。在所有其他图中,数字表示指示门中的像元百分比。该图显示了5个实验中的1个代表性实验。在每个实验中,我们分析了来自3只小鼠的混合BM细胞。 请点击此处查看此图的放大版本。

图3:来自LNs和脾脏的抗原特异性CD8 T细胞的分析。用Chad3-gag肌内(i.m.)启动Balb / c小鼠,并用MVA-gag增强i.m。在增强后的第3天,从接种疫苗和未治疗的对照小鼠引流LN和脾脏细胞通过流式细胞术染色和分析。(A)门控策略的方案,分五步识别单个细胞(步骤1);活细胞(步骤2);淋巴细胞(步骤3);CD8 T细胞(步骤4);和呕吐特异性细胞(步骤5)。(B-C)图例:分析来自未处理(上)和接种疫苗(下)小鼠的(B)LNs和(C)脾脏的细胞。 我们在步骤1的DNA-A / -W图上鉴定了单个细胞。然后,在步骤2中,我们通过死细胞染料排除选择活细胞。在步骤3中,我们对淋巴细胞使用了非规范的"放松"门。在步骤4中,我们通过CD3和CD8的双重表达来鉴定CD8 T细胞。然后,我们在步骤5中鉴定出gag特异性细胞和非gag特异性细胞,基于它们分别结合荧光染料标记的H-2kd-gag-Pentamer(Pent-gag)和H-2kd-gag-Tetramer(Tetr-gag)的能力。(D)如上所述门控后,gag特异性(蓝色)和非gag特异性(灰色)细胞的FSC-A / SSC-A轮廓。数字表示指示的门中的单元格百分比。该图显示了5个实验中的1个代表性实验。在每个实验中,我们分析了来自3只接种疫苗的小鼠和3只未治疗小鼠的合并脾脏和混合LN细胞。请点击此处查看此图的放大版本。

图4:抗原特异性CD8 T细胞的细胞周期分析。小鼠接种如图3所示,并在增强后的第3天进行gag特异性细胞的细胞周期分析,如图3所示,在5个步骤中门控后进行。(A)来自接种小鼠的LNs(顶部)和脾脏(底部)的gag特异性CD8 T细胞的细胞周期分析示例。细胞周期阶段如图2B所示。这些面板代表G0、G1、S-G2/M(左)和Ki67 mAb(右)的荧光减一(FMO)控制中的细胞。数字表示指示的门中的单元格百分比。(B)FSC-A / SSC-A点图显示S-G2/ M阶段(红色)中的gag特异性CD8 T细胞覆盖在来自接种小鼠的LNs(顶部)和脾脏(底部)的总CD3+CD8+ T细胞(灰色)上。(C)偏移直方图显示来自接种小鼠的LNs(顶部)和脾脏(底部)的G0(绿色),G1(蓝色)和S-G 2 / M(红色)中gg特异性CD8 T细胞的CD62L表达。y 轴表示规范化的事件数。该图显示了总共15只小鼠的5个独立实验中的1个代表性示例。请点击此处查看此图的放大版本。
补充材料:流式细胞仪设置。 请点击此处下载此文件。
Discussion
尽管已经对T细胞克隆扩增进行了深入研究,但某些方面仍然未知,主要是因为可用于研究它的工具很少,并且有其自身的缺点。从这个角度来看,我们建立了一种高灵敏度的流式细胞术方法,以分析小鼠模型中疫苗接种后早期抗原特异性CD8 T细胞的细胞周期。该协议基于Ki67和DNA染色的组合,其先前用于分析小鼠BM造血细胞的细胞周期13,14。为了使方案适应抗原特异性CD8 T细胞,我们必须考虑一些关键问题,包括DNA染料的选择,在不同样品中获得可比DNA染色的适当条件,以及数据分析的门控策略。
许多染料可用于DNA染色,包括碘化丙啶和7-氨基放线菌素D;我们之所以选择Hoechst,是因为它与膜染色和Ki67染色所需的温和固定/透化方案兼容。同时,使用Hoechst染色使我们能够获得高质量的DNA直方图,即G0/ G1 和G2/ M DNA峰的变异系数(CV)远低于通常使用其他DNA染料获得的DNA峰,例如DRAQ519。事实上,Hoechst甚至可以在活细胞中染色DNA20。
使用一些策略来避免同一实验的不同样品中Hoechst强度的波动。在流式细胞仪采集样品之前进行Hoechst染色,以最大限度地减少染料强度随时间的下降。对于那些有兴趣在大量样品的大型实验中重现该协议的人,我们建议一次对几个样品进行Hoechst染色。另一个缺点是,在与染料孵育期间,Hoechst强度可能受到细胞数量的严重影响。因此,我们强烈建议始终使用相同数量的细胞和相同的体积的每个样品进行DNA染色。如果在流式细胞仪上采集需要大量细胞,我们建议制备两个或多个相同的样品,然后在Hoechst染色步骤之前将它们合并。
该协议的一个关键点是用于数据分析的门控策略。我们最近发表了一种在免疫应答早期进行T细胞分析的新策略,这使我们能够提高抗原特异性T细胞检测的灵敏度12。我们将此策略应用于此处显示的数据,如下所示。首先,我们在DNA-A / W图中排除了细胞聚集体。其次,在门控死细胞后,我们在FSC / SSC图中使用了相当大的淋巴细胞门("放松门")。通过这种策略,我们能够在S-G2/ M中包括高度活化的抗原特异性CD8 T细胞,这些细胞通常被当前的门控策略所遗漏,因为这些细胞具有高FSC-A和SSC-A。总之,数据分析代表了该方法的关键部分,这对于获得活化/增殖抗原特异性T细胞的灵敏检测至关重要。
该方法可防止在免疫应答的早期阶段丢失关键T细胞数据的可能性,并为T细胞免疫监测开辟了新的视角。未来的改进可能是包括磷酸化组蛋白3的染色,这将允许区分G2 和M21。目前的限制是细胞必须固定和透化才能染色核标记物Ki67。因此,细胞不能用于其他类型的分析,例如排序和随后的功能分析。此外,DNA染料,包括Hoechst,通常会干扰基因组DNA分析,不适合这种类型的评估。鉴定与不同细胞周期相相关并且可以在活细胞上染色的膜标志物可以克服这一限制。总之,该方法在疫苗接种,感染,免疫介导的疾病和免疫治疗等几种情况下评估活化/增殖的T细胞具有巨大的潜力。
Disclosures
A. Folgori和S. Capone是Reithera Srl. A. Nicosia在专利申请WO 2005071093(A3)"黑猩猩腺病毒疫苗载体"中被任命为发明人。其他作者没有什么可披露的。
Acknowledgments
这项工作得到了Reithera,MIUR项目2017K55HLC_006以及意大利Ricerca sul Cancro协会(AIRC)的5×1000赠款的支持。以下四聚体是通过NIH四聚体设施获得的:APC偶联的H-2K(d)HIV gag 197-205 AMQMLKETI。
Materials
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
References
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- Mouse phenotype. , Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020).
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Tags
免疫学和感染,第167期,抗原特异性CD8 T细胞,细胞周期,Ki67,DNA染料,流式细胞术,脾脏,淋巴结,小鼠Erratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021.
Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
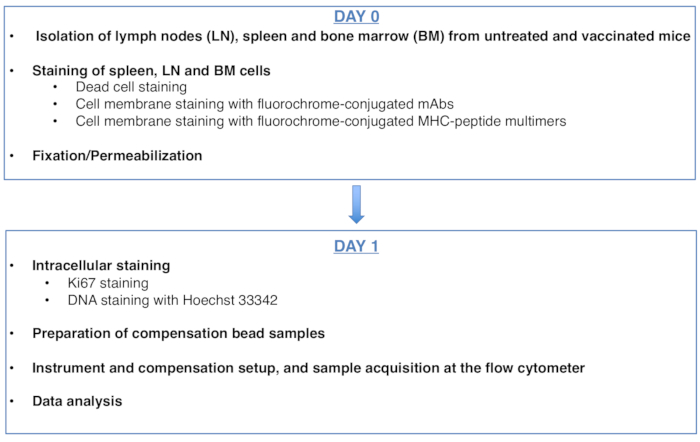
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:
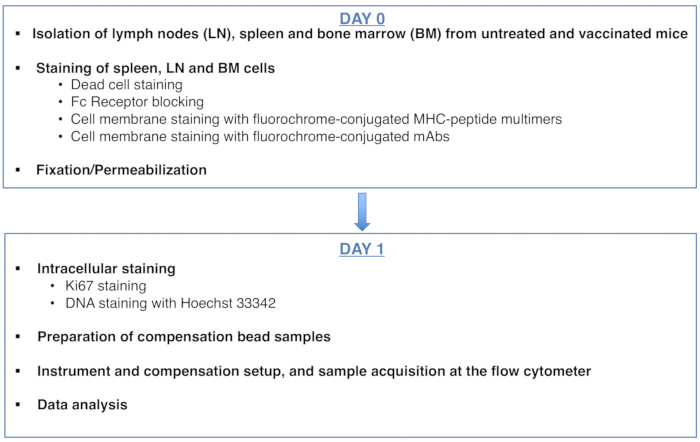
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.



