ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
क्लोनल विस्तार एंटीजन-विशिष्ट टी सेल प्रतिक्रिया की एक प्रमुख विशेषता है। हालांकि, एंटीजन-प्रतिक्रिया टी कोशिकाओं के सेल चक्र खराब जांच की गई है, आंशिक रूप से तकनीकी सीमाओं की वजह से । हम टीका लगाए गए चूहों की तिल्ली और लिम्फ नोड्स में एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं के क्लोनी विस्तार का विश्लेषण करने के लिए एक प्रवाह साइटोमेट्रिक विधि का वर्णन करते हैं।
Abstract
वीवो में एंटीजन-विशिष्ट टी कोशिकाओं के कोशिका चक्र की जांच कुछ तरीकों का उपयोग करके की गई है, जिनमें से सभी के पास कुछ सीमाएं हैं। ब्रोमोडेऑक्सीयूरिडीन (BrdU) उन कोशिकाओं को चिह्नित करता है जो हाल ही में पूरे किए गए एस-चरण में हैं, और कार्बोक्सीफ्लोरेसेइन एससीआईनिमिडिल एस्टर (सीएफएसई) विभाजन के बाद बेटी कोशिकाओं का पता लगाता है। हालांकि, ये रंग विश्लेषण के समय सेल चक्र चरण की पहचान की अनुमति नहीं देते हैं। एक वैकल्पिक दृष्टिकोण Ki67 का फायदा उठाने के लिए है, एक मार्कर जो शांत चरण जी 0 को छोड़कर कोशिका चक्र के सभी चरणों में कोशिकाओं द्वारा अत्यधिक व्यक्त कियाजाताहै। दुर्भाग्य से, Ki67 आगे भेदभाव की अनुमति नहीं देता है क्योंकि यह एस-चरण में कोशिकाओं को अलग नहीं करता है जोजी-1 में उन लोगों से माइटोसिस के लिए प्रतिबद्ध हैं जो इस चरण में रह सकतेहैं, साइकिल िंग में आगे बढ़ें, या जी0में चले जाएं।
यहां, हम माउस माध्यमिक लिम्फाइड अंगों में विभिन्न कोशिका चक्र चरणों में टी कोशिकाओं के "स्नैपशॉट" पर कब्जा करने के लिए एक प्रवाह साइटोमेट्रिक विधि का वर्णन करते हैं। विधि प्रमुख हिस्टोकम्पैटिबिलिटी कॉम्प्लेक्स (एमएचसी) -पेप्टाइड-मल्टीमर स्टेनिंग और एक अभिनव गेटिंग रणनीति के साथ Ki67 और डीएनए धुंधला को जोड़ती है, हमें सफलतापूर्वक जी 0 में एंटीजन विशिष्ट सीडी8टी कोशिकाओं के बीच अंतर करने की अनुमति, जी1 में और एस-जी2/एमचरणों में तिल्ली में कोशिका चक्र के और वायरल वैक्टर के साथ टीकाकरण के बाद चूहों के लिम्फ नोड्स को मानव इम्यूनोडेफिशिएंसी वायरस (एचआईवी)-1 के मॉडल एंटीजन गैग ले जाने के बाद ।
विधि के महत्वपूर्ण कदम डीएनए डाई और गेटिंग रणनीति का चुनाव परख संवेदनशीलता को बढ़ाने के लिए और अत्यधिक सक्रिय/प्रसार एंटीजन विशिष्ट टी कोशिकाओं है कि विश्लेषण के वर्तमान मापदंड से याद किया गया होता शामिल थे । डीएनए डाई, Hoechst ३३३४२, हमें जी0/जी 1और जी2/एमडीएनए चोटियों के एक उच्च गुणवत्ता वाले भेदभाव प्राप्त करने के लिए सक्षम है, जबकि झिल्ली और इंट्रासेलुलर धुंधला संरक्षण । विधि वीवो में टी सेल प्रतिक्रिया के बारे में ज्ञान बढ़ाने के लिए और इम्यूनो निगरानी विश्लेषण में सुधार करने के लिए महान क्षमता है ।
Introduction
भोले टी कोशिकाओं क्लोनल विस्तार और एंटीजन-प्राइमिंग पर भेदभाव से गुजरना । विभेदित टी कोशिकाएं प्रभावकार कार्यों को प्रदर्शित करती हैं जो एंटीजन क्लीयरेंस के लिए आवश्यक हैं और एंटीजन-विशिष्ट स्मृति के रखरखाव के लिए, जो लंबे समय तक चलने वाली सुरक्षा के लिए महत्वपूर्ण है। प्राथमिक प्रतिक्रिया के पहले चरणों के दौरान, लिम्फोइड अंगों में विशेष निकस के भीतर एंटीजन-पेश करने वाली कोशिकाओं (एपीसीएस) के साथ भोली टी सेल बातचीत विशाल टी सेल प्रसार को प्रेरित करने के लिए महत्वपूर्ण है जो क्लोनल विस्तार चरण1,2,3की विशेषता है। टी सेल-एपीसी इंटरैक्शन को एंटीजन, सह-उत्तेजक संकेतों, और घुलनशील कारकों (साइटोकिन्स और केमोकिन्स) की एकाग्रता और दृढ़ता से बारीकी से विनियमित किया जाता है जोटी सेल क्लोनल संतान4,5,6,7की मात्रा और गुणवत्ता को प्रभावित करते हैं।
टी सेल क्लोनल विस्तार के गहन अध्ययन के बावजूद, यह अभी भी ज्ञात नहीं है कि एंटीजन-प्राइमेड टी कोशिकाएं एंटीजन मान्यता की साइट पर अपने पूरे सेल चक्र को पूरा करती हैं, या क्या वे कोशिका चक्र प्रगति के दौरान अन्य अंगों में स्थानांतरित होते हैं। ज्ञान की यह कमी सेल चक्र विश्लेषण के लिए उपलब्ध उपकरणों के गुणों के कारण है। इनमें परमाणु मार्कर, Ki67 और सेल रंगों के लिए विशिष्ट मोनोक्लोनल एंटीबॉडी (एमएबी) शामिल हैं जो या तो कोशिका चक्र (जैसे ब्रोमोडेऑक्सीयूरिडीन (BrdU)) के एस-चरण से गुजरने वाली कोशिकाओं की पहचान करते हैं या बेटी कोशिकाओं और उनके पूर्वजों (जैसे, कार्बोक्सीफ्लोरेसिन सुसिनिमिडिल एस्टर (CFSE)) के बीच भेदभाव करते हैं।
हालांकि, सीएफएसई और ब्रेडू जैसे सेल-लेबलिंग रंग इस दृढ़ संकल्प की अनुमति नहीं देते हैं कि क्या किसी विशेष अंग में पाई जाने वाली कोशिकाएं स्थानीय रूप से बढ़ी हैं या बल्कि डिवीजन8,9के बाद इस साइट पर चले गए थे। इसके अलावा, इंट्रान्यूक्लियर प्रोटीन, Ki67, केवल किसी भी अन्य कोशिका चक्र चरण (Ki67-सकारात्मक कोशिकाओं) में उन लोगों से जी0 (Ki67-नकारात्मक कोशिकाओं) में कोशिकाओं को अलग करने में सक्षम है । इस प्रकार, Ki67 विश्लेषण सक्रिय प्रसार में कोशिकाओं को अलग नहीं करता है (यानी, एस, जी2,या एम में) जी1में उन लोगों से, जो या तो जल्दी से विभाजन के लिए प्रगति या जी1 में लंबी अवधि के लिए रह सकते है या quiescence10, 11को वापस ।
यहां, हम टीका लगाए गए चूहों (चित्रा1) की तिल्ली और लिम्फ नोड्स (एलएनएस) से एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं 12 के सेल चक्र विश्लेषण के लिए एक नई प्रवाह साइटोमेट्रिक विधि का वर्णनकरते हैं। यह विधि Ki67 और डीएनए धुंधला के संयोजन का शोषण करती है जिसका उपयोग पहले माउस बोन मैरो (बीएम) हेमेटोपोइटिक कोशिकाओं13,14के कोशिका चक्र का विश्लेषण करने के लिए किया जाता था । यहां, हमने सीडी 8 टी सेल क्लोनल विस्तार के विश्लेषण के लिए हाल ही में प्रकाशित अभिनव गेटिंग रणनीति12के साथ-साथ Ki67 प्लस डीएनए धुंधला को सफलतापूर्वक लागू किया। हम जी 0 में एंटीजन-विशिष्ट सीडी8टी कोशिकाओं के बीच स्पष्ट रूप से भेदभाव करने में सक्षम थे, जी1में, और एस-जी2/एमचरणों में तिल्ली में और टीका लगाया चूहों के एलएन draining ।
Protocol
चूहों को Plaisant पशु सुविधा में रखे गए थे, और यह काम इटली के स्वास्थ्य प्राधिकरण संख्या 1065/2015-पीआर मंत्रालय के तहत किया गया था । प्रोटोकॉल राष्ट्रीय और अंतरराष्ट्रीय कानूनों और नीतियों (UE निर्देश 2010/63/UE) के अनुसार पशु देखभाल दिशा निर्देशों का पालन किया; इतालवी विधायी डिक्री 26/2014) ।
1. मध्यम और धुंधला समाधान की तैयारी
- तैयार करें पूरा माध्यम: रोसवेल पार्क मेमोरियल इंस्टीट्यूट (आरपीएमआई) माध्यम 2 एमएमएम ग्लूटामाइन के साथ, 100 यू/एमएल पेनिसिलिन/स्ट्रेप्टोमाइसिन, 50 माइक्रोन बीटा-मर्केप्टोथेनॉल, और भ्रूण गोजातीय सीरम (एफबीएस) की 10% मात्रा/मात्रा (v/v)
- दाग बफर तैयार करें: 1% वजन/मात्रा (w/v) गोजातीय सीरम एल्बुमिन (बीएसए) और 2 एमएम एथिलीनडाइमाएंट्रेएक्टिक एसिड डिसोडियम नमक (EDTA) के साथ Ca2 +/mg2 + (PBS) के बिना फॉस्फेट-बफर खारा
2. माउस उपचार
- प्राइम 7-8-वीक, मादा बाल्ब/सी चूहों द्वारा इंट्रामस्क्युलर (i.m)) मानव इम्यूनोडेफिशिएंसी वायरस (एचआईवी) के क्वाड्रिसेप्स में इंजेक्शन-1-गैग-एक्सप्रेसिंग-चिंपांजी एडेनोविरल वेक्टर (ChAd3-gag) १०७ वायरल कणों की खुराक के साथ ।
- भड़काना के बाद 1-4 महीने में, एक बार एचआईवी के .m इंजेक्शन द्वारा चूहों को बढ़ावा-1-चुप-संशोधित vaccinia अंकारा वायरस (MVA-चुप) १०६ पट्टिका बनाने इकाइयों की एक खुराक के साथ व्यक्त ।
- बढ़ावा देने के बाद 3 दिन में, गर्भाशय ग्रीवा अव्यवस्था द्वारा बढ़ाया चूहों का बलिदान, और अनुपचारित चूहों के साथ समानांतर में उनका विश्लेषण।
- क्वाड्रिसेप्स (इलियाक, पॉपलाइटल, और इंगिनल) और बूस्ट और अनुपचारित चूहों से प्लीहा को निकालने वाले एलएनएस को फसल करें। इसके अलावा, अनुपचारित चूहों से दो पिछले पैरों से बीएम इकट्ठा करें, और प्रवाह साइटोमीटर सेटिंग्स के लिए और सेल चक्र विश्लेषण(चित्रा 2)के लिए सकारात्मक नियंत्रण के रूप में इस बीएम का उपयोग करें।
नोट:पहले12, 15, 16, 17वर्णित ChAd3-gag और एमवीए-गैग वैक्टर उत्पन्न करें ।
3. एलएन, तिल्ली और बीएम कोशिकाओं को निकालने का अलगाव
- तिल्ली और एलएन कोशिकाओं का अलगाव
- दो 15 एमएल ट्यूबों में से प्रत्येक में पूर्ण माध्यम के 5 एमएल रखें, और अंगों को एकत्र करने के लिए तैयार, उन्हें बर्फ पर रखें।
- सर्वाइकल अव्यवस्था द्वारा एक वयस्क माउस का बलिदान करें।
- माउस को अपनी पीठ पर रखें, और 70% v/v इथेनॉल के साथ त्वचा की सतह को निष्फल करें।
- इंजिनियल एलएन इकट्ठा करने के लिए, कैंची के साथ पेट पर ~ 1 सेमी अनुदैर्ध्य चीरा बनाएं, और संदंश के साथ चीरा फैलाएं।
- त्वचा की आंतरिक सतह पर इंगिनल एलएन की कल्पना करें, और उन्हें संदंश के साथ फसल करें। स्टेप 3.1.1 में तैयार दो 15 एमएल ट्यूब में से एक में इंजिनियल एलएन रखें।
- तिल्ली इकट्ठा करने के लिए कैंची से पेरिटोनियल चीरा लगाकर तिल्ली निकाल लें। आसपास के कनेक्टिव टिश्यू को काटने के बाद, प्लीहा को दूसरे 15 एमएल ट्यूब में रखें जो चरण 3.1.1 में तैयार किया गया है।
- इलियाक एलएन इकट्ठा करने के लिए, आंतों को एक तरफ ले जाएं और अवर वेना कावा के करीब इलियाक एलएन की कल्पना करें, और फिर उन्हें संदंश का उपयोग करके इकट्ठा करें। इलियाक एलएन को एक ही ट्यूब में रखें जिसमें इंजिनियल एलएन हो।
नोट: धुंधला (धारा 4 देखें) के लिए पर्याप्त एलएन कोशिकाओं को प्राप्त करने के लिए, अक्सर एक माउस से पॉपलाइटल, इंगिनल और इलियाक एलएन पूल करना आवश्यक होता है। ये सभी एलएन क्वाड्रिसेप्स (आई.m टीकाकरण की साइट) को सूखा रहे हैं। यह प्रोटोकॉल पूल किए गए एलएन की केवल एक 15 एमएल ट्यूब का उपयोग करता है। - पॉपलाइटल एलएन इकट्ठा करने के लिए, पिछले पैरों की त्वचा को समझें और मांसपेशियों को उजागर करने के लिए धीरे-धीरे इसे नीचे की ओर खींचें। फिर, घुटने के जोड़ के नीचे मांसपेशियों के बीच संदंश डालें, और पॉपलाइटल एलएन एकत्र करें। पॉपलाइटल एलएन को एक ही ट्यूब में रखें जिसमें इंजिनियल और इलियाक एलएन होते हैं।
नोट: 3.1.7 के बाद नोट देखें। - तिल्ली को 5 मिमी संस्कृति के 5 एमएल से भरे 60 मिमी संस्कृति पकवान के भीतर 70 माइक्रोन सेल छलनी में रखें। 5 एमएल सिरिंज प्लंजर का उपयोग करके, धीरे-धीरे अंग को तब तक मैश करें जब तक कि इसकी पूरी डिसैगरेशन न हो जाए।
- छलनी निकालें, और सेल निलंबन को एक साफ 15 एमएल ट्यूब में स्थानांतरित करें।
- संस्कृति पकवान के लिए पूरा माध्यम के 5 एमएल जोड़ें, और ध्यान से पकवान और छन्नी धोने के लिए सुनिश्चित करें कि सभी कोशिकाओं को बरामद किया गया है । 15 एमएल ट्यूब में तिल्ली सेल निलंबन के बाकी के साथ पूल।
- पूल्ड इंगिनल, इलियाक और पॉपलाइटल एलएन के लिए, तिल्ली के लिए चरण 3.1.9 से 3.1.11 में उपयोग की जाने वाली इसी प्रक्रिया के बाद एक एकल सेल निलंबन तैयार करें।
- 4 डिग्री सेल्सियस पर 10 मिनट के लिए 400 × ग्राम पर सेंट्रलाइज कोशिकाएं। सुपरनैंट को त्यागें, और पीबीएस में सेल छर्रों को फिर से खर्च करें।
- लाल रक्त कोशिका लाइसिस बफर और पीबीएस में 0.04% v/v ट्राइपैन ब्लू का उपयोग करके न्यूबॉर चैंबर के साथ कोशिकाओं की गणना करें।
- बीएम कोशिकाओं का अलगाव
- 15 एमएल ट्यूब में 5 एमएल कंप्लीट मीडियम रखें, और इसे बर्फ पर रखें, जो पिछले पैरों के कलेक्शन के लिए तैयार है ।
- सर्वाइकल अव्यवस्था द्वारा एक वयस्क माउस का बलिदान करें।
- 70% v/v इथेनॉल के साथ त्वचा की सतह को स्टरलाइज करें।
- कैंची के साथ वेंट्रल त्वचा पर ~ 1 सेमी ट्रांसवर्स चीरा बनाएं, कट के दोनों किनारों पर त्वचा को दृढ़ता से समझें, और पिछले पैरों की मांसपेशियों को उजागर करने के लिए धीरे-धीरे नीचे की ओर खींचें।
- पिछले पैरों के पीछे से त्वचा को खत्म करने के लिए, माउस को एक रीढ़ की स्थिति में रखते हुए, क्लैंप को घुटने के नीचे रखें, और मांसपेशियों को बेनकाब करने के लिए ऊपर की ओर खींचें।
- एक हिंद पैर के दो छोर पर हड्डियों को काटें: श्रोणि/कूल्हे के जोड़ और टखने ।
- दोनों पिछले पैरों को चरण 3.2.1 में तैयार 15 एमएल ट्यूब पर स्थानांतरित करें। ट्यूब को बर्फ पर रखें।
- 15 एमएल ट्यूब से पिछले पैरों को लें और उन्हें टिश्यू पेपर में स्थानांतरित करें। टिबिया को दूर करने के लिए घुटने के जोड़ के ठीक नीचे पिछले पैरों को काट लें। आसपास की मांसपेशियों से फीमर और टिबिया को विच्छेदन करें, कैंची का उपयोग करके अतिरिक्त ऊतक निकालें, और ऊतक पेपर को गीला करें।
- आंतरिक मज्जा शाफ्ट का पर्दाफाश करने के लिए कैंची के साथ हड्डी समाप्त होता है काटें। बीएम निष्कर्षण ट्यूब में टिबिया और फीमर डालें (नीचे सबसे व्यापक अंत के साथ 3.2.9.1-3.2.2.18 में तैयारी देखें।
- टिप के अंत के ठीक ऊपर और 100 माइक्रोल लाइन पर लाइन पर 200 माइक्रोन पिपेट टिप काटें।
- मध्य भाग को टिप के ऊपरी, बड़े हिस्से में रखें, और इसे 1.5 एमएल माइक्रोफ्यूज ट्यूब में रखें।
- 1 मिनट के लिए 800 × जी पर बीएम निष्कर्षण ट्यूब स्पिन।
- हड्डी को त्यागें, और किसी भी समूह को हटाने के लिए पूर्ण माध्यम के 1 एमएल में गोली को सख्ती से फिर से खर्च करें। 15 एमएल ट्यूब के शीर्ष पर रखे गए 70 माइक्रोन फ़िल्टर के माध्यम से सेल निलंबन को फ़िल्टर करें।
- बीएम निष्कर्षण ट्यूब को हर बार 1 एमएल पूर्ण माध्यम से धोएं। एक 70 माइक्रोन फ़िल्टर के माध्यम से फ़िल्टर करें, और चरण 3.2.11 में प्राप्त शेष सेल निलंबन के साथ वॉल्यूम पूल करें।
नोट: एक एकल 15 एमएल ट्यूब में माउस के दोनों पिछले पैरों से कोशिकाएं होंगी। - 4 डिग्री सेल्सियस पर 10 मिनट के लिए 400 × ग्राम पर सेंट्रलाइज कोशिकाएं। सुपरनेट को त्यागें, और पीबीएस में सेल पेलेट को फिर से खर्च करें।
- लाल रक्त कोशिका लाइसिस बफर और पीबीएस में 0.04% v/v ट्राइपैन ब्लू का उपयोग करके न्यूबॉर चैंबर के साथ कोशिकाओं की गणना करें।
4. तिल्ली, एलएन, और बीएम कोशिकाओं का धुंधला
- सेल नमूनों को 3 उपसमूहों में दाग दिया जाएगा: मुआवजे के लिए सेल नमूने,अनुपचारित चूहों से बीएम कोशिकाओं सहित केवल Hoechst ३३३४२ के साथ दाग हो (अब से Hoechst के रूप में संदर्भित) और अनुपचारित चूहों से तिल्ली कोशिकाओं को मृत सेल डाई मुआवजे के लिए एक मृत/लाइव सेल मिश्रण तैयार करने के लिए इस्तेमाल किया जाएगा; कोशिका चक्र विश्लेषण के लिए सकारात्मक नियंत्रण,अनुपचारित चूहों से बीएम नमूना शामिल है; और अनुपचारित और टीका लगाया चूहों से तिल्ली और एलएन नमूनों युक्त प्रयोगात्मक नमूने ।
नोट: सुनिश्चित करें कि गैग-विशिष्ट सीडी 8 टी कोशिकाओं की पर्याप्त संख्या के विश्लेषण के लिए पर्याप्त तिल्ली और एलएन कोशिकाएं हैं। यह अक्सर पूल तिल्ली कोशिकाओं का उपयोग करें और 3 टीका लगाया चूहों से पूल एलएन कोशिकाओं का उपयोग करने के लिए आवश्यक है और पूल्ड कोशिकाओं के दो या अधिक समान नमूनों दाग, प्रत्येक 3 × १०६ कोशिकाओं युक्त । Hoechst धुंधला के कदम पर समान नमूनों को मर्ज करें। इसी तरह, दाग 3 अनुपचारित चूहों से तिल्ली कोशिकाओं और एलएन कोशिकाओं पूल, और अंत में समान नमूनों विलय । उपकरण और क्षतिपूर्ति सेटअप के लिए उपयोग किए जाने वाले अनुपचारित माउस से तिल्ली कोशिकाओं का एक अन दाग नमूना अलग सेट करें। - मृत सेल डाई क्षतिपूर्ति के लिए मृत/लाइव सेल मिश्रण तैयार करें (कोशिकाओं का यह मिश्रण केवल मृत सेल डाई के साथ दाग दिया जाएगा)।
- 65 डिग्री सेल्सियस पर पानी स्नान गर्म करें।
- तिल्ली कोशिकाओं का एक aliquot ले लो (~ 3 × 106)।
- सेल सस्पेंशन को माइक्रोफ्यूज ट्यूब में ट्रांसफर करें, इसे 5 मिनट के लिए 65 डिग्री सेल्सियस पर पानी के स्नान में रखें, और फिर तुरंत इसे 10 मिनट के लिए बर्फ पर रखें।
- 1:1 के अनुपात में जीवित तिल्ली कोशिकाओं (~ 3 ×10 6)के साथ गर्मी से मारे गए कोशिकाओं को मिलाएं, और मिश्रण के आधे हिस्से को 96 अच्छी तरह से गोल नीचे की प्लेट में स्थानांतरित करें (~ 3 × 106 कोशिकाएं/
- प्रायोगिक नमूनों की मृत कोशिका धुंधला, सेल चक्र विश्लेषण के लिए सकारात्मक नियंत्रण और मृत/
- तिल्ली, एलएन, बीएम कोशिकाओं (3 × १० ६ कोशिकाओं/अच्छीतरह से) हस्तांतरण, और मृत/लाइव सेल मिश्रण (धारा ४.२) ९६ में अच्छी तरह से गोल नीचे थाली, धुंधला योजना (कदम ४.१) के अनुसार, और ४०० × जी पर 4 डिग्री सेल्सियस पर 3 मिनट के लिए अपकेंद्रित्र ।
- पीबीएस में पतला मृत सेल डाई के 50 माइक्रोन में प्रत्येक सेल गोली को फिर से रीसुस्ट करें, और तुरंत 3 बार ऊपर और नीचे पाइपिंग करके पुनर्व्यवित करें।
- 4 डिग्री सेल्सियस पर 30 मिनट के लिए इनक्यूबेट, प्रकाश से संरक्षित।
- धुंधला बफर के साथ 2 बार कोशिकाओं को धोएं; 200 माइक्रोन के साथ पहली बार और दूसरी बार 250 माइक्रोन के साथ। प्रत्येक धोने के लिए 4 डिग्री सेल्सियस पर 3 मिनट के लिए 400 × जी पर प्लेट अपकेंद्रित्र करें।
- सुपरनेट को त्यागें, और पीबीएस के 20 माइक्रोन में सेल पेलेट को फिर से खर्च करें।
- प्रमुख हिस्टोकंप्यूटिबिलिटी कॉम्प्लेक्स (एमएचसी)-पेप्टाइड मल्टीमर्स और एमएबी के साथ झिल्ली सेल धुंधला ।
- स्टेनिंग स्कीम (फ्लो साइटोमीटर सेटिंग्स, टेबल 1)के अनुसार आवश्यक मात्रा को ध्यान में रखते हुए, निम्नलिखित रिएजेंट तैयार करें:
- उचित कमजोर पड़ने के अनुसार धुंधला बफर में तनु mAb 2.4G2 (सामग्री की तालिकादेखें); प्रत्येक नमूने को दाग लगाने के लिए, इस कमजोर पड़ने के 10 माइक्रोन का उपयोग करें।
नोट: 2.4G2 mAb ब्लॉक गैर-एंटीजन-विशिष्ट FcγIII और FcγII रिसेप्टर्स के लिए इम्यूनोग्लोबुलिन के बाध्यकारी । - उचित कमजोर पड़ने (सामग्रीकी तालिका देखें) प्राप्त करने के लिए धुंधला बफर में एच-2k (d) AMQMLKETI allophycocyanin (एपीसी) लेबल टेट्रामर (Tetr-gag) पतला; प्रत्येक नमूने को दागदार करने के लिए, इस कमजोर पड़ने के 20 माइक्रोन का उपयोग करें।
- उपयुक्त कमजोर पड़ने के अनुसार धुंधला बफर में एमएबी को पतला करके एंटीबॉडी मिश्रण तैयार करें (सामग्री की तालिकादेखें) जो पहले टिटरेशन प्रयोगों में निर्धारित किया गया है; प्रत्येक नमूने को दाग लगाने के लिए, इस एंटीबॉडी मिश्रण के 20 माइक्रोन का उपयोग करें।
नोट: यहां, एंटी-सीडी3ई पेरिडिन क्लोरोफिल प्रोटीन (PerCP-Cy5.5) (क्लोन 145-2C11), एंटी-सीडी 8ए शानदार पराबैंगनी (BUV805) (क्लोन 53-6.7), और एंटी-सीडी62एल फाइकोरीथ्रिन सायनिन7 (PECy7) (क्लोन मेल-14) का उपयोग किया गया ।
- उचित कमजोर पड़ने के अनुसार धुंधला बफर में तनु mAb 2.4G2 (सामग्री की तालिकादेखें); प्रत्येक नमूने को दाग लगाने के लिए, इस कमजोर पड़ने के 10 माइक्रोन का उपयोग करें।
- पहले पतला 2.4 G2 mAb (चरण 4.4.1.1) के 10 माइक्रोन जोड़ें, और प्रकाश से संरक्षित 4 डिग्री सेल्सियस पर 10 मिनट के लिए इनक्यूबेट करें।
- पहले पतला Tetr-gag एपीसी (कदम 4.4.1.2) और एच-2k (d) AMQMLKETI phycoerythrin (पीई) पेंटामर (दबी हुई झूठ) के 10μL जोड़ें । 4 डिग्री सेल्सियस पर 15 मिनट के लिए इनक्यूबेट, प्रकाश से संरक्षित।
- पहले से तैयार एंटीबॉडी मिश्रण (चरण 4.4.1.3) के 20 माइक्रोन जोड़ें, और प्रकाश से सुरक्षित 4 डिग्री सेल्सियस पर 15 मिनट इनक्यूबेट करें।
नोट: इसलिए, अंतिम मात्रा प्रति अच्छी तरह से 80 माइक्रोन है (चरण 4.3.5, चरण 4.4.2 से 4.4.4)। - धुंधला बफर के 200 माइक्रोन के साथ कोशिकाओं को धोएं। 4 डिग्री सेल्सियस पर 5 मिनट के लिए 400 × जी पर सेंट्रलाइज।
- 250 माइक्रोन में सेल पेलेट को फिर से रीसुस्ल करें, और सेल सस्पेंशन को 5 एमएल ट्यूब में स्थानांतरित करें। ट्यूब में 1 एमएल धुंधला बफर जोड़ें, और 4 डिग्री सेल्सियस पर 5 मिनट के लिए 400 × जी पर अपकेंद्रित्र करें।
- बीएम कोशिकाओं के aliquot ले लो (3 × १०६ कोशिकाओं) (सेल नमूनों की सूची देखें, धारा ४.१) Hoechst चैनल क्षतिपूर्ति करने के लिए इस्तेमाल किया जा करने के लिए (Hoechst ३३३४२ एक पराबैंगनी लेजर (प्रवाह साइटोमीटर सेटिंग्स(तालिका 2))से उत्साहित है, और सेल निलंबन एक 5 mL ट्यूब में स्थानांतरित । ट्यूब में 1 एमएल धुंधला बफर जोड़ें, और 4 डिग्री सेल्सियस पर 5 मिनट के लिए 400 × जी अपकेंद्रित्र करें।
- स्टेनिंग स्कीम (फ्लो साइटोमीटर सेटिंग्स, टेबल 1)के अनुसार आवश्यक मात्रा को ध्यान में रखते हुए, निम्नलिखित रिएजेंट तैयार करें:
5. फिक्सेशन/पारमेबिलाइजेशन
- निर्माता के निर्देशों के अनुसार निर्धारण/पारमेबिलाइजेशन मंद के 3 भागों के साथ निर्धारण/पारमेबिलाइजेशन ध्यान के 1 भाग को कमजोर करके नए सिरे से निर्धारण/पारमेबिलाइजेशन बफर तैयार करें ।
- पैलेट और नाड़ी भंवर को पैलेट को पूरी तरह से अलग करने के लिए नमूनों को त्यागें।
- प्रत्येक ट्यूब में ताजा तैयार निर्धारण/पारमेबिलाइजेशन बफर के 1 एमएल जोड़ें, जिसमें अन दाग तिल्ली कोशिकाओं के साथ एक ट्यूब (3 x 106,सेल नमूनों की सूची देखें, धारा 4.1), और भंवर।
- 4 डिग्री सेल्सियस पर 16 घंटे के लिए इनक्यूबेट।
नोट: प्रोटोकॉल यहां रोका जा सकता है ।
6. इंट्रासेलुलर धुंधला
- Ki67 धुंधला
- निर्माता के निर्देशों के अनुसार, आसुत पानी के साथ पारशीलीकरण बफर 10x को पतला करके ताजा पारमेबिलाइजेशन बफर 1x तैयार करें। उपयोग से पहले, पारमेबिलाइजेशन बफर 1x को समुच्चय को खत्म करने के लिए 0.45 माइक्रोन फ़िल्टर के माध्यम से फ़िल्टर किया जाना चाहिए।
- पतला mAb Ki67 फ्लोरोसेइन आइसोथिओसाइनेट (फिटसी) (क्लोन सोला15) में परमेबिलाइजेशन बफर 1x (सामग्री की तालिकादेखें), जैसा कि पहले टिटरेशन प्रयोगों (प्रति नमूना 100 माइक्रोल की अंतिम मात्रा) में निर्धारित किया गया था।
- प्रत्येक ट्यूब में परमीबिलाइजेशन बफर 1x का 3 एमएल जोड़ें, और कमरे के तापमान (आरटी) पर 5 मिनट के लिए 400 × ग्राम पर अपकेंद्रित्र करें।
- सुपरनेट को त्यागें और चरण 6.1.3 दोहराएं।
- सुपरनैंट को त्यागें, और पहले पतला mAb Ki67 FITC (चरण 6.1.2) के 100 माइक्रोल में सेल पेलेट को फिर से खर्च करें।
- आरटी में 30 मिनट के लिए इनक्यूबेट, प्रकाश से संरक्षित।
- परिधि बफर 1x के 4 एमएल के साथ 2 बार कोशिकाओं को धोएं। आरटी में 5 मिनट के लिए 400 × जी पर प्रत्येक धोने अपकेंद्रित्र के लिए।
- निम्नलिखित खंडों पर विचार करते हुए पीबीएस में सेल पेलेट को फिर से रीसुस्ल करें: नमूनों के लिए पीबीएस के 350 माइक्रोन प्रवाह साइटोमीटर पर सीधे प्राप्त किए जाएंगे; नमूनों के लिए पीबीएस के 250 μL प्रवाह साइटोमेट्री (धारा 6.2) से पहले शीघ्र ही Hoechst के साथ इनक्यूबेटेड किया जाएगा।
- डीएनए धुंधला
- प्रत्येक नमूने में पीबीएस में 4 μg/mL Hoechst के २५० μL जोड़ें (Hoechst की अंतिम एकाग्रता 2 μg/mL है) ।
नोट: यदि पीबीएस में 250 माइक्रोन के दो या अधिक समान नमूने तैयार किए गए थे, तो उन्हें इस चरण में मर्ज करें, और पीबीएस में 4 माइक्रोन/एमएल होचस्ट समाधान की समान मात्रा जोड़ें (Hoechst की अंतिम एकाग्रता 2 μg/l है)। कोशिकाओं की संख्या डीएनए धुंधला कदम को बहुत प्रभावित करती है। प्रत्येक नमूने में एक ही सेल नंबर का उपयोग करें। ध्यान रखें कि यहां तक कि थोड़ा कम सेल संख्या (उदाहरण के लिए, पिछले धोने के चरणों में सेल हानि के कारण) डीएनए और उच्च Hoechst तीव्रता के लिए बाध्यकारी उच्च Hoechst में परिणाम है । - आरटी में 15 मिनट के लिए इनक्यूबेट, प्रकाश से संरक्षित।
- 400 × जी पर नमूनों को आरटी में 5 मिनट के लिए सेंट्रलाइज करें।
- पीबीएस के 350 माइक्रोन में सेल पेलेट को फिर से रीस्ब करें।
- प्रत्येक नमूने में पीबीएस में 4 μg/mL Hoechst के २५० μL जोड़ें (Hoechst की अंतिम एकाग्रता 2 μg/mL है) ।
7. मुआवजा मनका नमूनों की तैयारी
- धुंधला बफर में उचित रूप से एमएबी को पतला करके एंटीबॉडी के 5 माइक्रोन तैयार करें।
नोट: प्रयोग में उपयोग किए जाने वाले प्रत्येक फ्लोरोक्रोम-संयुग्मित एमएबी के लिए, इसकी इसी क्षतिपूर्ति मनका नमूना तैयार करें। - भंवर नकारात्मक नियंत्रण और विरोधी चूहा/हम्सटर आईजी, उपयोग से पहले Comp मोती ।
- प्रत्येक नमूने के लिए, नकारात्मक नियंत्रण CompBeads की एक बूंद (~ 20 μL) और विरोधी चूहा की एक बूंद/
- ट्यूब में पूर्वारक्षित एंटीबॉडी (चरण 7.1) के 5 माइक्रोन जोड़ें, और ऊपर और नीचे पिपेट करें।
- 4 डिग्री सेल्सियस पर 15 मिनट के लिए इनक्यूबेट, प्रकाश से संरक्षित।
- धुंधला बफर के 2 एमएल के साथ नमूने धोएं। 4 डिग्री सेल्सियस पर 5 मिनट के लिए 400 × जी पर सेंट्रलाइज।
- सुपरनेट को त्यागें, और प्रत्येक ट्यूब और भंवर में पीबीएस के 500 माइक्रोन जोड़कर गोली को फिर से खर्च करें।
8. उपकरण और मुआवजा सेटअप और प्रवाह साइटोमीटर पर प्रयोगात्मक नमूना अधिग्रहण
नोट: साइटोमीटर कॉन्फ़िगरेशन के लिए फ्लो साइटोमीटर सेटिंग्स(टेबल 2)को देखें।
- सामान्य साधन और मुआवजा सेटअप
- नमूना अधिग्रहण (सामग्री की तालिकादेखें) के लिए सॉफ्टवेयर खोलें, और कार्यक्षेत्र रिबन अनुभाग में नए प्रयोग पर क्लिक करके और नए खाली प्रयोगका चयन करके एक नया प्रयोग बनाएं।
- इसे खोलने के लिए बनाए गए प्रयोग पर डबल क्लिक करें।
- साइटोमीटर सेटिंग्स विंडो में, मापदंडों पर क्लिक करें और फॉरवर्ड स्कैटर (एफएससी) और साइड स्कैटर (एसएससी) पैरामीटर सहित स्टेनिंग पैनल में उपयोग किए जाने वाले सभी चैनलों (जैसे, पीई, एपीसी, आदि) का चयन करें।
- लॉग स्केल को अनचेक करके एक होचस्ट पैरामीटर के रूप में रैखिक पैमाने का चयन करें, और एफसीएस, एसएससी और होचस्ट के लिए वोल्टेज पल्स की चौड़ाई (डब्ल्यू) की जांच करें।
नोट: सभी पैरामीटर लॉगरिथमिक (लॉग) स्केल में डिफ़ॉल्ट रूप से दिखाए जाते हैं, सिवाय एफएससी और एसएससी जो रैखिक पैमाने पर हैं। सभी मापदंडों का विश्लेषण वोल्टेज पल्स के क्षेत्र (ए) और ऊंचाई (एच) द्वारा किया जाता है। - ग्लोबल वर्कशीटपर वाई-एक्सिस पर एक्स-एक्सिस और एसएससी-ए पर एफएससी-ए के साथ डॉट प्लॉट बनाएं ।
- अधिग्रहण डैशबोर्ड पर अधिग्रहण डेटा पर क्लिक करके अन दाग तिल्ली नमूना चलाएं।
- पैरामीटर अनुभाग में वोल्टेज मूल्यों को संशोधित करके कोशिकाओं की कल्पना करने के लिए उपयुक्त एफएससी और एसएससी सेटिंग्स सेट करें, और ग्लोबल वर्कशीटके कार्यक्षेत्र टूलबार पर बहुभुज गेट पर क्लिक करके एफएससी-ए/एसएससी-ए डॉट प्लॉट में प्रदर्शित सभी कोशिकाओं का चयन करने के लिए एक गेट बनाएं।
- एक्स-एक्सिस पर प्रत्येक फ्लोरेसेंस पैरामीटर के साथ हिस्टोग्राम में गेटेड कोशिकाओं को प्रदर्शित करें।
- प्रत्येक फ्लोरेसेंस पैरामीटर के लिए दाग कोशिकाओं के नकारात्मक और सकारात्मक संकेतों के बीच एक स्पष्ट जुदाई है फ्लोरेसेंस डिटेक्टर (पीएमटी) को समायोजित करने के लिए दागदार और पूरी तरह से दाग तिल्ली नमूनों को चलाएं।
- क्षतिपूर्ति सेटअप करने के लिए, कार्यक्षेत्र रिबन में प्रयोग पर क्लिक करें और क्षतिपूर्ति सेटअप अनुभाग के तहत, क्षतिपूर्ति नियंत्रण बनाएंका चयन करें। अनियंत्रित अनग्रेस्ड कंट्रोल ट्यूब/वेल और क्लिक करें ठीक हैशामिल हैं ।
नोट: इस ऑपरेशन के परिणामस्वरूप मुआवजा नियंत्रण नाम के एक नमूने और प्रत्येक चयनित पैरामीटर के अनुरूप कई शीट युक्त एक सामान्य वर्कशीट का निर्माण होगा। - मुआवजे के मोतियों का एक नमूना चलाएं (धारा 7 देखें); साइटोमीटर विंडो में एफएससी मापदंडों पर वोल्टेज मूल्यों और 5,000 की अधिग्रहण सीमा को संशोधित करके मोतियों की कल्पना करने के लिए उपयुक्त एफएससी और एसएससी सेटिंग्स सेट करें।
- मनका आबादी पर P1 गेट समायोजित करें, और जांच करें कि सकारात्मक और नकारात्मक चोटियों दोनों एक्स-अक्ष पर दिखाई दे रहे हैं। प्रत्येक मुआवजा मनका नमूने के लिए इस ऑपरेशन को दोहराएं, और अंत में अधिग्रहण डैशबोर्ड पर रिकॉर्ड डेटा पर क्लिक करके प्रत्येक नमूना फ़ाइल रिकॉर्ड करें (प्रत्येक नमूने के लिए कम से कम 5,000 घटनाओं को रिकॉर्ड करें)।
- प्रत्येक दर्ज मनका नमूना के लिए, क्रमशः सकारात्मक और नकारात्मक चोटियों पर P2 और P3 फाटकों सेट ।
- मुआवजे के लिए सेल नमूने चलाएं (चरण 4.2 और 4.4.7 देखें, और धारा 5 और 6)। एफएससी और एसएससी वोल्टेज को संशोधित करें और कोशिकाओं की कल्पना करने के लिए दहलीज मूल्य, P1 गेट को समायोजित करें, और अंत में प्रत्येक नमूना फ़ाइल को रिकॉर्ड करें (कम से कम 10,000 घटनाओं को रिकॉर्ड करें)। क्रमशः सकारात्मक और नकारात्मक चोटियों पर P2 और P3 गेट्स सेट करें ।
नोट: Hoechst चैनल के मुआवजे के लिए, नकारात्मक चोटी (P3) और जी 2 /M के रूप में सकारात्मक एक(P2)के रूप में जी0/जी1 का उपयोग करें । - कार्यक्षेत्र रिबन अनुभाग में प्रयोग पर क्लिक करें और मुआवजा सेटअप अनुभाग में, मुआवजे की गणना काचयन करें ।
- निर्मित क्षतिपूर्ति सेटिंग, लिंक का नाम दें, और इसे वर्तमान प्रयोग में सहेजें।
- प्रायोगिक नमूना अधिग्रहण
- ब्राउज़र टूलबार पर नए नमूने पर क्लिक करके एक नमूना खोलें, और ग्लोबल वर्कशीटमें गेटिंग रणनीति बनाएं।
नोट: नमूना अधिग्रहण की गेटिंग रणनीति नमूना विश्लेषण के समान है, जिसे चित्र 3 और धारा 9 में वर्णित किया गया है। - एक्स-एक्सिस पर सीडी 3-ए के साथ एक हिस्टोग्राम में सभी इवेंट पॉपुलेशन प्रदर्शित करें। केवल सीडी 3+ कोशिकाओं का चयन करने के लिए एक अंतराल गेट बनाएं।
- अधिग्रहण डैशबोर्डपर, एलएन नमूनों के लिए सभी घटनाओं के रूप में भंडारण गेट का चयन करें, और या तो सभी घटनाओं या सीडी 3+ कोशिकाओं तिल्ली नमूनों के लिए ।
- कम गति से प्रयोगात्मक नमूनों को चलाने के लिए, और अंत में टीका लगाया चूहों से प्रत्येक नमूने के लिए कम से कम 100-200 एंटीजन विशिष्ट सीडी 8 टी कोशिकाओं को इकट्ठा करने के लिए सुनिश्चित करने के लिए सभी फ़ाइलों को रिकॉर्ड।
नोट: प्रयोगात्मक नमूनों की फाइल आकार आमतौर पर बड़ा (30-120 एमबी) होता है, खासकर जब एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं की आवृत्ति कम होती है। इसलिए, घटनाओं की उच्च संख्या (> 1 × 106)को कम से कम 100-200 एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं को रिकॉर्ड करने के लिए एकत्र किया जाना चाहिए। बड़ी फाइलें बाद की डेटा विश्लेषण प्रक्रिया को धीमा कर सकती हैं। तिल्ली के नमूनों में केवल CD3+ कोशिकाओं का अधिग्रहण (ऊपर चरण 8.2.2 देखें) फ़ाइल आकार को छोटा रखने के लिए उपयोगी है। - चलाएं और कक्ष चक्र विश्लेषण के लिए सकारात्मक नियंत्रण रिकॉर्ड करें, यानी अनुपचारित चूहों से बीएम नमूना।
- ब्राउज़र टूलबार पर नए नमूने पर क्लिक करके एक नमूना खोलें, और ग्लोबल वर्कशीटमें गेटिंग रणनीति बनाएं।
9. डेटा विश्लेषण
- सॉफ्टवेयर खोलें (सामग्रियों की तालिकादेखें), और कार्यक्षेत्र रिबन अनुभाग में क्रिएट ग्रुप पर क्लिक करके विश्लेषण किए जाने वाले विभिन्न अंगों के अनुरूप विभिन्न समूह बनाएं (यानी, समूह "ए-एलएन" बनाएं; "बी-तिल्ली"; "सी-बीएम") ।
नोट: नव निर्मित समूह समूह सूची में दिखाई देंगे, जबकि "मुआवजा" समूह स्वचालित रूप से सॉफ्टवेयर द्वारा उत्पन्न होता है। - समूह के नाम पर डबल क्लिक करके संशोधित समूह विंडो खोलें, और जांच करें कि नवनिर्मित समूह सिंक्रोनाइज्ड हैं। यदि नहीं, तो फ़ंक्शन सिंक्रोनाइज्डपर एक चेकमार्क डालें।
- अपने संबंधित समूह में प्रत्येक .fcs फ़ाइल खींचें।
- "ए-एलएनएस" समूह से शुरू होने वाली गेटिंग रणनीति बनाएं।
- ग्राफ विंडो खोलने के लिए समूह में पूरी तरह से दाग वाले नमूने पर डबल क्लिक करें; एक्स-एंड वाई-एक्सिस को एफसीएस फाइल्स (फ्लो साइटोमीटर सेटिंग्स, टेबल 2देखें) के रूप में लेबल किया गया है।
- इस नमूने के लिए प्राप्त कुल घटनाओं को एक्स-एक्सिस पर डीएनए-ए और वाई-एक्सिस पर डीएनए-डब्ल्यू के साथ एक डॉट प्लॉट में प्रदर्शित करें।
- ग्राफ विंडो के गेटिंग टूल सेक्शन में आयत पर क्लिक करके केवल एकल सेल आबादी का चयन करें।
नोट: एकल कोशिकाओं में डीएनए-ए मान इस प्रकार होता है: 2N (कम): 2N और 4N (मध्यवर्ती) के बीच, या 4N (उच्च) के बराबर है, जबकि डीएनए-डब्ल्यू मूल्य उन सभी के लिए समान हैं (चित्र 3के चरण 1)। - आयताकार गेट के केंद्र में डबल क्लिक करें वाई-एक्सिस पर एक्स-एक्सिस और डेड सेल डाई पर एफएससी-ए पैरामीटर के साथ डॉट प्लॉट में सिंगल सेल प्रदर्शित करें ।
- ग्राफ विंडो के गेटिंग टूल सेक्शन में पॉलीगॉन पर क्लिक करके केवल लाइव सेल पॉपुलेशन चुनें। मृत कोशिका डाई (चित्र 3के चरण 2) के लिए लाइव कोशिकाएं नकारात्मक हैं।
- पॉलीगोनल गेट के केंद्र में डबल क्लिक करें वाई-एक्सिस पर एक्स-एक्सिस और एसएससी-ए पैरामीटर पर एफएससी-ए पैरामीटर के साथ एक डॉट प्लॉट में कोशिकाओं को प्रदर्शित करने के लिए।
- आयतपर क्लिक करें, और उस ग्राफ12 (चित्र 3के चरण 3) में सभी एकल जीवित कोशिकाओं को शामिल करने के लिए एक "आराम" गेट बनाएं।
- वाई-एक्सिस पर एक्स-एक्सिस और सीडी 8 पर सीडी 3 के साथ एक डॉट प्लॉट में कोशिकाओं को प्रदर्शित करने के लिए "आराम" गेट के केंद्र में डबल क्लिक करें।
- पॉलीगॉन (चित्रा 3के चरण 4) पर क्लिक करके CD3+CD8+ कोशिकाओं का चयन करें ।
- सीडी 3+सीडी8+ गेट के केंद्र में डबल क्लिक करें एक्स-एक्सिस पर टेटर-गैग और वाई-एक्सिस पर पेंट-गैग के साथ एक डॉट प्लॉट में कोशिकाओं को प्रदर्शित करने के लिए।
- पॉलीगॉन (चित्र 3के चरण 5) पर क्लिक करके एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं (टेटर-गैग और पेंट-गैग दोनों के लिए सकारात्मक) का चयन करें।
- वाई-एक्सिस(चित्रा 4)पर एक्स-एक्सिस और Ki67 पर डीएनए-ए के साथ एक डॉट प्लॉट में कोशिकाओं को प्रदर्शित करने के लिए गैग-स्पेसिफिक गेट के केंद्र में डबल क्लिक करें।
- ग्राफ विंडो के गेटिंग टूल सेक्शन में क्वाड पर क्लिक करके विभिन्न सेल चक्र चरणों में कोशिकाओं का चयन करें।
नोट: जी0 चरण में सेल Ki67neg-डीएनए कम कोशिकाओं (नीचे छोड़ दिया चतुर्भुज) हैं; जी1 में कोशिकाएं Ki67pos-डीएनए कम (ऊपरी बाएं चतुर्भुज) हैं; एस-जी2/एममें कोशिकाएं Ki67pos-DNA मध्यवर्ती/उच्च (शीर्ष सही चतुर्भुज)(चित्रा 4)हैं । - समूह के सभी नमूनों के लिए फाटक लागू करने के लिए इसी समूह के लिए एक नमूने में बनाई गई गेटिंग रणनीति की प्रतिलिपि।
- "ए-एलएन समूह" के लिए चरण 9.5 से 9.18 तक दोहराएं।
- जांच करें कि सभी द्वार "बी-तिल्ली" समूह के प्रत्येक नमूने के लिए उपयुक्त हैं। बीएम कोशिकाओं (सकारात्मक नियंत्रण) के बीच सेल चक्र का विश्लेषण करने के लिए, वाई-एक्सिस पर एक्स-एक्सिस और Ki67 पर डीएनए-ए के साथ एक डॉट प्लॉट में कोशिकाओं को प्रदर्शित करने के लिए "आराम" गेट के केंद्र में क्लिक करें।
- जांच करें कि सभी गेट 3 समूहों के प्रत्येक नमूने के लिए उपयुक्त हैं (यानी, तिल्ली, एलएन और बीएम से कोशिकाओं के लिए)।
नोट: एकल सेल जनसंख्या गेट (चरण 9.7) और सेल चक्र के लिए क्वाड गेट (चरण 9.17) में विभिन्न नमूनों में अलग-अलग गेट निर्देशांक हो सकते हैं, मुख्य रूप से नमूनों (धारा 6.2) के बीच होचस्ट डाई तीव्रता के संभावित मामूली अंतर के कारण। इस कारण से, प्रत्येक नमूने में सेल चक्र के लिए एकल सेल जनसंख्या गेट और क्वाड गेट को संशोधित करना आवश्यक हो सकता है। यह इस प्रकार किया जाएगा: समूह के नाम पर डबल क्लिक करें, और समूह संपत्तियों से सिंक्रोनाइजेशन को हटा दें। यह ऑपरेशन समूह के अन्य सभी नमूनों में एक ही फाटक को संशोधित किए बिना एक नमूने में फाटकों के संशोधन की अनुमति देता है। सिंक्रोनाइजेशन हटाने के बाद, जहां आवश्यक हो, फाटकों को संशोधित करें। - इस विश्लेषण द्वारा प्राप्त परिणामों की कल्पना करने के लिए, इसे खोलने के लिए कार्यक्षेत्र रिबन अनुभाग में लेआउट एडिटर पर क्लिक करें। लेआउट एडिटर को सैंपल फलक में गेटिंग स्ट्रैटजी के हर गेट को खींचें और गेटिंग स्ट्रैटजी के सीक्वेंस के हिसाब से प्लॉट्स रखें । यदि आवश्यक हो, तो लेआउट में संबंधित भूखंड पर डबल क्लिक करके ग्राफ प्रकार बदलें और ग्राफ परिभाषा विंडो में उपयुक्त प्रकार का चयन करें।
- समूह पर क्लिक करें और प्रत्येक अंग में प्राप्त परिणामों की कल्पना करने के लिए लेआउट रिबन पर कार्यों द्वारा फिर से शुरू करें, और विभिन्न नमूनों की तुलना करें।
Representative Results
तिल्ली, एलएन, और बाल्ब/सी चूहों के बीएम से कोशिकाओं के कोशिका चक्र चरणों फ्लोरोसेंट डीएनए डाई, Hoechst, और एक विरोधी Ki67 mAb का उपयोग कर विश्लेषण किया गया, चित्रा 1में संक्षेप प्रोटोकॉल के अनुसार । इस धुंधला कोशिका चक्र के निम्नलिखित चरणों में कोशिकाओं के भेदभाव की अनुमति दी: जी0 (Ki67neg, डीएनए के 2N के साथ DNAlow के रूप में परिभाषित), जी1 (Ki67pos, DNAlow), और एस-जी2/एम(Ki67pos, एक डीएनए सामग्री के साथ 2N और 4N के बीच शामिल है, या DNAmediate के रूप में परिभाषित डीएनए के 4N के बराबर/
हमने पहले प्रकाशित परिणाम13, 14 को पुन: पेश करने के लिए बीएम कोशिकाओं का सेल चक्र विश्लेषण किया और फिर ब्याज की कोशिकाओं यानी सीडी 8 टी कोशिकाओं का विश्लेषण किया। चित्रा 2 बीएम कोशिकाओं(चित्रा 2 ए)के सेल चक्र विश्लेषण का एक विशिष्ट उदाहरण दिखाता है। प्रोटोकॉल में जी0/जी1 और जी 2/एम डीएनए चोटियों के भिन्नता (सीवी) का कम गुणांक निकला, जो डीएनए धुंधला(चित्रा 2B)की उत्कृष्ट गुणवत्ता का संकेत है, जो सीवी < २.५ के साथ एक उदाहरण दिखा रहा है; सीवी हमेशा सभी प्रयोगों में 5 < था) ।
हम तो टीका लगाया चूहों से एंटीजन विशिष्ट सीडी 8 टी कोशिकाओं के लिए एक ही प्रोटोकॉल लागू किया । BALB/c चूहों को बढ़ाने के लिए भड़काना और एमवीए-गैग के लिए Chad3-gag का उपयोग करके एचआईवी-1 के एंटीजन गैग के खिलाफ टीका लगाया गया था, दोनों एचआईवी-1 झूठ ले जाने के लिए इंजीनियर । दिन (घ) 3 के बाद बढ़ावा देने पर, हम तिल्ली से चुप विशिष्ट सीडी 8 टी कोशिकाओं की आवृत्ति का विश्लेषण किया और LNs draining । हम प्रतिरक्षा प्रतिक्रिया के प्रारंभिक चरण में टी कोशिकाओं के लिए नए परिभाषित गेटिंग रणनीति का लाभ उठाया है, जो पारंपरिक रणनीति के विपरीत, अत्यधिक सक्रिय एंटीजन प्रतिक्रिया CD8 टी कोशिकाओं12का पता लगाने के लिए उपयुक्त है । हमने उपन्यास की रणनीति को बाद के पांच चरणों में निष्पादित किया। चरण 1 में, हमने डीएनए-ए/डब्ल्यू गेट द्वारा डबल्स या समुच्चय को बाहर रखा, और चरण 2 में, हमने मृत सेल मार्कर अपवर्जन द्वारा लाइव कोशिकाओं की पहचान की। स्टेप 3 में, हमने विहित संकीर्ण लिम्फोसाइट गेट12के बजाय एक गैर-पारंपरिक "आराम" एफएससी-ए/एसएससी-ए गेट(चित्रा 3 ए)का उपयोग करके ब्याज की आबादी की पहचान की । CD3+CD8+ कोशिकाओं (चित्रा 3Aके चरण 4) पर गेटिंग के बाद, हमने दो अलग-अलग MHC मल्टीमर्स, यानी पेंट-गैग और टेटर-गैग (चित्र 3 एके चरण 5) का उपयोग करके गैग-विशिष्ट सीडी 8 टी कोशिकाओं की पहचान की। हमने टीका लगाए गए चूहों में गैग-स्पेसिफिक सीडी8 टी सेल डिटेक्शन की संवेदनशीलता में सुधार करने के लिए एक के बजाय दो मल्टीमर्स का इस्तेमाल किया, अनुपचारित चूहों में धुंधला पृष्ठभूमि को बढ़ाए बिना(चित्रा 3B और सी,चरण 5)। इस प्रकार, हमने टीका लगाए गए चूहों (क्रमशः 0.46% और 0.29% एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं, क्रमशः एलएन और प्लीहा) से अनुपचारित चूहों (0.00% एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं, क्रमशः, चित्रा 3 बी और सी)से प्रतिष्ठित किया।
विशेष रूप से, प्रोटोकॉल ने हमें एलएनएस के एंटीजन-विशिष्ट सीडी 8 टी सेल गेट और अनुपचारित चूहों की तिल्ली (आमतौर पर 0.00% और अधिकतम 0.02% पर) में बेहद कम पृष्ठभूमि रखने की अनुमति दी। गैग-स्पेसिफिक और नॉट गैग-स्पेसिफिक एफएससी-ए/एसएससी-ए प्लॉट्स की तुलना से पता चला कि गैग-स्पेसेज में उच्च एसएससी-ए और एफएससी-ए(चित्रा 3डी)थे, जो इन कोशिकाओं को पकड़ने के लिए "आराम" एफएससी-ए/एसएससी-ए गेट का उपयोग करने की आवश्यकता की पुष्टि करते हैं । इसके बाद हमने विभिन्न सेल चक्र चरणों(चित्रा 4A)में गैग-विशिष्ट सीडी 8 टी कोशिकाओं के प्रतिशत का मूल्यांकन किया। हमने पाया है कि प्लीहा में चुप विशिष्ट सीडी 8 टी कोशिकाओं और भी अधिक निकासी एलएन में एस-जी2/एमचरणों में कोशिकाओं का एक उच्च अनुपात दिन में 3 के बाद बढ़ावा (१८.६०% और ३३.५२%, क्रमशः) निहित ।
इसके अलावा, हमने पाया कि एस-जी2/एमचरणों में गैग-विशिष्ट सीडी 8 टी कोशिकाओं में उच्च एफएससी-ए और एसएससी-ए था, जब एक ही अंग(चित्रा 4B)से कुल सीडी 8 टी कोशिकाओं पर मढ़ा गया था । गैग-विशिष्ट सीडी 8 टी कोशिकाओं द्वारा CD62L अभिव्यक्ति कम थी, जैसा कि सक्रिय टी कोशिकाओं के लिए उम्मीद थी, एलएन(चित्रा 4 सी)में जी0 में कुछ कोशिकाओं को छोड़कर। कुल मिलाकर, इन परिणामों ने पुष्टि की कि "आराम" गेट (चित्रा 3 ए, बी और सीका चरण 3) को सभी प्रोलाइफिंग एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओंको शामिलकरने की आवश्यकता थी। प्रोटोकॉल विश्लेषण के समय एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं के सेल चक्र चरणों के "स्नैपशॉट" मूल्यांकन और विभिन्न कोशिका चक्र चरणों में कोशिकाओं द्वारा सीडी 62एल अभिव्यक्ति के लिए अत्यंत मूल्यवान था।

चित्रा 1: एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं के सेल चक्र विश्लेषण के लिए प्रोटोकॉल की योजना। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

चित्रा 2:बीएम कोशिकाओं का सेल चक्र विश्लेषण। अनुपचारित बाल्ब/सी चूहों से बीएम कोशिकाओं को दाग और प्रवाह साइटोमेट्री द्वारा विश्लेषण किया गया । (क) गेटिंग रणनीति का उदाहरण । हम डीएनए में एकल कोशिकाओं पर gated-एक/W साजिश (बाएं) और बाद में मृत सेल डाई बहिष्कार (मध्य) द्वारा जीवित कोशिकाओं पर । फिर, एक "आराम" FSC-A/SSC-एक गेट सभी बीएम कोशिकाओं (दाएं) के लिए इस्तेमाल किया गया था । (ख) बीएम कोशिकाओं के कोशिका चक्र विश्लेषण का उदाहरण (बाएं) । हमने कोशिका चक्र के निम्नलिखित चरणों में कोशिकाओं की पहचान करने के लिए Ki67 और डीएनए धुंधला के संयोजन का उपयोग किया: जी0 (नीचे बाएं चतुर्भुज, Ki67neg-DNAlow कोशिकाएं), जी1 (ऊपर बाएं चतुर्भुज, Ki67pos-DNAlow), एस-जी2/M (शीर्ष सही चतुर्भुज, Ki67pos-DNAintermediate/high) । फ्लोरेसेंस माइनस वन (एफएमओ) Ki67 mAb (मध्य) और डीएनए हिस्टोग्राम (दाएं) का नियंत्रण दिखाया गया है। डीएनए हिस्टोग्राम प्लॉट में, बाएं और दाएं द्वार क्रमशः जी0/जी 1और जी2/एमडीएनए पीक के अनुरूप हैं, और संख्या प्रत्येक चोटी के भिन्नता (सीवी) के गुणांक का प्रतिनिधित्व करती है । अन्य सभी भूखंडों में, संख्या संकेतित द्वारों में सेल प्रतिशत का प्रतिनिधित्व करती है। आंकड़ा 5 में से 1 प्रतिनिधि प्रयोग दिखाता है । प्रत्येक प्रयोग में, हमने 3 चूहों से पूल्ड बीएम कोशिकाओं का विश्लेषण किया। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

चित्र 3:एलएन और तिल्ली से एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं का विश्लेषण। Balb/c चूहों को Chad3-gag के साथ इंट्रामस्क्युलर (i.m.) को प्राइम किया गया था और एमवीए-गैग के साथ i.m बढ़ाया गया था। दिन में 3 के बाद बढ़ावा, टीका लगाया और अनुपचारित नियंत्रण चूहों से एलएन और तिल्ली कोशिकाओं draining दाग और प्रवाह साइटोमेट्री द्वारा विश्लेषण किया गया । (क) एकल कोशिकाओं (चरण 1) की पहचान करने के लिए पांच चरणों में गेटिंग रणनीति की योजना; लाइव कोशिकाएं (चरण 2); लिम्फोसाइट्स (चरण 3); सीडी 8 टी कोशिकाएं (चरण 4); और झूठ-विशिष्ट कोशिकाएं (चरण 5)। (B-C) भूखंडों का उदाहरण: (बी) एलएन और (सी) अनुपचारित (ऊपर) और टीका लगाया (नीचे) चूहों की तिल्ली से कोशिकाओं का विश्लेषण । हमने चरण 1 में डीएनए-ए/डब्ल्यू प्लॉट पर एकल कोशिकाओं की पहचान की । फिर, चरण 2 में, हमने मृत सेल डाई अपवर्जन द्वारा लाइव कोशिकाओं का चयन किया। चरण 3 में, हमने लिम्फोसाइट्स के लिए एक गैर-विहित "आराम" गेट का उपयोग किया। चरण 4 में, हमने सीडी 3 और सीडी 8 की दोहरी अभिव्यक्ति से सीडी 8 टी कोशिकाओं की पहचान की। हम तो चुप विशिष्ट कोशिकाओं की पहचान की और नहीं चुप-5 में विशिष्ट, उनकी क्षमता के आधार पर फ्लोरोक्रोम-लेबल एच-2kd-चुप-Pentamer (पेंट-गैग) और एच-2kd-चुप-Tetramer (Tetr-gag), या नहीं, क्रमशः बांध । (घ) एफएससी-ए/एसएससी-गैग-स्पेसिफिक (ब्लू) का प्रोफाइल और ऊपर वर्णित गेटिंग के बाद गैग-स्पेसिफिक (ग्रे) कोशिकाएं नहीं । संख्या इंगित द्वारों में सेल प्रतिशत का प्रतिनिधित्व करते हैं। आंकड़ा 5 में से 1 प्रतिनिधि प्रयोग दिखाता है । प्रत्येक प्रयोग में, हमने 3 टीका लगाए गए चूहों और 3 अनुपचारित चूहों से पूल तिल्ली और पूल्ड एलएन कोशिकाओं का विश्लेषण किया। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

चित्रा 4:एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं का सेल चक्र विश्लेषण। चूहों को चित्र 3 के रूप में टीका लगाया गया था और गैग-विशिष्ट कोशिकाओं का सेल चक्र विश्लेषण 3 के बाद बढ़ावा देने के दिन में किया गया था, चित्रा 3के रूप में 5 चरणों में गेटिंग के बाद । (क)एलएन (ऊपर) से गैग-विशिष्ट सीडी8 टी कोशिकाओं के कोशिका चक्र विश्लेषण और टीका लगाए गए चूहों की तिल्ली (नीचे) का उदाहरण । सेल चक्र चरणों की पहचान चित्रा 2बीके रूप में की गई थी । पैनल जी0में कोशिकाओं का प्रतिनिधित्व करते हैं, जी1में, और एस-जी 2 /एम(बाएं)और फ्लोरेसेंस माइनस वन (एफएमओ) कि67 एमएबी (दाएं) के नियंत्रण में। संख्या इंगित द्वारों में सेल प्रतिशत का प्रतिनिधित्व करते हैं। (ख) एफएससी-ए/एसएससी-ए डॉट भूखंड एस-जी2/एमचरणों में गैग-विशिष्ट सीडी8 टी कोशिकाओं को दिखाते हुए (लाल रंग में) कुल सीडी 3+सीडी 8+ टी कोशिकाओं (ग्रे में) से एलएनएस (ऊपर) और टीका लगाए गए चूहों की तिल्ली (नीचे) पर मढ़ा । (ग) जी0 (हरे) में गैग-विशिष्ट सीडी8 टी कोशिकाओं द्वारा सीडी 62एल अभिव्यक्ति दिखाने वाले हिस्टोग्राम को ऑफसेट करें, जी1 (नीला) में, और एस-जी2/एम (लाल) में एलएनएस (ऊपर) और टीका लगाए गए चूहों की तिल्ली (नीचे) से । वाई-कुल्हाड़ियों से घटनाओं की सामान्य संख्या का संकेत मिलता है। यह आंकड़ा कुल 15 चूहों के साथ 5 स्वतंत्र प्रयोगों में से 1 प्रतिनिधि उदाहरण दिखाता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
अनुपूरक सामग्री: फ्लो साइटोमीटर सेटिंग्स। इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
Discussion
हालांकि टी सेल क्लोनल विस्तार का गहनता से अध्ययन किया गया है, कुछ पहलू अज्ञात रहते हैं, ज्यादातर क्योंकि इसकी जांच करने के लिए उपलब्ध उपकरण कुछ हैं और उनकी अपनी कमियां हैं। इस नजरिए से, हम एक माउस मॉडल में टीकाकरण के बाद शुरुआती समय में एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं के कोशिका चक्र का विश्लेषण करने के लिए एक अत्यधिक संवेदनशील प्रवाह साइटोमेट्रिक विधि स्थापित करते हैं। प्रोटोकॉल Ki67 और डीएनए धुंधला के संयोजन पर आधारित है, जिसका उपयोग पहले चूहों13, 14में बीएम हेमेटोपोइटिक कोशिकाओं के कोशिका चक्र का विश्लेषण करने के लिए कियागयाथा। एंटीजन-विशिष्ट सीडी 8 टी कोशिकाओं के लिए प्रोटोकॉल को अनुकूलित करने के लिए, हमें डीएनए डाई का चुनाव, विभिन्न नमूनों में तुलनीय डीएनए धुंधला प्राप्त करने के लिए उपयुक्त शर्तों और डेटा विश्लेषण के लिए गेटिंग रणनीति सहित कुछ महत्वपूर्ण मुद्दों पर विचार करना पड़ा।
डीएनए स्टेनिंग के लिए कई रंग उपलब्ध हैं, जिनमें प्रोपिडियम आयोडाइड और 7-अमीनोएक्टिनोमाइसिन डी शामिल हैं; हमने Hoechst को चुना क्योंकि यह झिल्ली धुंधला और Ki67 धुंधला के लिए आवश्यक हल्के निर्धारण/ इसके साथ ही, Hoechst के साथ धुंधला हमें उत्कृष्ट गुणवत्ता का एक डीएनए हिस्टोग्राम प्राप्त करने की अनुमति दी, यानी, जी0/जी1 और जी2/एमडीएनए चोटियों में आमतौर पर अन्य डीएनए रंगों के साथ प्राप्त डीएनए चोटियों की तुलना में भिन्नता (सीवी) का बहुत कम गुणांक था, उदाहरण के लिए, DRAQ519। दरअसल, Hoechst20जीवित कोशिकाओं में भी डीएनए दाग कर सकते हैं ।
एक ही प्रयोग के विभिन्न नमूनों में Hoechst तीव्रता में उतार-चढ़ाव से बचने के लिए कुछ रणनीतियों का उपयोग किया गया था। समय के दौरान डाई तीव्रता की गिरावट को कम करने के लिए प्रवाह साइटोमीटर पर नमूना अधिग्रहण से ठीक पहले Hoechst धुंधला प्रदर्शन किया गया था। कई नमूनों के साथ बड़े प्रयोगों में प्रोटोकॉल को पुन: उत्पन्न करने में रुचि रखने वालों के लिए, हम एक समय में कुछ नमूनों पर Hoechst धुंधला प्रदर्शन करने की सलाह देते हैं। एक अन्य दोष यह है कि डाई के साथ इनक्यूबेशन के दौरान होचस्ट तीव्रता को सेल नंबर से भारी प्रभावित किया जा सकता है। इस कारण से, हम दृढ़ता से हमेशा कोशिकाओं की एक ही संख्या और डीएनए धुंधला के लिए प्रति नमूना एक ही मात्रा का उपयोग करने की सलाह देते हैं । यदि प्रवाह साइटोमीटर पर अधिग्रहण के लिए कोशिकाओं की एक उच्च संख्या की आवश्यकता होती है, तो हम दो या अधिक समान नमूने तैयार करने की सलाह देते हैं और फिर होचस्ट स्टेनिंग चरण से ठीक पहले उन्हें विलय करते हैं।
प्रोटोकॉल का एक प्रमुख बिंदु डेटा विश्लेषण के लिए गेटिंग रणनीति है। हमने हाल ही में प्रतिरक्षा प्रतिक्रिया के शुरुआती समय में टी सेल विश्लेषण के लिए एक उपन्यास रणनीति प्रकाशित की है, जिसने हमें एंटीजन-विशिष्ट टी कोशिकाओं12का पता लगाने की संवेदनशीलता को बढ़ाने की अनुमति दी। हमने इस रणनीति को इस प्रकार दिखाए गए डेटा पर लागू किया है। सबसे पहले, हम डीएनए-ए/डब्ल्यू साजिश में सेल समुच्चय को बाहर रखा । दूसरा, मृत कोशिकाओं को बाहर निकालने के बाद, हमने एफएससी/एसएससी प्लॉट ("आराम से गेट") में काफी बड़े लिम्फोसाइट गेट का इस्तेमाल किया। इस रणनीति के द्वारा, हम एस-जी 2 /एम में अत्यधिक सक्रिय एंटीजन-विशिष्टसीडी8टी कोशिकाओं को शामिल करने में सक्षम थे जो आमतौर पर वर्तमान गेटिंग रणनीतियों से चूक जाते हैं, क्योंकि इन कोशिकाओं में उच्च एफएससी-ए और एसएससी-ए होते हैं। संक्षेप में, डेटा विश्लेषण विधि के एक महत्वपूर्ण भाग का प्रतिनिधित्व करता है, जो सक्रिय/एंटीजन-विशिष्ट टी कोशिकाओं का एक संवेदनशील पता लगाने को प्राप्त करने के लिए आवश्यक है ।
विधि प्रतिरक्षा प्रतिक्रिया के प्रारंभिक चरणों में महत्वपूर्ण टी सेल डेटा लापता होने की संभावना को रोकता है और टी सेल इम्यूनो-मॉनिटरिंग के लिए नए दृष्टिकोण खोलता है। भविष्य में सुधार के लिए फॉस्फो-हिस्टोन 3 के लिए धुंधला शामिल हो सकता है जो जी2 और एम21के बीच भेदभाव की अनुमति देगा। एक वर्तमान सीमा यह है कि कोशिकाओं को तय किया जाना है और परमाणु मार्कर, Ki67 के लिए दाग के लिए व्याप्त है । इस प्रकार, कोशिकाओं का उपयोग अन्य प्रकार के विश्लेषण जैसे छंटाई और बाद के कार्यात्मक विश्लेषण के लिए नहीं किया जा सकता है। इसके अलावा, Hoechst सहित डीएनए रंग, आमतौर पर जीनोमिक डीएनए विश्लेषण के साथ हस्तक्षेप और मूल्यांकन के इस प्रकार के लिए उपयुक्त नहीं हैं । झिल्ली मार्कर की पहचान जो विभिन्न कोशिका चक्र चरणों से सहसंबंधित होती है और जो जीवित कोशिकाओं पर दाग लगाया जा सकता है, इस सीमा को दूर कर सकता है। अंत में, इस विधि में टीकाकरण, संक्रमण, इम्यूनो-मध्यस्थता रोगों और इम्यूनो-थेरेपी जैसे कई संदर्भों में सक्रिय/प्रसार ति कोशिकाओं के मूल्यांकन की अपार संभावनाएं हैं ।
Disclosures
ए फोल्गोरी और एस कपोन Reithera Srl के कर्मचारी हैं । ए निकोसिया पेटेंट आवेदन WO 2005071093 (A3) "चिंपांजी adenovirus वैक्सीन वाहक पर आविष्कारक नामित किया गया है." अन्य लेखकों के पास खुलासा करने के लिए कुछ नहीं है ।
Acknowledgments
इस काम को रीटहेरा द्वारा समर्थित किया गया था, एमआईयूआर परियोजना 2017K55HLC_006 द्वारा, और 5 × 1000 अनुदान से एसोसियाज़िओन इटालियना राइसर्का सुल कैन्रो (एआईआरसी)। निम्नलिखित टेट्रामर को एनआईएच टेट्रामर सुविधा के माध्यम से प्राप्त किया गया था: एपीसी-संयुग्मित एच-2के (डी) एचआईवी गैग 197-205 AMQMLKETI।
Materials
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
References
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- Mouse phenotype. , Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020).
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Tags
इम्यूनोलॉजी और संक्रमण अंक 167 एंटीजन-विशिष्ट सीडी 8 टी कोशिकाएं सेल चक्र Ki67 डीएनए डाई प्रवाह साइटोमेट्री तिल्ली लिम्फ नोड्स माउसErratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021.
Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
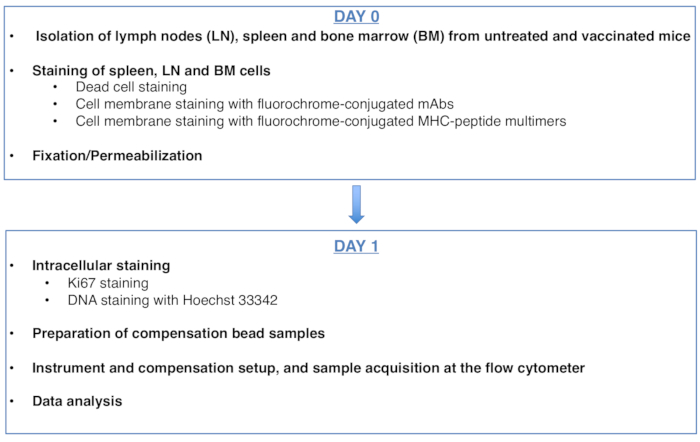
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:
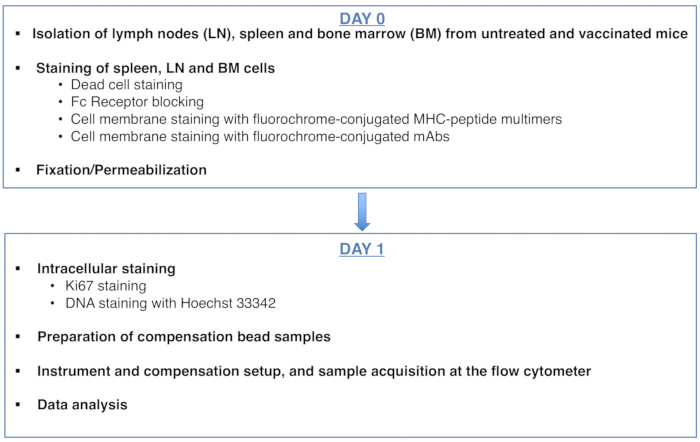
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.



