Schnelle Fraktionierung und Isolierung von Vollblutkomponenten in Proben aus einer Gemeinschaft basierende Einstellung Erhalten
Summary
We outline a methodology for the processing of whole blood to obtain a variety of components for further analysis. We have optimized a streamlined protocol that enables rapid, high-throughput simultaneous processing of whole blood samples in a non-clinical setting.
Abstract
Erhebung und Verarbeitung von Vollblut-Proben in einem nicht-klinischen Umfeld bietet eine einzigartige Gelegenheit, selbstständig lebenden Personen mit und ohne Vorerkrankungen zu bewerten. Schnelle Verarbeitung dieser Proben ist wichtig, um eine Verschlechterung der zellulären Schlüsselkomponenten zu vermeiden. Dazu zählen Verfahren zur gleichzeitigen Pbmc (PBMC), DNA, RNA und Serum-Isolierung aus einer einzigen Blutprobe in den Häusern der zustimmenden Teilnehmer in einem Ballungsgebiet durchgeführt, mit der Verarbeitung innerhalb von 2 h Sammlung initiiert. Wir haben diese Techniken verwendet werden, um mehr als 1.600 Blutproben ergeben konsistente, hochwertige Materialien, die später in erfolgreiche DNA-Methylierung, Genotypisierung, Genexpression verwendet worden ist und Strömungsanalysen Zytometrie verarbeiten. Einige der verwendeten Methoden sind Standard; Wenn sie jedoch in der beschriebenen Weise kombiniert werden, eine effiziente Verarbeitung von Proben von Teilnehmern populations und / oder gemeinschafts ermöglichen siebasierte Studien, die normalerweise nicht in einer klinischen Umgebung ausgewertet würde. Daher hat dieses Protokoll die Möglichkeit, Proben (und anschließend Daten), die mehr repräsentativ für die allgemeine Bevölkerung zu erhalten.
Introduction
Mehrere Studien haben Unterschiede in der Genexpression, DNA-Methylierung und Zell-Untergruppe im Blut bei Menschen mit und ohne geistige (oder anderen) Erkrankungen 1-4 gekennzeichnet. Diese Studien haben jedoch in klinischen Settings, in denen krankheitsassoziierten Unterschiede können aufgrund des allgemein schwerer Natur der Krankheiten, für die Patienten, die Behandlung vergrößert werden erhalten. Aufgrund der Fortschritte in der "Omics" Ansätze hat die letzten zehn Jahre eine Explosion von Interesse an biologischen Proben aus Community und / oder epidemiologische Einstellungen 5-7, um populationsbasierte Schätzungen der Prävalenz der Krankheit geben und ein umfassenderes Bild des Gesehenen Umweltfaktoren dieser geistigen und / oder körperlichen Krankheiten.
Eine der größten Herausforderungen in dieser Hinsicht ist die Voraussetzung für eine schnelle Verarbeitung der gesammelten Proben. Abbau von mononuklearen Zellen, Schlüssel Immunsystem Komponenten, die frequentl sindy verwendet werden, um beurteilen die Gesundheit einer Person, beginnt unmittelbar nach Blutentnahme mit einer signifikanten Abnahme der Wiederaufnahme nach 2 Stunden der Sammlung 8-10. Um dieser Herausforderung zu begegnen, stellen wir Ihnen ein optimiertes Protokoll, bei dem mehrere Komponenten von menschlichem Vollblut gleichzeitig von Proben in den Häusern von Themen, die in einem großen Stadtgebiet erhalten wurde, isoliert. Das Protokoll ist nach unserer Zusammenstellung und Modifikation der aktuellen Techniken, einschließlich der Speicherung aller "extra" Fraktionen bei Zukunftstechniken ermöglichen eine weitere Isolierung der Basis / Analysen. Alternative Verfahren oder Kits können anstelle der hier beschriebenen einzelnen Methoden diejenigen umrissen eingesetzt werden haben sich als zuverlässige und effiziente Einrichtung zum Verarbeiten von Proben in einem Hochdurchsatz-Art sein. Hochwertige Fraktionen (PBMCs, DNA, Serum und RNA) von frischem Blut kann innerhalb von 2 Stunden der Sammlung und alle Test-ready Proben produzierten verfügbar innerhalb von 2 Tagen (Abbildung 1 sein).
Dieses Protokoll wurde entwickelt, um die effiziente Verarbeitung von Proben aus Community-Wohnung, erwachsenen Einwohner der Stadt von Detroit für die Prüfung in der Detroit Neighborhood Health Study gesammelt aktivieren (DNHS; DA022720, RC1MH088283, DA022720-05-S1), eine bevölkerungsbezogene Studie über die sozialen und biologischen Determinanten der posttraumatischen Belastungsstörung (PTSD) und anderen psychischen Erkrankungen. Die Prävalenz von PTSD in Detroit mehr als zweimal der nationale Durchschnitt 11,12. Identifizierung von biologischen Determinanten von PTBS in dieser Population kann helfen, geeignete pharmakologische und / oder kognitiv-verhaltenstherapeutische Maßnahmen, um diejenigen, die an der Krankheit zu unterstützen entwickeln, sowohl in diesem städtischen Bevölkerung und in anderen Hochrisikogruppen (zB der Rückkehr Kriegsveteranen). Unser Labor, die zuvor an der Wayne State University in Detroit, Michigan, wurde für die Verarbeitung basierend auf unserem Know-how im Umgang mit frischen Gewebeproben von einer Vielzahl abgeleitet gewähltvon Quellen, um die Notwendigkeit, die Bearbeitung der Proben innerhalb von 2 Stunden der Sammlung zu beginnen, und unsere Nähe zu den Sammelstellen. Mit dieser einzigartigen Gelegenheit, bei der Hand, war es unser Ziel, um die Verarbeitung für höchste Ausbeute von DNA, RNA, Serum und peripheren mononukleären Blutzellen (PBMCs) von jeder Probe (insgesamt N = 1.639 Proben über 5 Wellen der Probenentnahme) zu optimieren. Die hier beschriebenen Verfahren können gleichzeitig in einer nicht-klinischen Umfeld durchgeführt werden, wodurch Ausgangsmaterial (siehe Tabelle 1 für die Durchschnittserträge) für eine Vielzahl von nachgelagerten Anwendungen, einschließlich Microarray, epigenetischen, Echtzeit-RT-PCR und Durchflusszytometrie analysiert.
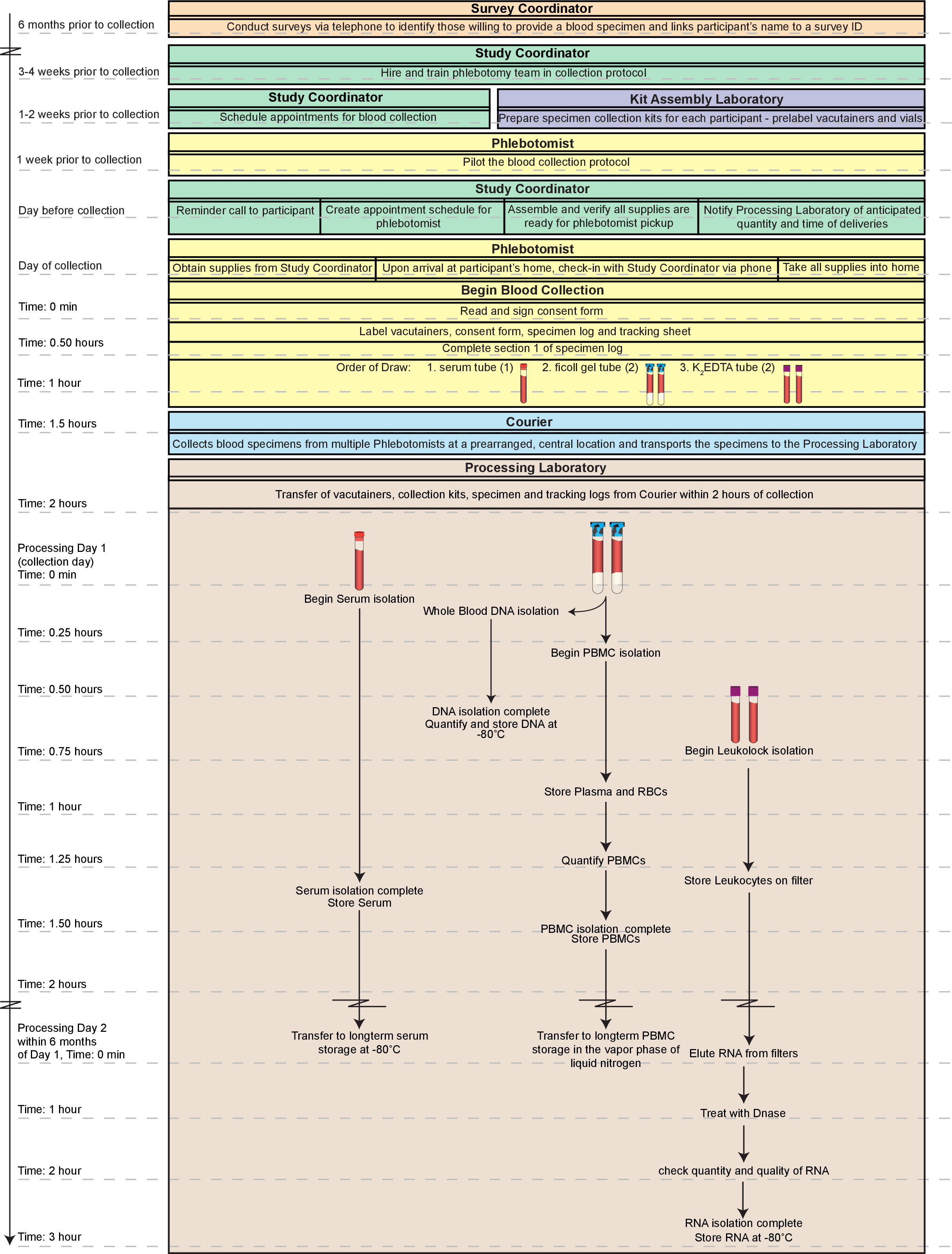
Abbildung 1. Gesamtarbeitsablauf. Die hier dargestellte Gesamtprozess umfasst die Logistik der Gewinnung der Blutproben von der Identifizierung zustimmenden particnehmer in die Blutabnahme selbst. Hochwertige, Fraktionen (periphere mononukleäre Blutzellen; PBMCs, DNA, Serum und RNA) von frischem Vollblut kann innerhalb von 2 Stunden der Sammlung produziert werden und alle Test-ready Proben können innerhalb von 2 Tagen zur Verfügung stehen. Darüber hinaus sind die durch dieses Verfahren hergestellten Fraktionen geeignet für Langzeitspeicherung, wenn die Proben nicht sofort getestet werden. Der gesamte Zeitachse hier skizzierten konnte an einem einzigen Tag (~ 5 Stunden insgesamt) abgeschlossen sein. Allerdings würde ein solcher Tag extrem arbeitsintensiv insbesondere für einen Monteur mit umfassender Erfahrung mit den Techniken. Daher empfehlen wir, Aufteilen der Verfahren an Tag 1 zwischen mindestens zwei Techniker und Abschluss der RNA-Prozessierung am Tag 2. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Protocol
Representative Results
Discussion
Wir haben eine schlanke Protokoll, das bereits erfolgreich angewendet wurde, um mehr als 1.600 Vollblut-Proben in der Detroit Neighborhood Health Study bearbeiten beschrieben. Obwohl viele dieser Verfahren sind in der vorhandenen Literatur zur Verfügung, unsere Schritt-für-Schritt-Zusammenstellung, einschließlich der genauen zeitlichen Änderungen zwischen jedem Schritt, spiegelt eine optimierte effizientes Protokoll erfolgreich produziert eine Vielzahl von biologischen Proben mit einer Vielzahl von Downstream-Anwend…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We would like to thank Henriette Mair-Meijers for invaluable attention to detail and hours devoted to processing the blood collections. We are grateful for the graphic design expertise of Natalie Jameson Kiesling. We also appreciate the approval of the manufacturers (Qiagen (Valencia, CA), BD Biosciences (San Jose, CA), Life Technologies (Grand Island, NY)) mentioned herein to publish the use of their products as described. Funding for this work was generously provided by the National Institutes of Health award numbers DA022720, RC1MH088283, and DA022720-05-S1.
Materials
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Day 1: DNA isolation |
| Phosphate-buffered saline (PBS) | Sigma | P5493-1L | Day 1: PBMC isolation |
| 5 ¾” Pasteur pipets | Fisher | 13-678-6A | Day 1: PBMC isolation |
| Fetal Bovine Serum (FBS), heat inactivated | Life Technologies | 10082147 | Day 1: PBMC isolation |
| Dimethyl Sulfoxide (DMSO) | Sigma | D8418-500ml | Day 1: PBMC isolation |
| RPMI Medium 1640, liquid | Invitrogen | 11875119 | Day 1: PBMC isolation |
| 0.4% trypan blue stain | Invitrogen | T10282 | Day 1: PBMC isolation |
| Countess Cell Counting Chamber | Invitrogen | C10283 | Day 1: PBMC isolation |
| Countess Automated Cell Counter or cell counting device such as a microscope and hemocytometer | Invitrogen | C10281 | Day 1: PBMC isolation |
| LeukoLOCK Fractionation & Stabilization Kit | Ambion | 1933 | Day 1: Leukocyte RNA isolation |
| 25G x 5/8 in. needles | Becton Dickinson | 305122 | Day 1: Leukocyte RNA isolation |
| Syringes (5ml) | Becton Dickinson | 309646 | Days 1 and 2: Leukocyte RNA isolation |
| Denaturing Lysis Solution | Ambion | 8540G | Day 2: Leukocyte RNA isolation |
| 5M NaCl | Life Technologies | 24740011 | Day 2: Leukocyte RNA isolation |
| TRI Reagent | Ambion | 9738 | Day 2: Leukocyte RNA isolation |
| Bromo-3-chloro-propane (BCP) | Sigma | B-9673 | Day 2: Leukocyte RNA isolation |
| spin cartridges | Ambion | 10051G | Day 2: Leukocyte RNA isolation |
| 0.1mM EDTA | Ambion | 9912 | Day 2: Leukocyte RNA isolation |
| DNA-free Kit | Ambion | AM1960 | Day 2: DNase treament |
| RNA 6000 Ladder | Agilent | 5067-1529 | Day 2: Bioanalyzer analysis |
| RNA 6000 Nano Series II Kit | Agilent | 5067-1511 | Day 2: Bioanalyzer analysis |
| RNaseZAP | Ambion | AM8782 | Day 2: Bioanalyzer analysis |
| Ethanol >99% | Sigma | E7023-500ml | |
| Isopropanol >99% | Sigma | I9516-500ml | |
| Nuclease-free ultra pure water | Invitrogen | 9938 | |
| Pipette tips (nuclease-free) | Eppendorf | 22491253 | |
| Pipetter (serological) | Eppendorf | 2223020-4 | |
| Pipetters (for volumes under 1ml) | Eppendorf | 3120000-054 | |
| Pipettes (serological) | Fisher | 13-678-27E | |
| Controlled rate freezing containers | Nalgene | 5100-0001 | |
| Cryoboxes (to hold 2ml and 5ml cryovials and 1.5ml microcentrifuge tubes) | Fisher | 03-395-464 | |
| Test tube rack | Thermo Scientific | 14-804-134 | |
| 15ml polypropylene tubes | Fisher | 14-959-49D | |
| 1.5ml and 0.65 microcentrifuge tubes | Fisher | 07-200-534 and 07-200-185 | |
| 2ml and 5ml cryovials | Fisher | 10-500-26 and 10-269-88F | |
| 8ml CPT vacutainer | BD Biosciences | 362761 | 2 tubes |
| 6ml K2 EDTA vacutainer | BD Biosciences | 367863 | 2 tubes |
| 8.5ml SST vacutainer | BD Biosciences | 367988 | 1 tube |
| Vortexer | Fisher | 2215365 | |
| Dry bath incubator with heating block for microcentrifuge tubes | Fisher | 11-715-1250 | |
| Filtration/vacuum system for use within the cell culture hood | Fisher | 01-257-87 | |
| Fixed-angle rotor for microcentrifuge tubes with aerosol-tight lid | Eppendorf | 22637002 | |
| Refrigerated centrifuge with a swing-bucket rotor and aerosol-tight caps for 16 x 125mm vacutainers and 15ml polypropylene tubes | Eppendorf | 22628157 | 2, one does not need to be refrigerated |
| Nanodrop 2000 (recommended for accuracy of small volumes) or other spectrophotometric device | Fisher | 13-400-411 | |
| Agilent Bioanalyzer | Agilent Technologies | G2940CA | |
| Liquid nitrogen tank | Thermo Scientific | 11-676-56 | |
| -80 ˚C freezer | Thermo Scientific | 992RAK | |
| Sharps container | Fisher | 22-037-970 | |
| Biological waste container | Thermo Scientific | 1223P52 | |
| Biosafety Level 2 certified cell culture hood | Thermo Scientific | 13-261-315 |
References
- Hernandez, M. E., Martinez-Fong, D., Perez-Tapia, M., Estrada-Garcia, I., Estrada-Parra, S., Pavon, L. Evaluation of the effect of selective serotonin-reuptake inhibitors on lymphocyte subsets in patients with a major depressive disorder. Eur Neuropsychopharmacol. 20, 88-95 (2010).
- Weigelt, K., et al. TREM-1 and DAP12 expression in monocytes of patients with severe psychiatric disorders EGR3, ATF3 and PU.1 as important transcription factors. Brain Behav Immun. 25, 1162-1169 (2011).
- Robertson, M. J., et al. Lymphocyte subset differences in patients with chronic fatigue syndrome, multiple sclerosis and major depression. Clin Exp Immunol. 141, 326-332 (2005).
- Rotter, A., Asemann, R., Decker, A., Kornhuber, J., Biermann, T. Orexin expression and promoter-methylation in peripheral blood of patients suffering from major depressive disorder. J Affect Disord. 131, 186-192 (2011).
- Klengel, T., et al. Allele-specific FKBP5 DNA demethylation mediates gene-childhood trauma interactions. Nat Neurosci. 16, 33-41 (2013).
- Segman, R. H., Shefi, N., Goltser-Dubner, T., Friedman, N., Kaminski, N., Shalev, A. Y. Peripheral blood mononuclear cell gene expression profiles identify emergent post-traumatic stress disorder among trauma survivors. Mol Psychiatry. 10, 500-513 (2005).
- Smith, B. H., et al. Cohort Profile: Generation Scotland: Scottish Family Health Study (GS:SFHS). The study, its participants and their potential for genetic research on health and illness. Int J Epidemiol. 42, 689-700 (2013).
- Mallone, R., et al. Isolation and preservation of peripheral blood mononuclear cells for analysis of islet antigen-reactive T cell responses: position statement of the T-Cell Workshop Committee of the Immunology of Diabetes Society. Clin Exp Immunol. 163, 33-49 (2011).
- Duvigneau, J. C., Hartl, R. T., Teinfalt, M., Gemeiner, M. Delay in processing porcine whole blood affects cytokine expression. J Immunol Methods. 272, 11-21 (2003).
- Debey, S., et al. Comparison of different isolation techniques prior gene expression profiling of blood derived cells: impact on physiological responses, on overall expression and the role of different cell types. Pharmacogenomics J. 4, 193-207 (2004).
- Uddin, M., et al. Epigenetic and immune function profiles associated with posttraumatic stress disorder. Proc Natl Acad Sci U S A. 107, 9470-9475 (2010).
- Kessler, R. C., Wang, P. S. The descriptive epidemiology of commonly occurring mental disorders in the United States. Annu Rev Public Health. 29, 115-129 (2008).
- . QIAamp® DNA Mini and Blood Mini Handbook Available from: https://www.qiagen.com/us/resources/resourcedetail?id=67893a91-946f-49b5-8033-394fa5d752ea (2010)
- Koenen, K. C., et al. SLC6A4 methylation modifies the effect of the number of traumatic events on risk for posttraumatic stress disorder. Depress Anxiety. 28, 639-647 (2011).
- Walsh, K., Uddin, M., Soliven, R., Wildman, D. E., Bradley, B. Associations between the SS variant of 5-HTTLPR and PTSD among adults with histories of childhood emotional abuse: Results from two African American independent samples. J Affect Disord. 161, 91-96 (2014).
- Bustamante, A. C., et al. Childhood maltreatment is associated with epigenetic differences in hypothalamic-pituitary-adrenal (HPA) axis genes in the Detroit Neighborhood Health Study. , (2013).
- Sipahi, L., et al. Longitudinal epigenetic variation of DNA methyltransferase genes is associated with vulnerability to post-traumatic stress disorder. Psychol Med. 44, 3165-3179 (2014).
- Uddin, M., Koenen, K. C., Aiello, A. E., Wildman, D., de los Santos, R., Galea, S. Epigenetic and inflammatory marker profiles associated with depression in a community-based epidemiologic sample. Psychol Med. 41, 997-1007 (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M. Biological signatures of post-traumatic stress disorder in the Detroit Neighborhood Health Study. , (2010).
- Aiello, A. E. Cytomegalovirus antibodies as a marker of immunosenescence in the Detroit Neighborhood Health Study. , (2010).
- Bustamante, A. C., et al. . Distinct gene expression profiles characterize lifetime PTSD and childhood maltreatment in the Detroit Neighborhood Health Study. , (2013).

