Frazionamento rapida e isolamento dei componenti sangue intero in campioni ottenuti da una impostazione basata su Comunità
Summary
We outline a methodology for the processing of whole blood to obtain a variety of components for further analysis. We have optimized a streamlined protocol that enables rapid, high-throughput simultaneous processing of whole blood samples in a non-clinical setting.
Abstract
Raccolta e lavorazione di campioni di sangue intero in un contesto non clinico offre un'opportunità unica per valutare residenti in comunità gli individui con e senza condizioni preesistenti. Elaborazione rapida di questi campioni è essenziale per evitare la degradazione dei componenti cellulari chiave. Vi sono incluse le modalità di simultanea periferico delle cellule mononucleate del sangue (PBMC), DNA, RNA e l'isolamento del siero da un unico prelievo di sangue effettuato nelle case dei partecipanti consentendo all'interno delle aree metropolitane, con l'elaborazione iniziata entro 2 ore di raccolta. Abbiamo usato queste tecniche per elaborare oltre 1.600 campioni di sangue cedevole, materiale costante qualità elevata, che è stato successivamente utilizzato in metilazione del DNA, la genotipizzazione, espressione genica successo e citometria a flusso analisi. Alcuni dei metodi utilizzati sono di serie; tuttavia, se combinati nel modo descritto, consentono un'efficiente elaborazione dei campioni di partecipanti di population- e / o community-studi basati che normalmente non verrebbero valutati in un ambiente clinico. Pertanto, questo protocollo ha il potenziale per ottenere campioni (e, successivamente, dati) che sono più rappresentativi della popolazione generale.
Introduction
Molteplici studi hanno caratterizzato le differenze di espressione genica, la metilazione del DNA e sottoinsieme di cellule nel sangue tra gli individui con e senza mentale (o altro) malattie 1-4. Questi studi, tuttavia, sono stati ottenuti da situazioni cliniche in cui le differenze malattie associate possono essere amplificate a causa della natura generalmente più grave delle malattie per le quali pazienti stanno cercando di trattamento. Grazie ai progressi in tecnologie "omiche", l'ultimo decennio ha visto un'esplosione di interesse ad ottenere campioni biologici provenienti da comunità e / o le impostazioni epidemiologici 5-7, al fine di fornire stime basate sulla popolazione di prevalenza della malattia e di un quadro più ampio della determinanti ambientali di queste malattie mentali e / o fisici.
Una sfida chiave in questo senso è il requisito per una rapida elaborazione dei campioni raccolti. Degradazione delle cellule mononucleate, componenti chiave del sistema immunitario che sono frequently utilizzato per valutare la salute di un individuo, inizia subito dopo prelievo di sangue con una diminuzione significativa recupero dopo 2 ore di raccolta 8-10. Per affrontare questa sfida, vi presentiamo un protocollo ottimizzato in cui più componenti del sangue umano intero sono contemporaneamente isolati da campioni ottenuti nelle case di soggetti che vivono in una grande area metropolitana. Il protocollo si basa sulla nostra compilazione e modifica delle tecniche attuali, compreso lo stoccaggio di tutte le frazioni "extra" in caso di tecniche future permettono di ulteriore isolamento / analisi. Mentre i metodi o kit alternativi possono essere impiegati al posto dei singoli metodi qui descritti, quelli delineati hanno dimostrato di essere un mezzo affidabile ed efficiente per la lavorazione dei campioni in maniera high-throughput. Frazioni di alta qualità (PBMC, DNA, siero, e RNA) di sangue fresco possono essere prodotti in 2 ore di raccolta e di tutti i campioni del test-ready può essere disponibile entro 2 giorni (Figura 1).
Questo protocollo è stato sviluppato per consentire il trattamento efficiente dei campioni raccolti da residenti in comunità, adulti residenti della città di Detroit per la sperimentazione in Detroit Quartiere Health Study (DNHS; DA022720, RC1MH088283, DA022720-05-S1) un basato sulla popolazione, studio dei determinanti sociali e biologiche del disturbo post-traumatico da stress (PTSD) e di altre malattie mentali. La prevalenza di PTSD a Detroit è più del doppio della media nazionale 11,12. Identificare determinanti biologiche di PTSD in questa popolazione può aiutare a sviluppare farmacologico adeguato e / o interventi cognitivo-comportamentali per aiutare coloro che soffrono di questo disturbo, sia in questa popolazione urbana, e in altre popolazioni ad alto rischio (ad esempio, tornando veterani militari). Il nostro laboratorio, precedentemente situato presso la Wayne State University di Detroit, Michigan, è stato selezionato per l'elaborazione sulla base della nostra esperienza nella gestione di campioni di tessuto fresche provenienti da una varietàdelle fonti, la necessità di iniziare il trattamento dei campioni in 2 ore di raccolta, e la nostra vicinanza ai siti di raccolta. Con questa opportunità unica a portata di mano, il nostro obiettivo era quello di ottimizzare il trattamento per il massimo rendimento di DNA, RNA, siero e le cellule mononucleate del sangue periferico (PBMC) da ciascun campione (per un totale di N = 1.639 campioni in 5 ondate di raccolta del campione). Le procedure descritte qui possono essere eseguite contemporaneamente in un contesto non clinico, producendo materiale (vedi Tabella 1 per rendimenti medi) di partenza per un gran numero di applicazioni a valle, tra cui microarray, epigenetica, real-time RT-PCR, e citometria a flusso analisi.
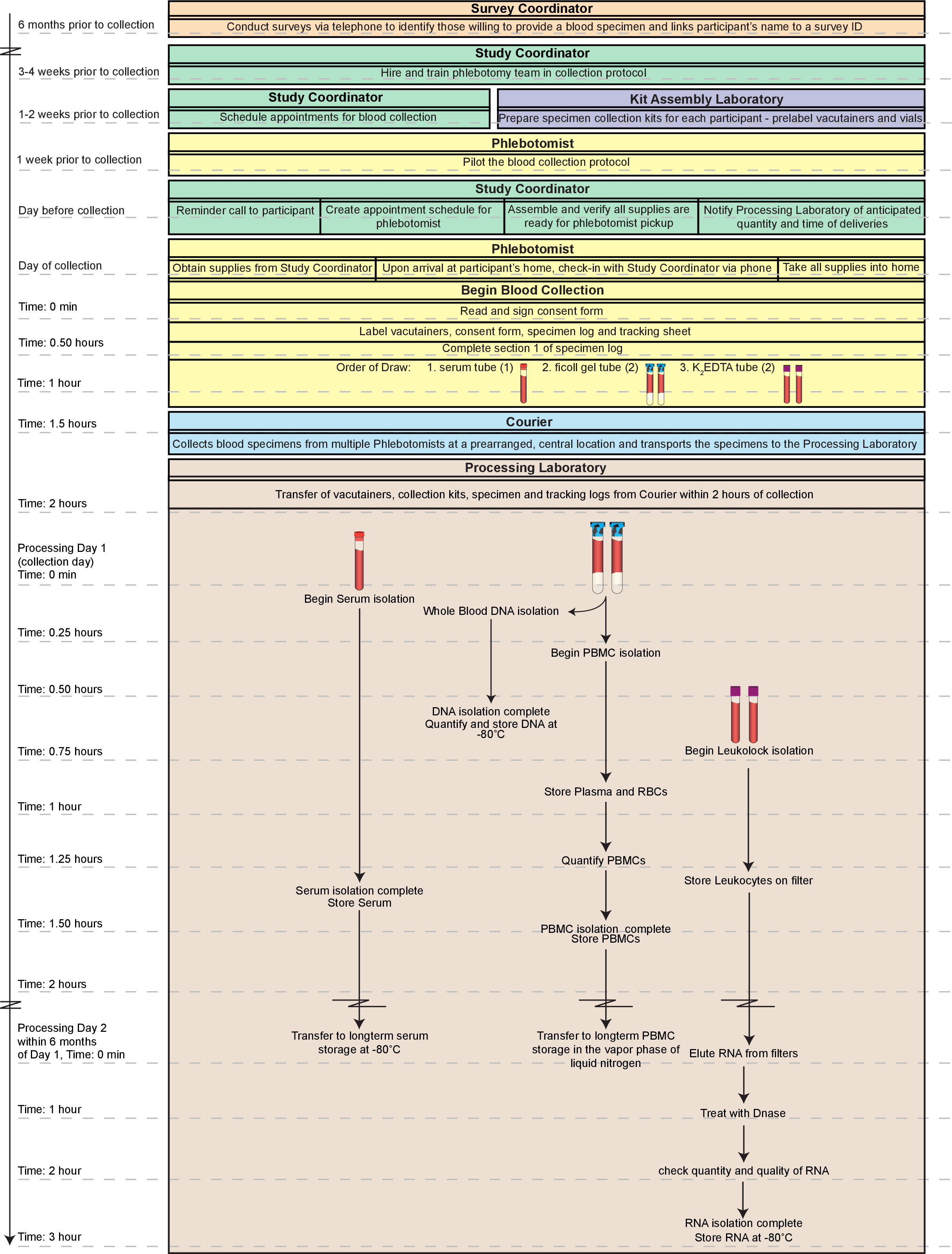
Figura 1. flusso di lavoro complessivo. Il processo globale raffigurato qui comprende la logistica di ottenere i campioni di sangue da identificare consenzienti particparte- al sangue disegnare sé. Di alta qualità, le frazioni (cellule mononucleate del sangue periferico, PBMC, DNA, siero, e RNA) di sangue intero fresco può essere prodotto in 2 ore di raccolta e di tutti i campioni del test-ready può essere disponibile entro 2 giorni. Inoltre, le frazioni preparati con questo metodo sono adatti per lo stoccaggio a lungo termine se i campioni non devono essere testati immediatamente. L'intera timeline qui delineato potrebbe essere completata in un solo giorno (~ 5 ore in totale). Tuttavia, un tale giorno sarebbe estremamente laborioso soprattutto per un singolo tecnico con esperienza sostanziale con le tecniche. Pertanto, si consiglia di dividere le procedure il giorno 1 tra almeno due tecnici e il completamento del trattamento RNA Day 2. Cliccate qui per vedere una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Abbiamo descritto un protocollo semplificato che è stato applicato con successo a elaborare più di 1.600 campioni di sangue intero in Health Study Detroit Quartiere. Sebbene molte di queste tecniche sono disponibili in letteratura, il nostro compilazione passo-passo, comprese le modifiche temporizzati precisamente ogni aumento, riflette una ottimizzata, protocollo efficiente che produce con successo una varietà di campioni biologici con una vasta gamma di applicazioni a valle, tra cui la metilazione del DNA, espressi…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We would like to thank Henriette Mair-Meijers for invaluable attention to detail and hours devoted to processing the blood collections. We are grateful for the graphic design expertise of Natalie Jameson Kiesling. We also appreciate the approval of the manufacturers (Qiagen (Valencia, CA), BD Biosciences (San Jose, CA), Life Technologies (Grand Island, NY)) mentioned herein to publish the use of their products as described. Funding for this work was generously provided by the National Institutes of Health award numbers DA022720, RC1MH088283, and DA022720-05-S1.
Materials
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Day 1: DNA isolation |
| Phosphate-buffered saline (PBS) | Sigma | P5493-1L | Day 1: PBMC isolation |
| 5 ¾” Pasteur pipets | Fisher | 13-678-6A | Day 1: PBMC isolation |
| Fetal Bovine Serum (FBS), heat inactivated | Life Technologies | 10082147 | Day 1: PBMC isolation |
| Dimethyl Sulfoxide (DMSO) | Sigma | D8418-500ml | Day 1: PBMC isolation |
| RPMI Medium 1640, liquid | Invitrogen | 11875119 | Day 1: PBMC isolation |
| 0.4% trypan blue stain | Invitrogen | T10282 | Day 1: PBMC isolation |
| Countess Cell Counting Chamber | Invitrogen | C10283 | Day 1: PBMC isolation |
| Countess Automated Cell Counter or cell counting device such as a microscope and hemocytometer | Invitrogen | C10281 | Day 1: PBMC isolation |
| LeukoLOCK Fractionation & Stabilization Kit | Ambion | 1933 | Day 1: Leukocyte RNA isolation |
| 25G x 5/8 in. needles | Becton Dickinson | 305122 | Day 1: Leukocyte RNA isolation |
| Syringes (5ml) | Becton Dickinson | 309646 | Days 1 and 2: Leukocyte RNA isolation |
| Denaturing Lysis Solution | Ambion | 8540G | Day 2: Leukocyte RNA isolation |
| 5M NaCl | Life Technologies | 24740011 | Day 2: Leukocyte RNA isolation |
| TRI Reagent | Ambion | 9738 | Day 2: Leukocyte RNA isolation |
| Bromo-3-chloro-propane (BCP) | Sigma | B-9673 | Day 2: Leukocyte RNA isolation |
| spin cartridges | Ambion | 10051G | Day 2: Leukocyte RNA isolation |
| 0.1mM EDTA | Ambion | 9912 | Day 2: Leukocyte RNA isolation |
| DNA-free Kit | Ambion | AM1960 | Day 2: DNase treament |
| RNA 6000 Ladder | Agilent | 5067-1529 | Day 2: Bioanalyzer analysis |
| RNA 6000 Nano Series II Kit | Agilent | 5067-1511 | Day 2: Bioanalyzer analysis |
| RNaseZAP | Ambion | AM8782 | Day 2: Bioanalyzer analysis |
| Ethanol >99% | Sigma | E7023-500ml | |
| Isopropanol >99% | Sigma | I9516-500ml | |
| Nuclease-free ultra pure water | Invitrogen | 9938 | |
| Pipette tips (nuclease-free) | Eppendorf | 22491253 | |
| Pipetter (serological) | Eppendorf | 2223020-4 | |
| Pipetters (for volumes under 1ml) | Eppendorf | 3120000-054 | |
| Pipettes (serological) | Fisher | 13-678-27E | |
| Controlled rate freezing containers | Nalgene | 5100-0001 | |
| Cryoboxes (to hold 2ml and 5ml cryovials and 1.5ml microcentrifuge tubes) | Fisher | 03-395-464 | |
| Test tube rack | Thermo Scientific | 14-804-134 | |
| 15ml polypropylene tubes | Fisher | 14-959-49D | |
| 1.5ml and 0.65 microcentrifuge tubes | Fisher | 07-200-534 and 07-200-185 | |
| 2ml and 5ml cryovials | Fisher | 10-500-26 and 10-269-88F | |
| 8ml CPT vacutainer | BD Biosciences | 362761 | 2 tubes |
| 6ml K2 EDTA vacutainer | BD Biosciences | 367863 | 2 tubes |
| 8.5ml SST vacutainer | BD Biosciences | 367988 | 1 tube |
| Vortexer | Fisher | 2215365 | |
| Dry bath incubator with heating block for microcentrifuge tubes | Fisher | 11-715-1250 | |
| Filtration/vacuum system for use within the cell culture hood | Fisher | 01-257-87 | |
| Fixed-angle rotor for microcentrifuge tubes with aerosol-tight lid | Eppendorf | 22637002 | |
| Refrigerated centrifuge with a swing-bucket rotor and aerosol-tight caps for 16 x 125mm vacutainers and 15ml polypropylene tubes | Eppendorf | 22628157 | 2, one does not need to be refrigerated |
| Nanodrop 2000 (recommended for accuracy of small volumes) or other spectrophotometric device | Fisher | 13-400-411 | |
| Agilent Bioanalyzer | Agilent Technologies | G2940CA | |
| Liquid nitrogen tank | Thermo Scientific | 11-676-56 | |
| -80 ˚C freezer | Thermo Scientific | 992RAK | |
| Sharps container | Fisher | 22-037-970 | |
| Biological waste container | Thermo Scientific | 1223P52 | |
| Biosafety Level 2 certified cell culture hood | Thermo Scientific | 13-261-315 |
References
- Hernandez, M. E., Martinez-Fong, D., Perez-Tapia, M., Estrada-Garcia, I., Estrada-Parra, S., Pavon, L. Evaluation of the effect of selective serotonin-reuptake inhibitors on lymphocyte subsets in patients with a major depressive disorder. Eur Neuropsychopharmacol. 20, 88-95 (2010).
- Weigelt, K., et al. TREM-1 and DAP12 expression in monocytes of patients with severe psychiatric disorders EGR3, ATF3 and PU.1 as important transcription factors. Brain Behav Immun. 25, 1162-1169 (2011).
- Robertson, M. J., et al. Lymphocyte subset differences in patients with chronic fatigue syndrome, multiple sclerosis and major depression. Clin Exp Immunol. 141, 326-332 (2005).
- Rotter, A., Asemann, R., Decker, A., Kornhuber, J., Biermann, T. Orexin expression and promoter-methylation in peripheral blood of patients suffering from major depressive disorder. J Affect Disord. 131, 186-192 (2011).
- Klengel, T., et al. Allele-specific FKBP5 DNA demethylation mediates gene-childhood trauma interactions. Nat Neurosci. 16, 33-41 (2013).
- Segman, R. H., Shefi, N., Goltser-Dubner, T., Friedman, N., Kaminski, N., Shalev, A. Y. Peripheral blood mononuclear cell gene expression profiles identify emergent post-traumatic stress disorder among trauma survivors. Mol Psychiatry. 10, 500-513 (2005).
- Smith, B. H., et al. Cohort Profile: Generation Scotland: Scottish Family Health Study (GS:SFHS). The study, its participants and their potential for genetic research on health and illness. Int J Epidemiol. 42, 689-700 (2013).
- Mallone, R., et al. Isolation and preservation of peripheral blood mononuclear cells for analysis of islet antigen-reactive T cell responses: position statement of the T-Cell Workshop Committee of the Immunology of Diabetes Society. Clin Exp Immunol. 163, 33-49 (2011).
- Duvigneau, J. C., Hartl, R. T., Teinfalt, M., Gemeiner, M. Delay in processing porcine whole blood affects cytokine expression. J Immunol Methods. 272, 11-21 (2003).
- Debey, S., et al. Comparison of different isolation techniques prior gene expression profiling of blood derived cells: impact on physiological responses, on overall expression and the role of different cell types. Pharmacogenomics J. 4, 193-207 (2004).
- Uddin, M., et al. Epigenetic and immune function profiles associated with posttraumatic stress disorder. Proc Natl Acad Sci U S A. 107, 9470-9475 (2010).
- Kessler, R. C., Wang, P. S. The descriptive epidemiology of commonly occurring mental disorders in the United States. Annu Rev Public Health. 29, 115-129 (2008).
- . QIAamp® DNA Mini and Blood Mini Handbook Available from: https://www.qiagen.com/us/resources/resourcedetail?id=67893a91-946f-49b5-8033-394fa5d752ea (2010)
- Koenen, K. C., et al. SLC6A4 methylation modifies the effect of the number of traumatic events on risk for posttraumatic stress disorder. Depress Anxiety. 28, 639-647 (2011).
- Walsh, K., Uddin, M., Soliven, R., Wildman, D. E., Bradley, B. Associations between the SS variant of 5-HTTLPR and PTSD among adults with histories of childhood emotional abuse: Results from two African American independent samples. J Affect Disord. 161, 91-96 (2014).
- Bustamante, A. C., et al. Childhood maltreatment is associated with epigenetic differences in hypothalamic-pituitary-adrenal (HPA) axis genes in the Detroit Neighborhood Health Study. , (2013).
- Sipahi, L., et al. Longitudinal epigenetic variation of DNA methyltransferase genes is associated with vulnerability to post-traumatic stress disorder. Psychol Med. 44, 3165-3179 (2014).
- Uddin, M., Koenen, K. C., Aiello, A. E., Wildman, D., de los Santos, R., Galea, S. Epigenetic and inflammatory marker profiles associated with depression in a community-based epidemiologic sample. Psychol Med. 41, 997-1007 (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M. Biological signatures of post-traumatic stress disorder in the Detroit Neighborhood Health Study. , (2010).
- Aiello, A. E. Cytomegalovirus antibodies as a marker of immunosenescence in the Detroit Neighborhood Health Study. , (2010).
- Bustamante, A. C., et al. . Distinct gene expression profiles characterize lifetime PTSD and childhood maltreatment in the Detroit Neighborhood Health Study. , (2013).

