Fracionamento rápido e isolamento de componentes de sangue total em amostras obtidas a partir de uma configuração de base comunitária
Summary
We outline a methodology for the processing of whole blood to obtain a variety of components for further analysis. We have optimized a streamlined protocol that enables rapid, high-throughput simultaneous processing of whole blood samples in a non-clinical setting.
Abstract
Coleta e processamento de amostras de sangue total em um ambiente não-clínica oferece uma oportunidade única para avaliar indivíduos residentes na comunidade com e sem condições pré-existentes. Transformação rápida destas amostras é essencial para evitar a degradação dos componentes celulares principais. Incluem-se aqui métodos para simultânea de células mononucleares do sangue periférico (PBMC), DNA, RNA e isolamento de soro a partir de uma única coleta de sangue realizados nas casas dos consentindo participantes através de uma área metropolitana, com o processamento iniciado no prazo de 2 horas da coleta. Temos usado essas técnicas para processar mais de 1.600 amostras de sangue produzindo, materiais de alta qualidade consistente, o que foi posteriormente utilizado na metilação do DNA, genotipagem, expressão gênica bem sucedido e citometria de fluxo analisa. Alguns dos métodos empregados são padrão; No entanto, quando combinados da maneira descrita, permitem um processamento eficiente das amostras a partir de participantes de populacional e / ou comunitáriaestudos baseados que normalmente não seriam avaliadas em um ambiente clínico. Por conseguinte, este protocolo tem o potencial de se obter amostras de dados (e, posteriormente), que são mais representativas da população geral.
Introduction
Vários estudos têm caracterizado diferenças na expressão genética, a metilação do DNA e subconjunto de células no sangue entre os indivíduos com e sem mental (ou outras) doenças 1-4. Esses estudos, no entanto, foram obtidos a partir de situações clínicas em que as diferenças associados à doença podem ser ampliadas devido à natureza geralmente mais grave das doenças para as quais os pacientes procuram tratamento. Devido aos avanços na "ómicas" abordagens, a última década assistiu a uma explosão de interesse em obter amostras biológicas de comunidade e / ou contextos epidemiológicos 5-7, a fim de fornecer estimativas populacionais de prevalência da doença e um quadro mais amplo do determinantes ambientais destas doenças mentais e / ou físicas.
Um desafio fundamental a este respeito é a exigência de um rápido processamento dos espécimes coletados. Degradação de células mononucleares, os componentes do sistema imunológico que são fundamentais frequently utilizado para avaliar a saúde de um indivíduo, começa imediatamente após a colheita de sangue com uma diminuição significativa na recuperação após 2 horas de recolha de 8-10. Para enfrentar esse desafio, apresentamos um protocolo otimizado em que vários componentes do sangue humano são simultaneamente isolado a partir de amostras obtidas nas casas de indivíduos que vivem em uma grande área metropolitana. O protocolo é baseado em nossa compilação e modificação das técnicas atuais, incluindo o armazenamento de todas as frações "extra" em caso futuras técnicas permitem um maior isolamento / análises. Enquanto os métodos alternativos ou estojos podem ser utilizados no lugar dos métodos descritos aqui individuais, aqueles descritos provaram ser um meio fiável e eficiente para o processamento de amostras de um modo de alto rendimento. Frações de alta qualidade (PBMC, DNA, soro e RNA) de sangue fresco pode ser produzido dentro de 2 horas de coleta e todos os espécimes de ensaio-pronto pode estar disponível dentro de 2 dias (Figura 1).
Este protocolo foi desenvolvido para permitir o processamento eficiente de amostras coletadas de comunidade-moradia, adultos residentes na cidade de Detroit para testes no Estudo de Saúde Bairro Detroit (DNHS; DA022720, RC1MH088283, DA022720-05-S1) uma base populacional, estudo dos determinantes sociais e biológicas do transtorno de estresse pós-traumático (PTSD) e outras doenças mentais. A prevalência de TEPT em Detroit é mais do que o dobro da média nacional 11,12. Identificar os determinantes biológicos de PTSD nesta população pode ajudar a desenvolver farmacológico adequado e / ou intervenções cognitivo-comportamentais para ajudar aqueles que sofrem de transtorno, tanto na população urbana, e em outras populações de alto risco (por exemplo, retornando veteranos militares). Nosso laboratório, anteriormente localizado na Wayne State University, em Detroit, Michigan, foi selecionado para o processamento com base em nossa experiência em lidar com amostras de tecido fresco derivadas de uma variedadede fontes, a necessidade de começar a processar as amostras no prazo de 2 horas da coleta, e nossa proximidade com os locais de coleta. Com esta oportunidade única na mão, o nosso objetivo foi otimizar o processamento para maior rendimento de DNA, RNA, soro e células mononucleares do sangue periférico (PBMC) a partir de cada amostra (de um total de n = 1.639 amostras ao longo de 5 ondas de coleta de amostra). Os procedimentos descritos aqui podem ser realizadas simultaneamente num ambiente não-clínica, produzindo, assim, o material (ver Tabela 1 para os rendimentos médios) a partir de um grande número de aplicações a jusante, incluindo microarrays, epigenética, em tempo real de RT-PCR, e a citometria de fluxo análises.
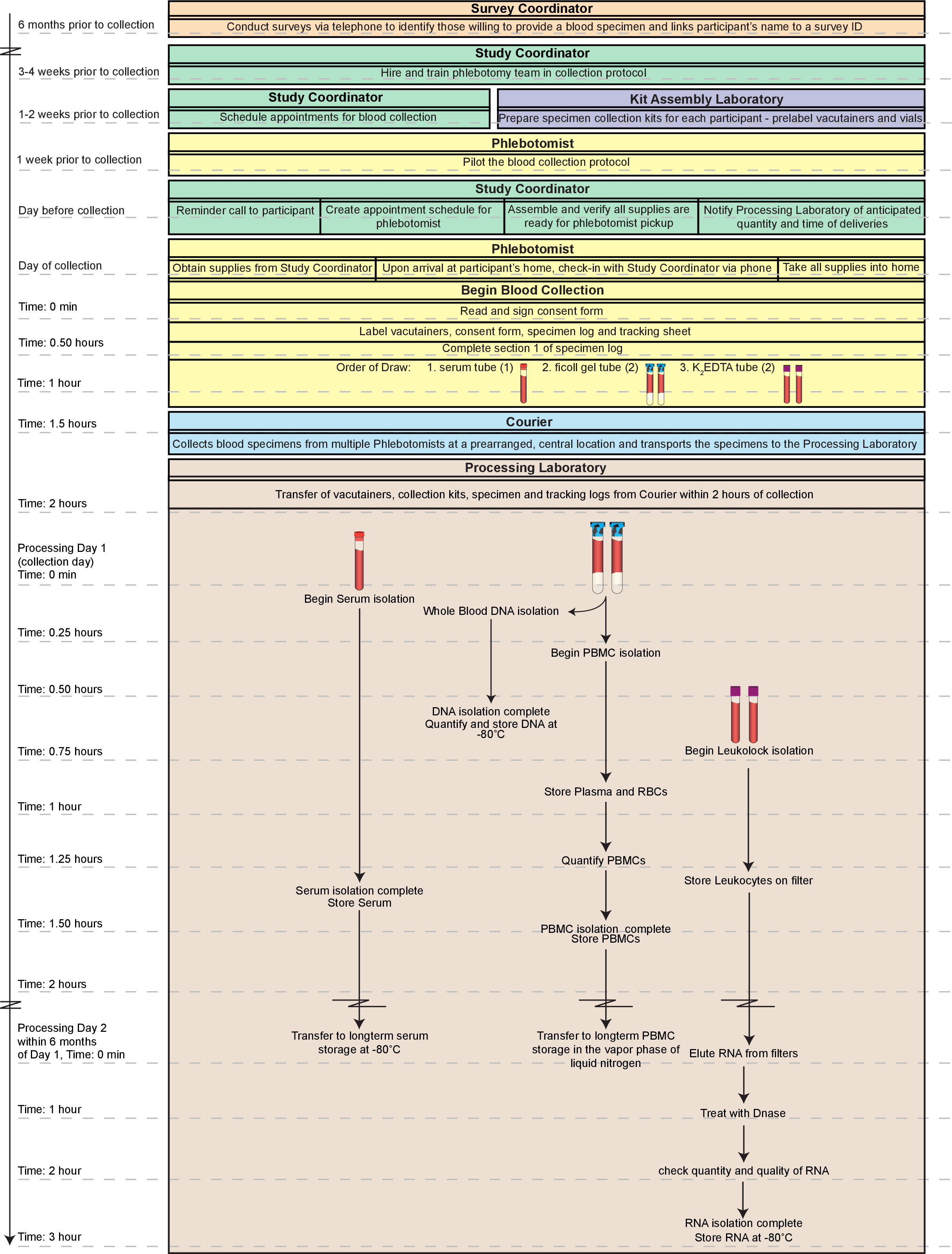
Figura 1. fluxo geral de trabalho. O processo global descrito aqui inclui a logística de obtenção das amostras de sangue de identificar consentindo participants para o sangue desenhar a si próprio. De alta qualidade, as fracções (células mononucleares do sangue periférico; PBMC, ADN, soro, ARN) e de sangue inteiro fresco pode ser produzido dentro de 2 horas de recolha e todas as amostras de ensaio prontos podem estar disponíveis no prazo de 2 dias. Além disso, as fracções preparadas por este método são adequados para armazenamento a longo prazo se as amostras não são para ser testada imediatamente. Todo o cronograma descrito aqui pode ser concluído em um único dia (~ 5 horas total). No entanto, um tal dia seria extremamente trabalhoso, especialmente para um único técnico com experiência substancial com as técnicas. Assim, recomendamos dividindo os procedimentos no dia 1 entre pelo menos dois técnicos e concluir o processamento do RNA no dia 2. Por favor clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Nós descrevemos um protocolo simplificado que tem sido aplicado com sucesso para processar mais de 1.600 amostras de sangue total no Estudo de Saúde Detroit Neighborhood. Apesar de muitas destas técnicas estão disponíveis na literatura existente, a compilação passo-a-passo, incluindo alterações precisamente programado entre cada passo, reflecte, um protocolo optimizado eficiente que produz com sucesso uma variedade de amostras biológicas com uma ampla gama de aplicações a jusante, incluindo a metilação do …
Disclosures
The authors have nothing to disclose.
Acknowledgements
We would like to thank Henriette Mair-Meijers for invaluable attention to detail and hours devoted to processing the blood collections. We are grateful for the graphic design expertise of Natalie Jameson Kiesling. We also appreciate the approval of the manufacturers (Qiagen (Valencia, CA), BD Biosciences (San Jose, CA), Life Technologies (Grand Island, NY)) mentioned herein to publish the use of their products as described. Funding for this work was generously provided by the National Institutes of Health award numbers DA022720, RC1MH088283, and DA022720-05-S1.
Materials
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Day 1: DNA isolation |
| Phosphate-buffered saline (PBS) | Sigma | P5493-1L | Day 1: PBMC isolation |
| 5 ¾” Pasteur pipets | Fisher | 13-678-6A | Day 1: PBMC isolation |
| Fetal Bovine Serum (FBS), heat inactivated | Life Technologies | 10082147 | Day 1: PBMC isolation |
| Dimethyl Sulfoxide (DMSO) | Sigma | D8418-500ml | Day 1: PBMC isolation |
| RPMI Medium 1640, liquid | Invitrogen | 11875119 | Day 1: PBMC isolation |
| 0.4% trypan blue stain | Invitrogen | T10282 | Day 1: PBMC isolation |
| Countess Cell Counting Chamber | Invitrogen | C10283 | Day 1: PBMC isolation |
| Countess Automated Cell Counter or cell counting device such as a microscope and hemocytometer | Invitrogen | C10281 | Day 1: PBMC isolation |
| LeukoLOCK Fractionation & Stabilization Kit | Ambion | 1933 | Day 1: Leukocyte RNA isolation |
| 25G x 5/8 in. needles | Becton Dickinson | 305122 | Day 1: Leukocyte RNA isolation |
| Syringes (5ml) | Becton Dickinson | 309646 | Days 1 and 2: Leukocyte RNA isolation |
| Denaturing Lysis Solution | Ambion | 8540G | Day 2: Leukocyte RNA isolation |
| 5M NaCl | Life Technologies | 24740011 | Day 2: Leukocyte RNA isolation |
| TRI Reagent | Ambion | 9738 | Day 2: Leukocyte RNA isolation |
| Bromo-3-chloro-propane (BCP) | Sigma | B-9673 | Day 2: Leukocyte RNA isolation |
| spin cartridges | Ambion | 10051G | Day 2: Leukocyte RNA isolation |
| 0.1mM EDTA | Ambion | 9912 | Day 2: Leukocyte RNA isolation |
| DNA-free Kit | Ambion | AM1960 | Day 2: DNase treament |
| RNA 6000 Ladder | Agilent | 5067-1529 | Day 2: Bioanalyzer analysis |
| RNA 6000 Nano Series II Kit | Agilent | 5067-1511 | Day 2: Bioanalyzer analysis |
| RNaseZAP | Ambion | AM8782 | Day 2: Bioanalyzer analysis |
| Ethanol >99% | Sigma | E7023-500ml | |
| Isopropanol >99% | Sigma | I9516-500ml | |
| Nuclease-free ultra pure water | Invitrogen | 9938 | |
| Pipette tips (nuclease-free) | Eppendorf | 22491253 | |
| Pipetter (serological) | Eppendorf | 2223020-4 | |
| Pipetters (for volumes under 1ml) | Eppendorf | 3120000-054 | |
| Pipettes (serological) | Fisher | 13-678-27E | |
| Controlled rate freezing containers | Nalgene | 5100-0001 | |
| Cryoboxes (to hold 2ml and 5ml cryovials and 1.5ml microcentrifuge tubes) | Fisher | 03-395-464 | |
| Test tube rack | Thermo Scientific | 14-804-134 | |
| 15ml polypropylene tubes | Fisher | 14-959-49D | |
| 1.5ml and 0.65 microcentrifuge tubes | Fisher | 07-200-534 and 07-200-185 | |
| 2ml and 5ml cryovials | Fisher | 10-500-26 and 10-269-88F | |
| 8ml CPT vacutainer | BD Biosciences | 362761 | 2 tubes |
| 6ml K2 EDTA vacutainer | BD Biosciences | 367863 | 2 tubes |
| 8.5ml SST vacutainer | BD Biosciences | 367988 | 1 tube |
| Vortexer | Fisher | 2215365 | |
| Dry bath incubator with heating block for microcentrifuge tubes | Fisher | 11-715-1250 | |
| Filtration/vacuum system for use within the cell culture hood | Fisher | 01-257-87 | |
| Fixed-angle rotor for microcentrifuge tubes with aerosol-tight lid | Eppendorf | 22637002 | |
| Refrigerated centrifuge with a swing-bucket rotor and aerosol-tight caps for 16 x 125mm vacutainers and 15ml polypropylene tubes | Eppendorf | 22628157 | 2, one does not need to be refrigerated |
| Nanodrop 2000 (recommended for accuracy of small volumes) or other spectrophotometric device | Fisher | 13-400-411 | |
| Agilent Bioanalyzer | Agilent Technologies | G2940CA | |
| Liquid nitrogen tank | Thermo Scientific | 11-676-56 | |
| -80 ˚C freezer | Thermo Scientific | 992RAK | |
| Sharps container | Fisher | 22-037-970 | |
| Biological waste container | Thermo Scientific | 1223P52 | |
| Biosafety Level 2 certified cell culture hood | Thermo Scientific | 13-261-315 |
References
- Hernandez, M. E., Martinez-Fong, D., Perez-Tapia, M., Estrada-Garcia, I., Estrada-Parra, S., Pavon, L. Evaluation of the effect of selective serotonin-reuptake inhibitors on lymphocyte subsets in patients with a major depressive disorder. Eur Neuropsychopharmacol. 20, 88-95 (2010).
- Weigelt, K., et al. TREM-1 and DAP12 expression in monocytes of patients with severe psychiatric disorders EGR3, ATF3 and PU.1 as important transcription factors. Brain Behav Immun. 25, 1162-1169 (2011).
- Robertson, M. J., et al. Lymphocyte subset differences in patients with chronic fatigue syndrome, multiple sclerosis and major depression. Clin Exp Immunol. 141, 326-332 (2005).
- Rotter, A., Asemann, R., Decker, A., Kornhuber, J., Biermann, T. Orexin expression and promoter-methylation in peripheral blood of patients suffering from major depressive disorder. J Affect Disord. 131, 186-192 (2011).
- Klengel, T., et al. Allele-specific FKBP5 DNA demethylation mediates gene-childhood trauma interactions. Nat Neurosci. 16, 33-41 (2013).
- Segman, R. H., Shefi, N., Goltser-Dubner, T., Friedman, N., Kaminski, N., Shalev, A. Y. Peripheral blood mononuclear cell gene expression profiles identify emergent post-traumatic stress disorder among trauma survivors. Mol Psychiatry. 10, 500-513 (2005).
- Smith, B. H., et al. Cohort Profile: Generation Scotland: Scottish Family Health Study (GS:SFHS). The study, its participants and their potential for genetic research on health and illness. Int J Epidemiol. 42, 689-700 (2013).
- Mallone, R., et al. Isolation and preservation of peripheral blood mononuclear cells for analysis of islet antigen-reactive T cell responses: position statement of the T-Cell Workshop Committee of the Immunology of Diabetes Society. Clin Exp Immunol. 163, 33-49 (2011).
- Duvigneau, J. C., Hartl, R. T., Teinfalt, M., Gemeiner, M. Delay in processing porcine whole blood affects cytokine expression. J Immunol Methods. 272, 11-21 (2003).
- Debey, S., et al. Comparison of different isolation techniques prior gene expression profiling of blood derived cells: impact on physiological responses, on overall expression and the role of different cell types. Pharmacogenomics J. 4, 193-207 (2004).
- Uddin, M., et al. Epigenetic and immune function profiles associated with posttraumatic stress disorder. Proc Natl Acad Sci U S A. 107, 9470-9475 (2010).
- Kessler, R. C., Wang, P. S. The descriptive epidemiology of commonly occurring mental disorders in the United States. Annu Rev Public Health. 29, 115-129 (2008).
- . QIAamp® DNA Mini and Blood Mini Handbook Available from: https://www.qiagen.com/us/resources/resourcedetail?id=67893a91-946f-49b5-8033-394fa5d752ea (2010)
- Koenen, K. C., et al. SLC6A4 methylation modifies the effect of the number of traumatic events on risk for posttraumatic stress disorder. Depress Anxiety. 28, 639-647 (2011).
- Walsh, K., Uddin, M., Soliven, R., Wildman, D. E., Bradley, B. Associations between the SS variant of 5-HTTLPR and PTSD among adults with histories of childhood emotional abuse: Results from two African American independent samples. J Affect Disord. 161, 91-96 (2014).
- Bustamante, A. C., et al. Childhood maltreatment is associated with epigenetic differences in hypothalamic-pituitary-adrenal (HPA) axis genes in the Detroit Neighborhood Health Study. , (2013).
- Sipahi, L., et al. Longitudinal epigenetic variation of DNA methyltransferase genes is associated with vulnerability to post-traumatic stress disorder. Psychol Med. 44, 3165-3179 (2014).
- Uddin, M., Koenen, K. C., Aiello, A. E., Wildman, D., de los Santos, R., Galea, S. Epigenetic and inflammatory marker profiles associated with depression in a community-based epidemiologic sample. Psychol Med. 41, 997-1007 (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M., et al. Post-traumatic stress disorder is associated with immunosenescent T cell phenotypes in the Detroit Neighborhood Health Study. , (2011).
- Uddin, M. Biological signatures of post-traumatic stress disorder in the Detroit Neighborhood Health Study. , (2010).
- Aiello, A. E. Cytomegalovirus antibodies as a marker of immunosenescence in the Detroit Neighborhood Health Study. , (2010).
- Bustamante, A. C., et al. . Distinct gene expression profiles characterize lifetime PTSD and childhood maltreatment in the Detroit Neighborhood Health Study. , (2013).

