Summary
ويرد وصف لطريقة متوازية للغاية لقياس الانقسام الموقع محددة من الحمض النووي في مستوى جزيء واحد. يوضح هذا البروتوكول الأسلوب باستخدام NdeI قيد endonuclease. يمكن بسهولة تعديل هذه الطريقة لدراسة أي عملية تؤدي إلى شق الحمض النووي في الموقع.
Abstract
إن شق الحمض النووي الخاص بالموقع (SSDC) هو خطوة رئيسية في العديد من العمليات الخلوية ، وهو أمر بالغ الأهمية لتحرير الجينات. يصف هذا العمل اختبارًا حركيًا قادرًا على قياس SSDC في العديد من جزيئات الحمض النووي الفردية في وقت واحد. يتم إعداد DSNrate الركيزة المربوطة، وتحتوي كل منها على نسخة واحدة من التسلسل المستهدف، في قناة تدفق ميكروفلويديك. المغناطيس الخارجي ينطبق قوة ضعيفة على الخرز paramagnetic. يمكن مراقبة سلامة ما يصل إلى 1000 DNAs الفردية من خلال تصور الميكروبات تحت التصوير darkfield باستخدام حقل واسع ، هدف التكبير المنخفض. حقن endonuclease تقييد، NdeI، يبدأ رد فعل الانقسام. يستخدم المجهر الفيديو لتسجيل اللحظة الدقيقة لكل انشقاق الحمض النووي من خلال مراقبة الإطار الذي يتحرك فيه الخرزة المرتبطة لأعلى وخارج المستوى البؤري للهدف. يحدد عد حبة الإطار حسب الإطار مقدار التفاعل، ويحدد الاحتواء الأسي معدل التفاعل. هذه الطريقة تسمح بجمع البيانات الكمية والناحصة إحصائيا على جزيء واحد SSDC التفاعلات في تجربة واحدة.
Introduction
إن شق الحمض النووي الخاص بالموقع (SSDC) هو خطوة رئيسية في العديد من المعاملات الجينومية. على سبيل المثال، تحمي أنظمة التقييد البكتيرية -التعديل (RM)1 و CRISPR2 الخلايا من الهجوم بواسطة phages و plasmids من خلال التعرف على الحمض النووي الأجنبي وشقه في تسلسلات محددة. في النوع الثاني RM، تقييد endonucleases (REs) التعرف على قصيرة 4-8 قاعدة الزوج (bp) تسلسلات عبر البروتين النووي التفاعلات حمض3. ترتبطوريس المرتبطة بـ CRISPR، مثل Cas9، بالمواقع عبر تهجين الموقع المستهدف مع ربط crRNAs إلى endonucleases4. إنشاء موقع محدد فواصل مزدوجة الذين تقطعت بهم السبل (DSBs) هي أيضا الخطوة الأولى في العديد من أحداث إعادة التهيئة DNA5. على سبيل المثال، يتطلب تنوع مناطق ربط المستضدات التي أنشأها إعادة التركيب V(D)J الاعتراف وشق مواقع مستهدفة محددة6. ومن المعروف أن بعض transposons لاستهداف تسلسلات الحمض النووي محددة, وكذلك7. ليس من المستغرب أن العديد من النوى الخاصة بالموقع التي تشارك في هذه العمليات ، مثل Cas9 ، هي مكون رئيسي لتقنيات تحرير الجينات8. وبالإضافة إلى ذلك، تم أيضا تصميم رواية endonucleases موقع محدد (أي، زنك النيوكلاليس إصبع9 و تالنس10)كما تم تصميمها لتحرير الجينوم.
وقد استخدمت العديد من الأساليب لقياس حركة شق الموقع المحدد للأحماض النووية. وتشمل هذه التحليل هلام، fluorescence11،12، والتسلسل على أساس الأساليب13. وقد تحقق تقدم كبير مع ربط الميكروبات ، والذي يسمح DSBs في جزيئات واحدة من الحمض النووي ليتم الكشف عنها من قبل حركة حبة بعد فصل حبلا. في هذه الأساليب، يتم استخدام أنواع مختلفة من القوات لضمان فصل حبلا والحركة من الخرزة ما بعد الانقسام. في حالة واحدة، وقد استخدمت الفخاخ البصرية لقياس الانقسام من الحمض النووي من قبل EcoRV14. في هذه التجارب، البحث الهدف هو الهدف من التحقيق، مع الشروط الأمثل بحيث ربط موقع معين هو معدل الحد من الخطوة. عيب واحد من الفخاخ البصرية هو أنه يمكن ملاحظة الحمض النووي واحد فقط في وقت واحد. وبالإضافة إلى ذلك، يجب تطبيق قوة سحب كبيرة دورية لاختبار فصل حبلا.
تقنية أخرى يستخدم مزيج من تدفق وضعف القوى المغناطيسية لسحب على حبة بطريقة مستمرة15. وبهذه الطريقة، يتم قياس الانقسام الانتشار المحدود بواسطة NdeI. وتسمح الطريقة المستخدمة بالقياس المتزامن لعدة مئات من السنوات من النايـزات DNN في آن واحد، مما يتيح تحقيق الأهمية الإحصائية في تجربة واحدة. كما استخدمت التجارب القائمة على ملاقط مغناطيسية. في إحدى هذه الدراسات، تمت دراسة إنتيجراس فيروسي من خلال تضمين DSB في أوليجونوكليوتيد16. نتج عن الاندماج الناجح دمج DSB في الحمض النووي المربوط وفقدان الخرزة المرفقة. وفي دراسة مماثلة للنوع الثالث المعتمد على ATP للاندليس Endonuclease EcoPI، لوحظ عشرات من DN أنياب في تجربة واحدة17. ملاقط المغناطيسي عقد ميزة أن التوتر، فضلا عن حلقات الحمض النووي، يمكن السيطرة عليها ورصدها أثناء التفاعل.
تظهر هنا طريقة جزيء واحد متوازي للغاية لقياس حركية SSDC ، والتي تستفيد من التحسينات الحديثة في الربط على نطاق واسع من DN. هذا الأسلوب هو تحسين وتمديد الطرق السابقة المستخدمة لقياس النسخ المتماثل الحمض النووي18، طول كفاف من الحمض النووي19، والانقسام بواسطة REs15. في هذه التقنية، يتم إعداد DNAs الخطية التي تحتوي على نسخة واحدة من تسلسل التعرف مع البيوتين في نهاية واحدة وdidoxigenin في الطرف الآخر. يربط البيوتين streptavidin ، الذي يرتبط بشكل مُت بارادييدي شبه مُجَاَنِي. يتم حقن مجمعات حبة الحمض النووي في قناة microfluidic التي تم وظيفية مع شظايا مضادة للحفرية. ثم الحبال الحمض النووي إلى نقاط مرفق السطح عن طريق ربط digoxigenin لشظايا القوات المسلحة البوروندية الممزتزة. القوى المغناطيسية ضعيفة تطبيقها مع المغناطيس الدائم الحفاظ على حبة من التمسك غير على وجه التحديد إلى السطح. يمكن حقن العينات بسرعة (< 30 s) في قناة التدفق لتنشيط تفاعل الانقسام. يتم إيقاف تشغيل التدفق أثناء تجميع البيانات. كما هو مشقوق كل الحمض النووي، يمكن تحديد الوقت الدقيق للشق عن طريق تسجيل الإطار الذي يتحرك الخرز صعودا وخارج خطة التنسيق من الهدف، وبالتالي تختفي من سجل الفيديو. ويمكن استخدام عدد الإطارات حسب الإطارات من الخرز المتبقية لقياس التقدم في التفاعل.
ويرد أدناه البروتوكول الكامل وكذلك البيانات التي تم جمعها باستخدام NdeI. وكمثال على كيفية تطبيق هذه التقنية، يتم قياس معدلات الانقسام لمجموعة من تركيزات البروتين في تركيزين مختلفين من المغنيسيوم، وهو عامل مساعد معدني أساسي. على الرغم من أن هذا التطبيق للبروتوكول يستخدم NdeI، يمكن تكييف الأسلوب للاستخدام مع أي نواة خاصة بالموقع عن طريق تغيير تصميم الركيزة الحمض النووي.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. جعل خلية تدفق
- غسل الأغطية
- مكان يغطي في تلطيخ الجرار و sonicate مع الإيثانول (EtOH)، ثم مع 1 M KOH (لمدة 30 دقيقة لكل من). لتجنب هطول الأمطار كوه في EtOH، شطف جيدا مع ddH2س بين يغسل.
- كرر كل من EtOH وOH غسل الخطوات 1x لمجموع عدد من أربعة يغسل (اثنين EtOH واثنين من KOH). تخزين أغطية نظيفة في ddH2O في تلطيخ الجرار.
- قطع أنابيب التحميل والخروج (8 سم طويلة) باستخدام شفرة حلاقة نظيفة وإدراجها في ثقوب في شريحة زجاجية نظيفة. استخدم PE-20 للمدخل وPE-60 للمنفذ. الايبوكسي لمدة 5 دقائق لتأمين أنابيب وتقليم قبالة أي أنابيب الزائدة.
ملاحظة: الشرائح الزجاجية قياس 2'' × 3'' × 1 ملم. يتم حفر الثقوب في أزواج 15 مم عن بعضها البعض. كل زوج النماذج إما نهاية قناة واحدة. يتم استخدام أنابيب مع معرف أصغر للمدخل، وهذا يقلل من حجم الميت المنبع وبالتالي وقت الخلط المطلوبة (الشكل 1). - خط حتى وتطبيق الشريط قبل قطع على الوجهين مع نمط قناة قطع أكثر من الثقوب على الشريحة الزجاجية. سلاسة الخروج مع ملقط من البلاستيك لتحقيق ختم جيد.
ملاحظة: يبلغ سمك الشريط المزدوج الوجه المستخدم في هذه التجربة 120 ميكرومتر، و طول القنوات 2 مم وطوله 15 مم. يتم قطع القنوات باستخدام طابعة سكين (جدول المواد). من الممكن أن تناسب ما يصل إلى أربع قنوات على غطاء واحد. انظر الشكل 1 للحصول على صورة لخلية التدفق. - بعد تقشير قبالة دعم، وتطبيق غطاء نظيف (المجففة مع الهواء المضغوط) على الشريط وسلس مرة أخرى مع ملقط من البلاستيك لختم جيد.
- الايبوكسي على حافة قبالة الغطاء لختم الخلية تدفق والسماح لها علاج.
2. إعداد الحمض النووي المسمى للربط
- في أنبوب PCR، إعداد 50 ميكرولتر من مزيج تفاعل PCR التي تحتوي على 0.02 U/μL بوليميراز الحمض النووي عالية الدقة، 200 μM dNTPs، 0.5 μM التمهيدي إلى الأمام، 0.5 μM التمهيدي العكسي، و 250 نانوغرام من الحمض النووي M13mp18 المتجه.
ملاحظة: هنا، تم اختيار التمهيدي الأمامي (البيوتين-CCAACTTACGCCTTGC) واوبامير عكس (digoxigenin-TGACCATTAGATACATTCC) لتضخيم منطقة ما يقرب من 1،000 bp طويلة، تمتد من المواقع 6338 إلى 107 في الجينوم الدائري للحمض النووي M13mp18. هناك موقع واحد NdeI في وسط المنطقة تضخيم. التمهيدي الأمامي هو 5 2000 توصف مع ديجوكسيجينين، الذي يربط المضادة للديجوجينين على غطاء. و هو 5 5 مع البيوتين، الذي يربط الخرز المغلفة streptavidin. - أدخل أنبوب PCR في ثيرموساير وتتبع الدورة كما هو مبين في الجدول 1.
- تنقية المنتج PCR مع مجموعة تنظيف PCR بعد بروتوكول الشركة المصنعة.
ملاحظة: باستخدام مجموعة المحددة في جدول المواد، فإن العائد من الحمض النووي النموذجي هو ~ 2 ميكروغرام.
3. الربط من الحمض النووي والخرز
- إعداد 10 مل من العازلة A (1 م تريس-HCl [pH = 7.5] ، 50 mM NaCl ، 2 mM MgCl2، 1 ملغ / مل β - كيسين ، 1 ملغم / mL Pluronic F-127). ديغا في جهاز اجسائي للغوط على الأقل 1 ساعة.
- لإضفاء الطابع الوظيفي على خلية التدفق، قم بإدخال 25 ميكرولتر من شظايا مضادة للدوجوجينين من فاب (20 ميكروغرام/مل) في قناة التدفق. استخدم نصائح تحميل الجل لتلائم أنابيب PE-60. احتضان في درجة حرارة الغرفة (RT) لمدة 30 دقيقة.
- بعد الحضانة، دافق القناة عن طريق سحب 0.5 مل من العازلة A من خلال القناة باستخدام حقنة. الحرص على عدم إدخال الهواء في القناة.
- بعد وظيفية، جبل الخلية تدفق على المجهر المقلوب. ربط أنبوب منفذ إلى مضخة حقنة ووضع أنبوب مدخل في أنبوب microcentrifuge تحتوي على العازلة A.
- يدويا سحب ما لا يقل عن 0.5 مل من المخزن المؤقت أ لطرد النظام ورئيس المضخة. السماح لتشغيل المضخة في 10 μL / دقيقة لمدة 5 دقيقة على الأقل لتكدير النظام.
- لإعداد الخرز (جدول المواد)، دوامة زجاجة الأسهم من الخرز والماصات 1.6 ميكرولتر من 10 ملغ / مل الخرز الأسهم في 50 ميكرولتر من العازلة A ، ثم دوامة.
- باستخدام فاصل المغناطيسي، ماصة خارج العازلة و resuspend في 50 ميكرولتر من العازلة A، ثم دوامة.
- كرر الخطوة 3.7 2x لمجموع ثلاثة يغسل. في الغسيل الأخير، إعادة التركيز في 100 ميكرولتر من العازلة A ودوامة لتحقيق تركيز نهائي من 160 ميكروغرام /مل.
- إلى تعقيد الحمض النووي والخرز، أولا إعداد 480 ميكرولتر من 0.5 درجة كلمتر المسمى الركيزة الحمض النووي في العازلة A. ثم، ماصة في 20 ميكرولتر من 160 ميكروغرام / مل تعليق الخرز، والتأكد من دوامة الخرز قبل الأنابيب. ضعه على دُورّة لمدة 3 دقائق.
- بعد 3 دقائق، على الفور تحميل في قناة بمعدل تدفق 10 ميكرولتر / دقيقة لمدة 15 دقيقة ~ أو حتى يتم ملاحظة ربط حبة كافية.
ملاحظة: لا ينبغي أن تكون الخرز معبأة بكثافة بحيث تتفاعل مع بعضها البعض على السطح (انظر قسم المناقشة). - لغسل قناة جميع الخرزات الحرة، قم بتبديل أنبوب المدخل إلى أنبوب جديد من العازلة A وتدفقه في 50 ميكرولتر/دقيقة لمدة 10 دقائق على الأقل أو حتى لا يتم ملاحظة الخرز الفضفاض.
4 - جمع البيانات وتحليلها
- للتحضير لجمع البيانات، ضع أنبوب مدخل في أنبوب microcentrifuge يحتوي على ما لا يقل عن 100 ميكرولتر من ندي (0.25-4.00 U/mL) في المخزن المؤقت A. قم بخفض المغناطيس الدائم عبر قناة التدفق، ووضع مصدر الضوء قبالة محور التصوير في الحقول المظلمة.
ملاحظة: يتم عقد اثنين من المغناطيس الأرض النادرة الحلقية، epoxied معا، 8 مم فوق السطح النشط للقناة تدفق باستخدام وظيفة البصرية cantilevered أثناء جمع البيانات. يتم استخدام مصباح الرقبة أوزة ل غير محور ضوء الدورة. - استخدام المجهر التجاري وكاميرا الفيديو وبرنامج جمع البيانات(جدول المواد). في البرنامج، انقر فوق علامة التبويب"التعرض"،ثم قم بتعيين"وقت التعرض"إلى 10 مللي ثانية. انقر على "Timelapse" علامة التبويب وتعيين "عدد الصور" إلى 600 ،"المدة"إلى 20 دقيقة ، و "الفاصل الزمني" إلى 2 ثانية. انقر فوق "تشغيل" لبدء جمع البيانات.
- على مضخة حقنة، تعيين معدل التدفق إلى 150 ميكرولتر/ دقيقة وحجم الحقن إلى 80 ميكرولتر. اضغط على "تشغيل" في 1 دقيقة في جمع البيانات. بعد الحقن، قم بإيقاف تشغيل المضخة وإغلاق الصمام لمنع تدفقها أثناء جمع البيانات.
- مرة واحدة يتم جمع البيانات، فتح برنامج تحليل الصور(جدول المواد). ضمن "ملف" علامة التبويب ، اختر "استيراد" | "تسلسل الصورة". حدد موقع ملفات الصور في القائمة المنبثقة وانقر فوق "فتح".
- تعيين عتبة عن طريق اختيار "ضبط Theshold" تحت "صورة" القائمة المنسدلة. استخدم شريط شريط التمرير لتعيين قيمة العتبة لتحديد النقاط المضيئة المطابقة للخرزات في الصورة.
- عد النقاط المضيئة في كل إطار بالنقر فوق "تحليل الجسيمات" في "تحليل" القائمة المنسدلة. انقر على"موافق"، ثم اختر"نعم"لمعالجة جميع الصور. حفظ ملف النتائج.
ملاحظة: هذا سيتم حفظ ملف بيانات يحتوي على عدد الخرز في كل إطار فيديو مسجل. - فتح برنامج تحليل البيانات(جدول المواد)واستيراد ملف النتائج بالنقر فوق "استيراد من ملف نصي" في "ملف" القائمة المنسدلة. رسم بيانات العد حبة مقابل الوقت.
- تناسب عدد من الخرز مقابل الوقت عن طريق النقر على "منحنى صالح" في "تحليل" القائمة المنسدلة. اختر المعادلة "الأس الطبيعي" وانقر على "Try Fit" | "حسنا".
ملاحظة: يجب أن تدرج فقط البيانات المسجلة بعد حقن العينة في منطقة المناسب. المعلمة تناسب في الأس من وظيفة المناسب سيكون معدل الانقسام.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
باستخدام هذه التقنية، تم قياس معدلات SSDC من NdeI لمجموعة من تركيزات البروتين (0.25-4.00 U/mL) في تركيزين مختلفين من المغنيسيوم (2 mM و 4 mM). وقد تكرر كل من هذه الشروط مرتين على الأقل، مع بضع مئات إلى 1000 DNAs مربوطة لكل تجربة. ويصف الشكل 2 التصميم التجريبي. ويبين الشكل 3 أمثلة على تفاصيل جمع البيانات وتحليلها. يوضح الشكل 4 كيف يعتمد المعدل على تركيز البروتين في تركيزين من المغنيسيوم. ويمكن ملاحظة أن تركيزات البروتين منخفضة بما فيه الكفاية، ومعدل يتناسب مع البروتين ومستقلة عن المغنيسيوم. بالنسبة لتركيزات البروتين عالية بما فيه الكفاية، يعتمد المعدل على المغنيسيوم ولكن مستقل عن تركيز البروتين.
| خطوه | وصف | درجة الحرارة (درجة مئوية) | الوقت (ق) |
| 1 | التشبع بالدن | 98 | 30 |
| 2 | تذوب | 98 | 10 |
| 3 | آنال | 60 | 30 |
| 4 | توسيع | 72 | 30 |
| 5 | تمديد النهائي | 72 | 120 |
الجدول 1: معلمات PCR. تظهر درجات الحرارة و فترات خطوات برنامج الدراجات الحرارية المستخدمة في الخطوة 2.2 من البروتوكول. يتم تكرار الخطوات تذوب، والحنّ، وتمدد (الخطوات 2 و3 و4) 30x.
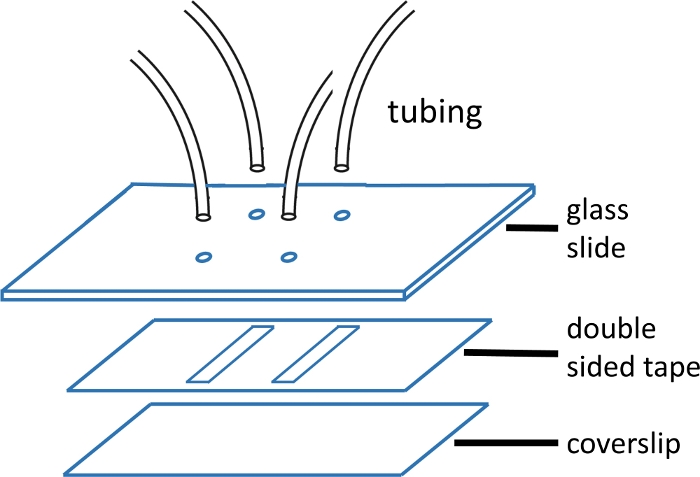
الشكل 1: بناء خلايا التدفق الدقيق.
الشريحة الزجاجية العلوية (2'× 3''، 1 مم) تم حفرها مسبقًا مع فتحات مطابقة لنمط القناة. يتم إدخال أنابيب مدخل ومخرج في الثقوب وثابتة مع الايبوكسي قبل إرفاق الشريط والزجاج غطاء. الشريط على الوجهين هو قبل قطع مع نمط القناة. الشريحة السفلية (#1 أو #1.5 غطاء الزجاج) سابقاً تنظيف باستخدام البروتوكول الموضح في النص الرئيسي. تجميعها مرة واحدة، يتم ختم حافة الزجاج غطاء مع الايبوكسي. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 2: التصميم التجريبي.
(أ)طريقة ربط الحمض النووي. يتم إرفاق الحمض النووي المزدوج (1 كيلو بايت) المسمى بالديوكسيجينين على الطرف الخامس على سطح خلية التدفق عن طريق التفاعل بين antidigoxigenin-digoxigenin. يتم إرفاق الطرف 3 من الحمض النووي، المسمى بالبيوتين، بميكروبات عبر تفاعل streptavidin-biotin. موقع انشقاق NdeI يقع في وسط الحمض النووي. (B) الإعداد التجريبي أثناء جمع البيانات يظهر المغناطيس والموقف الموضوعي. يحافظ المغناطيس الدائم على قوة صاعدة ضعيفة على الخرزة أثناء تفاعل الانقسام. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
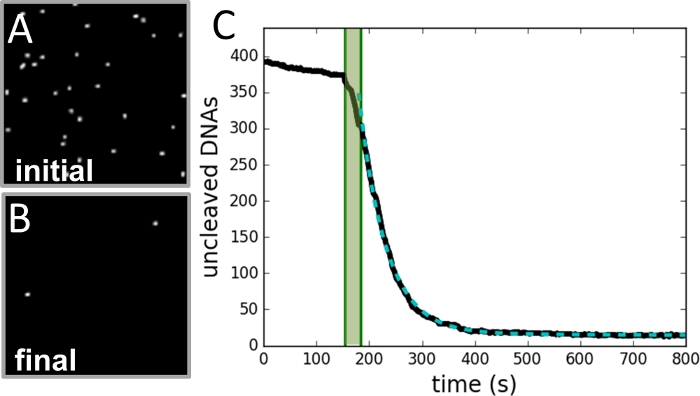
الشكل 3: مثال على جمع البيانات وتحليلها.
(أ) صورة لمنطقة الخرز التي اتخذت قبل بدء التفاعل الانقسام. (B) صورة لنفس المنطقة بعد اكتمال التفاعل. (C) رسم عدد من الخرز مقابل الوقت (منحنى أسود) على النحو المحدد من كل إطار من سجل الفيديو. المنطقة الخضراء المظللة علامات فترة حقن انزيم وغير المدرجة في صالح. وتناسب البيانات منحنى أسي واحد (منحنى أخضر متقطع)، مع ثابت اضمحلال يساوي معدل التفاعل. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 4: يعتمد انشقاق NdeI على تركيزات البروتين والمغنيسيوم.
مؤامرة من قياس SSDC معدل NdeI لمجموعة من تركيزات البروتين في تركيزين مختلفين من المغنيسيوم: 2 mM (الدوائر الزرقاء) و 4 mM (المربعات الخضراء). تمثل أشرطة الخطأ SEM. المنحنيات المتقطعة هي خطوط اتجاه ولا تمثل تناسب النظرية. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
ويمكن استخدام البروتوكول لقياس حركية أي نظام SSDC، شريطة أن يتم ملاحظة فصل الخيوط أثناء التجربة. يتأثر الكشف عن الانقسام من خلال مراقبة انفصال الخرز المربوط ، وبالتالي يمثل لحظة انفصال حبلا. تحدث كافة الخطوات السابقة قبل الكشف عن الانقسام; وهكذا، يتم تسجيل وقت العبور الإجمالي فقط.
يتم وظيفية غطاء الخلية تدفق عن طريق الامتزاز غير محددة من بروتين الأجسام المضادة إلى الزجاج النظيف. قد يؤثر الزجاج غير الكافي الذي يتم تنظيفه على ربط الجسم المضاد. في الربط، يجب أن تكون كثافة الخرز منخفضة بما فيه الكفاية بحيث لا تتفاعل الخرز. يمكن التحكم في كثافة السطح من نقاط المرفقات من قبل تركيز الأجسام المضادة أثناء وظيفية. يعتمد إجمالي عدد الخرز على حجم مجال الرؤية. في هذه الحالة، كانت بضع مئات إلى 1000 حبة مربوطة كافية للإحصاءات الجيدة وتجنب التفاعلات حبة حبة. أثناء حقن الخرز، تم رصد الربط السطحي عبر الفيديو المباشر. تم إيقاف حقن الخرز عندما كان عدد الخرز المقدر ما بين 500 و 1000 حبة.
إن أسرع معدلات الانقسام التي يمكن قياسها بدقة محدودة من خلال وقت الخلط في خلية التدفق. يتأثر وقت الخلط في خلايا تدفق اللامينار بعدة عوامل. إن الانتشار إلى السطح هو خطوة رئيسية؛ لذلك، فإن وقت الخلط يعتمد على معامل الانتشار من reactant. القص الكبير الذي يحدث في أنابيب المدخل، والذي ينقل العينة إلى قناة التدفق من خزان العينة، يمكن أن يزيد من الوقت اللازم لضمان خلط كاف في سطح التفاعل في القناة. ووجد أن وقت الخلط يمكن أن يخفض عن طريق تقليل حجم المنبع الميت وزيادة معدل التدفق. مع القطر الداخلي من 380 ميكرومتر والحد الأقصى طول 8 سم لمواهب مدخل (ومعدل تدفق 150 ميكرولتر / دقيقة)، ووجد أنه يمكن تخفيض وقت الحقن إلى ~ 20 s دون التأثير على معدل الانقسام قياس. منذ وقت الخلط يعتمد على معامل الانتشار من reactant، ينبغي أن تحدد بشكل منفصل لكل انزيم أو شق المنشط درس.
تسمح طريقة الربط بتمزق الحبل غير المحدد ، ويفترض أنه يرجع إما إلى تفكك مجمع الأجسام المضادة للديجوجينين أو إطلاق الجسم المضاد من السطح. وهذا يؤدي إلى إعادة إنتاجها معدل فقدان حبة الخلفية الحالية قبل حقن انزيم ~ 3 × 10-4 ق-1. ويمكن تصحيح هذا التأثير المنهجي إما عن طريق طرح معدل الخلفية من معدل الانقسام المقاس، أو عن طريق نمّجة الخلفية في المعادلة المناسبة. ومع ذلك، فإن معدلات الانقسام أقل من هذا الحد الأدنى ستكون أقل قياساً موثوقاً.
يمكن أن يؤدي تخميل السطح غير المثالي إلى الربط غير السليم. وهذا يؤدي إما إلى زيادة كسور من الخرز "العالقة"، التي لا تزول أثناء the التجربة، أو إلى الخرز المربوط بشكل غير صحيح، والتي تنفصل عن السطح ببطء شديد. يؤدي هذا إلى إنشاء خط أساس أعلى وربما منحدر في البيانات المعالجة. ووجد أنه مع زلات الغطاء تنظيفها بشكل صحيح وحديثا β القضيه حل الأسهم، وكانت هذه الآثار ضئيلة بالنسبة لمعظم مجموعات البيانات. بالنسبة لمجموعات البيانات العرضية التي تظهر هذا، يمكن تعديل وظيفة التركيب (لتضمين خط أساس مائل) لتصحيح هذا التأثير.
يمكن توسيع البروتوكول الحالي بعدة طرق. ويمكن إجراء مزيد من العزلة للخطوات الآلية بعد ربط الموقع المستهدف باستخدام تنسيق ما قبل الربط ، حيث يتم حقن البروتين في غياب العوامل المساعدة الأساسية. يتم اختبار هذه الفكرة عن طريق حقن NdeI في غياب المغنيسيوم. في ظل هذه الظروف ، يرتبط البروتين بموقعه المشترك ولكنه لا يشد الحمض النووي. حقن المغنيسيوم بعد هذه الخطوة ملزمة ينشط الانقسام مما أدى إلى فقدان حبة سريعة. الإعداد التجريبي يسمح أيضا للسيطرة على التشكل الحمض النووي والتوتر من خلال تغيير تكوين المغناطيس أو إضافة تدفق. تحت القوى المنخفضة في هذه التجارب، يصبح الحمض النووي ملفوفًا جزئيًا. تغيير القوات قليلا يمكن أن يكون لها تأثير كبير على التشكل من الحمض النووي. على سبيل المثال، في ظل ظروف العازلة التي يتم فيها تحديد معدل البحث المستهدف، يمكن أن يؤدي اختلاف مطابقة الحمض النووي إلى اختبار تأثير القفز على البحث عن الهدف. في ظل الظروف العازلة التي تكون فيها خطوة التحليل المائي تحد من معدل، يمكن أن تُفَدَّد القوة من تأثير توتر الحمض النووي على التحلل المائي للسندات الفوسفاتية. وتجدر الإشارة إلى أنه في ظل التكبير المنخفض المستخدم، لا يمكن ملاحظة هذه التغيرات التكتكونية. يجب تتبع الحركات الصغيرة الناتجة في وضعية الخرزة تحت التكبير العالي للتحقق من إمكانية التحكم في مطابقة الحمض النووي.
ويمكن توسيع نطاق تحليل البيانات بطرق متنوعة. يطبق هذا العمل أسلوباً بسيطاً لحساب حبة متبوعاً بتركيب منحنى لوظيفة أسية واحدة. ويمكن استخدام أساليب تستند إلى تحليل وقت الإقامة، وكذلك20. ويمكن بعد ذلك تحليل توزيعات أوقات الإقامة من DNAs الفردية عن طريق تركيب منحنى أو باستخدام تقنيات أكثر تطورا، مثل الأسلوب المعمم لحظات21.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
ولا يوجد لدى أصحاب البلاغ أي تضارب في المصالح للكشف عنه.
Acknowledgments
وقد دعم هذا العمل من قبل المؤسسة الوطنية للعلوم منحة MCB-1715317.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



