Summary
En svært parallell metode for måling av stedsspesifikk spaltning av DNA på enkeltmolekylnivå er beskrevet. Denne protokollen viser teknikken ved hjelp av restriksjonen endonuclease NdeI. Metoden kan enkelt endres for å studere enhver prosess som resulterer i stedsspesifikk DNA-spalting.
Abstract
Stedsspesifikk DNA-spalting (SSDC) er et viktig skritt i mange cellulære prosesser, og det er avgjørende for genredigering. Dette arbeidet beskriver en kinetisk analyse som kan måle SSDC i mange enkelt DNA-molekyler samtidig. Bead-tethered substrat DNAs, som hver inneholder en enkelt kopi av målsekvensen, er utarbeidet i en mikrofluidisk strømningskanal. En ekstern magnet bruker en svak kraft på paramagnetiske perler. Integriteten til opptil 1000 individuelle DNAer kan overvåkes ved å visualisere mikroperlene under darkfield-avbildning ved hjelp av et bredt felt, lavt forstørrelsesmål. Injeksjon av en begrensning endonukleær, NdeI, initierer spaltingreaksjonen. Videomikroskopi brukes til å registrere det nøyaktige øyeblikket for hver DNA-spalting ved å observere rammen der den tilknyttede perlen beveger seg opp og ut av fokusplanet til målet. Frame-by-frame perle telling kvantifiserer reaksjonen, og en eksponentiell passform bestemmer reaksjonshastigheten. Denne metoden tillater innsamling av kvantitative og statistisk signifikante data om enkeltmolekylSSDC-reaksjoner i ett enkelt eksperiment.
Introduction
Stedsspesifikk DNA-spalting (SSDC) er et viktig skritt i mange genomiske transaksjoner. Bakteriell begrensningsmodifisering (RM)1- og CRISPR2-systemer beskytter for eksempel celler mot angrep fra falider og plasmider ved å gjenkjenne og spalte utenlandsk DNA ved bestemte sekvenser. I type II RM gjenkjenner restriksjonsetdonukleer (NE) korte 4–8 basisparsekvenser (bp) via proteinkjernesyreinteraksjoner3. CRISPR-tilknyttede endonukleærer, som Cas9, binder seg til nettsteder via hybridisering av målområdet med crRNAs bundet til endonucleases4. Etableringen av stedsspesifikke dobbelt strandede pauser (DSBs) er også det første trinnet i mange DNA-rekombinasjonshendelser5. Mangfoldet av antigenbindingsområder som er opprettet av V(D)J-rekombinasjon, krever for eksempel anerkjennelse og spaltning av bestemte målområder6. Noen transposoner er kjent for å målrette spesifikke DNA-sekvenser, samt7. Ikke overraskende er mange stedsspesifikke kjerner involvert i disse prosessene, for eksempel Cas9, en viktig komponent i genredigeringsteknologier8. I tillegg er nye stedsspesifikke endonukleer (det vil si sinkfingerkjerner9 og TALENS10)også blitt konstruert for å redigere genomer.
Mange metoder har blitt brukt for å måle kinetikken til stedsspesifikk spaltning av nukleinsyrer. Disse inkluderer gelanalyse, fluorescens11,,12og sekvensering baserte metoder13. Et stort fremskritt ble oppnådd med tethering av mikroperler, noe som gjør at DSB i enkeltmolekyler av DNA kan oppdages ved bevegelse av en perle etter strandseparasjon. I disse metodene er ulike typer krefter ansatt for å sikre strandseparasjon og bevegelse av perlen etter spalting. I ett tilfelle har optiske feller blitt brukt til å måle spaltning av DNA av EcoRV14. I disse eksperimentene er målsøk målet for undersøkelsen, med betingelser optimalisert slik at nettstedsspesifikk binding er det begrensende trinnet. En ulempe med optiske feller er at bare et enkelt DNA kan observeres om gangen. I tillegg må en periodisk stor trekkraft brukes til å teste for strandseparasjonen.
En annen teknikk bruker en kombinasjon av strømning og svake magnetiske krefter til å trekke på perlen på en kontinuerligmåte 15. På denne måten måles diffusjonsbegrenset spaltning av NdeI. Metoden som brukes gjør det mulig å samtidig måle flere hundre DNAer samtidig, slik at statistisk signifikans kan nås i et enkelt eksperiment. Eksperimenter basert på magnetiske pinsett har også blitt brukt. I en slik studie ble en retroviral integrase studert ved å inkludere en DSB i innsetting oligonukleotid16. Vellykket integrasjon resulterte i inkorporering av DSB i bundet DNA og tap av den vedlagte perlen. I en lignende studie av den ATP-avhengige type III-begrensningen endonuklese EcoPI ble titalls DNAer observert i et enkelt eksperiment17. Magnetiske pinsett har fordelen av at spenning, samt DNA looping, kan kontrolleres og overvåkes under reaksjonen.
Presentert her er en svært parallell enkeltmolekylmetode for måling av SSDC-kinetikk, som utnytter nylige forbedringer i storskala tilknytning av DNAer. Denne metoden er en forbedring og utvidelse av tidligere metoder som brukes til å måleDNA-replikering 18,konturlengde på DNA19og spalting av REs15. I denne teknikken er lineære DNAer som inneholder en enkelt kopi av anerkjennelsessekvensen utarbeidet med biotin i den ene enden og digoksigenin i den andre. Biotin binder streptavidin, som er kovalent festet til en paramagnetisk mikroperle. DNA-perlekompleksene injiseres i en mikrofluidisk kanal som har blitt funksjonalisert med anti-digoksigenin FAB fragmenter. DNA-et festepunkter til overflatetilbehørspunktene via binding av digoksigeninen til de adssorberte FAB-fragmentene. Svake magnetiske krefter påført med en permanent magnet holder perlen fra å stikke ikke-spesielt til overflaten. Prøver kan injiseres raskt (<30 s) inn i strømningskanalen for å aktivere spaltingsreaksjonen. Flow er deaktivert under datainnsamling. Etter hvert som hvert DNA er spaltet, kan den nøyaktige tiden for spalting bestemmes ved å registrere rammen der perlen beveger seg opp og ut av fokusplanen for målet, og dermed forsvinner fra videoopptaket. En ramme-for-ramme antall gjenværende perler kan brukes til å kvantifisere reaksjonsfremdriften.
Nedenfor er den komplette protokollen samt eksempeldata samlet inn ved hjelp av NdeI. Som et eksempel på hvordan teknikken kan brukes, måles spaltingshastigheter for en rekke proteinkonsentrasjoner ved to forskjellige konsentrasjoner av magnesium, en essensiell metallkofaktor. Selv om denne anvendelsen av protokollen bruker NdeI, kan metoden tilpasses for bruk med en hvilken som helst stedsspesifikk kjerne ved å variere DNA-substratdesignen.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Lage strømningscellen
- Vaske dekkslips
- Plasser dekkglass i fargeglass og soniker med etanol (EtOH), deretter med 1 M KOH (i 30 min hver). For å unngå KOH-nedbør i EtOH, skyll grundig med ddH2O mellom vaskene.
- Gjenta både EtOH og KOH vasketrinn 1x for totalt fire vasker (to EtOH og to KOH). Oppbevar deslipper i ddH2O i fargeglass.
- Skjær lasting og utgangsrør (8 cm lange) med en ren barberhøvel og sett inn i hull i et rent glasssklie. Bruk PE-20 for innløp og PE-60 for utløp. Epoxy i 5 min for å sikre slangen og trimme av overflødig rør.
MERK: Glasssklier måler 2'' x 3'' x 1 mm. Hull bores par 15 mm fra hverandre. Hvert par danner hver ende av en enkelt kanal. Slangen med mindre ID brukes til innløpet, da dette reduserer oppstrøms dødvolum og dermed blandetiden som kreves (figur 1). - Linje opp og påfør precut dobbeltsidig tape med kanalmønster skåret ut over hull på glasslysbildet. Glatt ut med plast tang for å oppnå en god forsegling.
MERK: Det dobbeltsidige båndet som brukes i dette eksperimentet er 120 μm tykk, og kanalene er 2 mm brede og 15 mm lange. Kanaler kuttes ved hjelp av en knivskriver (Materials tabell). Det er mulig å få plass til opptil fire kanaler på en enkelt dekkslipp. Se Figur 1 for et bilde av flytcellen. - Etter peeling av baksiden, påfør en ren dekkslips (tørket med trykkluft) over båndet og glatt ut igjen med plast tang for en god forsegling.
- Epoxy kanten av dekkslips for å forsegle strømningscellen og la den kurere.
2. Fremstilling av merket DNA for tilknytning
- I et PCR-rør klargjør du 50 μL PCR-reaksjonsblanding som inneholder 0,02 U/μL high fidelity DNA-polymerase, 200 μM dNTPer, 0,5 μM fremover primer, 0,5 μM omvendt primer og 250 ng av M13mp18 vektor DNA.
MERK: Her ble den fremre primeren (biotin-CCAACTTAATCGCCTTGC) og omvendt primer (digoksigenin-TGACCATTAGATACATTTCGC) valgt for å forsterke en region ca. 1000 bp lang, som strekker seg fra posisjoner 6338 til 107 i det sirkulære genomet til M13mp18 DNA. Det er et enkelt NdeI-område midt i den forsterkede regionen. Den fremre primeren er 50 merket med digoksigenin, som binder anti-digoksigenin på dekkslippen. Den omvendte primeren er 50 merket med biotin, som binder de streptavidinbelagte perlene. - Sett inn PCR-røret i termocycleren og følg syklusen som vist i tabell 1.
- Rens PCR-produktet med et PCR-oppryddingssett etter produsentens protokoll.
MERK: Ved hjelp av settet som er angitt i materialtabellen,er det typiske DNA-utbyttet ~2 μg.
3. Internett-hering av DNA og perler
- Forbered 10 ml buffer A (1 M Tris-HCl [pH = 7,5], 50 mM NaCl, 2 mM MgCl2,1 mg/ml β-Casein, 1 mg/ml pluronic F-127). Degas i en vakuumdesiccator i minst 1 time.
- For å funksjonalisere strømningscellen, injiser 25 μL anti-digoksigenin FAB fragmenter (20 μg/ml) i PBS inn i strømningskanalen. Bruk gel lasting tips for å passe inn i PE-60 slangen. Inkuber ved romtemperatur (RT) i 30 min.
- Etter inkubasjon spyler du kanalen ved å trekke 0,5 ml buffer A gjennom kanalen ved hjelp av en sprøyte. Vær forsiktig så du ikke introduserer luft inn i kanalen.
- Etter funksjonalisering monterer du strømningscellen på et omvendt mikroskop. Koble utløpsrøret til en sprøytepumpe og sett innløpsrøret i et mikrocentrifugerør som inneholder buffer A.
- Trekk manuelt minst 0,5 ml buffer A for å skylle systemet og prime pumpen. La pumpen gå ved 10 μL/min i minst 5 min for å likevekte systemet.
- For å forberede perlene (Table of Materials), vortex lagerflasken med perler og pipette 1,6 μL av 10 mg / ml lagerperler i 50 μL buffer A, deretter virvel.
- Bruk en magnetisk separator, pipette ut bufferen og resuspend i 50 μL buffer A, deretter virvel.
- Gjenta trinn 3,7 2x for totalt tre vasker. For siste vask, resuspend i 100 μL buffer A og virvel for å oppnå en endelig konsentrasjon på 160 μg / ml.
- For å kompleksere DNA og perler, først forberede 480 μL av 0,5 pM merket DNA substrat i buffer A. Deretter pipette i 20 μL av 160 μg / ml perle suspensjon, sørg for å vortex perlene før pipettering. Plasser på en rotator i 3 min.
- Etter 3 min, umiddelbart laste inn i kanal med en strømningshastighet på 10 μL / min i ~ 15 min eller til tilstrekkelig perle tethering er observert.
MERK: Perler bør ikke være så tettpakket at de samhandler med hverandre på overflaten (se diskusjonsdelen). - For å vaske kanalen til alle frie perler, bytt innløpsrøret til et friskt rør av buffer A og flyt inn ved 50 μL / min i minst 10 min eller til ingen løse perler observeres.
4. Datainnsamling og analyse
- For å forberede datainnsamling, plasser innløpsrøret i et mikrocentrifugerør som inneholder minst 100 μL ndei (0,25–4,00 E/ml) i buffer A. Senk den permanente magneten over strømningskanalen, og plasser lyskilden av aksen for darkfield imaging.
MERK: To ringformede sjeldne jordmagneter, som er epoxied sammen, holdes 8 mm over den aktive overflaten av strømningskanalen ved hjelp av en cantilevered optisk post under datainnsamling. En gåse hals lampe brukes for off-axis lys kurs. - Bruk et kommersielt mikroskop, videokamera og datainnsamlingsprogramvare (Table of Materials). I programvaren klikker du kategorien "Eksponering" og setter "Eksponeringstid"til 10 ms. Klikk på "Timelapse" tab og sett "Image Count" til 600, "Varighet" til 20 min, og "Intervall" til 2 s. Klikk "Kjør" for å starte datainnsamling.
- På en sprøytepumpe setter du strømningshastigheten til 150 μL/min og injeksjonsvolum til 80 μL. Trykk på "Kjør" ved 1 min inn i datainnsamling. Etter injeksjon, slå av pumpen og lukk ventilen for å hindre flyt under datainnsamling.
- Når data er samlet inn, åpen bildeanalyse programvare (Table of Materials). Under kategorien"Fil"velger du "Importer" | "Bildesekvens". Finn bildefilene i lokalmenyen og klikk på "Åpne".
- Still inn terskelen ved å velge" Juster Theshold" under "Bilde"-nedtrekksmenyen. Bruk glidebryteren til å angi terskelverdien for å identifisere lyse flekker som tilsvarer perler i bildet.
- Telle de lyse flekkene i hver ramme ved å klikke "Analyser partikler" i "Analyser" nedtrekksmenyen. Klikk "OK", og velg deretter "Ja" for å behandle alle bilder. Lagre resultatfilen.
MERK: Dette vil lagre en datafil som inneholder antall perler i hver innspilte videoramme. - Åpne dataanalyse programvare (Table of Materials) og importere resultatfilen ved å klikke på"Importer fra Text File" i "File" --nedtrekksmenyen. Plott perletellingsdataene vs. tid.
- Monter antall perler vs. tid ved å klikke "Curve Fit" i"Analyser" nedtrekksmenyen. Velg"Natural Exponent"ligningen og klikk "Prøv Fit" | "OK".
MERK: Bare dataene som er registrert etter injeksjonen av prøven, skal inkluderes i tilpasningsområdet. Tilpasningsparameteren i eksponenten for tilpasningsfunksjonen vil være spaltingshastigheten.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Ved hjelp av denne teknikken ble SSDC-frekvensen av NdeI målt for en rekke proteinkonsentrasjoner (0,25–4,00 E/ml) ved to forskjellige konsentrasjoner av magnesium (2 mM og 4 mM). Hver av disse forholdene ble replikert minst to ganger, med noen få hundre til 1000 bundet DNAer per eksperiment. Figur 2 beskriver eksperimentell design. Figur 3 viser eksempler på datainnsamling og analysedetaljer. Figur 4 illustrerer hvordan hastigheten avhenger av proteinkonsentrasjon ved de to konsentrasjonene av magnesium. Det kan observeres at ved tilstrekkelig lave proteinkonsentrasjoner er frekvensen proporsjonal med protein og uavhengig av magnesium. For tilstrekkelig høye proteinkonsentrasjoner er frekvensen avhengig av magnesium, men uavhengig av proteinkonsentrasjon.
| Trinn | Beskrivelse | Temperatur (°C) | Tid (er) |
| 1 | Denning | 98 | 30 |
| 2 | Smelte | 98 | 10 |
| 3 | Anneal | 60 | 30 |
| 4 | Utvide | 72 | 30 |
| 5 | Siste utviding | 72 | 120 |
Tabell 1: PCR-parametere. Vist er temperaturer og varigheter av termocycler program trinnene som brukes i trinn 2.2 av protokollen. Smelte-, gløde- og forlengetrinnene (trinn 2, 3 og 4) gjentas 30x.
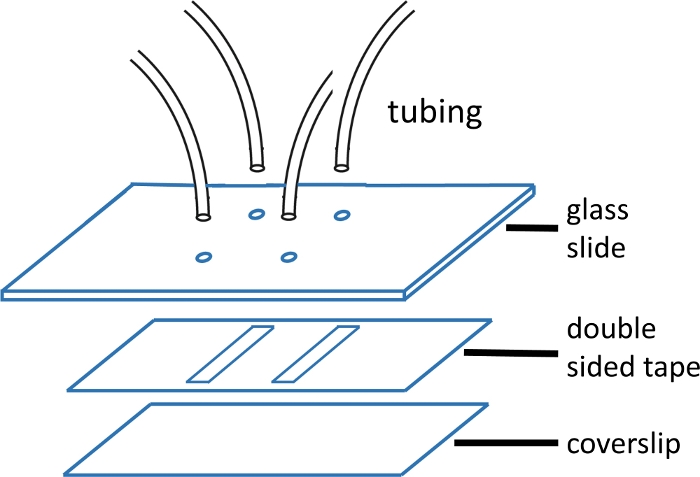
Figur 1: Mikrofluidisk strømningscellekonstruksjon.
Det øverste glasslysbildet (2'' x 3'', 1 mm tykk) er forhåndsboret med hull som samsvarer med kanalmønsteret. Innløps- og utløpsrørene settes inn i hullene og festes med epoksy før du fester tape og dekkglass. Den dobbeltsidige tapen er forhåndsskåret med kanalmønster. Det nederste lysbildet (#1 eller #1,5 omslagsglass) rengjøres tidligere ved hjelp av protokollen som er beskrevet i hovedteksten. Når det er montert, er kanten av dekkglasset forseglet med epoksy. Vennligst klikk her for å se en større versjon av denne figuren.

Figur 2: Eksperimentell design.
(A) Metode for DNA-tilknytning. Dobbelttrådet DNA (1 kbp) merket med digoksigenin på 50-enden er festet til overflaten av strømningscellen via antidigoksigenin-digokigenininteraksjon. Den 300 enden av DNA, merket med biotin, er festet til en mikroperle via streptavidin-biotin interaksjon. NdeI cleavage området ligger i sentrum av DNA. (B)Eksperimentelt oppsett under datainnsamling som viser magnet og objektiv posisjon. Den permanente magneten opprettholder en svak oppadgående kraft på perlen under spaltingsreaksjonen. Vennligst klikk her for å se en større versjon av denne figuren.
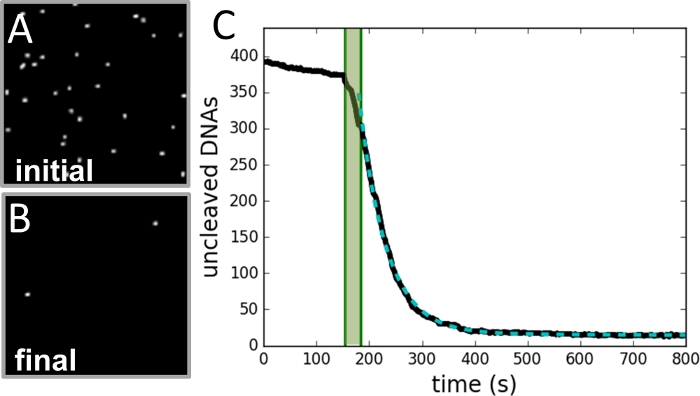
Figur 3: Eksempel på datainnsamling og analyse.
(A) Bilde av regionen av perler tatt før cleavage reaksjon er initiert. (B) Bilde av samme region etter at reaksjonen er fullført. (C) Plot av antall perler vs. tid (svart kurve) som bestemmes fra hver ramme av videoopptaket. Det skyggelagte grønne området markerer injeksjonsperioden for enzym og er ikke inkludert i passformen. Dataene passer til en enkelt eksponentiell kurve (stiplet grønn kurve), med en forfall konstant lik reaksjonshastigheten. Vennligst klikk her for å se en større versjon av denne figuren.

Figur 4: NdeI cleavage er avhengig av konsentrasjoner av protein og magnesium.
Tomten av den målte SSDC-frekvensen av NdeI for en rekke proteinkonsentrasjoner ved to forskjellige konsentrasjoner av magnesium: 2 mM (blå sirkler) og 4 mM (grønne firkanter). Feilfelt representerer SEM. De stiplede kurvene er trendlinjer og representerer ikke passer til teorien. Vennligst klikk her for å se en større versjon av denne figuren.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Protokollen kan brukes til å måle kinetikken til et hvilket som helst SSDC-system, forutsatt at strandseparasjonen observeres under eksperimentet. Påvisning av spalting påvirkes ved å observere løsrivelse av den bundet perle og markerer derfor øyeblikket for strandseparasjon. Alle foregående trinn oppstår før påvisning av spaltningen; dermed registreres bare den totale transittiden.
Strømningscelledekslipen er funksjonalisert via ikke-spesifikk adsorpsjon av antistoffprotein til det rene glasset. Utilstrekkelig rengjort glass kan påvirke bindingen av antistoffet. Ved tethering bør perletetthet være lav nok til at perler ikke samhandler. Overflatetettheten av festepunkter kan kontrolleres av konsentrasjonen av antistoff under funksjonalisering. Det totale antallet perler avhenger av størrelsen på synsfeltet. I dette tilfellet var noen få hundre til 1000 bundet perler tilstrekkelig for god statistikk og unngikk perleperle interaksjoner. Under perleinnsprøytning ble overflateteter overvåket via live video. Perleinjeksjon ble stoppet da estimert perletelling var mellom 500 og 1000 perler.
De raskeste spaltingshastighetene som kan måles nøyaktig, er begrenset av blandetiden til strømningscellen. Blandetiden i laminærstrømningsceller påvirkes av flere faktorer. Diffusjon til overflaten er et viktig skritt; Derfor avhenger blandetiden av diffusjonskoeffisienten til reaktanten. Den betydelige skjær som oppstår i inngangsslangen, som transporterer prøven til strømningskanalen fra prøvereservoaret, kan øke tiden som trengs for å sikre tilstrekkelig blanding på reaksjonsoverflaten i kanalen. Det ble funnet at blandetiden kunne reduseres ved å redusere oppstrøms dødvolum og øke strømningshastigheten. Med en indre diameter på 380 μm og maksimal lengde på 8 cm for innløpsslangen (og strømningshastighet på 150 μL/min), ble det funnet at injeksjonstiden kunne reduseres til ~20 s uten å påvirke den målte spaltingshastigheten. Siden blandetiden avhenger av diffusjonskoeffisienten til reaktanten, bør den bestemmes separat for hvert enzym eller spaltingsaktivator som studeres.
Tetheringsmetoden muliggjør ikke-spesifikk tetherbrudd, antagelig på grunn av enten dissosiasjon av digoksigenin-antistoffkomplekset eller frigjøring av antistoffet fra overflaten. Dette resulterer i en reproduserbar bakgrunn perle tapsrate tilstede før injeksjon av enzym på ~ 3 x 10-4 s-1. Denne systematiske effekten kan korrigeres enten ved å trekke bakgrunnshastigheten fra den målte spaltingshastigheten, eller ved å modellere bakgrunnen i tilpasningsligningen. Spaltingshastigheter lavere enn denne nedre grensen vil imidlertid bli mindre pålitelig målt.
Ufullkommen overflate passivisering kan føre til feil internetttering. Dette fører enten til økte fraksjoner av "fast" perler, som ikke går bort under the eksperimentet, eller til feil bundet perler, som dissosierer fra overflaten veldig sakte. Dette skaper en høyere og muligens skrånende grunnlinje i de behandlede dataene. Det ble funnet at med riktig rengjort cover slips og nylaget β-casein lagerløsning, disse effektene var minimal for de fleste datasett. For sporadiske datasett som viser dette, kan endring av tilpasningsfunksjonen (for å inkludere en skrånende opprinnelig plan) korrigere for denne effekten.
Den gjeldende protokollen kan utvides på flere måter. Ytterligere isolering av mekanistiske skritt etter binding på målområdet kan utføres ved hjelp av et prebindingsformat, der protein injiseres i fravær av essensielle kofaktorer. Denne ideen er testet ved å injisere NdeI i fravær av magnesium. Under disse forholdene binder proteinet seg til sitt kognatested, men holder ikke DNA. Injisere magnesium etter dette bindende trinnet aktiverer spaltningen som resulterer i raskt perletap. activates Det eksperimentelle oppsettet muliggjør også kontroll av DNA-konformasjon og spenning ved å variere magnetkonfigurasjonen eller legge til flyt. Under de lave kreftene i disse eksperimentene blir DNA delvis kveilet. Å endre kreftene litt kan ha en dramatisk effekt på konformasjonen av DNA. For eksempel, under bufferforhold der målsøk er hastighetsbegrensende, kan varierende samsvar av DNA teste for effekten av å hoppe på målsøket. Under bufferforhold der hydrolysetrinnet er hastighetsbegrensende, kan varierende kraft undersøke effekten av DNA-spenningen på fosfoderbindinghydrolyse. Det bør bemerkes at under den lave forstørrelsen som brukes, kan disse konformasjonsendringene ikke observeres. De resulterende små bevegelsene i perleposisjon må spores under høyere forstørrelse for å bekrefte at DNA-konformasjon kan kontrolleres.
Dataanalyse kan utvides på en rekke måter. Dette arbeidet gjelder en enkel perletellingsmetode etterfulgt av kurvemontering av en enkelt eksponentiell funksjon. Metoder basert på boligtidsanalyse kan brukes, samt20. Distribusjoner av oppholdstider for individuelle DNAer kan deretter analyseres via kurvemontering eller ved å bruke mer sofistikerte teknikker, for eksempel den generaliserte metoden for øyeblikk21.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne har ingen interessekonflikter å avsløre.
Acknowledgments
Dette arbeidet ble støttet av National Science Foundation grant MCB-1715317.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



