Summary
Une méthode très parallèle pour mesurer le clivage spécifique au site de l’ADN au niveau de la molécule unique est décrite. Ce protocole démontre la technique utilisant la restriction endonuclease NdeI. La méthode peut facilement être modifiée pour étudier n’importe quel processus qui aboutit à un clivage d’ADN spécifique au site.
Abstract
Le clivage d’ADN spécifique au site (SSDC) est une étape clé dans de nombreux processus cellulaires, et il est crucial pour l’édition de gènes. Ce travail décrit un test cinétique capable de mesurer SSDC dans de nombreuses molécules d’ADN unique simultanément. Les ADN de substrat attachés aux perles, contenant chacun une seule copie de la séquence cible, sont préparés dans un canal de flux microfluidique. Un aimant externe applique une force faible aux perles paramagnétiques. L’intégrité de jusqu’à 1 000 DNA individuels peut être surveillée en visualisant les microbilles sous l’imagerie darkfield à l’aide d’un objectif de grossissement à large champ et faible. L’injection d’une endonuclease de restriction, NdeI, initie la réaction de clivage. La microscopie vidéo est utilisée pour enregistrer le moment exact de chaque clivage d’ADN en observant le cadre dans lequel la perle associée se déplace de haut en haut du plan focal de l’objectif. Le comptage des perles image par image quantifie la réaction, et un ajustement exponentiel détermine le taux de réaction. Cette méthode permet la collecte de données quantitatives et statistiquement significatives sur les réactions de la SSDC à molécule unique dans une seule expérience.
Introduction
Le clivage d’ADN spécifique au site (SSDC) est une étape clé dans de nombreuses transactions génomiques. Par exemple, les systèmes de modification des restrictions bactériennes (RM)1 et CRISPR2 protègent les cellules contre les attaques par les phages et les plasmides en reconnaissant et en coupant l’ADN étranger à des séquences spécifiques. Dans le type II RM, les endonuclées de restriction (RE) reconnaissent les courtes séquences de paires de base (bp) de 4 à 8 par l’intermédiaire d’interactions protéine-nucléique3. Les endonucléles associés à CRISPR, tels que Cas9, se lient aux sites par hybridation du site cible avec des crRNA liés aux endonucléases4. La création de ruptures à double brin spécifiques au site (DSB) sont également la première étape dans de nombreux événements de recombinaison de l’ADN5. Par exemple, la diversité des régions liant les antigènes créées par la recombinaison V(D)J nécessite la reconnaissance et le clivage de sites cibles spécifiques6. Certains transposons sont connus pour cibler des séquences d’ADN spécifiques, ainsique 7. Il n’est pas surprenant que de nombreuses nucléases spécifiques à un site impliquées dans ces processus, comme Cas9, constituent un élément clé des technologies d’édition génétique8. En outre, de nouvelles endonuclées spécifiques au site (c.-à-d. nucléases des doigts de zinc9 et TALENS10)ont également été conçues pour modifier les génomes.
De nombreuses méthodes ont été employées pour mesurer la cinétique du clivage spécifique au site des acides nucléiques. Il s’agit notamment de l’analyse du gel, de la fluorescence11,12et des méthodes basées sur le séquençage13. Un progrès majeur a été réalisé avec l’attachement des microbilles, qui permet aux DSBs dans des molécules simples de l’ADN d’être détectés par le mouvement d’une perle après séparation de brin. Dans ces méthodes, différents types de forces sont utilisés pour assurer la séparation des brins et le mouvement de la perle post-clivage. Dans un cas, des pièges optiques ont été utilisés pour mesurer le clivage de l’ADN par EcoRV14. Dans ces expériences, la recherche de cibles est l’objectif de l’enquête, avec des conditions optimisées de sorte que la liaison spécifique au site est l’étape limitant le taux. Un inconvénient des pièges optiques est qu’un seul ADN peut être observé à la fois. En outre, une force de traction importante périodique doit être appliquée pour tester la séparation du brin.
Une autre technique utilise une combinaison de flux et de forces magnétiques faibles pour tirer sur la perle d’une manière continue15. De cette façon, la diffusion du clivage limité par NdeI est mesurée. La méthode utilisée permet la mesure simultanée de plusieurs centaines d’ARN à la fois, permettant d’atteindre une signification statistique dans une seule expérience. Des expériences basées sur des pinces magnétiques ont également été utilisées. Dans une telle étude, une intégrase rétrovirale a été étudiée en incluant un DSB dans l’oligonucléotide d’insertion16. L’intégration réussie a eu comme conséquence l’incorporation de dsb dans l’ADN attaché et la perte de la perle attachée. Dans une étude similaire de l’EcoPI de restriction de type III dépendant de l’ATP, des dizaines d’ANN ont été observés dans une seule expérience17. Les pinces magnétiques détiennent l’avantage que la tension, ainsi que les boucles d’ADN, peuvent être contrôlées et surveillées pendant la réaction.
Présenté ici est une méthode très parallèle molécule unique pour mesurer la cinétique SSDC, qui tire parti des améliorations récentes dans l’attachement à grande échelle des DNA. Cette méthode est une amélioration et une extension des méthodes précédentes utilisées pour mesurer la réplication de l’ADN18, la longueur de contour de l’ADN19, et le clivage par REs15. Dans cette technique, les ADN linéaires contenant une seule copie de la séquence de reconnaissance sont préparés avec de la biotine à une extrémité et de la digoxigenine à l’autre. La biotine lie la streptavidine, qui est covalently attachée à une microbille paramagnétique. Les complexes de perles d’ADN sont injectés dans un canal microfluidique qui a été fonctionnalisé avec des fragments de FAB anti-digoxigenin. L’ADN s’attache ensuite aux points de fixation de surface par liaison de la digoxigenine aux fragments adsorbés fab. Les forces magnétiques faibles appliquées avec un aimant permanent empêchent la perle de coller non spécifiquement à la surface. Les échantillons peuvent être injectés rapidement (<30 s) dans le canal d’écoulement pour activer la réaction de clivage. Le flux est désactivé pendant la collecte de données. Comme chaque ADN est fendu, l’heure exacte du clivage peut être déterminée en enregistrant le cadre dans lequel la perle se déplace de haut en haut du plan focal de l’objectif, disparaissant ainsi de l’enregistrement vidéo. Un nombre image par image des perles restantes peut être utilisé pour quantifier la progression de la réaction.
Présenté ci-dessous est le protocole complet ainsi que des exemples de données recueillies à l’aide de NdeI. À titre d’exemple de la façon dont la technique peut être appliquée, les taux de clivage pour une gamme de concentrations de protéines sont mesurés à deux concentrations différentes de magnésium, un cofacteur métallique essentiel. Bien que cette application du protocole utilise NdeI, la méthode peut être adaptée pour une utilisation avec n’importe quelle nucléase spécifique au site en variant la conception du substrat d’ADN.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Fabrication de la cellule d’écoulement
- Lavage des couvercles
- Placez les couvercles dans les pots de coloration et sonicate avec de l’éthanol (EtOH), puis avec 1 M KOH (pour 30 min chacun). Pour éviter les précipitations koh dans EtOH, rincer soigneusement avec ddH2O entre les lavages.
- Répétez à la fois EtOH et les étapes de lavage KOH 1x pour un nombre total de quatre lavages (deux EtOH et deux KOH). Conserver les couvercles nettoyés dans ddH2O dans des pots de coloration.
- Couper les tubes de chargement et de sortie (8 cm de long) à l’aide d’un rasoir propre et insérer dans les trous dans une lame de verre propre. Utilisez le PE-20 pour l’entrée et le PE-60 pour la prise. Époxy pendant 5 min pour fixer le tube et couper tout tube excédentaire.
REMARQUE : Les lames en verre mesurent 2'' x 3'' x 1 mm. Les trous sont percés par paires à 15 mm l’un de l’autre. Chaque paire forme l’une ou l’autre extrémité d’un seul canal. Le tube avec la plus petite pièce d’identité est utilisé pour l’entrée, car cela réduit le volume mort en amont et donc le temps de mélange requis (Figure 1). - Alignez et appliquez du ruban double face précoupé avec un motif de canal découpé sur des trous sur la glissière en verre. Lisser avec des forceps en plastique pour obtenir un bon joint.
REMARQUE : Le ruban double face utilisé dans cette expérience est de 120 μm d’épaisseur, et les canaux sont de 2 mm de large et 15 mm de long. Les canaux sont coupés à l’aide d’une imprimante à couteaux (Table des matériaux). Il est possible d’adapter jusqu’à quatre canaux sur un seul couvercle. Voir la figure 1 pour une image de la cellule d’écoulement. - Après avoir épluché le support, appliquer un couvercle propre (séché avec de l’air comprimé) sur le ruban adhésif et lisser à nouveau avec des forceps en plastique pour un bon joint.
- Époxy le bord de la couverture pour sceller la cellule d’écoulement et le laisser guérir.
2. Préparation de l’ADN étiqueté pour l’attache
- Dans un tube PCR, préparer 50 μL de mélange de réaction PCR contenant 0,02 polymérase d’ADN haute fidélité U/μL, 200 dNTPs μM, 0,5 μM d’amorce avant, 0,5 μM d’amorce inverse et 250 ng de vecteur d’ADN M13mp18.
NOTE : Ici, l’amorce avant (biotine-CCAACTTATCGCCTCCTGC) et l’amorce inverse (digoxigenin-TGACCATTAGATACATTTCGCC) ont été choisies pour amplifier une région d’environ 1 000 pb de long, allant des positions 6338 à 107 dans le génome circulaire de l’ADN M13mp18. Il y a un seul site NdeI au milieu de la région amplifiée. L’amorce avant est 50 étiqueté avec la digoxigenine, qui lie l’anti-digoxigenin sur le couvercle. L’amorce inverse est étiquetée 5o avec de la biotine, qui lie les perles enduites de streptavidine. - Insérez le tube PCR dans le thermocycler et suivez le cycle tel qu’illustré au tableau 1.
- Purifier le produit PCR avec un kit de nettoyage PCR suivant le protocole du fabricant.
REMARQUE : À l’aide du kit spécifié dans le tableau des matériaux,le rendement typique de l’ADN est d’environ 2 μg.
3. Attachement de l’ADN et des perles
- Préparer 10 mL de tampon A (1 M Tris-HCl [pH = 7,5], 50 mM NaCl, 2 mM MgCl2, 1 mg/mL β-Caséine, 1 mg/mL Pluronic F-127). Dégazer dans un desiccateur sous vide pendant au moins 1 h.
- Pour fonctionnaliser la cellule d’écoulement, injectez 25 μL de fragments d’anti-digoxigenine FAB (20 μg/mL) dans le canal d’écoulement. Utilisez des pointes de chargement de gel pour s’insérer dans le tube PE-60. Incuber à température ambiante (RT) pendant 30 min.
- Après l’incubation, rincer le canal en tirant 0,5 mL de tampon A à travers le canal à l’aide d’une seringue. Veillez à ne pas introduire l’air dans le canal.
- Après la fonctionnalisation, monter la cellule d’écoulement sur un microscope inversé. Branchez le tube de sortie à une pompe à seringues et placez le tube d’entrée dans un tube de microcentrifuge contenant le tampon A.
- Tirez manuellement au moins 0,5 mL de tampon A pour rincer le système et amorcer la pompe. Laissez la pompe fonctionner à 10 μL/min pendant au moins 5 min pour équilibrer le système.
- Pour préparer les perles (Table des Matériaux),vortex la bouteille de stock de perles et pipette 1,6 μL des perles de stock de 10 mg/mL en 50 μL de tampon A, puis vortex.
- À l’aide d’un séparateur magnétique, pipette sur le tampon et resuspend dans 50 μL de tampon A, puis vortex.
- Répétez l’étape 3.7 2x pour un total de trois lavages. Pour le dernier lavage, resuspendez dans 100 μL de tampon A et vortex pour atteindre une concentration finale de 160 μg/mL.
- Pour complexer l’ADN et les perles, préparez d’abord 480 μL de 0,5 pM de substrat d’ADN étiqueté dans le tampon A. Ensuite, la pipette dans 20 μL de suspension de perles de 160 μg/mL, en veillant à vortexer les perles avant de pipetage. Déposer sur un rotateur pendant 3 min.
- Après 3 min, chargez immédiatement dans le canal à un débit de 10 μL/min pendant ~15 min ou jusqu’à ce qu’on observe suffisamment d’attache de perles.
REMARQUE : Les perles ne doivent pas être si denses qu’elles interagissent les unes avec les autres à la surface (voir la section de discussion). - Pour laver le canal de toutes les perles libres, passer le tube d’entrée à un tube frais de tampon A et couler à 50 μL/min pendant au moins 10 min ou jusqu’à ce qu’aucune perle lâche ne soit observée.
4. Collecte et analyse de données
- Pour se préparer à la collecte de données, placez le tube d’entrée dans un tube de microcentrifuge contenant au moins 100 μL de NdeI (0,25–4,00 U/mL) dans la mémoire tampon A. Abaisser l’aimant permanent au-dessus du canal d’écoulement et positionner la source lumineuse hors de l’axe pour l’imagerie des champs sombres.
REMARQUE : Deux aimants annulaires de terres rares, epoxied ensemble, sont maintenus à 8 mm au-dessus de la surface active du canal d’écoulement à l’aide d’un poteau optique en porte-à-faux pendant la collecte des données. Une lampe à cou d’oie est utilisée pour le cours de lumière hors axe. - Utilisez un microscope commercial, une caméra vidéo et un logiciel de collecte de données(Tableau des matériaux). Dans le logiciel, cliquez sur l’onglet «Exposition» et définissez «Temps d’exposition» sur 10 ms. Cliquez sur "Timelapse" onglet et définir "Compte d’image" à 600, "Durée" à 20 min, et "Intervalle" à 2 s. Cliquez sur "Exécuter" pour démarrer la collecte de données.
- Sur une pompe à seringues, réglez le débit à 150 μL/min et le volume d’injection à 80 μL. Appuyez sur «Exécuter» à 1 min dans la collecte de données. Après l’injection, éteignez la pompe et fermez la vanne pour empêcher le débit pendant la collecte des données.
- Une fois les données collectées, logiciel d’analysed’imagesouverts ( Table des matériaux ). Sous l’onglet "Fichier« , choisissez "Importer" | «Séquence d’image« . Recherchez les fichiers d’image dans le menu contextuel et cliquez sur « Ouvrir ».
- Définissez le seuil en choisissant «Ajuster Theshold» sous le menu déroulant «Image». Utilisez la barre de curseur pour définir la valeur seuil afin d’identifier les points lumineux correspondant aux perles de l’image.
- Comptez les points lumineux de chaque image en cliquant sur "Analyser les particules" dans le menu déroulant "Analyser« . Cliquez sur "OK« , puis choisissez "Oui" pour traiter toutes les images. Enregistrez le fichier de résultats.
REMARQUE : Cela permettra d’enregistrer un fichier de données qui contient le nombre de perles dans chaque image vidéo enregistrée. - Logiciel d’analyse de données ouvertes (Tableau des matériaux) et importez le fichier de résultats en cliquant sur « Importer à partir du fichiertexte» dans le menu déroulant «Fichier». Tracez les données de comptage des perles par rapport au temps.
- Ajustez le nombre de perles par rapport au temps en cliquant sur "Curve Fit" dans le menu "Analyser" pull-down. Choisissez l’équation «Natural Exponent» et cliquez sur «Try Fit» | «OK« .
REMARQUE : Seules les données enregistrées après l’injection de l’échantillon doivent être incluses dans la région d’ajustement. Le paramètre d’ajustement dans l’exposant de la fonction d’ajustement sera le taux de clivage.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
À l’aide de cette technique, les taux de SSDC de NdeI ont été mesurés pour une gamme de concentrations de protéines (0,25–4,00 U/mL) à deux concentrations différentes de magnésium (2 mM et 4 mM). Chacune de ces conditions a été reproduite au moins deux fois, avec quelques centaines à 1 000 DNA attachés par expérience. La figure 2 décrit la conception expérimentale. La figure 3 montre des exemples de détails de collecte et d’analyse de données. La figure 4 illustre comment le taux dépend de la concentration de protéines aux deux concentrations de magnésium. On peut observer qu’à des concentrations de protéines suffisamment faibles, le taux est proportionnel aux protéines et indépendant du magnésium. Pour des concentrations de protéines suffisamment élevées, le taux dépend du magnésium mais indépendant de la concentration de protéines.
| Étape | Description | Température (°C) | Heure (s) |
| 1 | Dénaturation | 98 | 30 |
| 2 | Fondre | 98 | 10 |
| 3 | Anneal | 60 | 30 |
| 4 | Étendre | 72 | 30 |
| 5 | Extension finale | 72 | 120 |
Tableau 1 : Paramètres PCR. Les températures et les durées des étapes du programme thermocycler utilisées à l’étape 2.2 du protocole sont indiquées. Les étapes de fusion, d’anne et d’extension (étapes 2, 3 et 4) sont répétées 30x.
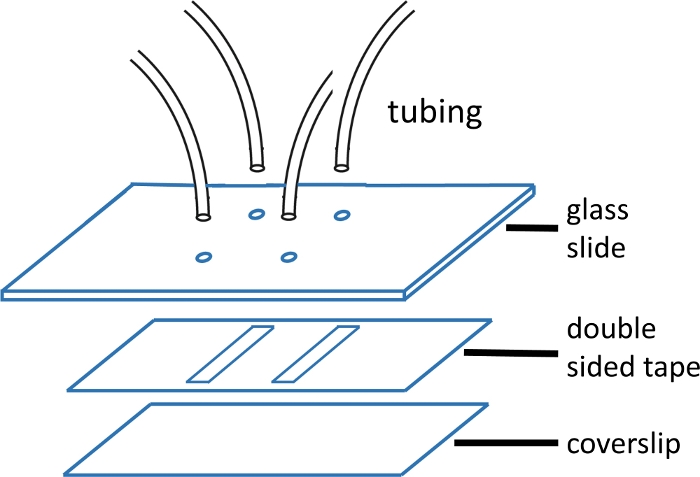
Figure 1 : Construction de cellules d’écoulement microfluidique.
La lame de verre supérieure (2'' x 3'', 1 mm d’épaisseur) est pré-forée avec des trous correspondant au motif du canal. Les tubes d’entrée et de sortie sont insérés dans les trous et fixés avec de l’époxy avant de fixer du ruban adhésif et de couvrir le verre. La bande à double face est prédécoupée avec le modèle de canal. La diapositive inférieure (#1 ou #1,5 verre de couverture) est préalablement nettoyée à l’aide du protocole décrit dans le texte principal. Une fois assemblé, le bord du verre de couverture est scellé avec de l’époxy. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Conception expérimentale.
(A) Méthode d’attachement à l’ADN. L’ADN à double brin (1 kbp) étiqueté avec la digoxigenine à l’extrémité 5 est fixé à la surface de la cellule d’écoulement par l’interaction antidigoxigenine-digoxigenin. L’extrémité de l’ADN, étiquetée avec de la biotine, est attachée à une microbille par l’interaction streptavidin-biotine. Le site de clivage NdeI est situé au centre de l’ADN. (B) Configuration expérimentale pendant la collecte de données montrant la position d’aimant et d’objectif. L’aimant permanent maintient une faible force ascendante sur la perle pendant la réaction de clivage. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
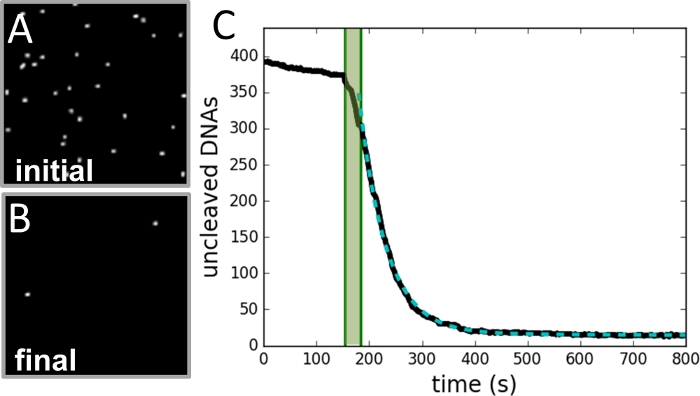
Figure 3 : Exemple de collecte et d’analyse de données.
(A) Image de la région des perles prises avant la réaction de clivage est lancée. (B) Image de la même région après la réaction est terminée. (C) Parcelle de nombre de perles vs temps (courbe noire) tel que déterminé à partir de chaque image de l’enregistrement vidéo. La zone verte ombragée marque la période d’injection d’enzymes et n’est pas incluse dans l’ajustement. Les données s’adaptent à une seule courbe exponentielle (courbe verte pointillée), avec une constante de décomposition égale au taux de réaction. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Le clivage NdeI dépend des concentrations de protéines et de magnésium.
Parcelle du taux mesuré de SSDC de NdeI pour une gamme de concentrations de protéines à deux concentrations différentes de magnésium : 2 mM (cercles bleus) et 4 mM (carrés verts). Les barres d’erreur représentent SEM. Les courbes pointillées sont des lignes de tendance et ne représentent pas des ajustements à la théorie. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Le protocole peut être utilisé pour mesurer la cinétique de n’importe quel système SSDC, à condition que la séparation des brins soit observée au cours de l’expérience. La détection du clivage est affectée par l’observation du détachement de la perle attachée et marque donc l’instant de la séparation des brins. Toutes les étapes précédentes se produisent avant la détection du clivage; ainsi, seul le temps total de transit est enregistré.
La cellule d’écoulement coverslip est fonctionnalisé par l’adsorption non spécifique de la protéine d’anticorps au verre propre. Un verre mal nettoyé peut affecter la liaison de l’anticorps. Dans l’attachement, la densité des perles doit être assez faible pour que les perles n’interagissent pas. La densité de surface des points d’attachement peut être contrôlée par la concentration d’anticorps pendant la fonctionnalisation. Le nombre total de perles dépend de la taille du champ de vision. Dans ce cas, quelques centaines à 1 000 perles attachées étaient suffisantes pour de bonnes statistiques et évitaient les interactions perle-perle. Pendant l’injection de perles, l’attachement de surface a été surveillé par vidéo en direct. L’injection de perles a été arrêtée lorsque le nombre estimé de perles était entre 500 et 1000 perles.
Les taux de clivage les plus rapides qui peuvent être mesurés avec précision sont limités par le temps de mélange de la cellule d’écoulement. Le temps de mélange dans les cellules de flux laminaires est influencé par plusieurs facteurs. La diffusion à la surface est une étape clé; par conséquent, le temps de mélange dépend du coefficient de diffusion du réactif. Le cisaillement significatif qui se produit dans le tube d’entrée, qui transporte l’échantillon au canal d’écoulement du réservoir d’échantillon, peut augmenter le temps nécessaire pour assurer un mélange adéquat à la surface de réaction dans le canal. Il a été constaté que le temps de mélange pouvait être réduit en réduisant le volume mort en amont et en augmentant le débit. D’un diamètre intérieur de 380 μm et d’une longueur maximale de 8 cm pour les tubes d’entrée (et un débit de 150 μL/min), il a été constaté que le temps d’injection pouvait être réduit à ~20 s sans affecter le taux de clivage mesuré. Puisque le temps de mélange dépend du coefficient de diffusion du réactif, il doit être déterminé séparément pour chaque enzyme ou activateur de clivage étudié.
La méthode d’attache permet une rupture d’attache non spécifique, vraisemblablement due soit à la dissociation du complexe digoxigenin-anticorps, soit à la libération de l’anticorps de la surface. Il en résulte un taux de perte de perles de fond reproductible présent avant l’injection d’enzymes de ~3 x 10-4 s-1. Cet effet systématique peut être corrigé soit en soustrayant le taux d’arrière-plan du taux de clivage mesuré, soit en modélisant l’arrière-plan de l’équation appropriée. Toutefois, les taux de clivage inférieurs à cette limite inférieure seront mesurés de façon moins fiable.
La passivation de surface imparfaite peut conduire à une mauvaise attache. Cela conduit soit à une augmentation des fractions de perles « oinnée », qui ne disparaissent pas pendant l’expérience, the ou à des perles mal attachées, qui se dissocient de la surface très lentement. improperly tethered Cela crée une ligne de base plus élevée et peut-être en pente dans les données traitées. Il a été constaté qu’avec des feuillets de couverture bien nettoyés et une solution de stock de β-caséine fraîchement préparé, ces effets étaient minimes pour la plupart des ensembles de données. Pour les ensembles de données occasionnels qui montrent cela, la modification de la fonction d’ajustement (pour inclure une ligne de base en pente) peut corriger cet effet.
Le protocole actuel peut être étendu de plusieurs façons. Un isolement supplémentaire des étapes mécanistes après la liaison du site cible peut être effectué à l’aide d’un format pré-contraignant, dans lequel la protéine est injectée en l’absence de cofacteurs essentiels. Cette idée est testée en injectant NdeI en l’absence de magnésium. Dans ces conditions, la protéine se lie à son site cognate mais ne coupe pas l’ADN. L’injection de magnésium après cette étape de liaison active le clivage entraînant une perte rapide de perles. La configuration expérimentale permet également de contrôler la conformation et la tension de l’ADN en variant la configuration de l’aimant ou en ajoutant le flux. Sous les forces basses de ces expériences, l’ADN devient partiellement enroulé. Changer légèrement les forces peut avoir un effet dramatique sur la conformation de l’ADN. Par exemple, dans des conditions tampons où la recherche cible limite le taux, la modification de la conformation de l’ADN peut tester l’effet du saut sur la recherche cible. Dans des conditions tampons où l’étape d’hydrolyse limite les taux, la force peut être variable pour sonder l’effet de la tension de l’ADN sur l’hydrolyse des liaisons phosphodiester. Il convient de noter que sous le faible grossissement utilisé, ces changements conformationnels ne peuvent pas être observés. Les petits mouvements en position de perle qui en résultent doivent être suivis sous un grossissement plus élevé pour vérifier que la conformation de l’ADN peut être contrôlée.
L’analyse des données peut être étendue de diverses façons. Ce travail applique une méthode simple de comptage des perles suivie d’un ajustement de courbe d’une seule fonction exponentielle. Des méthodes basées sur l’analyse du temps de résidence peuvent être utilisées, ainsique 20. Les distributions des temps de résidence des ADN individuels peuvent ensuite être analysées par l’intermédiaire d’un ajustement de courbe ou en utilisant des techniques plus sophistiquées, telles que la méthode généralisée des moments21.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Les auteurs n’ont pas de conflits d’intérêts à divulguer.
Acknowledgments
Ce travail a été soutenu par la Subvention de la Fondation nationale des sciences MCB-1715317.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



