Summary
단일 분자 수준에서 DNA의 부위 별 분열을 측정하는 매우 병렬적인 방법이 기재된다. 이 프로토콜은 제한 엔도니올리스 NdeI를 사용하는 기술을 보여 줍니다. 이 방법은 사이트별 DNA 분열의 결과를 초래하는 모든 과정을 연구하기 위해 쉽게 수정될 수 있다.
Abstract
사이트별 DNA 분열(SSDC)은 많은 세포 과정의 핵심 단계이며 유전자 편집에 매우 중요합니다. 이 작품은 많은 단일 DNA 분자에서 SSDC를 동시에 측정할 수 있는 운동 분석을 설명합니다. 비드 테더드 기판 DNA는 각각 표적 서열의 단일 복사본을 포함하는 미세 유체 흐름 채널에서 제조된다. 외부 자석은 파라자성 구슬에 약한 힘을 적용합니다. 최대 1,000개의 개별 DN의 무결성은 넓은 분야의 낮은 배율 목표를 사용하여 암필드 이미징 에서 마이크로비드를 시각화하여 모니터링할 수 있습니다. 제한 엔도니엔탈리스의 주입, NdeI, 분열 반응을 시작합니다. 비디오 현미경 검사는 관련 비드가 목표의 초점 평면에서 위아래로 이동하는 프레임을 관찰하여 각 DNA 분열의 정확한 순간을 기록하는 데 사용됩니다. 프레임별 비드 카운팅은 반응을 정량화하고 지수핏으로 반응 속도를 결정합니다. 이 방법을 사용하면 단일 실험에서 단일 분자 SSDC 반응에 대한 정량적 및 통계적으로 유의한 데이터를 수집할 수 있습니다.
Introduction
사이트별 DNA 분열(SSDC)은 많은 게놈 트랜잭션의 핵심 단계이다. 예를 들어, 세균 제한 수정(RM)1 및 CRISPR2 시스템은 특정 서열에서 외국 DNA를 인식하고 c떠나서 파지 및 플라스미드에 의한 공격으로부터 세포를 보호합니다. II형 RM에서, 제한 엔도니션리스(REs)는 단백질-핵산 상호작용3을통해 짧은 4-8염기쌍(bp) 서열을 인식한다. Cas9과 같은 CRISPR 관련 엔도누리스는 crRNA가 엔도니클리스4에바인딩된 대상 사이트의 혼성화를 통해 사이트에 바인딩됩니다. 사이트별 이중 좌초 휴식(DSBs)의 생성은 또한 많은 DNA 재조합 이벤트5의첫 번째 단계이다. 예를 들어, V(D)J 재조합에 의해 생성된 항원 결합 영역의 다양성은 특정 표적 부위6의인식 및 분열을 필요로 한다. 일부 트랜스포슨은 7뿐만아니라특정 DNA 서열을 표적으로 하는 것으로 알려져 있다. 당연히 Cas9와 같은 이러한 공정에 관여하는 많은 사이트 별 뉴클레아제는 유전자 편집 기술의 핵심 구성 요소입니다8. 또한, 새로운 사이트별 엔도나클레스(즉, 아연 핑거 뉴클레아제9 및 TALENS10)도게놈을 편집하도록 설계되었다.
많은 방법은 핵산의 사이트 별 분열의 운동학을 측정하기 위하여 채택되었습니다. 여기에는 겔 분석,형광(11,,12)및 시퀀싱 기반방법(13)이포함됩니다. DNA의 단일 분자에서 DSBs가 가닥 분리 후 비드의 움직임에 의해 검출될 수 있도록 하는 마이크로비드의 테더링으로 중요한 발전이 이루어졌습니다. 이러한 방법에서, 다른 유형의 힘은 비드 포스트 분열의 가닥 분리 및 움직임을 보장하기 위해 사용된다. 한 경우, 광학 트랩은 EcoRV14에의해 DNA의 분열을 측정하는 데 사용되었습니다. 이러한 실험에서 대상 검색은 조사의 목적이며, 사이트별 바인딩이 속도 제한 단계되도록 조건이 최적화되어 있습니다. 광학 트랩의 한 가지 단점은 한 번에 단일 DNA만 관찰할 수 있다는 것입니다. 또한, 가닥 분리를 테스트하기 위해 주기적인 큰 당기는 힘을 적용해야 한다.
또 다른 기술은 흐름과 약한 자기력의 조합을 사용하여 연속적인 방식으로 구드를당긴다(15). 이러한 방식으로, NdeI에 의한 확산 제한 분열이 측정된다. 이 방법을 사용하면 한 번에 수백 개의 DN을 동시에 측정할 수 있으므로 단일 실험에서 통계적 유의를 얻을 수 있습니다. 자기 핀셋을 기반으로 한 실험도 사용되었습니다. 이러한 연구에서는, 레트로바이러스 인테그라스는 삽입 올리고뉴클레오티드(16)에 DSB를 포함시킴으로써 연구되었다.16 성공적인 통합은 묶인 DNA에 DSB를 통합하고 부착된 비드의 손실을 초래했습니다. ATP 의존형 III 제한 엔토누리스 에코피의 유사한 연구에서, 수십 개의 DNA가 단일실험(17)에서관찰되었다. 자기 핀셋은 긴장뿐만 아니라 DNA 루핑이 반응 중에 제어및 모니터링될 수 있다는 이점을 보유하고 있습니다.
여기에 제시된 DNA의 대규모 테더링의 최근 개선을 활용하는 SSDC 운동학을 측정하기 위한 매우 병렬적인 단일 분자 방법이 있습니다. 본 방법은 DNA복제(18)및 DNA 19의 윤곽 길이, 및REs(15)에의한 절단을 측정하는 데 사용되는 이전 방법의 개선 및 확장이다.19 이 기술에서, 인식 서열의 단일 사본을 포함하는 선형 DNA는 한쪽 끝에 비오틴으로 제조되고 다른 쪽은 디곡시게닌을 제조한다. 비오틴은 파라마그네틱 마이크로비드에 공동으로 부착된 스트렙타비딘을 결합합니다. DNA 비드 복합체는 항 디곡시겐 FAB 단편으로 기능화된 미세유체 채널로 주입됩니다. 그런 다음 DNA는 디곡시게닌의 결합을 통해 표면 부착 점에 결합하여 흡착된 FAB 단편에 결합합니다. 영구 자석으로 가해지는 약한 자기력은 비포장물이 표면에 특별히 달라붙지 않도록 합니다. 시료는 골짜기 반응을 활성화하기 위해 유동 채널에 빠르게 주입(&30 s)할 수 있다. 데이터 수집 중에 흐름이 꺼져 있습니다. 각 DNA가 갈라질 때, 비드가 목표의 초점 계획에서 위아래로 이동하는 프레임을 기록하여 정확한 분열 시간을 결정할 수 있으므로 비디오 레코드에서 사라집니다. 남은 구슬의 프레임별 개구부는 반응 진행 상황을 정량화하는 데 사용될 수 있다.
다음은 NdeI를 사용하여 수집된 전체 프로토콜과 예제 데이터입니다. 이 기술을 어떻게 적용할 수 있는지의 예로, 단백질 농도의 범위에 대한 절단 비율은 필수 금속 보조인인 마그네슘의 두 가지 농도로 측정됩니다. 프로토콜의 이 응용은 NdeI를 사용하지만, DNA 기판 설계를 변화시킴으로써 임의의 부위 별 뉴클레아제와 함께 사용하기 위해 방법을 조정할 수 있다.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. 유동 셀 만들기
- 커버립 세척
- 커버를 염색 항아리에 넣고 에탄올 (EtOH)으로 초음파 처리한 다음 1 M KOH (각 30 분)로 초음파 처리합니다. EtOH에서 KOH 강수량을 피하기 위해 세척 사이에 ddH2O로 철저히 헹구십시오.
- EtOH와 KOH 세척 단계는 총 4개의 세척(EtOH 2개, KOH 2개)에 대해 1x로 반복합니다. 염색 항아리에 ddH2O에 청소 커버립을 저장합니다.
- 깨끗한 면도기를 사용하여 로딩 및 출구 튜브 (8cm 길이)를 자르고 깨끗한 유리 슬라이드의 구멍에 삽입하십시오. 입구에 PE-20을 사용하고 PE-60을 콘센트에 사용하십시오. 에폭시 5 분 튜브를 확보하고 여분의 튜브를 잘라.
참고: 유리 슬라이드는 2'' x 3'' x 1mm를 측정합니다. 구멍은 15mm 간격으로 쌍으로 드릴링됩니다. 각 쌍은 단일 채널의 양쪽 끝을 형성합니다. 더 작은 ID를 가진 튜브는 인렛에 사용되며, 이는 업스트림 데드 볼륨을 감소시키고 따라서 필요한 혼합시간(그림 1)을감소시킨다. - 줄을 서서 유리 슬라이드의 구멍 위로 잘라 채널 패턴이있는 미리 절단 양면 테이프를 적용합니다. 좋은 밀봉을 달성하기 위해 플라스틱 집게로 부드럽게.
참고: 이 실험에 사용된 양면 테이프는 두께120μm이며 채널의 너비는 2mm이고 길이는 15mm입니다. 채널은 칼 프린터(재료의 표)를사용하여 절단됩니다. 하나의 커버슬립에 최대 4개의 채널을 맞출 수 있습니다. 플로우 셀의 이미지는 그림 1을 참조하십시오. - 백킹을 벗겨낸 후 테이프 위에 깨끗한 커버슬립(압축 공기로 건조)을 바르고 플라스틱 집게로 다시 부드럽게 하여 좋은 밀봉을 합니다.
- 에폭시 플로우 셀을 밀봉하고 치료할 수 있도록 커버슬립에서 가장자리를 제거합니다.
2. 테더링을 위한 표지된 DNA의 준비
- PCR 튜브에서 0.02 U/μL 고충실도 DNA 폴리머라제, 200 μM dNTPs, 0.5 μM 전방 프라이머, 0.5 μM 역 프라이머 및 M13mp18 벡터 DNA의 250 ng를 포함하는 PCR 반응 믹스 50 μL을 준비한다.
참고: 여기서, 전방 프라이머(비오틴-CCAACTTAATCGCCTTGC) 및 역프라이머(디고시겐닌-TGACCAT타타타카타택트CGC)는 M13mp18 DNA의 원형 게놈에서 6338에서 107까지 의 위치를 아우르는 약 1,000bp 길이의 영역을 증폭하도록 선택되었다. 증폭 된 영역의 중간에 단일 NdeI 사이트가 있습니다. 포워드 프라이머는 5~디곡시게닌으로 표시되어 있으며, 이는 커버슬립에 항디곡시게닌을 결합합니다. 역프라이머는 5개의 비틴으로 표시되어 있으며, 이는 스트렙타비딘 코팅 구슬을 묶습니다. - 열순환기에서 PCR 튜브를 삽입하고 표 1에표시된 대로 사이클을 따릅니다.
- 제조업체의 프로토콜에 따라 PCR 정리 키트로 PCR 제품을 정화합니다.
참고: 재료 표에지정된 키트를 사용하여 일반적인 DNA 수율은 ~2 μg입니다.
3. DNA와 구슬의 테더링
- 완충 A 10mL 준비 (1 M Tris-HCl [pH = 7.5], 50 mM NaCl, 2 mM MgCl2,1 mg /mL β 카제인, 1 mg / mL 플로론 F-127). 진공 건조기에서 적어도 1 h.
- 유동 셀을 기능화하려면 PBS에 25 μL의 항디곡시겐 FAB 단편(20 μg/mL)을 유량 채널에 주입합니다. 젤 로딩 팁을 사용하여 PE-60 튜브에 맞춥니까. 실온(RT)에서 30분 동안 배양합니다.
- 인큐베이션 후 주사기를 사용하여 채널을 통해 0.5 mL의 버퍼 A를 당겨 채널을 플러시한다. 채널에 공기를 도입하지 않도록주의하십시오.
- 기능화 후, 역현미경에 유동셀을 장착한다. 콘센트 튜브를 주사기 펌프에 연결하고 입구 튜브를 완충액 A가 함유된 미세원심분리기 튜브에 넣습니다.
- 수동으로 0.5mL 이상의 버퍼 A를 당겨 시스템을 플러시하고 펌프를 프라임합니다. 펌프를 10 μL/분에서 최소 5분 동안 실행하여 시스템을 상형화하게 합니다.
- 구슬(재료의표)을준비하기 위해, 10 mg/mL 스톡 구슬의 구슬과 파이펫 1.6 μL의 스톡 병을 완충 A의 50 μL로, 다음 소용돌이로 소용돌이.
- 자기 분리기를 사용하여 버퍼를 피펫하고 버퍼 A의 50 μL에서 다시 중단한 다음 소용돌이를 반복합니다.
- 총 3개의 세차재에 대해 3.7 2배의 반복단계를 반복합니다. 마지막 세척을 위해 완충 A및 소용돌이의 100 μL에서 다시 중단하여 160 μg/mL의 최종 농도를 달성하십시오.
- DNA와 구슬을 복잡하게 하기 위해 먼저 완충A에서 0.5 pM의 DNA 기판을 480 μL을 준비한다. 그런 다음 160 μg/mL 비드 서스펜션의 20 μL에서 파이펫을 피펫팅하기 전에 구슬을 소용돌이시게 합니다. 회전에 3 분 동안 놓습니다.
- 3분 후, ~15분 동안 또는 충분한 비드 테더링이 관찰될 때까지 10 μL/min의 유속으로 즉시 채널로 로드합니다.
참고: 구슬을 너무 조밀하게 포장하여 표면에서 서로 상호 작용해서는 안 됩니다(토론 섹션 참조). - 모든 무료 구슬의 채널을 세척하려면 입구 튜브를 완충 A의 신선한 튜브로 전환하고 50 μL/min에서 최소 10분 또는 느슨한 구슬이 관찰되지 않을 때까지 흐릅니다.
4. 데이터 수집 및 분석
- 데이터 수집을 준비하기 위해 입구 튜브를 최소 100μL(0.25-4.00 U/mL)의 NdeI(0.25-4.00 U/mL)를 포함하는 마이크로센트심분리기 튜브에 넣고, 유량 채널을 통해 영구 자석을 낮추고, 암필드 이미징을 위한 광원을 축에서 분리한다.
참고: 함께 epoxied 두 개의 환상희토 자석은 데이터 수집 중에 캔틸레버 광학 포스트를 사용하여 유동 채널의 활성 표면 에서 8mm 를 유지합니다. 오프축 라이트 코스에는 거위 넥 램프가 사용됩니다. - 상용 현미경, 비디오 카메라 및 데이터 수집소프트웨어(재료 표)를 사용합니다. 소프트웨어에서"노출"탭을 클릭하고"노출 시간"을10MS로 설정합니다. "타임랩스" 탭 및 설정 "이미지 카운트" 600,"지속 시간"~20 분,"간격"~ 2 초. "실행"을클릭하여 데이터 수집을 시작합니다.
- 주사기 펌프에서 유속량을 150 μL/min으로 설정하고 사출 부피를 80RunμL로 설정합니다. 주입 후 펌프를 끄고 밸브를 닫아 데이터 수집 중에 흐름을 방지합니다.
- 데이터가 수집되면 개방형 이미지 분석소프트웨어(재료 표). "파일"탭 에서 "가져오기" | "이미지 시퀀스". 팝업 메뉴에서 이미지 파일을 찾아 "열기"를 클릭합니다.
- "이미지"풀다운 메뉴 아래에"Theshold" 조정"을선택하여 임계값을 설정합니다. 슬라이더 막대를 사용하여 임계값 값을 설정하여 이미지의 구슬에 해당하는 밝은 반점을 식별합니다.
- "분석"풀다운 메뉴에서"파티클 분석"을클릭하여 각 프레임의 밝은 반점을 계산합니다. "확인"을클릭한 다음"예"를선택하여 모든 이미지를 처리합니다. 결과 파일을 저장합니다.
참고: 이렇게 하면 녹화된 각 비디오 프레임에 구슬 수가 포함된 데이터 파일이 저장됩니다. - 데이터 분석소프트웨어(자료 표)를열고"파일"풀다운메뉴에서 "텍스트 파일에서 가져오기"를클릭하여 결과 파일을 가져옵니다. 비드 카운트 데이터를 대 시간 플롯합니다.
- "분석" 풀다운 메뉴에서"곡선 맞춤"을클릭하여Analyze구슬 수와 시간을 맞춥습니다. "자연지수" 방정식을 선택하고 클릭 "적합시도" | "괜찮아요"
참고: 샘플 주입 후 기록된 데이터만 피팅 영역에 포함해야 합니다. 피팅 함수의 지수에 맞는 매개 변수는 절단 속도가 될 것입니다.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
이 기술을 이용하여, NdeI의 SSDC 비율은 마그네슘(2mM 및 4mMMM)의 2개의 상이한 농도에서 단백질 농도(0.25-4.00 U/mL)의 범위에 대해 측정되었다. 이러한 각 조건은 실험당 수백에서 1,000개의 Tetheed DNA와 함께 적어도 두 번 복제되었습니다. 그림 2는 실험 설계를 설명합니다. 그림 3에서는 데이터 수집 및 분석 세부 정보의 예제를 보여 주습니다. 도 4는 이 비율이 마그네슘의 두 농도에서 단백질 농도에 어떻게 의존하는지 보여줍니다. 단백질 농도가 충분히 낮을 때, 비율은 단백질에 비례하고 마그네슘과 무관하다는 것을 관찰할 수 있다. 충분히 높은 단백질 농도의 경우, 속도는 마그네슘에 의존하지만 단백질 농도와 는 무관합니다.
| 단계 | 설명 | 온도(°C) | 시간 (들) |
| 1 | 변성 | 98 | 30 |
| 2 | 녹아 | 98 | 10 |
| 3 | 안닐 () | 60 | 30 |
| 4 | 확장 | 72 | 30 |
| 5 | 최종 확장 | 72 | 120 |
표 1: PCR 매개 변수. 프로토콜의 2.2 단계에서 사용되는 열순환기 프로그램 단계의 온도 및 지속 시간이 표시됩니다. 용융, 음막 및 확장 단계(단계 2, 3 및 4)는 30배 반복됩니다.
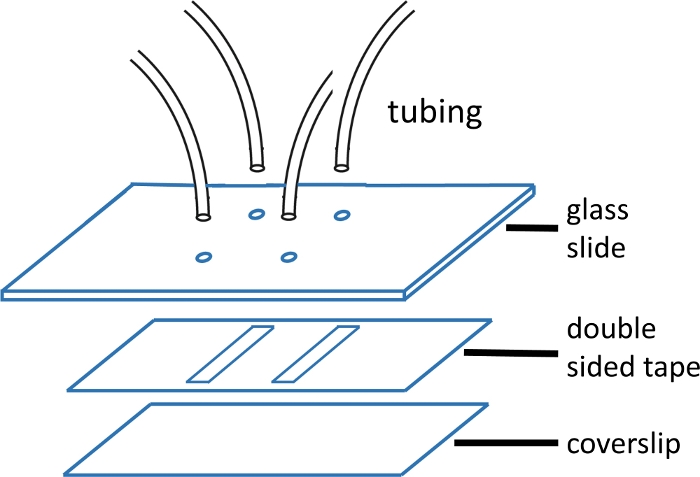
그림 1: 미세 유체 흐름 셀 구조.
상단 유리 슬라이드(2'' x 3',두께 1mm)는 채널 패턴과 일치하는 구멍으로 사전 드릴링됩니다. 입구와 콘센트 튜브는 구멍에 삽입되고 테이프와 커버 유리를 부착하기 전에 에폭시로 고정됩니다. 양면 테이프는 채널 패턴으로 미리 절단됩니다. 하단 슬라이드(#1 또는 #1.5 커버 글래스)는 이전에 주 텍스트에 설명된 프로토콜을 사용하여 세척됩니다. 조립되면 커버 유리의 가장자리는 에폭시로 밀봉됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 실험 설계.
(A)DNA 테더링 방법. 5에 디곡시게닌으로 표지된 이중 좌초 DNA(1kbp)는 항디곡시겐-디곡시겐 상호작용을 통해 유량세포의 표면에 부착된다. 비오틴으로 표지된 DNA의 3엔끝은 스트렙타비딘-비오틴 상호작용을 통해 미생물에 부착된다. NdeI 분열 부위는 DNA의 중심에 위치하고 있습니다. (B)자석과 객관적위치를 보여주는 데이터 수집 중 실험설정. 영구 자석은 분열 반응 중에 비드에 약한 상향 력을 유지합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
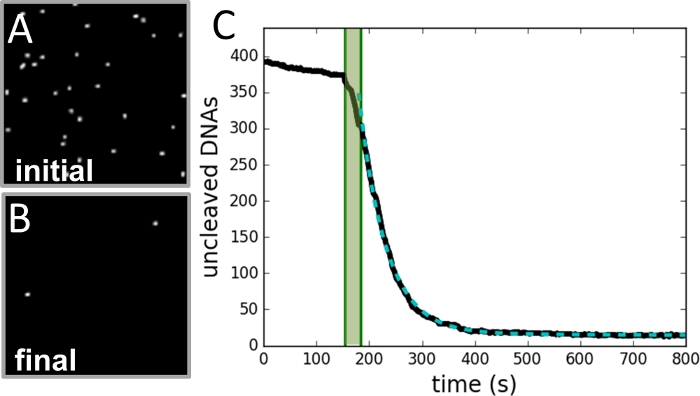
그림 3: 데이터 수집 및 분석의 예입니다.
(A)분열 반응이 시작되기 전에 찍은 구슬 의 영역의 이미지. (B)반응 완료 후 동일한 영역의 이미지. (C)비디오 레코드의 각 프레임에서 결정된 구슬 수 대 시간(검정 곡선)의 플롯. 그늘진 녹색 영역은 효소의 주입 기간을 표시하고 적합에 포함되지 않습니다. 데이터는 반응 속도와 동일한 붕괴 상수와 단일 지수 곡선(파선 된 녹색 곡선)에 적합합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

도 4: NdeI 분열은 단백질과 마그네슘의 농도에 의존한다.
측정된 SSDC 비율의 플롯은 2mM(블루 원) 및 4mM(녹색 사각형)의 두 가지 다른 농도에서 단백질 농도의 범위에 대한 값입니다. 오류 막대는 SEM을 나타냅니다. 파선 곡선은 추세선이며 이론에 적합하지 않습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
프로토콜은 실험 중에 가닥 분리가 관찰되는 경우 모든 SSDC 시스템의 역학을 측정하는 데 사용할 수 있습니다. 분열의 검출은 테더드 비드의 분리를 관찰하여 영향을 받아 가닥 분리의 순간을 표시한다. 모든 앞단계는 골짜기를 감지하기 전에 발생합니다. 따라서 총 전송 시간만 기록됩니다.
유동 세포 커버슬립은 항체 단백질을 깨끗한 유리에 비특이적으로 흡착하여 기능화된다. 불충분하게 세척된 유리는 항체의 결합에 영향을 미칠 수 있습니다. 테더링에서 비드 밀도는 충분히 낮아서 구슬이 상호 작용하지 않도록해야 합니다. 부착점의 표면 밀도는 기능화 중에 항체의 농도에 의해 조절될 수 있다. 구슬의 총 수는 시야의 크기에 따라 다릅니다. 이 경우, 수백에서 1,000개의 묶인 구슬은 좋은 통계를 위해 충분했으며 비드 비드 상호 작용을 피했습니다. 비드 주입 하는 동안, 표면 테더링은 라이브 비디오를 통해 모니터링 되었다. 비드 주사는 예상 된 비드 수가 사이 있을 때 중지 되었다 500 그리고 1,000 구슬.
정확하게 측정할 수 있는 가장 빠른 절단 속도는 유동 셀의 혼합 시간에 의해 제한됩니다. 라미나르 유량 셀의 혼합 시간은 여러 요인에 의해 영향을 받습니다. 표면에 확산하는 것이 핵심 단계입니다. 따라서 혼합 시간은 반응제의 확산 계수에 따라 달라집니다. 샘플 저장소에서 유동 채널로 샘플을 운반하는 입구 튜브에서 발생하는 중요한 전단은 채널의 반응 표면에서 적절한 혼합을 보장하는 데 필요한 시간을 증가시킬 수 있습니다. 업스트림 데드 볼륨을 줄이고 유량을 증가시킴으로써 혼합 시간을 줄일 수 있는 것으로 나타났다. 내지름 380 μm및 유입구 튜브(및 150μL/min의 유량)에 대한 최대 길이 8cm로 측정된 골짜기 속도에 영향을 주지 않으면서 주입 시간을 ~20초로 줄일 수 있는 것으로 나타났다. 혼합 시간은 반응제의 확산 계수에 따라 달라지므로, 각 효소 또는 절단 활성제에 대해 별도로 결정되어야 한다.
테더링 방법은 비특이적 밧줄 파열을 허용하며, 아마도 디곡시겐 항체 복합체의 해리 또는 표면으로부터 항체의 방출로 인한 것으로 추정된다. 이는 ~3 x10-4 s-1의효소 를 주입하기 전에 재현 가능한 배경 비드 손실률을 초래한다. 이러한 체계적인 효과는 측정된 절단 속도에서 배경 속도를 빼거나 피팅 방정식의 배경을 모델링하여 수정할 수 있습니다. 그러나, 이 낮은 바운드보다 낮은 분열 속도는 덜 안정적으로 측정될 것이다.
불완전한 표면 통과는 부적절한 테더링으로 이어질 수 있습니다. 이것은 실험 도중 멀리 가지 않는 "붙어 있는" 구슬의 증가한 분수로 이끌어 the 내거나, 표면에서 아주 천천히 해리하는 부적절하게 테더드 구슬에 연결됩니다. 이렇게 하면 처리된 데이터에서 더 높고 경사진 기준이 생성됩니다. 제대로 청소 커버 슬립과 갓 만든 β 카제인 재고 솔루션으로, 이러한 효과는 대부분의 데이터 세트에 대한 최소한의 것으로 나타났습니다. 이 것을 표시하는 가끔 데이터 집합의 경우 피팅 함수를 수정(경사진 기준선을 포함)이 이 효과를 수정할 수 있습니다.
현재 프로토콜은 여러 가지 방법으로 확장할 수 있습니다. 대상 부위 결합 후 기계론 적 단계의 추가 격리는 필수 보조 인자의 부재에서 단백질을 주입하는 사전 결합 형식을 사용하여 수행 될 수있다. 이 아이디어는 마그네슘이 없는 경우 NdeI를 주입하여 테스트합니다. 이러한 조건하에서, 단백질은 그것의 cognate 사이트에 결합하지만 DNA를 갈라지지 않습니다. 이 결합 단계 후 마그네슘을 주입하면 분열이 활성화되어 신속한 비드 손실이 발생합니다. 실험 용 설정은 또한 자석 구성을 변화하거나 흐름을 추가하여 DNA 형성 및 장력의 제어를 허용합니다. 이 실험에 있는 낮은 힘에서, DNA는 부분적으로 코일된다. 힘을 약간 변경하면 DNA의 형성에 극적인 영향을 미칠 수 있습니다. 예를 들어, 표적 검색이 속도 제한되는 완충 조건하에서 DNA의 변형을 변화시켜 대상 검색에 점프하는 효과를 테스트할 수 있습니다. 가수분해 단계가 속도 제한되는 완충 조건하에서, 힘이 변화하는 것은 인포디스터 결합 가수분해에 대한 DNA 장력의 효과를 조사할 수 있다. 사용되는 낮은 배율 하에서 이러한 형태 변화를 관찰할 수 없다는 점에 유의해야 합니다. 비드 위치에 있는 결과 작은 움직임은 DNA 형성이 통제될 수 있다는 것을 확인하기 위하여 더 높은 배율의 밑에 추적되어야 합니다.
데이터 분석은 다양한 방법으로 확장될 수 있습니다. 이 작업은 단일 지수 함수의 곡선 피팅 뒤에 간단한 구단 계수 방법을 적용합니다. 거주 시간 분석을 기반으로 하는 방법도20개뿐만아니라 사용할 수 있습니다. 개별 DNA의 거주 시간 분포는 커브 피팅을 통해 또는모멘트(21)의일반화 된 방법과 같은 보다 정교한 기술을 사용하여 분석 할 수 있습니다.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
저자는 공개 할 이해 상충이 없습니다.
Acknowledgments
이 작품은 국립 과학 재단 보조금 MCB-1715317에 의해 지원되었다.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



