Summary
Beschrieben wird eine hochparallele Methode zur Messung der standortspezifischen DNA-Spaltung auf einzelmolekülebene. Dieses Protokoll demonstriert die Technik mithilfe der Einschränkung endonuklease NdeI. Die Methode kann leicht modifiziert werden, um jeden Prozess zu untersuchen, der zu einer standortspezifischen DNA-Spaltung führt.
Abstract
Standortspezifische DNA-Spaltung (SSDC) ist ein wichtiger Schritt in vielen zellulären Prozessen, und es ist entscheidend für die Genbearbeitung. Diese Arbeit beschreibt einen kinetischen Assay, der sSDC in vielen einzelnen DNA-Molekülen gleichzeitig messen kann. Bead-tethered Substrat DNAs, die jeweils eine einzige Kopie der Zielsequenz enthalten, werden in einem mikrofluidischen Durchflusskanal hergestellt. Ein äußerer Magnet wendet eine schwache Kraft auf die paramagnetischen Perlen an. Die Integrität von bis zu 1.000 einzelnen DNAs kann überwacht werden, indem die Mikroperlen unter Dunkelfeld-Bildgebung mit einem Breitfeld-Objektiv mit geringer Vergrößerung visualisiert werden. Die Injektion einer Restriktionsendonuklease, NdeI, löst die Spaltungsreaktion aus. Die Videomikroskopie wird verwendet, um das genaue Moment jeder DNA-Spaltung aufzuzeichnen, indem der Rahmen beobachtet wird, in dem sich die zugehörige Perle auf und ab der Brennebene des Objektivs bewegt. Frame-by-Frame-Perlenzählung quantifiziert die Reaktion, und eine exponentielle Anpassung bestimmt die Reaktionsgeschwindigkeit. Diese Methode ermöglicht die Erfassung quantitativer und statistisch signifikanter Daten über Einzelne Molekül-SSDC-Reaktionen in einem einzigen Experiment.
Introduction
Standortspezifische DNA-Spaltung (SSDC) ist ein wichtiger Schritt in vielen genomischen Transaktionen. Beispielsweise schützen bakterielle Restriktionsmodifikationssysteme (RM)1 und CRISPR2 Zellen vor Angriffen durch Phagen und Plasmide, indem sie fremde DNA in bestimmten Sequenzen erkennen und spalten. In Typ II RM erkennen Restriktionsendonukleasen (REs) kurze 4–8 Basenpaarsequenzen (bp) über Protein-Nukleinsäure-Wechselwirkungen3. CRISPR-assoziierte Endonukleasen, wie Cas9, binden über die Hybridisierung des Zielstandorts an Standorte mit crRNAs, die an die Endonukleases4gebunden sind. Die Erstellung von ortsspezifischen Doppelsträngigenbrüchen (DSBs) ist auch der erste Schritt bei vielen DNA-Rekombinationsereignissen5. Beispielsweise erfordert die Vielfalt der Durch V(D)J-Rekombination geschaffenen Antigenbindungsbereiche die Erkennung und Spaltung bestimmter Zielstandorte6. Einige Transposons sind dafür bekannt, bestimmte DNA-Sequenzen anzusprechen, sowie7. Es überrascht nicht, dass viele standortspezifische Nukleasen, die an diesen Prozessen beteiligt sind, wie Cas9, eine Schlüsselkomponente von Gen-Editing-Technologiensind 8. Darüber hinaus wurden auch neuartige ortsspezifische Endonukleasen (d. h. Zinkfingernukleasen9 und TALENS10) entwickelt, um Genome zu bearbeiten.
Viele Methoden wurden eingesetzt, um die Kinetik der standortspezifischen Spaltung von Nukleinsäuren zu messen. Dazu gehören Gelanalyse, Fluoreszenz11,12und Sequenzierung basierende Methoden13. Ein großer Fortschritt wurde mit dem Ankleben von Mikroperlen erreicht, wodurch DSBs in einzelnen DNA-Molekülen durch die Bewegung einer Perle nach der Strangtrennung nachgewiesen werden können. Bei diesen Methoden werden verschiedene Arten von Kräften eingesetzt, um die Strangtrennung und Bewegung der Perle nach der Spaltung zu gewährleisten. In einem Fall wurden optische Fallen verwendet, um die Spaltung der DNA durch EcoRV14zu messen. In diesen Experimenten ist die Zielsuche das Ziel der Untersuchung, wobei die Bedingungen so optimiert sind, dass die standortspezifische Bindung der Schritt zur Preisbegrenzung ist. Ein Nachteil optischer Fallen ist, dass jeweils nur eine einzige DNA beobachtet werden kann. Darüber hinaus muss eine periodisch große Zugkraft angewendet werden, um die Strangtrennung zu testen.
Eine andere Technik verwendet eine Kombination aus Strömung und schwachen magnetischen Kräften, um die Perle kontinuierlich zu ziehen15. Auf diese Weise wird die Diffusionsbegrenzte Spaltung durch NdeI gemessen. Die angewandte Methode ermöglicht die gleichzeitige Messung von mehreren hundert DNAs auf einmal, so dass die statistische Signifikanz in einem einzigen Experiment erreicht werden kann. Experimente auf Basis magnetischer Pinzetten wurden ebenfalls verwendet. In einer solchen Studie wurde ein retrovirales Integras untersucht, indem ein DSB in das Insertionsoligonukleotid16eingebunden wurde. Eine erfolgreiche Integration führte zur Einbindung von DSB in die gebundene DNA und zum Verlust der angehängten Perle. In einer ähnlichen Studie der ATP-abhängigen Typ-III-Beschränkung Endonuklease EcoPI wurden Dutzende von DNAs in einem einzigen Experiment beobachtet17. Magnetische Pinzette haben den Vorteil, dass Spannung, sowie DNA-Looping, während der Reaktion gesteuert und überwacht werden können.
Hier wird eine hochparallele Einzelmolekülmethode zur Messung der SSDC-Kinetik vorgestellt, die die jüngsten Verbesserungen bei der großflächigen Tethering von DNAs nutzt. Diese Methode ist eine Verbesserung und Erweiterung der bisherigen Methoden zur Messung der DNA-Replikation18, Konturlänge von DNA19und Spaltung durch REs15. Bei dieser Technik werden lineare DNAs, die eine einzige Kopie der Erkennungssequenz enthalten, mit Biotin an einem Ende und Digoxigenin am anderen Ende hergestellt. Das Biotin bindet Streptavidin, das kovalent an einer paramagnetischen Mikroperle befestigt ist. Die DNA-Perlenkomplexe werden in einen mikrofluidischen Kanal injiziert, der mit Anti-Digoxigenin-FAB-Fragmenten funktionalisiert wurde. Die DNA bindet sich dann über die Bindung des Digoxigenins an die adsorbierten FAB-Fragmente an die Oberflächenbefestigungspunkte. Schwache magnetische Kräfte, die mit einem Permanentmagneten aufgebracht werden, halten die Perle davon ab, nicht spezifisch an der Oberfläche zu kleben. Proben können schnell (<30 s) in den Durchflusskanal injiziert werden, um die Spaltreaktion zu aktivieren. Der Flow wird während der Datenerfassung deaktiviert. Da jede DNA spaltet wird, kann der genaue Zeitpunkt der Spaltung bestimmt werden, indem der Rahmen aufgezeichnet wird, in dem sich die Perle nach oben und aus dem Brennplan des Objektivs bewegt und somit aus dem Videoaufgenommen verschwindet. Eine Frame-by-Frame-Anzahl der verbleibenden Perlen kann verwendet werden, um den Reaktionsfortschritt zu quantifizieren.
Im Folgenden wird das komplette Protokoll sowie Beispieldaten dargestellt, die mit NdeI gesammelt werden. Als Beispiel dafür, wie die Technik angewendet werden kann, werden die Spaltraten für eine Reihe von Proteinkonzentrationen in zwei verschiedenen Konzentrationen von Magnesium, einem essentiellen Metallkofaktor, gemessen. Obwohl diese Anwendung des Protokolls NdeI verwendet, kann das Verfahren für die Verwendung mit jeder standortspezifischen Nuklease angepasst werden, indem das DNA-Substratdesign variiert wird.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Herstellung der Durchflusszelle
- Waschen der Abdeckungen
- Deckelinen in Färbegefäße geben und mit Ethanol (EtOH) beschallen, dann mit 1 M KOH (jeweils 30 min). Um KOH-Fällung in EtOH zu vermeiden, spülen Sie gründlich mit ddH2O zwischen den Wärjahren.
- Wiederholen Sie sowohl EtOH als auch die KOH-Waschschritte 1x für insgesamt vier Waschungen (zwei EtOH und zwei KOH). Gereinigte Deckelinen in ddH2O in Färbegefäßen aufbewahren.
- Be- und Austrittsrohre (8 cm lang) mit einem sauberen Rasiermesser schneiden und in Löcher in einem sauberen Glasschlitten einfügen. Verwenden Sie PE-20 für Einlass und PE-60 für Auslass. Epoxy für 5 min, um Schläuche zu sichern und überschüssige Schläuche abzuschneiden.
HINWEIS: Glasschlitten messen 2'' x 3'' x 1 mm. Die Löcher werden paarweise im Abstand von 15 mm gebohrt. Jedes Paar bildet entweder das Ende eines einzelnen Kanals. Für den Einlass werden die Schläuche mit der kleineren ID verwendet, da dadurch das vorgeschaltete Totvolumen und damit die benötigte Mischzeit reduziert wird (Abbildung 1). - Line up und tragen vorgeschnittene doppelseitige Klebeband mit Kanalmuster über Löcher auf der Glasrutsche geschnitten. Glätten Sie mit Kunststoffzangen, um eine gute Dichtung zu erreichen.
HINWEIS: Das in diesem Experiment verwendete doppelseitige Band ist 120 m dick, und die Kanäle sind 2 mm breit und 15 mm lang. Die Kanäle werden mit einem Messerdrucker (Tabelle der Materialien) geschnitten. Es ist möglich, bis zu vier Kanäle auf einem einzigen Coverslip zu montieren. Siehe Abbildung 1 für ein Bild der Durchflusszelle. - Nach dem Abschälen der Unterlage einen sauberen Deckelschlupf (mit Druckluft getrocknet) über das Band auftragen und mit Kunststoffzangen für eine gute Abdichtung wieder glätten.
- Epoxy die Kante aus dem Deckelrutsch, um die Durchflusszelle zu versiegeln und lassen Sie es aushärten.
2. Herstellung von markierter DNA zum Tethering
- Bereiten Sie in einer PCR-Röhre einen PCR-Reaktionsmix von 50 l vor, der 0,02 U/L-DNA-Polymerase mit hoher Genauigkeit, 200 M dNTPs, 0,5 M Vorwärtsprimer, 0,5 -M-Reverseprimer und 250 ng M13mp18-Vektor-DNA enthält.
HINWEIS: Hier wurden der Vorwärtsprimer (Biotin-CCAACTTAATCGCCTTGC) und der Reverse Primer (digoxigenin-TGACCATTAGATACATTTCGC) ausgewählt, um eine Region von etwa 1.000 bp Länge zu verstärken, die sich von den Positionen 6338 bis 107 im kreisförmigen Genom der M13mp18 DNA erstreckt. Es gibt einen einzigen NdeI-Standort in der Mitte der verstärkten Region. Die Vordergrundzahl ist 5ʹ mit Digoxigenin beschriftet, das das Anti-Digoxigenin auf dem Coverslip bindet. Die Reverseprimer ist 5ʹ mit Biotin beschriftet, das die mit Streptavidin beschichteten Perlen bindet. - Setzen Sie die PCR-Röhre in thermocycler ein und folgen Sie dem Zyklus, wie in Tabelle 1dargestellt.
- Reinigen Sie das PCR-Produkt mit einem PCR-Bereinigungskit nach dem Herstellerprotokoll.
ANMERKUNG: Mit dem in der Materialtabelleangegebenen Kit beträgt die typische DNA-Ausbeute 2 g.
3. Tethering von DNA und Perlen
- 10 ml Puffer A vorbereiten (1 M Tris-HCl [pH = 7,5], 50 mM NaCl, 2 mM MgCl2, 1 mg/ml β-Casein, 1 mg/ml Pluronisch F-127). Degas in einem Vakuum-Austrocknungor für mindestens 1 h.
- Um die Durchflusszelle zu funktionalisieren, injizieren Sie 25 L Anti-Digoxigenin-FAB-Fragmente (20 g/ml) in PBS in den Durchflusskanal. Verwenden Sie Gel-Ladespitzen, um in die PE-60-Schläuche zu passen. Inkubieren bei Raumtemperatur (RT) für 30 min.
- Nach der Inkubation spülen Sie den Kanal, indem Sie 0,5 ml Puffer A mit einer Spritze durch den Kanal ziehen. Achten Sie darauf, keine Luft in den Kanal einzuführen.
- Montieren Sie die Durchflusszelle nach der Funktionalisierung auf einem invertierten Mikroskop. Schließen Sie das Auslassrohr an eine Spritzenpumpe an und legen Sie das Einlassrohr in ein Mikrozentrifugenrohr mit Puffer A.
- Ziehen Sie manuell mindestens 0,5 ml Puffer A, um das System zu spülen und die Pumpe zu grundieren. Lassen Sie die Pumpe mindestens 5 min bei 10 l/min laufen, um das System auszustatten.
- Zur Herstellung der Perlen (Tabelle der Materialien), Wirbel die Lagerflasche von Perlen und Pipette 1,6 l der 10 mg/ml Stockperlen in 50 l Puffer A, dann Wirbel.
- Mit einem magnetischen Separator, Pipette aus dem Puffer und resuspend in 50 l Puffer A, dann Wirbel.
- Wiederholen Sie Schritt 3.7 2x für insgesamt drei Wähbe. Für die letzte Wäsche in 100 l Puffer A und Wirbel wieder aufsetzen, um eine Endkonzentration von 160 g/ml zu erreichen.
- Um die DNA und Perlen zu komplexieren, bereiten Sie zunächst 480 l 0,5 pM markiertes DNA-Substrat in Puffer A vor. Dann, Pipette in 20 l von 160 g/mL Perlensuspension, um sicherzustellen, dass die Perlen vor dem Pipetieren wirbeln. Legen Sie auf einen Rotator für 3 min.
- Nach 3 min sofort in den Kanal mit einer Durchflussrate von 10 l/min für ca. 15 min oder bis eine ausreichende Perlentethering beobachtet wird.
HINWEIS: Perlen sollten nicht so dicht gepackt sein, dass sie auf der Oberfläche miteinander interagieren (siehe Diskussionsabschnitt). - Um den Kanal aller freien Perlen zu waschen, schalten Sie das Einlassrohr auf ein frisches Rohr von Puffer A und fließen sie mindestens 10 min in 50 l/min ein oder bis keine losen Perlen beobachtet werden.
4. Datenerhebung und -analyse
- Zur Vorbereitung der Datenerfassung legen Sie ein Einlassrohr in ein Mikrozentrifugenrohr, das mindestens 100 l NdeI (0,25–4,00 U/ml) enthält, in Puffer A. Senken Sie den Permanentmagneten über dem Strömungskanal, und positionieren Sie die Lichtquelle von der Achse für die Dunkelfeld-Bildgebung.
HINWEIS: Zwei ringförmige Seltenerdmagnete, die zusammen vereist sind, werden während der Datenerfassung mit einem freischwingenden optischen Pfosten 8 mm über der aktiven Oberfläche des Strömungskanals gehalten. Für den Off-Axis-Lichtkurs wird eine Gänsehalslampe verwendet. - Verwenden Sie ein kommerzielles Mikroskop, eine Videokamera und eine Datenerfassungssoftware(Materialtabelle). Klicken Sie in der Software auf die Registerkarte "Belichtung" und legen Sie "Belichtungszeit" auf 10 ms fest. Klicken Sie auf "Timelapse" Tab und setzen Sie "Image Count" auf 600, "Duration" auf 20 min, und "Interval" auf 2 s. Klicken Sie auf "Ausführen", um die Datensammlung zu starten.
- Stellen Sie an einer Spritzenpumpe den Durchfluss auf 150 l/min und das Injektionsvolumen auf 80 l ein. Drücken Sie"Run" bei 1 min in die Datenerfassung. Schalten Sie nach dem Einspritzen die Pumpe aus und schließen Sie das Ventil, um den Fluss während der Datenerfassung zu verhindern.
- Sobald Daten gesammelt sind, öffnen Sie bildanalysesoftware (Tabelle der Materialien). Wählen Sie unter der Registerkarte "Datei" "Importieren | "Bildsequenz". Suchen Sie die Bilddateien im Popup-Menü und klicken Sie auf "Öffnen".
- Legen Sie den Schwellenwert fest, indem Sie "Thesholdanpassen " unter dem Pulldown-Menü "Image" auswählen. Verwenden Sie die Schiebereglerleiste, um den Schwellenwert festzulegen, um helle Flecken zu identifizieren, die Perlen im Bild entsprechen.
- Zählen Sie die hellen Flecken in jedem Frame, indem Sie im Pulldown-Menü "Analysieren" auf "Partikel analysieren" klicken. Klicken Sie auf "OK", und wählen Sie dann "Ja", um alle Bilder zu verarbeiten. Speichern Sie die Ergebnisdatei.
HINWEIS: Dadurch wird eine Datendatei gespeichert, die die Anzahl der Perlen in jedem aufgezeichneten Videorahmen enthält. - Öffnen Sie die Datenanalysesoftware (Tabelle der Materialien) und importieren Sie die Ergebnisdatei, indem Sie im Pulldown-Menü "Datei" auf "Aus Textdateiimportieren " klicken. Zeichnen Sie die Daten zur Anzahl der Perlen im Vergleich zur Zeit.
- Passen Sie die Anzahl der Perlen vs. Zeit an, indem Sie im Pulldown-Menü "Analysieren" auf "Kurvenanpassung" klicken. Wählen Sie die "Natural Exponent" Gleichung und klicken Sie auf "Try Fit" | "OK".
HINWEIS: Nur die Daten, die nach der Injektion der Probe aufgezeichnet wurden, sollten in den Anpassungsbereich einbezogen werden. Der Anpassungsparameter im Exponenten der Fitting-Funktion ist die Dekolletérate.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Mit dieser Technik wurden die SSDC-Raten von NdeI für eine Reihe von Proteinkonzentrationen (0,25–4,00 U/ml) bei zwei verschiedenen Magnesiumkonzentrationen (2 mM und 4 mM) gemessen. Jede dieser Bedingungen wurde mindestens zweimal repliziert, mit ein paar hundert bis 1.000 gebundenen DNAs pro Experiment. Abbildung 2 beschreibt den experimentellen Entwurf. Abbildung 3 zeigt Beispiele für Datenerfassungs- und Analysedetails. Abbildung 4 zeigt, wie die Rate von der Proteinkonzentration in den beiden Magnesiumkonzentrationen abhängt. Es kann beobachtet werden, dass bei ausreichend niedrigen Proteinkonzentrationen die Rate proportional zu Protein ist und unabhängig von Magnesium ist. Bei ausreichend hohen Proteinkonzentrationen ist die Rate von Magnesium abhängig, aber unabhängig von der Proteinkonzentration.
| Schritt | Beschreibung | Temperatur (°C) | Zeit (s) |
| 1 | Denaturierung | 98 | 30 |
| 2 | Schmelzen | 98 | 10 |
| 3 | Anneal | 60 | 30 |
| 4 | Erweitern | 72 | 30 |
| 5 | Final Extend | 72 | 120 |
Tabelle 1: PCR-Parameter. Gezeigt werden Temperaturen und Dauern der Thermocycler-Programmschritte, die in Schritt 2.2 des Protokolls verwendet werden. Die Schmelz-, Neal- und Verlängerungsschritte (Schritte 2, 3 und 4) werden 30x wiederholt.
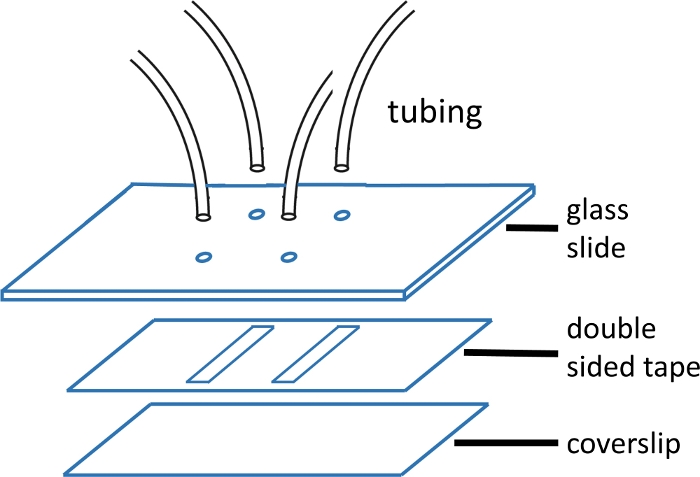
Abbildung 1: Mikrofluidische Durchflusszellenkonstruktion.
Die obere Glasrutsche (2'' x 3'', 1 mm dick) ist mit Löchern vorgebohrt, die dem Kanalmuster entsprechen. Die Ein- und Auslassrohre werden in die Löcher eingeführt und vor dem Anbringen von Klebeband und Abdeckglas mit Epoxid fixiert. Das doppelseitige Band ist mit Kanalmuster vorgeschnitten. Das untere Dia (#1 oder #1,5 Deckglas) wird zuvor mit dem im Haupttext beschriebenen Protokoll gereinigt. Nach der Montage wird die Kante des Deckglases mit Epoxid versiegelt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Experimentelles Design.
(A) Methode der DNA-Tethering. Doppelsträngige DNA (1 kbp), die mit Digoxigenin am 5ʹ Ende markiert ist, wird über Antidigoxigenin-Digoxigenin-Wechselwirkung an der Oberfläche der Durchflusszelle befestigt. Das 3ʹ Ende der DNA, mit Biotin gekennzeichnet, wird über die Streptavidin-Biotin-Wechselwirkung an einer Mikroperle befestigt. Die NdeI-Spaltungsstelle befindet sich im Zentrum der DNA. (B) Versuchsaufbau während der Datenerfassung, der Magnet und objektive Position zeigt. Der Permanentmagnet behält während der Dekolletéreaktion eine schwache Aufwärtskraft auf der Perle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
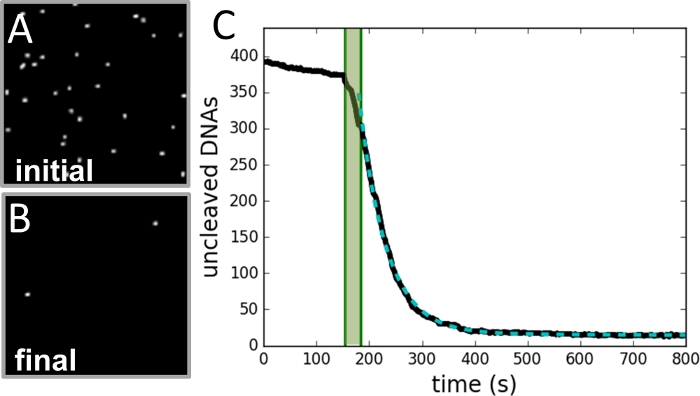
Abbildung 3: Beispiel für die Datenerfassung und -analyse.
(A) Bild der Region der Perlen, die vor Beginn der Spaltungsreaktion aufgenommen wurde. (B) Das Bild derselben Region nach der Reaktion ist abgeschlossen. (C) Diagramm der Anzahl der Perlen vs. Zeit (schwarze Kurve), wie aus jedem Frame des Videodatensatzes bestimmt. Die schattierte Grünfläche markiert die Injektionsdauer des Enzyms und ist nicht in der Passform enthalten. Die Daten passen zu einer einzelnen exponentiellen Kurve (gestrichelte grüne Kurve), wobei eine Zerfallskonstante der Reaktionsgeschwindigkeit entspricht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Die NdeI-Spaltung ist abhängig von Protein- und Magnesiumkonzentrationen.
Darstellung der gemessenen SSDC-Rate von NdeI für eine Reihe von Proteinkonzentrationen bei zwei verschiedenen Magnesiumkonzentrationen: 2 mM (blaue Kreise) und 4 mM (grüne Quadrate). Fehlerbalken stellen SEM dar. Die gestrichelten Kurven sind Trendlinien und stellen keine Passform zur Theorie dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Das Protokoll kann verwendet werden, um die Kinetik jedes SSDC-Systems zu messen, vorausgesetzt, dass die Strangtrennung während des Experiments beobachtet wird. Der Nachweis der Spaltung wird durch die Beobachtung der Ablösung der gefesselten Perle beeinflusst und markiert somit den Moment der Strangtrennung. Alle vorhergehenden Schritte erfolgen vor der Detektion der Spaltung; somit wird nur die gesamte Transitzeit erfasst.
Der Flow Cell Coverslip wird durch unspezifische Adsorption von Antikörperprotein an das saubere Glas funktionalisiert. Unzureichend gereinigtes Glas kann die Bindung des Antikörpers beeinträchtigen. Beim Tethering sollte die Perlendichte niedrig genug sein, damit Perlen nicht interagieren. Die Oberflächendichte von Befestigungspunkten kann durch die Konzentration von Antikörpern während der Funktionalisierung gesteuert werden. Die Gesamtanzahl der Perlen hängt von der Größe des Sichtfeldes ab. In diesem Fall reichten ein paar hundert bis 1.000 gefesselte Perlen für gute Statistiken aus und vermieden Perlen-Perlen-Wechselwirkungen. Während der Perleninjektion wurde das Ankleben der Oberfläche per Live-Video überwacht. Die Perleninjektion wurde gestoppt, als die geschätzte Perlenzahl zwischen 500 und 1.000 Perlen lag.
Die schnellsten Spaltraten, die genau gemessen werden können, werden durch die Mischzeit der Durchflusszelle begrenzt. Die Mischzeit in laminaren Durchflusszellen wird durch mehrere Faktoren beeinflusst. Die Diffusion an die Oberfläche ist ein wichtiger Schritt; Daher hängt die Mischzeit vom Diffusionskoeffizienten des Reaktanten ab. Die signifikante Scherung, die in den Eingangsschläuchen auftritt und die Probe vom Probenreservoir zum Strömungskanal transportiert, kann die Zeit erhöhen, die benötigt wird, um eine ausreichende Durchmischung an der Reaktionsfläche im Kanal zu gewährleisten. Es wurde festgestellt, dass die Mischzeit reduziert werden konnte, indem das vorgeschaltete Totvolumen reduziert und die Durchflussmenge erhöht wurde. Bei einem Innendurchmesser von 380 m und einer maximalen Länge von 8 cm für den Einlassschlauch (und einer Durchflussrate von 150 l/min) wurde festgestellt, dass die Injektionszeit auf 20 s reduziert werden konnte, ohne die gemessene Spaltrate zu beeinflussen. Da die Mischzeit vom Diffusionskoeffizienten des Reaktos abhängt, sollte sie für jedes untersuchte Enzym oder Dekolleté-Aktivator separat bestimmt werden.
Das Tethering-Verfahren ermöglicht einen unspezifischen Tetherbruch, vermutlich aufgrund der Dissoziation des Digoxigenin-Antikörper-Komplexes oder der Freisetzung des Antikörpers von der Oberfläche. Dies führt zu einer reproduzierbaren Hintergrund-Perlenverlustrate, die vor der Injektion des Enzyms von 3 x 10-4 s-1vorhanden ist. Dieser systematische Effekt kann entweder durch Subtraktion der Hintergrundrate von der gemessenen Spaltungsrate oder durch Modellierung des Hintergrunds in der Anpassungsgleichung korrigiert werden. Allerdings werden die Dekolletératen, die unter dieser Untergrenze liegen, weniger zuverlässig gemessen.
Eine unvollkommene Oberflächenpassivierung kann zu unsachgemäßem Ankleben führen. Dies führt entweder zu erhöhten Fraktionen von "stuck" Perlen, die während des Experiments nicht verschwinden, oder zu unsachgemäß gefesselten Perlen, die sich sehr langsam von der Oberfläche lösen. Dadurch wird eine höhere und möglicherweise schräge Basislinie in den verarbeiteten Daten erstellt. Es wurde festgestellt, dass mit ordnungsgemäß gereinigten Deckelscheinen und frisch hergestellten β-Casein-Lagerlösung diese Effekte für die meisten Datensätze minimal waren. Bei gelegentlichen Datensätzen, die dies anzeigen, kann das Ändern der Anpassungsfunktion (um eine geneigte Baseline einzuschließen) diesen Effekt korrigieren.
Das aktuelle Protokoll kann auf verschiedene Weise erweitert werden. Eine weitere Isolierung mechanistischer Schritte nach der Zielstandortbindung kann in einem Vorbindungsformat durchgeführt werden, in dem Protein in Ermangelung wesentlicher Kofaktoren injiziert wird. Diese Idee wird durch Injektion von NdeI in Abwesenheit von Magnesium getestet. Unter diesen Bedingungen bindet das Protein an seine Kognanstelle, spaltet die DNA aber nicht. Die Injektion von Magnesium nach diesem Bindungsschritt aktiviert die Spaltung, was zu einem schnellen Perlenverlust führt. Der Versuchsaufbau ermöglicht auch die Steuerung der DNA-Konformation und -spannung durch Variation der Magnetkonfiguration oder Addition des Durchflusses. Unter den geringen Kräften in diesen Experimenten wird die DNA teilweise aufgewickelt. Eine leichte Änderung der Kräfte kann dramatische Auswirkungen auf die Konformation der DNA haben. Beispielsweise kann unter Pufferbedingungen, unter denen die Zielsuche die Rate begrenzt ist, die Variation der Konformation der DNA auf die Wirkung eines Sprungs auf die Zielsuche testen. Unter Pufferbedingungen, unter denen der Hydrolyseschritt preisbegrenzend ist, kann die unterschiedliche Kraft die Wirkung der DNA-Spannung auf die Phosphodiester-Bindungshydrolyse untersuchen. Es sei darauf hingewiesen, dass bei der verwendeten geringen Vergrößerung diese Konformationsveränderungen nicht beobachtet werden können. Die resultierenden kleinen Bewegungen in Derenposition müssen unter höherer Vergrößerung verfolgt werden, um zu überprüfen, ob die DNA-Konformation gesteuert werden kann.
Die Datenanalyse kann auf verschiedene Weise erweitert werden. Diese Arbeit wendet eine einfache Perlenzählungsmethode an, gefolgt von der Kurvenanpassung einer einzelnen exponentiellen Funktion. Methoden auf der Grundlage der Verweilzeitanalyse können verwendet werden, sowie20. Die Verteilung der Verweilzeiten einzelner DNAs kann dann mittels Kurvenanpassung oder mit ausgefeilteren Techniken, wie der verallgemeinerten Methode der Momente21,analysiert werden.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Die Autoren haben keine Interessenkonflikte offenzulegen.
Acknowledgments
Diese Arbeit wurde von der National Science Foundation Stipendium MCB-1715317 unterstützt.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



