Summary
En meget parallel metode til måling af den stedspecifikke kavalergang af DNA på et enkelt molekyleniveau er beskrevet. Denne protokol demonstrerer teknikken ved hjælp af begrænsningen endonuclease NDEI. Metoden kan nemt ændres til at studere enhver proces, der resulterer i sted-specifikke DNA spaltning.
Abstract
Site-specifikke DNA spaltning (SSDC) er et vigtigt skridt i mange cellulære processer, og det er afgørende for genredigering. Dette arbejde beskriver en kinetisk analyse i stand til at måle SSDC i mange enkelt DNA-molekyler samtidigt. Perle-tøjret substrat DNAs, der hver indeholder en enkelt kopi af målsekvensen, fremstilles i en mikrofluidisk flowkanal. En ekstern magnet anvender en svag kraft til de paramagnetiske perler. Integriteten af op til 1.000 individuelle DNA'er kan overvåges ved at visualisere mikroperler under darkfield billeddannelse ved hjælp af et bredt felt, lav forstørrelse mål. Injektion af en begrænsning endonuclease, NDEI, initiere spaltning reaktion. Videomikroskopi bruges til at registrere det nøjagtige tidspunkt for hver DNA-spaltning ved at observere den ramme, hvor den tilknyttede perle bevæger sig op og ud af målets brændplan. Frame-by-frame perletælling kvantificerer reaktionen, og en eksponentiel pasform bestemmer reaktionshastigheden. Denne metode gør det muligt at indsamle kvantitative og statistisk signifikante data om enkeltmolekyleS SSDC-reaktioner i et enkelt eksperiment.
Introduction
Site-specifikke DNA spaltning (SSDC) er et vigtigt skridt i mange genomiske transaktioner. For eksempel beskytter bakteriel begrænsningsændring (RM)1- og CRISPR2-systemer celler mod angreb fra fager og plasmider ved at genkende og kløve fremmed DNA ved specifikke sekvenser. I type II RM genkender restriktionsendonukleaser (REs) korte 4-8 basisparsekvenser (bp) via protein-nukleinsyreinteraktioner3. CRISPR-associerede endonucleaser, såsom Cas9, binder sig til websteder via hybridisering af målwebstedet med crRNAs bundet til endonucleas4. Oprettelsen af stedsspecifikke dobbeltstrengede pauser (DSB'er) er også det første skridt i mange DNA-rekombinationshændelser5. F.eks. kræver mangfoldigheden af antigenbindingsregioner, der er oprettet ved hjælp af rekombination af V(D)J), anerkendelse og spaltning af specifikke målområder6. Nogle transposons er kendt for at målrette specifikke DNA-sekvenser, samt7. Ikke overraskende, mange site-specifikke nukleaser involveret i disse processer, såsom Cas9, er en vigtig komponent i genredigering teknologier8. Desuden er nye stedspecifikke endonucleaser (dvs. zinkfinger nukleaser9 og TALENS10) også blevet udviklet til at redigere genomer.
Mange metoder er blevet anvendt til at måle kinetik af stedsspecifikke spaltning af nukleinsyrer. Disse omfatter gelanalyse, fluorescens11,12og sekvenseringsbaserede metoder13. En stor fremgang blev opnået med tøjring af mikroperler, som gør det muligt DSBs i enkelte molekyler af DNA, der skal påvises ved bevægelse af en perle efter streng adskillelse. I disse metoder anvendes forskellige typer kræfter for at sikre strengadskillelse og bevægelse af perlen efter spaltningen. I et tilfælde er optiske fælder blevet brugt til at måle kavalergang af DNA ved EcoRV14. I disse eksperimenter er målsøgning formålet med undersøgelsen med betingelser, der er optimeret, så stedsspecifik binding er det hastighedsbegrænsende trin. En ulempe ved optiske fælder er, at kun en enkelt DNA kan observeres på et tidspunkt. Derudover skal der anvendes en periodisk stor trækkraft til at teste for strengadskillelsen.
En anden teknik bruger en kombination af flow og svage magnetiske kræfter til at trække på perle på en kontinuerlig måde15. På denne måde måles diffusion begrænset spaltning med NDEI. Den anvendte metode gør det muligt at foretage samtidig måling af flere hundrede DNA'er på én gang, hvilket giver mulighed for statistisk signifikans i et enkelt eksperiment. Der er også anvendt forsøg baseret på magnetiske pincet. I en sådan undersøgelse blev en retroviral integrase undersøgt ved at inkludere en DSB i indsættelsen oligonukleotid16. En vellykket integration resulterede i, at DSB blev indlemmret i det tøjlede DNA og tabet af den tilhørende perle. I en lignende undersøgelse af den ATP-afhængige type III-begrænsning endonuclease EcoPI blev der observeret snesevis af DNA'er i et enkelt eksperiment17. Magnetiske pincet har den fordel, at spændinger, samt DNA looping, kan kontrolleres og overvåges under reaktionen.
Præsenteret her er en meget parallel enkelt molekyle metode til måling af SSDC kinetik, som udnytter de seneste forbedringer i storstilet tøjring af DNAs. Denne metode er en forbedring og udvidelse af tidligere metoder , der anvendes til at måle DNA replikation18, konturlængden af DNA19og spaltning af REs15. I denne teknik fremstilles lineære DNA'er, der indeholder en enkelt kopi af genkendelsessekvensen, med biotin i den ene ende og digoxigenin i den anden. Den biotin binder streptavidin, som er kovalent knyttet til en paramagnetisk mikroperle. DNA-perlekomplekserne sprøjtes ind i en mikrofluidic kanal, der er blevet funktionaliseret med anti-digoxigenin FAB fragmenter. DNA'et binder derefter til overfladen vedhæftet fil punkter via binding af digoxigenin til adsorbed FAB fragmenter. Svage magnetiske kræfter påføres med en permanent magnet holde perle fra stikning ikke-specifikt til overfladen. Prøver kan hurtigt (<30 s) sprøjtes ind i flowkanalen for at aktivere spaltningsreaktionen. Flow er slået fra under dataindsamlingen. Da hvert DNA er kløvet, kan det nøjagtige tidspunkt for spaltning bestemmes ved at registrere den ramme, hvor iruden bevæger sig op og ud af målplanen for målet, og dermed forsvinder fra videooptagelsen. En frame-by-frame antal resterende perler kan bruges til at kvantificere reaktionfremdriften.
Præsenteret nedenfor er den komplette protokol samt eksempel data indsamlet ved hjælp af NDEI. Som et eksempel på, hvordan teknikken kan anvendes, måles spaltningshastigheden for en række proteinkoncentrationer ved to forskellige koncentrationer af magnesium, en essentiel metal cofaktor. Selv om denne anvendelse af protokollen bruger NDEI, kan metoden tilpasses til brug med enhver stedsspecifik nuklease ved at variere DNA-substratdesignet.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Sådan får du flowcellen
- Vask af dækslæberne
- Placer coverslips i farvning krukker og sonikere med ethanol (EtOH), derefter med 1 M KOH (i 30 min hver). For at undgå KOH-udfældning i EtOH skylles grundigt med ddH2O mellem vaskene.
- Gentag både EtOH og KOH vaske trin 1x for et samlet antal af fire vasker (to EtOH og to KOH). Opbevar rensede dækslip i ddH2O i farvningsglas.
- Skær læsse- og udgangsrør (8 cm lange) med en ren barbermaskine og sæt dem ind i huller i et rent glasskred. Brug PE-20 til indløb og PE-60 til udløb. Epoxy i 5 min for at sikre slange og trimme eventuelle overskydende slanger.
BEMÆRK: Glasdias måler 2'' x 3'' x 1 mm. Huller bores parvis med 15 mm fra hinanden. Hvert par danner hver ende af en enkelt kanal. Slangen med det mindre ID anvendes til indløbet, da dette reducerer opstrømsdøde volumen og dermed den nødvendige blandingstid (Figur 1). - Line up og anvende precut dobbeltsidet tape med kanal mønster skåret ud over huller på glasset dias. Glat ud med plastfæcesser for at opnå en god forsegling.
BEMÆRK: Den dobbeltsidede tape, der anvendes i dette eksperiment, er 120 μm tyk, og kanalerne er 2 mm brede og 15 mm lange. Kanaler skæres ved hjælp af en kniv printer( Tabel af Materialer). Det er muligt at montere op til fire kanaler på en enkelt coverslip. Se Figur 1 for et billede af flowcellen. - Efter peeling af bagside, anvende en ren coverslip (tørret med komprimeret luft) over båndet og glat ud igen med plastfæcefæcet for en god forsegling.
- Epoxy kanten fra dækslæbet for at forsegle flowcellen og lad den hærde.
2. Fremstilling af mærket DNA til tøjring
- I et PCR-rør forberedes 50 μL PCR-reaktionsblanding indeholdende 0,02 U/μL high fidelity DNA polymerase, 200 μM dNTPs, 0,5 μM fremadrettet primer, 0,5 μM omvendt primer og 250 ng M13mp18 DNA-vektor.
BEMÆRK: Her blev den forreste primer (biotin-CCAACTTAATCGCCTGC) og omvendt primer (digoxigenin-TGACCATTAGATACATTTCGC) valgt til at forstærke en region ca. 1.000 bp lang, der spænder fra position 6338 til 107 i det cirkulære genom af M13mp18 DNA. Der er et enkelt NDEI-sted midt i den forstærkede region. Den forreste primer er 5ʹ mærket med digoxigenin, som binder anti-digoxigenin på coverslip. Den omvendte primer 5ʹ mærket med biotin, som binder de streptavidin-belagte perler. - Sæt PCR-røret i termocykret, og følg cyklussen som vist i tabel 1.
- Rens PCR-produktet med et PCR-oprydningssæt efter producentens protokol.
BEMÆRK: Ved hjælp af det sæt, der er angivet i materialetabellen, er det typiske DNA-udbytte ~2 μg.
3. Tøjring af DNA og perler
- Der klargør 10 ml buffer A (1 M Tris-HCl [pH = 7,5], 50 mM NaCl, 2 mM MgCl2,1 mg/ml β-Casein, 1 mg/ml Pluronic F-127). Degas i vakuum ekssiccator i mindst 1 time.
- For at fungere flowcelle skal der injiceres 25 μL anti-digoxigenin FAB-fragmenter (20 μg/ml) i PBS i flowkanalen. Brug gellæssespidser til at passe ind i PE-60-slangen. Inkuber ved stuetemperatur (RT) i 30 min.
- Efter inkubation skylles kanalen ved at trække 0,5 ml buffer A gennem kanalen ved hjælp af en sprøjte. Pas på ikke at indføre luft i kanalen.
- Efter funktionalisering monteres flowcellen på et omvendt mikroskop. Sæt udløbsrøret på en sprøjtepumpe, og sæt indsugningsrøret i et mikrocentrifugerør, der indeholder buffer A.
- Træk manuelt mindst 0,5 ml buffer A for at skylle systemet og prime pumpen. Lad pumpen køre ved 10 μL/min i mindst 5 min. for at bringe systemet i ligevægt.
- For at forberede perlerne (Tabel af Materialer),vortex bestanden flaske perler og pipette 1,6 μL af de 10 mg / ml lager perler i 50 μL buffer A, så vortex.
- Ved hjælp af en magnetisk separator, pipette ud buffer og resuspendret i 50 μL buffer A, derefter vortex.
- Gentag trin 3,7 2x for i alt tre vaske. Ved den sidste vask skal der resuspendeges i 100 μL buffer A og vortex for at opnå en endelig koncentration på 160 μg/ml.
- For at komplekse DNA og perler, først forberede 480 μL af 0,5 pM mærket DNA substrat i buffer A. Derefter pipette i 20 μL af 160 μg / ml perle suspension, og sørg for at vortex perlerne før pipettering. Placer på en rotator i 3 min.
- Efter 3 min. indlad straks i kanalen ved en strømningshastighed på 10 μL/min. i ~15 min. eller indtil der observeres tilstrækkelig perlesting.
BEMÆRK: Perler bør ikke være så tæt pakket, at de interagerer med hinanden på overfladen (se diskussionsafsnit). - For at vaske kanalen for alle frie perler, skal du skifte indsugningsrøret til et frisk rør af buffer A og flyde ind ved 50 μL/min i mindst 10 min. eller indtil der ikke observeres løse perler.
4. Dataindsamling og -analyse
- For at forberede dataindsamlingen skal indsugningsrøret anbringes i et mikrocentrifugerør, der indeholder mindst 100 μL NDEI (0,25-4,00 U/ml) i buffer A. Sænk den permanente magnet over flowkanalen, og placer lyskilden fra aksen til darkfield-billeddannelse.
BEMÆRK: To ringformede sjældne jordarters magneter, epoxied sammen, holdes 8 mm over den aktive overflade af flowkanalen ved hjælp af en udkragede optisk post under dataindsamling. En gåsehalslampe bruges til off-axis lysbanen. - Brug et kommercielt mikroskop, videokamera og dataindsamlingssoftware (Tabel over materialer). Klik på fanen "Eksponering" i softwaren, og angiv "Eksponeringstid "til 10 ms. Klik på "Timelapse" fane og sæt "Billedoptælling" til 600, "Varighed" til 20 min og "Interval" til 2 s. Klik på "Kør" for at starte dataindsamlingen.
- På en sprøjtepumpe indstilles strømningshastigheden til 150 μL/min og injektionsvolumen til 80 μL. Tryk på "Kør" ved 1 minut i dataindsamling. Efter injektionen skal du slukke for pumpen og lukke ventilen for at forhindre flow under dataindsamling.
- Når data er indsamlet, åbne billede analyse software (Tabel over materialer). Under fanen "Filer" skal du vælge "Importér" | "Billedsekvens". Find billedfilerne i pop op-menuen, og klik på "Åbn".
- Angiv tærsklen ved at vælge "Juster Theshold" underrullemenuen" Image " . Brug skyderen til at indstille tærskelværdien for at identificere lyse pletter, der svarer til perler i billedet.
- Tæl de lyse pletter i hver ramme ved at klikke på "Analyze Particles" i "Analyze" pull-down-menuen. Klik på "OK", og vælg derefter "Ja" for at behandle alle billeder. Gem resultatfilen.
BEMÆRK: Dette vil gemme en datafil, der indeholder antallet af perler i hver optaget videoramme. - Åbn dataanalysesoftwaren (Tabel over Materialer), og importer resultatfilen ved at klikke på " Importer fratekstfil" i rullemenuen "File" . Afbild perletællingsdata vs. tid.
- Sæt antallet af perler i forhold til tiden ved at klikke på "Curve Fit" i "Analyze" pull-down-menuen. Vælg ligningen "Natural Exponent", og klik på "Prøv Fit" | "OK".
BEMÆRK: Kun de data, der registreres efter injektionen af prøven, bør indgå i monteringsområdet. Fit-parameteren i eksponenten for monteringsfunktionen vil være spaltningshastigheden.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Ved hjælp af denne teknik blev SSDC-satserne for NDEI målt for en række proteinkoncentrationer (0,25-4,00 U/ml) ved to forskellige koncentrationer af magnesium (2 mM og 4 mM). Hver af disse betingelser blev gentaget mindst to gange, med et par hundrede til 1.000 tøjrede DNAs pr eksperiment. Figur 2 beskriver det eksperimentelle design. Figur 3 viser eksempler på oplysninger om dataindsamling og analyse. Figur 4 illustrerer, hvordan hastigheden afhænger af proteinkoncentrationen ved de to koncentrationer af magnesium. Det kan bemærkes, at ved tilstrækkeligt lave proteinkoncentrationer er hastigheden proportional med protein og uafhængig af magnesium. For tilstrækkeligt høje proteinkoncentrationer afhænger hastigheden af magnesium, men uafhængig af proteinkoncentrationen.
| Trin | Beskrivelse | Temperatur (°C) | Tid (e) |
| 1 | Denaturering | 98 | 30 |
| 2 | Smelte | 98 | 10 |
| 3 | Anneal | 60 | 30 |
| 4 | Udvide | 72 | 30 |
| 5 | Endelig forlængelse | 72 | 120 |
Tabel 1: PCR-parametre. Vist er temperaturer og varigheder af termocyrprogrammet trin, der anvendes i trin 2.2 i protokollen. Smelte, gløde og udvide trin (trin 2, 3 og 4) gentages 30x.
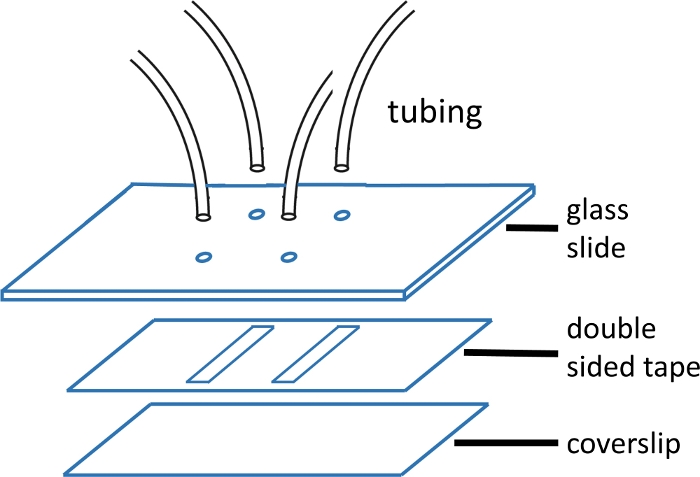
Figur 1: Konstruktion af mikrofluide flowceller.
Den øverste glasrutsjebane (2'' x 3'', 1 mm tyk) er forboret med huller, der matcher kanalmønsteret. Indløbs- og udløbsrørene indsættes i hullerne og fastgøres med epoxy, før tape og dækglas fastgøres. Det dobbeltsidede bånd er forskåret med kanalmønster. Det nederste dias (#1 eller #1,5 dækglas) renses tidligere ved hjælp af den protokol, der er beskrevet i hovedteksten. Når dækglasset er samlet, forsegles den med epoxy. Klik her for at se en større version af dette tal.

Figur 2: Eksperimentelt design.
AA) Metode til DNA-tethering. Dobbeltstrenget DNA (1 kbp) mærket med digoxigenin på 5ʹ ende er fastgjort til overfladen af flowcellen via antidigoxigenin-digoxigenin interaktion. Den 3ʹ ende af DNA, mærket med biotin, er fastgjort til en mikroperle via streptavidin-biotin interaktion. NDEI-spaltningsstedet er placeret i midten af DNA'et. BB) Eksperimentel opsætning under dataindsamling, der viser magnet og objektiv position. Den permanente magnet opretholder en svag opadgående kraft på perle under spaltningsreaktionen. Klik her for at se en større version af dette tal.
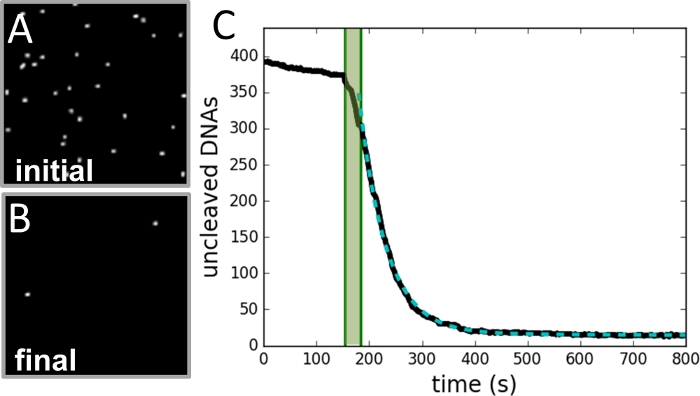
Figur 3: Eksempel på dataindsamling og -analyse.
a)Billedeaf det område af perler, der er taget, før spaltningsreaktionen påbegyndes. bB) Billede af samme region, når reaktionen er afsluttet. (C) Plot af antallet af perler vs tid (sort kurve) som bestemt fra hver ramme af videooptagelsen. Det skraverede grønne område markerer perioden med injektion af enzym og er ikke inkluderet i pasformen. Dataene passer til en enkelt eksponentiel kurve (stiplet grøn kurve) med en henfaldskonstant svarende til reaktionshastigheden. Klik her for at se en større version af dette tal.

Figur 4: NDEI spaltning er afhængig af koncentrationer af protein og magnesium.
Plot af den målte SSDC-satsen for NDEI for en række proteinkoncentrationer i to forskellige koncentrationer af magnesium: 2 mM (blå cirkler) og 4 mM (grønne firkanter). Fejllinjer repræsenterer SEM. De stiplede kurver er tendenslinjer og repræsenterer ikke passer til teorien. Klik her for at se en større version af dette tal.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Protokollen kan bruges til at måle kinetik i ethvert SSDC-system, forudsat at strengadskillelsen observeres under forsøget. Påvisning af spaltning påvirkes ved at observere løsrivelse af den tøjret perle og markerer derfor det øjeblik, hvor strengadskillelsen. Alle tidligere trin opstår før påvisning af spaltningen; Således registreres kun den samlede transittid.
Flowcellen coverslip er funktionaliseret via ikke-specifik adsorption af antistofprotein til det rene glas. Utilstrækkeligt renset glas kan påvirke bindingen af antistoffet. I tøjring bør perletætheden være lav nok til, at perlerne ikke interagerer. Overfladetætheden af fastgørelsespunkter kan styres af koncentrationen af antistof under funktionalisering. Det samlede antal perler afhænger af størrelsen af synsfeltet. I dette tilfælde var et par hundrede til 1.000 tøjlede perler tilstrækkelige til gode statistikker og undgik perle-perle interaktioner. Under injektion af perle blev overfladesting overvåget via livevideo. Perle injektion blev stoppet, når anslået perle tæller var mellem 500 og 1.000 perler.
De hurtigste spaltningshastigheder, der kan måles nøjagtigt, begrænses af flowcellens blandingstid. Blandingstiden i laminar flowceller påvirkes af flere faktorer. Diffusion til overfladen er et vigtigt skridt; Blandingstiden afhænger derfor af reaktantens diffusionskoefficient. Den betydelige forskydning, der forekommer i indgangsslangen, som transporterer prøven til strømningskanalen fra prøvebeholderen, kan øge den tid, der er nødvendig for at sikre tilstrækkelig blanding ved reaktionsoverfladen i kanalen. Det blev konstateret, at blandingstiden kunne reduceres ved at reducere den opstrøms døde volumen og øge strømningshastigheden. Med en indre diameter på 380 μm og en maksimal længde på 8 cm for indløbsslangen (og strømningshastighed på 150 μL/min) blev det konstateret, at injektionstiden kunne reduceres til ~20 s uden at påvirke den målte spaltningshastighed. Da blandingstiden afhænger af reaktantens diffusionskoefficient, bestemmes den separat for hvert enkelt enzym eller hver spademeraktivator, der undersøges.
Tøjringsmetoden giver mulighed for ikke-specifik brud, formentlig på grund af enten dissociation af digoxigenin-antistofkomplekset eller frigivelse af antistoffet fra overfladen. Dette resulterer i en reproducerbar baggrund perle tab sats til stede før injektion af enzym på ~ 3 x 10-4 s-1. Denne systematiske effekt kan korrigeres for enten ved at trække baggrundshastigheden fra den målte spaltningshastighed eller ved at modellere baggrunden i tilpasningsligningen. Spaltningshastigheder, der er lavere end denne nedre grænse, vil dog blive målt mindre pålideligt.
Ufuldkommen overflade passivation kan føre til forkert tøjring. Dette fører enten til øgede fraktioner af "fast" perler, som ikke går væk under the eksperimentet, eller til forkert tøjlede perler, som tager afstand fra overfladen meget langsomt. Dette skaber en højere og muligvis skrånende basislinje i de behandlede data. Det blev konstateret, at med korrekt rengjort dække glider og frisklavet β-casein lagerløsning, disse virkninger var minimale for de fleste datasæt. For de lejlighedsvise datasæt, der viser dette, kan en ændring af tilpasningsfunktionen (til at omfatte en skrånende grundlinje) korrigere for denne effekt.
Den aktuelle protokol kan udvides på flere måder. Yderligere isolering af mekanistiske trin efter målstedsbinding kan udføres ved hjælp af et præbindingsformat, hvor protein injiceres i mangel af væsentlige cofaktorer. Denne idé er testet ved at injicere NDEI i mangel af magnesium. Under disse betingelser binder proteinet sig til sit cognate-sted, men kløver ikke DNA'et. Indsprøjtning af magnesium efter dette bindingstrin aktiverer spaltningen, hvilket resulterer i hurtigt tab af perle. activates Den eksperimentelle opsætning giver også mulighed for kontrol af DNA-kropsbygning og spænding ved at variere magnetkonfigurationen eller tilføje flow. Under de lave kræfter i disse eksperimenter, dna bliver delvist oprullet. Ændring af kræfterne lidt kan have en dramatisk effekt på kropsbygning af DNA. For eksempel, under buffer betingelser, hvor målet søgning er sats begrænsende, varierende kropsbygning af DNA kan teste for effekten af at hoppe på målet søgning. Under buffer betingelser, hvor hydrolyse trin er rentebegrænsende, varierende kraft kan sonde effekten af DNA spænding på fosfordiester obligation hydrolyse. Det skal bemærkes, at under den lave forstørrelse anvendes, kan disse kropslige ændringer ikke overholdes. De resulterende små bevægelser i perle position skal spores under højere forstørrelse for at kontrollere, at DNA-kropsbygning kan kontrolleres.
Dataanalyse kan udvides på forskellige måder. Dette arbejde anvender en simpel perletællingsmetode efterfulgt af kurvemontering af en enkelt eksponentiel funktion. Metoder baseret på bopæl tid analyse kan anvendes, samt20. Fordeling af opholdstider for individuelle DNAs kan derefter analyseres via kurvemontering eller ved hjælp af mere avancerede teknikker, såsom den generelle metode til moments21.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne har ingen interessekonflikter at afsløre.
Acknowledgments
Dette arbejde blev støttet af National Science Foundation tilskud MCB-1715317.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



