Summary
Een zeer parallelle methode voor het meten van het site-specifieke decolleté van DNA op het niveau van één molecuul wordt beschreven. Dit protocol toont de techniek met behulp van de beperking endonuclease NdeI. De methode kan eenvoudig worden aangepast om elk proces dat resulteert in site-specifieke DNA-decolleté te bestuderen.
Abstract
Site-specifieke DNA decolleté (SSDC) is een belangrijke stap in veel cellulaire processen, en het is cruciaal voor genbewerking. Dit werk beschrijft een kinetische test die SSDC in veel afzonderlijke DNA-moleculen tegelijk kan meten. Kraal-gebonden substraat JA's, elk met een enkele kopie van de doelsequentie, worden bereid in een microfluïdisch stroomkanaal. Een externe magneet past een zwakke kracht toe op de paramagnetische kralen. De integriteit van maximaal 1.000 individuele AV's kan worden gecontroleerd door de microbeads onder darkfield imaging te visualiseren met behulp van een breedveld, lage vergrotingsdoelstelling. Het injecteren van een beperking endonuclease, NdeI, initieert de decolletéreactie. Videomicroscopie wordt gebruikt om het exacte moment van elk DNA-decolleté vast te leggen door het frame te observeren waarin de bijbehorende kraal omhoog en uit het brandpuntsvlak van het doel beweegt. Frame-voor-frame kraal tellen kwantificeert de reactie, en een exponentiële pasvorm bepaalt de reactiesnelheid. Deze methode maakt het verzamelen van kwantitatieve en statistisch significante gegevens over single molecule SSDC reacties in een enkel experiment.
Introduction
Site-specifieke DNA decolleté (SSDC) is een belangrijke stap in veel genomische transacties. Bijvoorbeeld, bacteriële beperking-modificatie (RM)1 en CRISPR2 systemen cellen te beschermen tegen aanvallen door fagen en plasmiden door het herkennen en klijmen van vreemd DNA op specifieke sequenties. In type II RM herkennen restrictie-endonucleases (REs) korte 4-8 basispaarsequenties (bp) via eiwit-nucleïnezuurinteracties3. CRISPR-geassocieerde endonucleases, zoals Cas9, binden zich aan sites via hybridisatie van de doelsite met crRNAs gebonden aan de endonucleases4. De creatie van site-specifieke double stranded breaks (DSB's) zijn ook de eerste stap in veel DNA-recombinatiegebeurtenissen5. De diversiteit van antigeenbindingsgebieden die door de recombinatie V(D)J worden gecreëerd, vereist bijvoorbeeld de erkenning en decolleté van specifieke doellocaties6. Sommige transposons zijn bekend om specifieke DNA-sequenties richten,evenals 7. Niet verrassend, veel site-specifieke nucleases die betrokken zijn bij deze processen, zoals Cas9, zijn een belangrijk onderdeel van genbewerkingstechnologieën8. Daarnaast zijn ook nieuwe site-specifieke endonucleases (d.w.z. zinkvinger nucleases9 en TALENS10)ontworpen om genomen te bewerken.
Veel methoden zijn gebruikt om de kinetiek van site-specifieke decolleté van nucleïnezuren te meten. Deze omvatten gelanalyse, fluorescentie11,12, en sequencing gebaseerde methoden13. Een belangrijke vooruitgang werd bereikt met de tethering van microbeads, waardoor DSB's in enkele moleculen van DNA kunnen worden gedetecteerd door de beweging van een kraal na de scheiding van de streng. Bij deze methoden worden verschillende soorten krachten gebruikt om strengscheiding en beweging van het kraal na decolleté te garanderen. In één geval zijn optische vallen gebruikt om het decolleté van DNA te meten door EcoRV14. In deze experimenten, doel zoeken is het doel van het onderzoek, met voorwaarden geoptimaliseerd, zodat site-specifieke binding is de tarief beperkende stap. Een nadeel van optische vallen is dat slechts een enkel DNA kan worden waargenomen op een moment. Daarnaast moet een periodieke grote trekkracht worden toegepast om te testen op de strengscheiding.
Een andere techniek maakt gebruik van een combinatie van stroom en zwakke magnetische krachten te trekken op de kraal op een continue manier15. Op deze manier wordt diffusie beperkt decolleté door NdeI gemeten. De gebruikte methode maakt het mogelijk om meerdere honderden NA's tegelijk te meten, waardoor statistische significantie in één experiment kan worden bereikt. Er zijn ook experimenten gebruikt op basis van magnetische pincetten. In een dergelijke studie werd een retrovirale integrase bestudeerd door een DSB op te nemen in de invoeging oligonucleotide16. Succesvolle integratie resulteerde in de integratie van DSB in het vastgebonden DNA en het verlies van de bijgevoegde kraal. In een soortgelijke studie naar de ATP-afhankelijke type III-beperking endonuclease EcoPI werden tientallen DNO's waargenomen in één experiment17. Magnetische pincet houden het voordeel dat spanning, evenals DNA-lus, kan worden gecontroleerd en gecontroleerd tijdens de reactie.
Hier gepresenteerd is een zeer parallelle single molecule methode voor het meten van SSDC kinetiek, die profiteert van recente verbeteringen in grootschalige tethering van DNAs. Deze methode is een verbetering en uitbreiding van eerdere methoden die worden gebruikt om DNA-replicatie18, contourlengte van DNA19en decolleté door REs15te meten . In deze techniek worden lineaire TINA's met één kopie van de herkenningssequentie bereid met biotine aan de ene kant en aan de andere kant. De biotine bindt streptavidin, die covalent is bevestigd aan een paramagnetische microbead. De DNA-kraalcomplexen worden geïnjecteerd in een microfluïdisch kanaal dat is gefunctionaliseerd met anti-digoxigenin FAB-fragmenten. Het DNA bindt vervolgens aan de oppervlaktebevestigingspunten via binding van de digoxigenine aan de adsorbeerde FAB-fragmenten. Zwakke magnetische krachten toegepast met een permanente magneet houden de kraal niet-specifiek aan het oppervlak. Monsters kunnen snel (<30 s) in het stroomkanaal worden geïnjecteerd om de decolletéreactie te activeren. Stroom wordt uitgeschakeld tijdens het verzamelen van gegevens. Aangezien elk DNA wordt gedes cleaved, kan de nauwkeurige tijd van decolleté door het kader registreren waarin de parel zich op en uit het brandpuntsplan van het doel beweegt, waardoor van de videoverslag verdwijnt. Een frame-voor-frame telling van de resterende kralen kan worden gebruikt om de reactievoortgang te kwantificeren.
Hieronder vindt u het volledige protocol en voorbeeldgegevens die met NdeI worden verzameld. Als voorbeeld van hoe de techniek kan worden toegepast, worden decolletésnelheden voor een reeks eiwitconcentraties gemeten in twee verschillende concentraties magnesium, een essentiële metaalcofactor. Hoewel deze toepassing van het protocol NdeI gebruikt, kan de methode worden aangepast voor gebruik met elke site-specifieke nuclease door het DNA-substraatontwerp te variëren.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Het maken van de stroomcel
- Het wassen van de coverslips
- Plaats coverslips in kleurpotten en sonicaat met ethanol (EtOH), dan met 1 M KOH (voor 30 min elk). Om KOH-neerslag in EtOH te voorkomen, spoel je grondig af met ddH2O tussen wasbeurten.
- Herhaal zowel EtOH als de KOH wasstappen 1x voor een totaal aantal van vier wasbeurten (twee EtOH en twee KOH). Bewaar gereinigde coverslips in ddH2O in kleurpotten.
- Snijd laad- en uitgangsbuizen (8 cm lang) met een schoon scheermes en steek in gaten in een schone glazen schuif. Gebruik PE-20 voor inlaat en PE-60 voor uitlaat. Epoxy gedurende 5 minuten om buizen te beveiligen en eventuele overtollige buizen af te knippen.
OPMERKING: Glazen glijbanen meten 2'' x 3'' x 1 mm. Gaten worden geboord in paren 15 mm uit elkaar. Elk paar vormt beide uiteindes van één kanaal. De slang met de kleinere ID wordt gebruikt voor de inlaat, omdat dit het upstream dode volume en dus de vereiste mengtijd vermindert (figuur 1). - Line-up en breng voorgesneden dubbelzijdige tape met kanaal patroon uitgesneden over gaten op de glazen glijbaan. Glad met plastic tangen om een goede afdichting te bereiken.
OPMERKING: De dubbelzijdige tape die in dit experiment wordt gebruikt, is 120 μm dik en de kanalen zijn 2 mm breed en 15 mm lang. Kanalen worden gesneden met behulp van een mes printer(Tabel van materialen). Het is mogelijk om maximaal vier kanalen op één coverslip te plaatsen. Zie figuur 1 voor een afbeelding van de stroomcel. - Na het afpellen van de rug, breng een schone coverslip (gedroogd met perslucht) over de tape en glad weer met plastic tang voor een goede afdichting.
- Epoxy de rand van de coverslip om de stroomcel te verzegelen en te laten genezen.
2. Bereiding van gelabeld DNA voor tethering
- Bereid in een PCR-buis 50 μL PCR-reactiemix voor met 0,02 U/μL high fidelity DNA polymerase, 200 μM dNTPs, 0,5 μM forward primer, 0,5 μM reverse primer en 250 ng M13mp18 vector-DNA.
OPMERKING: Hier werden de voorwaartse primer (biotine-CCAACTTAATCGCCTTGC) en omgekeerde primer (digoxigenin-TGACCATTAGATACATTTCGC) gekozen om een gebied van ongeveer 1.000 bp lang te versterken, verspreid van posities 6338 tot 107 in het circulaire genoom van het M13mp18 DNA. Er is een enkele NdeI-site in het midden van de versterkte regio. De voorwaartse primer is 5ー gelabeld met digoxigenine, die de anti-digoxigenine op de coverslip bindt. De omgekeerde primer is 5ー gelabeld met biotine, die de streptavidin-gecoate kralen bindt. - Plaats de PCR-buis in de thermocycler en volg de cyclus zoals weergegeven in tabel 1.
- Zuiver het PCR-product met een PCR-opruimkit volgens het protocol van de fabrikant.
OPMERKING: Met behulp van de kit die is opgegeven in de tabel van materialen,de typische DNA-opbrengst is ~ 2 μg.
3. Tethering van DNA en kralen
- Bereid 10 mL buffer A (1 M Tris-HCl [pH = 7,5], 50 mM NaCl, 2 mM MgCl2, 1 mg/mL β-Casein, 1 mg/mL Pluronic F-127). Ontgassen in een vacuüm desiccator voor ten minste 1 uur.
- Om de stroomcel te functionaliseren, injecteer u 25 μL anti-digoxigenin FAB-fragmenten (20 μg/mL) in PBS in het stroomkanaal. Gebruik gel laadtips om in de PE-60 buizen te passen. Incubeer bij kamertemperatuur (RT) gedurende 30 min.
- Na incubatie spoel je het kanaal door 0,5 mL buffer A door het kanaal te trekken met behulp van een spuit. Zorg ervoor dat u geen lucht in het kanaal introduceert.
- Na functionalisatie, monteer de stroomcel op een omgekeerde microscoop. Sluit de uitlaatbuis aan op een spuitpomp en plaats de inlaatbuis in een microcentrifugebuis met buffer A.
- Trek handmatig minstens 0,5 mL buffer A om het systeem door te spoelen en de pomp te primen. Laat de pomp minstens 5 minuten op 10 μL/min draaien om het systeem in evenwicht te brengen.
- Om de kralen (Tabel van materialen), vortex de voorraad fles kralen en pipet 1,6 μL van de 10 mg /mL voorraad kralen in 50 μL van buffer A, dan vortex.
- Met behulp van een magnetische separator, pipet uit de buffer en resuspend in 50 μL van buffer A, dan vortex.
- Herhaal stap 3.7 2x voor een totaal van drie wasbeurten. Voor de laatste wasbeurt, resuspend in 100 μL buffer A en vortex om een uiteindelijke concentratie van 160 μg/mL te bereiken.
- Om het DNA en de kralen te complexen, bereidt u eerst 480 μL van 0,5 pM gelabeld DNA-substraat in buffer A. Vervolgens pipet in 20 μL van 160 μg/mL kraal suspensie, om ervoor te zorgen dat de kralen vortex voordat pipetteren. Plaats op een rotator gedurende 3 min.
- Na 3 min, onmiddellijk laden in het kanaal bij een stroomsnelheid van 10 μL /min gedurende ~ 15 min of totdat voldoende kraal tethering is waargenomen.
OPMERKING: Kralen mogen niet zo dicht verpakt zijn dat ze op het oppervlak met elkaar communiceren (zie discussiesectie). - Om het kanaal van alle vrije kralen te wassen, schakelt u de inlaatbuis in op een verse buis van buffer A en stroomt u gedurende ten minste 10 minuten binnen bij 50 μL/min of totdat er geen losse kralen zijn waargenomen.
4. Het verzamelen en analyseren van gegevens
- Om zich voor te bereiden op het verzamelen van gegevens, plaatst u de inlaatbuis in een microcentrifugebuis met ten minste 100 μL NdeI (0,25–4,00 U/mL) in buffer A. Verlaag de permanente magneet over het stroomkanaal en plaats de lichtbron buiten de as voor donkere beeldvorming.
OPMERKING: Twee ringvormige zeldzame aardmagneten, samen geepoxideerd, worden 8 mm boven het actieve oppervlak van het stroomkanaal gehouden met behulp van een uitkragende optische post tijdens het verzamelen van gegevens. Een ganzenhalslamp wordt gebruikt voor de off-axis lichtcursus. - Gebruik een commerciële microscoop, videocamera en software voor het verzamelen van gegevens(Tabel van materialen). Klik in de software op het tabbladBelichtingen stel 'Belichtingstijd' in op 10 ms. Klik op het tabbladTimelapseen stel 'Afbeeldingstelling' in op 600,duurop 20 min enintervalop 2 s. Klik opUitvoerenom het verzamelen van gegevens te starten.
- Stel op een spuitpomp de debiet in op 150 μL/min en injectievolume op 80 μL. Druk op "Run" op 1 min in het verzamelen van gegevens. Schakel na de injectie de pomp uit en sluit de klep om stroom tijdens het verzamelen van gegevens te voorkomen.
- Zodra gegevens worden verzameld, open beeldanalyse software(Tabel van materialen). Kies onderFilehet tabblad Bestanddeoptie Importeren | "Beeldvolgorde". Zoek de afbeeldingsbestanden in het pop-upmenu en klik op 'Openen'.
- Stel de drempel in door"Theshold aanpassen" tekiezen onder het menu "Afbeelding". Gebruik de schuifbalk om de drempelwaarde in te stellen om heldere vlekken te identificeren die overeenkomen met kralen in de afbeelding.
- Tel de lichtpuntjes in elk frame door te klikken op "Deeltjes analyseren" in het "Analyseren" pull-down menu. Klik op' OK',en kies 'Ja'om alle afbeeldingen te verwerken. Sla het resultatenbestand op.
OPMERKING: Hiermee wordt een gegevensbestand opgeslagen dat het aantal kralen in elk opgenomen videoframe bevat. - Open data-analysesoftware (Tabel van materialen) en importeer het resultatenbestand door te klikken op " Importeren uittekstbestand" in het pull-downmenuBestand. Plot de gegevens van het aantal kraals versus tijd.
- Pas het aantal kralen versus tijd aan door op 'Curve Fit' te klikken in het uittrekmenu' Analyseren'. Kies de vergelijking" Natuurlijke Exponent" en klik op " ProbeerFit" | "OK".
OPMERKING: Alleen de gegevens die na de injectie van het monster zijn geregistreerd, moeten in het montagegebied worden opgenomen. De pasvormparameter in de exponent van de montagefunctie is de decolletésnelheid.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Met behulp van deze techniek werden de SSDC-percentages van NdeI gemeten voor een reeks eiwitconcentraties (0,25–4,00 U/mL) bij twee verschillende concentraties magnesium (2 mM en 4 mM). Elk van deze voorwaarden werd minstens tweemaal gerepliceerd, met een paar honderd tot 1.000 vastgebonden DNAs per experiment. Figuur 2 beschrijft het experimentele ontwerp. Figuur 3 toont voorbeelden van gegevensverzameling en analysedetails. Figuur 4 illustreert hoe de snelheid afhangt van de eiwitconcentratie bij de twee concentraties magnesium. Opgemerkt kan worden dat bij voldoende lage eiwitconcentraties de snelheid evenredig is met eiwit en onafhankelijk is van magnesium. Voor voldoende hoge eiwitconcentraties is de snelheid afhankelijk van magnesium, maar onafhankelijk van de eiwitconcentratie.
| Stap | Beschrijving | Temperatuur (°C) | Tijd (en) |
| 1 | Denaturatie | 98 | 30 |
| 2 | Smelten | 98 | 10 |
| 3 | Anneal | 60 | 30 |
| 4 | Uitbreiden | 72 | 30 |
| 5 | Laatste uitbreiding | 72 | 120 |
Tabel 1: PCR-parameters. Getoond zijn temperaturen en duur van de stappen van het thermocyclerprogramma die in stap 2.2 van het protocol worden gebruikt. De smelt-, anneal- en uitschuifstappen (stappen 2, 3 en 4) worden 30x herhaald.
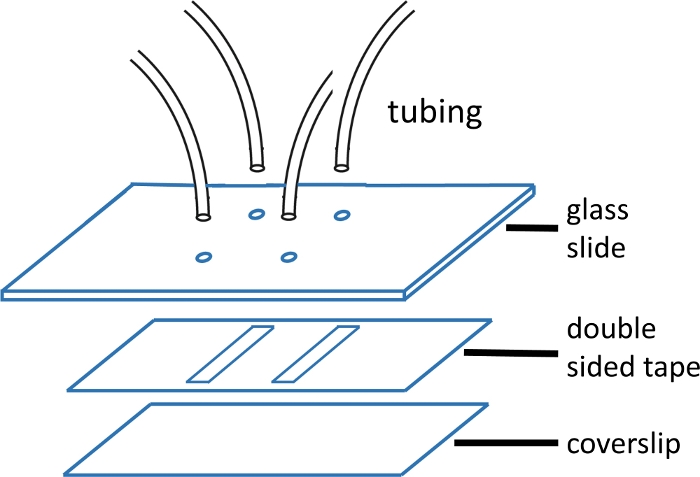
Figuur 1: Microfluïde stroomcelconstructie.
De bovenste glazen schuif (2'' x 3'', 1 mm dik) is voorgeboord met gaten die overeenkomen met het kanaalpatroon. De inlaat- en uitlaatbuizen worden in de gaten gestoken en met epoxy bevestigd voordat de tape en het afdekglas worden bevestigd. De dubbelzijdige tape is voorgesneden met kanaalpatroon. De onderste dia (#1 of #1.5 afdekglas) wordt eerder gereinigd met behulp van het protocol beschreven in de hoofdtekst. Eenmaal gemonteerd, wordt de rand van het afdekglas verzegeld met epoxy. Klik hier om een grotere versie van dit cijfer te bekijken.

Figuur 2: Experimenteel ontwerp.
(A) Methode van DNA-tethering. Dubbelstrengs DNA (1 kbp) gelabeld met digoxigenine aan het 5%-uiteinde is via antidigoxigenine-digoxigenine interactie aan het oppervlak van de stroomcel bevestigd. Het 30-eind van het DNA, gelabeld met biotine, is bevestigd aan een microbead via streptavidin-biotine interactie. De NdeI decolleté site is gelegen in het midden van het DNA. (B) Experimentele installatie tijdens het verzamelen van gegevens met magneet en objectieve positie. De permanente magneet behoudt een zwakke opwaartse kracht op de kraal tijdens de decolletéreactie. Klik hier om een grotere versie van dit cijfer te bekijken.
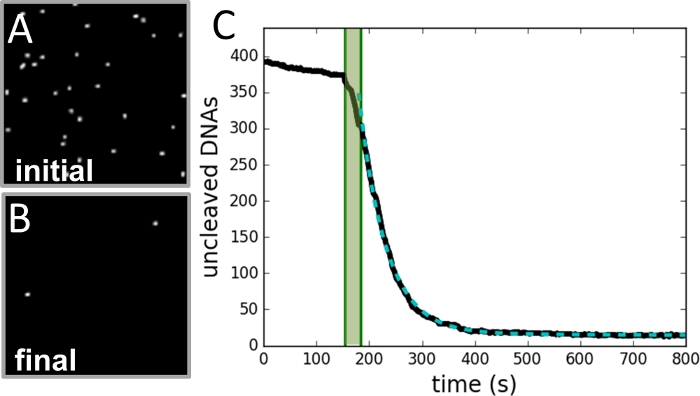
Figuur 3: Voorbeeld van het verzamelen en analyseren van gegevens.
(A) Beeld van het gebied van kralen genomen voordat decolleté reactie wordt gestart. (B) Beeld van dezelfde regio na reactie is voltooid. (C) Plot van het aantal kralen vs. tijd (zwarte curve) zoals bepaald uit elk frame van de video-record. Het schaduwrijke groene gebied markeert de periode van injectie van enzym en is niet inbegrepen in de pasvorm. De gegevens passen op een enkele exponentiële curve (gestreemmte groene curve), met een vervalconstante gelijk aan de reactiesnelheid. Klik hier om een grotere versie van dit cijfer te bekijken.

Figuur 4: NdeI decolleté is afhankelijk van concentraties eiwit en magnesium.
Plot van de gemeten SSDC-snelheid van NdeI voor een reeks eiwitconcentraties bij twee verschillende concentraties magnesium: 2 mM (blauwe cirkels) en 4 mM (groene vierkanten). Foutbalken vertegenwoordigen SEM. De gestippelde rondingen zijn trendlijnen en vertegenwoordigen geen past aan theorie. Klik hier om een grotere versie van dit cijfer te bekijken.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Het protocol kan worden gebruikt om de kinetiek van een SSDC-systeem te meten, op voorwaarde dat de strengscheiding tijdens het experiment wordt waargenomen. De detectie van decolleté wordt beïnvloed door het observeren van het loskoppelen van de vastgebonden kraal en markeert daarom het moment van strengscheiding. Alle voorgaande stappen vinden plaats vóór de detectie van het decolleté; zo wordt alleen de totale transittijd geregistreerd.
De flow cell coverslip wordt gefunctionaliseerd via niet-specifieke adsorptie van antilichaam eiwit aan het schone glas. Onvoldoende gereinigd glas kan de binding van het antilichaam beïnvloeden. Bij tethering moet de kraaldichtheid laag genoeg zijn, zodat kralen niet interageren. De oppervlaktedichtheid van bevestigingspunten kan worden gecontroleerd door de concentratie van antilichamen tijdens de functionalisatie. Het totale aantal kralen is afhankelijk van de grootte van het gezichtsveld. In dit geval waren een paar honderd tot 1.000 vastgebonden kralen voldoende voor goede statistieken en vermeden kraleninteracties. Tijdens de kraalinjectie werd oppervlaktetethering gemonitord via live video. Kraal injectie werd gestopt toen geschatte kraal tellen was tussen de 500 en 1.000 kralen.
De snelste decolletésnelheden die nauwkeurig kunnen worden gemeten, worden beperkt door de mengtijd van de stroomcel. De mengtijd in laminaire stroomcellen wordt beïnvloed door verschillende factoren. Verspreiding naar het oppervlak is een belangrijke stap; daarom is de mengtijd afhankelijk van de diffusiecoëfficiënt van de reactant. De significante afschuif die optreedt in de ingangsbuizen, die het monster naar het stroomkanaal van het monsterreservoir transporteert, kan de tijd die nodig is om voldoende mengen op het reactieoppervlak in het kanaal te garanderen, verhogen. Men vond dat de mengtijd kon worden verminderd door het upstream dode volume te verminderen en de stroomsnelheid te verhogen. Met een binnendiameter van 380 μm en een maximale lengte van 8 cm voor de inlaatbuizen (en de stroomsnelheid van 150 μL/min) bleek de injectietijd te kunnen worden teruggebracht tot ~20 s zonder de gemeten decolletésnelheid te beïnvloeden. Aangezien de mengtijd afhangt van de diffusiecoëfficiënt van de reactant, moet deze voor elk enzym of decolleté-activator afzonderlijk worden bepaald.
De tethering methode zorgt voor niet-specifieke kettingbreuk, vermoedelijk als gevolg van ofwel dissociatie van de digoxigenin-antilichaam complex of het vrijkomen van het antilichaam van het oppervlak. Dit resulteert in een reproduceerbare achtergrond kraal verlies tarief aanwezig voor de injectie van enzym van ~ 3 x 10-4 s-1. Dit systematische effect kan worden gecorrigeerd voor hetzij door de achtergrondsnelheid af te trekken van het gemeten decolletépercentage, of door de achtergrond in de passende vergelijking te modelleren. Echter, decolleté tarieven lager dan deze ondergrens zal minder betrouwbaar worden gemeten.
Onvolmaakte oppervlaktepassivatie kan leiden tot onjuiste tethering. Dit leidt ofwel tot verhoogde fracties van "vastzittende" kralen, die niet verdwijnen tijdens het the experiment, of tot onjuist vastgebonden kralen, die zich zeer langzaam van het oppervlak distantiëren. Hierdoor ontstaat een hogere en mogelijk schuine basislijn in de verwerkte gegevens. Men vond dat met behoorlijk schoongemaakte dekkingsslips en vers gemaakte β-caseïnevoorraadoplossing, deze gevolgen voor de meeste gegevensreeksen minimaal waren. Voor de incidentele gegevenssets die dit weergeven, kan het wijzigen van de montagefunctie (om een schuine basislijn op te nemen) dit effect corrigeren.
Het huidige protocol kan op verschillende manieren worden uitgebreid. Verdere isolatie van mechanistische stappen na doelsitebinding kan worden uitgevoerd met behulp van een pre-bindend formaat, waarbij eiwit wordt geïnjecteerd bij afwezigheid van essentiële cofactoren. Dit idee wordt getest door het injecteren van NdeI in de afwezigheid van magnesium. Onder deze omstandigheden bindt het eiwit zich aan zijn cognate-site, maar splijt het DNA niet. Het injecteren van magnesium na deze bindende stap activeert het decolleté wat resulteert in snel parelverlies. De experimentele opstelling zorgt ook voor de controle van DNA-conformatie en spanning door het variëren van de magneetconfiguratie of het toevoegen van flow. Onder de lage krachten in deze experimenten wordt het DNA gedeeltelijk opgerold. Het veranderen van de krachten enigszins kan een dramatisch effect hebben op de bevleesdheid van het DNA. Bijvoorbeeld, onder buffer omstandigheden waarin doel zoeken is tariefbeperkend, het variëren van de conformatie van het DNA kan testen op het effect van springen op het doel zoeken. Onder bufferomstandigheden waarin de hydrolysestap tariefbeperkend is, kan het variëren van de kracht het effect van de DNA-spanning op fosfodiesterbindingshydrlyse onderzoeken. Opgemerkt moet worden dat onder de lage vergroting die wordt gebruikt, deze conformatieveranderingen niet kunnen worden waargenomen. De resulterende kleine bewegingen in kraalpositie moeten onder hogere vergroting worden gevolgd om te controleren of dna-conformatie kan worden gecontroleerd.
Gegevensanalyse kan op verschillende manieren worden uitgebreid. Dit werk past een eenvoudige methode van het pareltelling toe, gevolgd door curvefitting van een enkele exponentiële functie. Methoden op basis van verblijfstijd analyse kan worden gebruikt,evenals 20. Distributies van verblijfstijden van individuele NC's kunnen vervolgens worden geanalyseerd via curve fitting of met behulp van meer geavanceerde technieken, zoals de gegeneraliseerde methode van momenten21.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
De auteurs hebben geen belangenconflicten te onthullen.
Acknowledgments
Dit werk werd ondersteund door de National Science Foundation subsidie MCB-1715317.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



