Summary
TEK molekül düzeyinde DNA'nın bölgeye özgü dekoltesini ölçmek için son derece paralel bir yöntem tanımlanmıştır. Bu protokol, kısıtlama enonuclease NdeI kullanarak tekniği göstermektedir. Yöntem kolayca siteye özgü DNA bölünmesi ile sonuçlanan herhangi bir süreci incelemek için değiştirilebilir.
Abstract
Siteye özgü DNA bölünmesi (SSDC) birçok hücresel süreçte önemli bir adımdır ve gen düzenleme için çok önemlidir. Bu çalışma, aynı anda birçok tek DNA moleküllerinde SSDC ölçme yeteneğine sahip bir kinetik töz açıklar. Her biri hedef sıranın tek bir kopyasını içeren boncuk-bağlı substrat DNA'ları mikroakışkan akış kanalında hazırlanır. Harici mıknatıs paramanyetik boncuklar için zayıf bir kuvvet uygular. 1.000'e kadar ayrı DNA'nın bütünlüğü, geniş alan, düşük büyütme hedefi kullanılarak karanlık alan görüntüleme siperinde mikroboncukların görselleştirilmesiyle izlenebilir. Bir kısıtlama endonükleaz enjekte, NdeI, dekolte reaksiyonu başlatır. Video mikroskobu, ilişkili boncuk hedefin odak düzleminin yukarı ve dışına hareket ettiği çerçeveyi gözlemleyerek her DNA bölünmesinin tam anını kaydetmek için kullanılır. Kare kare boncuk sayma reaksiyonu ölçer ve üstel uyum tepki hızını belirler. Bu yöntem, tek bir deneyde tek moleküllü SSDC reaksiyonları hakkında nicel ve istatistiksel olarak anlamlı verilerin toplanmasına olanak sağlar.
Introduction
Siteye özgü DNA bölünmesi (SSDC) birçok genomik işlemin önemli bir adımıdır. Örneğin, bakteriyel kısıtlama-modifikasyon (RM)1 ve CRISPR2 sistemleri belirli dizilerde yabancı DNA'yı tanıyarak ve yarıklayarak hücreleri faj ve plazmidlerin saldırısına karşı korur. Tip II RM'de, restriksiyon endokleazları (REs) protein-nükleik asit etkileşimleri ile kısa 4-8 baz çifti (bp) dizilerinitanırlar 3. Cas9 gibi CRISPR ilişkili ensonükleazlar, ensonükleazlara bağlı crRNA'lar ile hedef alanın hibridizasyonu yoluyla sitelere bağlanır4. Siteye özgü çift iplikçikli molaların (DSB'ler) oluşturulması da birçok DNA rekombinasyon olayının ilk adımıdır5. Örneğin, V(D)J rekombinasyon tarafından oluşturulan antijen bağlama bölgelerinin çeşitliliği belirli hedef sitelerin tanınmasını ve bölünmesini gerektirir6. Bazı transpozonlar belirli DNA dizileri hedef bilinmektedir, yanı sıra7. Beklendiği gibi, cas9 gibi bu süreçlerde yer alan birçok siteye özgü nükleazlar, gen düzenleme teknolojileri8önemli bir bileşenidir. Buna ek olarak, yeni siteye özgü endokleazlar (örneğin, çinko parmak nükleazları9 ve TALENS10)da genomları döşeyecek şekilde tasarlanmıştır.
Birçok yöntem nükleik asitlerin siteye özgü dekolte kinetik ölçmek için kullanılmıştır. Bu jel analizi dahil, floresan11,12, ve sıralama tabanlı yöntemler13. Mikroboncukların tethering ile büyük bir ilerleme sağlandı, hangi DNA'nın tek moleküllerinde DSB'ler iplikçik ayrımı ndan sonra bir boncuk hareketi ile tespit edilmesine olanak sağlar. Bu yöntemlerde, boncuk sonrası dekoltenin iplikçik ayrıştırılması ve hareketini sağlamak için farklı kuvvet türleri kullanılır. Bir durumda, optik tuzaklar EcoRV14tarafından DNA bölünmesi ölçmek için kullanılmıştır. Bu deneylerde, hedef arama araştırmanın amacıdır ve siteye özgü bağlamanın oranı sınırlayan adım olması için koşullar optimize edilebistir. Optik tuzakların bir dezavantajı da aynı anda sadece tek bir DNA'nın gözlemlenebildiğidir. Buna ek olarak, iplikçik ayrımı için test etmek için periyodik büyük çekme kuvveti uygulanmalıdır.
Başka bir teknik sürekli bir şekilde boncuk çekmek için akış ve zayıf manyetik kuvvetlerin bir arada kullanır15. Bu şekilde NdeI ile difüzyon sınırlı dekolte ölçülür. Kullanılan yöntem aynı anda birkaç yüz DA'nın eşzamanlı olarak ölçülmesine olanak sağlayarak, tek bir deneyde istatistiksel anlamlıya ulaşılmasını sağlar. Manyetik cımbız üzerine deneyler de kullanılmıştır. Bu tür bir çalışmada, bir retroviral integrase ekleme oligonükleotid bir DSB dahil edilerek çalışıldı16. Başarılı entegrasyon, DSB'nin bağlı DNA'ya dahil edilmesi ve ekli boncuk kaybıile sonuçlandı. ATP'ye bağlı tip III restriksiyon endokleaz EcoPI benzer bir çalışmada, tek bir deneyde onlarca DNA gözlenmiştir17. Manyetik cımbız, reaksiyon sırasında gerilimin yanı sıra DNA döngünün de kontrol edilebildiği ve izlenebileceği avantajını elinde tutar.
Burada sunulan SSDC kinetik ölçmek için son derece paralel tek molekülyöntemi, hangi BÜYÜK ÖLÇEKLI TEthering büyük ölçekli tethering son gelişmeler yararlanır. Bu yöntem, DNA replikasyonu18,DNA19kontur uzunluğu ve REs15ile dekolte ölçmek için kullanılan önceki yöntemlerin bir iyileşme ve uzantısıdır. Bu teknikte, tanıma dizisinin tek bir kopyasını içeren lineer DNA'lar bir uçta biotin, diğer ucunda digoksijenin ile hazırlanır. Biotin streptavidin bağlar, kovalent bir paramanyetik mikrop bağlı. DNA-boncuk kompleksleri anti-digoksigenin FAB parçaları ile işlevselleştirilmiş bir mikroakışkan kanal içine enjekte edilir. DNA daha sonra digoksigenin adsorbe FAB parçalarına bağlanması yoluyla yüzey eki noktalarına bağlanır. Kalıcı bir mıknatısla uygulanan zayıf manyetik kuvvetler, boncuk ların özellikle yüzeye yapışmamasını önlüyor. Numuneler dekolte reaksiyonu etkinleştirmek için akış kanalına hızla (<30 s) enjekte edilebilir. Veri toplama sırasında akış kapatılır. Her DNA'nın ayrılmasıyla, dekoltenin tam zamanı, reklamın hedefin odak planının yukarı ve dışına doğru hareket ettiği çerçevenin kaydedilmesi yle belirlenebilir ve böylece video kaydından kaybolur. Kalan boncukların kare kare sayısı reaksiyon ilerlemesini ölçmek için kullanılabilir.
Aşağıda sunulan tam protokol yanı sıra NdeI kullanılarak toplanan örnek veriler. Tekniğin nasıl uygulanabildiğini örnek olarak, bir dizi protein konsantrasyonu için bölünme oranları, önemli bir metal kofaktör olan iki farklı magnezyum konsantrasyonu ile ölçülür. Protokolün bu uygulaması NdeI'yi kullansa da, yöntem DNA substrat tasarımını değiştirerek herhangi bir yere özgü nükleaz ile kullanılmak üzere uyarlanabilir.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Akış hücresi yapma
- Kapakların yıkanması
- Boyama kavanozları ve etanol (EtOH) ile sonicate yerleştirin, sonra 1 M KOH (her biri için 30 dakika için). EtOH'da KOH yağışını önlemek için yıkarlar arasında ddH2O ile iyice durulayın.
- Toplam dört yıkama (iki EtOH ve iki KOH) sayısı için hem EtOH hem de KOH yıkama adımlarını 1x tekrarlayın. Temizlenmiş kapakları boyama kavanozlarında ddH2O'da saklayın.
- Temizleme bir jilet kullanarak yükleme ve çıkış tüplerini (8 cm uzunluğunda) kesin ve temiz bir cam kaydıraktaki deliklere yerleştirin. Giriş için PE-20 ve çıkış için PE-60 kullanın. Epoksi 5 dk için tüp güvenli ve herhangi bir fazla tüp kapalı kırpma.
NOT: Cam slaytlar 2'' x 3'' x 1 mm ölçülerindedir. Delikler 15 mm arayla çift olarak delinmiş. Her çift, tek bir kanalın her iki ucunu oluşturur. Daha küçük kimlikli borular giriş için kullanılır, çünkü bu, yukarı doğru ölü hacmi ve böylece gerekli karıştırma süresini azaltır (Şekil 1). - Hattı ve kanal deseni cam slayt üzerinde delikler üzerinde kesilmiş precut çift taraflı bant uygulayın. Iyi bir mühür elde etmek için plastik forceps ile düzleştirin.
NOT: Bu deneyde kullanılan çift taraflı bant 120 μm kalınlığında, kanallar 2 mm genişliğinde ve 15 mm uzunluğundadır. Kanallar bıçakyazıcı(Malzeme Tablosu)kullanılarak kesilir. Tek bir kapak kaymasına en fazla dört kanal sığdırmak mümkündür. Akış hücresinin görüntüsü için Şekil 1'e bakın. - Arka dan sıyrıldıktan sonra, bant üzerine temiz bir coverslip (basınçlı hava ile kurutulmuş) uygulayın ve iyi bir mühür için plastik forseps ile tekrar düzleştirin.
- Epoksi akış hücremühür ve tedavi sağlar kapak kapalı kenarı.
2. Tethering için etiketli DNA hazırlanması
- Bir PCR tüpünde, 0,02 U/μL yüksek doğrulukta DNA polimeraz, 200 μM dNTPs, 0,5 μM ileri astar, 0,5 μM ters astar ve 250 ng M13mp18 vektör DNA içeren 50 μL PCR reaksiyon karışımı hazırlayın.
NOT: Burada, ileri astar (biotin-CCAACTTAATCGCCTTGC) ve ters astar (digoksigenin-TGACCATTAGATACATTTCGC) m13mp18 DNA'nın dairesel genomunda 6338 ile 107 arasında değişen yaklaşık 1.000 bp uzunluğundaki bir bölgeyi yükseltmek için seçilmiştir. Yükseltilmiş bölgenin ortasında tek bir NdeI sitesi vardır. Ön astar 50 digoksigenin ile etiketlenmiş, hangi coverslip üzerinde anti-digoksigenin bağlar. Ters astar 50 biotin ile etiketlenmiş, hangi streptavidin kaplı boncukbağlar. - PCR tüpünü termocycler'a yerleştirin ve Tablo 1'degösterildiği gibi döngüyü takip edin.
- PCR ürününü, üreticinin protokolünü izleyerek bir PCR temizleme kiti yle arındırın.
NOT: Malzeme Tablosu'ndabelirtilen kiti kullanarak tipik DNA verimi ~2 μg'dir.
3. DNA ve boncukte tethering
- 10 mL tampon A (1 M Tris-HCl [pH = 7.5], 50 mM NaCl, 2 mM MgCl2, 1 mg/mL β-Kazein, 1 mg/mL Pluronik F-127) hazırlayın. En az 1 saat vakum desiccator degas.
- Akış hücresini işlevselleştirmek için PBS'deki 25 μL anti-digoksigenin FAB parçalarını (20 μg/mL) akış kanalına enjekte edin. PE-60 boruiçine sığacak jel yükleme ipuçları nı kullanın. 30 dakika oda sıcaklığında (RT) kuluçka.
- Kuluçkadan sonra, bir şırınga kullanarak kanaldan 0,5 mL tampon A çekerek kanalı temize çıkar. Kanala hava sokmamaya dikkat edin.
- İşlevselleştirmeden sonra, akış hücresini ters bir mikroskoba monte edin. Çıkış tüpünü şırınga pompasına bağlayın ve giriş tüpünü tampon A içeren mikrosantrifüj tüpüne yerleştirin.
- Sistemi yıkamak ve pompayı astarlamak için en az 0,5 mL tampon A'yı manuel olarak çekin. Pompanın sistemi dengelemek için en az 5 dk 10 μL/dk'da çalışmasını sağlar.
- Boncukları(Malzeme Tablosu)hazırlamak için, 10 mg/mL stok boncukların 10 mg/mL'lik stok boncuklarının 1.6 μL'lik stok şişesini 50 μL tampon A'ya, sonra girdap haline getirmek.
- Manyetik ayırıcı kullanarak, arabellek dışarı pipet ve tampon A 50 μL, sonra girdap içinde resuspend.
- Üç yıkıntı toplam için adım 3.7 2x tekrarlayın. Son yıkama için, 160 μg/mL'lik son konsantrasyonu elde etmek için 100 μL tampon A ve girdapta yeniden askıya alın.
- DNA ve boncukları karmaşıkleştirmek için, ilk olarak tampon A'da 0,5 pM etiketli DNA substratı 480 μL hazırlayın. Daha sonra, pipet 20 μL 160 μg/mL boncuk süspansiyon, pipetleme önce boncuk girdap emin olun. 3 dakika boyunca bir rotator üzerine yerleştirin.
- 3 dakika sonra, ~15 dk için 10 μL/dk akış hızında veya yeterli boncuk tethering gözlenene kadar kanala hemen yükleyin.
NOT: Boncuklar yüzeyde birbirleriyle etkileşime girmeyecek kadar yoğun bir şekilde paketlenmemelidir (bkz. tartışma bölümü). - Tüm serbest boncukların kanalını yıkamak için giriş tüpünü yeni bir tampon A tüpüne çevirin ve en az 10 dakika boyunca 50 μL/dk'da veya gevşek boncuk lar gözlenene kadar akın.
4. Veri toplama ve analiz
- Veri toplamaya hazırlanmak için giriş tüpünü en az 100 μL NdeI (0.25-4.00 U/mL) içeren mikrosentrifüj tüpüne yerleştirin.
NOT: İki anüler nadir toprak mıknatıslar, birlikte epoksi, veri toplama sırasında bir cantilevered optik sonrası kullanılarak akış kanalının aktif yüzeyi üzerinde 8 mm tutulur. Eksen dışı ışık seyrinde kaz boyun lambası kullanılır. - Ticari bir mikroskop, video kamera ve veri toplama yazılımı(Malzeme Tablosu) kullanın. Yazılımda "Pozlama" sekmesine tıklayın ve "Pozlama Süresi" seçeneğini 10 ms'e ayarlayın. "Timelapse" sekmesine tıklayın ve "Resim Sayısı" 600' e, "Süre" den 20 dakikaya ve "Interval" i 2'ye ayarlayın. Veri toplamaya başlamak için "Çalıştır"ı tıklatın.
- Bir şırınga pompasında, akış hızını 150 μL/dk'ya, enjeksiyonRunhacmini 80 μL'ye ayarlayın. Enjeksiyondan sonra, pompayı kapatın ve veri toplama sırasında akışı önlemek için vanayı kapatın.
- Veriler toplandıktan sonra, açık görüntü analiz yazılımı(Malzeme Tablosu). "Dosya" sekmesialtında , "Import" | "Görüntü Dizisi". Açılan menüdeki resim dosyalarını bulun ve "Aç"ı tıklatın.
- "Image" pull-down menüsüaltında "Adjust Theshold" seçeneğini seçerek eşiği ayarlayın. Görüntüdeki boncuklara karşılık gelen parlak noktaları belirlemek için eşik değerini ayarlamak için kaydırıcı çubuğunu kullanın.
- "Analyze" pull-down menüsünde "Parçacıkları Analiz Et" seçeneğini tıklayarak her karedeki parlak noktaları sayın. Tüm görüntüleri işlemek için "Tamam"'ı tıklatın, ardından "Evet" seçeneğini belirleyin. Sonuç dosyasını kaydedin.
NOT: Bu, kaydedilen her video karesinde boncuk sayısını içeren bir veri dosyasını kaydeder. - Veri analiz yazılımı(Malzeme Tablosu)açın ve "Dosya" çekme menüsünde " TextFile "den Alma" butonuna tıklayarak sonuç dosyasını içeri aktarın. Boncuk sayısı verilerini zamana göre çizin.
- "Analyze" pull-down menüsünde "Curve Fit" butonuna tıklayarak boncuk ve zamana göre boncuk sayısını sığdırın. "Natural Expent" denklemini seçin ve "Try Fit" seçeneğini tıklayın | "Tamam".
NOT: Sadece numune enjeksiyonundan sonra kaydedilen veriler montaj bölgesine dahil edilmelidir. Montaj fonksiyonunun üstteki uygun parametresi dekolte hızı olacaktır.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Bu teknik kullanılarak, SSDC ndeI oranları iki farklı magnezyum konsantrasyonlarında (2 mM ve 4 mM) çeşitli protein konsantrasyonları (0.25-4.00 U/mL) olarak ölçüldü. Bu koşulların her biri deney başına birkaç yüz ila 1.000 bağlı NA'larla en az iki kez çoğaltıldı. Şekil 2 deneysel tasarımı tanımlar. Şekil 3 veri toplama ve analiz ayrıntıları örneklerini gösterir. Şekil 4, magnezyumun iki konsantrasyonundaki protein konsantrasyonuna nasıl bağlı olduğunu göstermektedir. Bu yeterince düşük protein konsantrasyonlarında, oranı protein ile orantılı ve magnezyum bağımsız olduğu görülebilir. Yeterince yüksek protein konsantrasyonları için, oranı magnezyum bağlıdır ama protein konsantrasyonu bağımsızdır.
| Adım | Açıklama | Sıcaklık (°C) | Zaman (lar) |
| 1 | Denatürasyon | 98 | 30 |
| 2 | Eritmek | 98 | 10 |
| 3 | Anneal | 60 | 30 |
| 4 | Genişlet -mek | 72 | 30 |
| 5 | Son Uzatma | 72 | 120 |
Tablo 1: PCR parametreleri. Protokolün 2.2. Erime, anneal ve genişletme adımları (adım 2, 3 ve 4) 30x tekrarlanır.
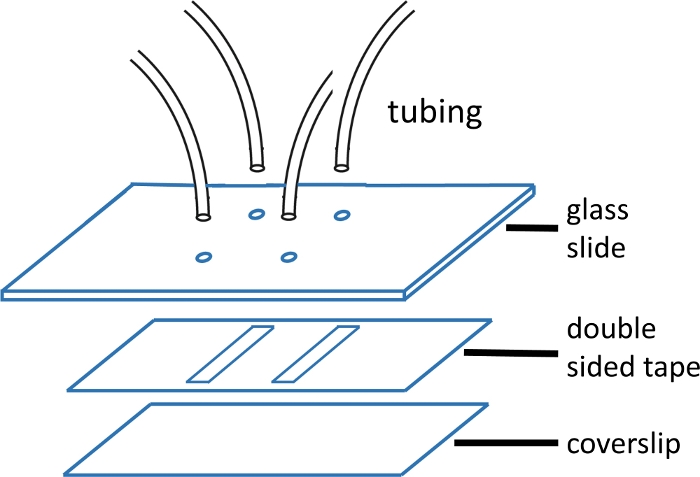
Şekil 1: Mikroakışkan akış hücresi yapımı.
Üst cam kaydırak (2'' x 3'', 1 mm kalınlığında) kanal desenine uyan delikler ile önceden delinmiş. Giriş ve çıkış tüpleri deliklere yerleştirilir ve bant ve kapak camı takmadan önce epoksi ile sabitlenir. Çift taraflı bant kanal deseni ile önceden kesilir. Alt slayt (#1 veya #1,5 kapak camı) daha önce ana metinde açıklanan protokol kullanılarak temizlenir. Bir kez monte edildikten sonra, kapak cam kenarı epoksi ile mühürlenir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: Deneysel tasarım.
(A) DNA tethering yöntemi. 50 ucunda digoksigenin etiketli çift iplikli DNA (1 kbp) antidigoksigenin-digoksigenin etkileşimi ile akış hücresi yüzeyine bağlanır. Dna'nın 30 ucu, biotin ile etiketlenmiş, streptavidin-biotin etkileşimi ile bir mikrop eklenir. NdeI dekolte alanı DNA'nın merkezinde yer alır. (B) Mıknatıs ve nesnel konumu gösteren veri toplama sırasında deneysel kurulum. Daimi mıknatıs dekolte reaksiyonu sırasında boncuk üzerinde zayıf bir yukarı kuvvet tutar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
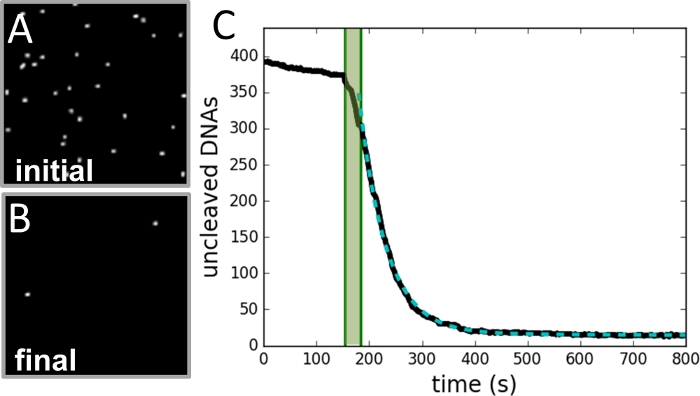
Şekil 3: Veri toplama ve analiz örneği.
(A) Dekolte reaksiyonu başlatılmadan önce çekilen boncuk ların bölgesinin görüntüsü. (B) Reaksiyon tamamlandıktan sonra aynı bölgenin görüntüsü. (C) Video kaydının her karesinden belirlenen boncuk sayısı ile zaman (siyah eğri) sayısının çizimi. Gölgeli yeşil alan enzim enjeksiyon uyşu dönemini işaretler ve uyum dahil değildir. Veriler, reaksiyon hızına eşit bir bozunma sabiti ile tek bir üstel eğriye (kesik yeşil eğri) uyar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 4: NdeI dekolte protein ve magnezyum konsantrasyonlarına bağlıdır.
2 mM (mavi daireler) ve 4 mM (yeşil kareler) magnezyum iki farklı konsantrasyonlarda protein konsantrasyonları bir dizi için NdeI ölçülen SSDC oranının arsa. Hata çubukları SEM'i temsil ediyor. Kesikli eğriler eğilim çizgileridir ve teoriye uygun değildir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Protokol, deney sırasında iplikçik ayrımının gözlenmiş olması koşuluyla, herhangi bir SSDC sisteminin kinetiklerini ölçmek için kullanılabilir. Dekolte tespiti, bağlı boncuk kopması gözlemleyerek etkilenir ve bu nedenle iplikçik ayırma anında işaretler. Tüm önceki adımlar dekolte tespitinden önce meydana gelir; böylece, sadece toplam transit süresi kaydedilir.
Akış hücre örtüsü temiz cama antikor proteininin spesifik olmayan adsorpsiyonu ile işlevselhale getirilmiştir. Yetersiz temizlenmiş cam antikor bağlama etkileyebilir. Tethering olarak, boncuk yoğunluğu boncuk etkileşim emzemez, böylece yeterince düşük olmalıdır. Ek noktalarının yüzey yoğunluğu işlevselleştirme sırasında antikor konsantrasyonu ile kontrol edilebilir. Toplam boncuk sayısı görüş alanının boyutuna bağlıdır. Bu durumda, birkaç yüz ila 1.000 bağlı boncuk iyi istatistikler için yeterli ve boncuk-boncuk etkileşimleri kaçınılmalıdır. Boncuk enjeksiyonu sırasında yüzey tethering canlı video ile izlendi. Boncuk enjeksiyonu, tahmini boncuk sayısı 500 ile 1000 boncuk arasında yken durduruldu.
Doğru bir şekilde ölçülebilen en hızlı bölünme oranları akış hücresinin karıştırma süresi ile sınırlıdır. Laminar akış hücrelerinde karıştırma süresi çeşitli faktörlerden etkilenir. Yüzeye difüzyon önemli bir adımdır; bu nedenle karıştırma süresi reaktanın difüzyon katsayısına bağlıdır. Numuneyi numune haznesinden akış kanalına taşıyan giriş borusunda oluşan önemli makas, kanaldaki reaksiyon yüzeyinde yeterli karıştırmayı sağlamak için gereken süreyi artırabilir. Bu karıştırma süresi upstream ölü hacmi azaltarak ve akış hızı artırarak azaltılabilir bulundu. Giriş borusu için 380 μm'lik iç çapı ve giriş borusu için maksimum uzunluğu 8 cm olan (ve 150 μL/dk akış hızı) ölçülen dekolte hızını etkilemeden enjeksiyon süresinin ~20 s'ye düşürülebileceği saptandı. Karıştırma süresi reaktanın difüzyon katsayısına bağlı olduğundan, çalışılan her enzim veya dekolte aktivörü için ayrı ayrı belirlenmelidir.
Tethering yöntemi non-spesifik tether rüptürü için izin verir, muhtemelen ya digoksigenin-antikor kompleksi veya yüzeyden antikor serbest bırakılması ayrıştırma nedeniyle. Bu da ~3 x 10-4 s-1enzim enjeksiyonundan önce tekrarlanabilir bir arka plan boncuk kaybı oranı ile sonuçlanır. Bu sistematik etki, arka plan hızını ölçülen bölünme hızından çıkararak veya montaj denkleminde arka planı modelleyerek düzeltilebilir. Ancak, bu alt sınırdan daha düşük dekolte oranları daha az güvenilir olarak ölçülecektir.
Kusurlu yüzey pasifasyonu yanlış tethering yol açabilir. Bu ya deney sırasında uzağa gitmez "sıkışmış" boncuk, artan fraksiyonları yol açar, the ya da çok yavaş yüzeyden ayrıştırmak yanlış bağlı boncuklar. Bu, işlenmiş verilerde daha yüksek ve büyük olasılıkla eğimli bir taban çizgisi oluşturur. Düzgün bir şekilde temizlenmiş kapak fişleri ve taze yapılmış β-kazein stok çözümü ile bu etkilerin çoğu veri seti için çok az olduğu bulunmuştur. Bunu gösteren ara sıra veri kümeleri için, montaj işlevini (eğimli bir taban çizgisi içerecek şekilde) değiştirmek bu etki için düzeltilebilir.
Geçerli protokol çeşitli şekillerde genişletilebilir. Hedef alan bağlama dan sonra mekanistik adımların daha fazla izolasyonu, temel kofaktörlerin yokluğunda proteinin enjekte edildiği bir ön bağlama biçimi kullanılarak gerçekleştirilebilir. Bu fikir magnezyum yokluğunda NdeI enjekte ederek test edilir. Bu koşullar altında, protein cognate sitesine bağlanır ama DNA cleave değildir. Bu bağlayıcı adımdan sonra magnezyum enjekte hızlı boncuk kaybı ile sonuçlanan dekolte aktive. Deneysel kurulum aynı zamanda mıknatıs konfigürasyon değiştirerek veya akış ekleyerek DNA konformasyonu ve gerginlik kontrolü için izin verir. Bu deneylerdeki düşük kuvvetler altında DNA kısmen kıvrılır. Kuvvetleri biraz değiştirmek DNA'nın konformasyonu üzerinde dramatik bir etki yaratabilir. Örneğin, hedef aramanın hızısınırlayıcı olduğu tampon koşullarda, DNA'nın konformasyonunu değiştirmek hedef aramada atlamanın etkisini test edebilir. Hidroliz adımının hız sınırlayıcı olduğu tampon koşullarda, kuvvetin değişmesi DNA geriliminin fosfodiester bağı hidrolizi üzerindeki etkisini araştırabilir. Kullanılan düşük büyütme altında bu konformasyonel değişikliklerin gözlenmemesi unutulmamalıdır. Boncuk pozisyonunda ortaya çıkan küçük hareketler, DNA konformasyonunkontrol edilebileni doğrulamak için daha yüksek büyütme altında izlenmeli.
Veri analizi çeşitli şekillerde genişletilebilir. Bu çalışma, basit bir boncuk sayma yöntemi ve ardından tek bir üstel fonksiyonun eğri montajı uygular. İkamet süresi analizine dayalı yöntemler kullanılabilir, yanı sıra20. Bireysel DAA'ların ikamet sürelerinin dağılımları daha sonra eğri montajı yoluyla veya momentlerin genelleştirilmiş yöntemi21gibi daha gelişmiş teknikler kullanılarak analiz edilebilir.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yazarların ifşa etmesi gereken çıkar çatışmaları yok.
Acknowledgments
Bu çalışma Ulusal Bilim Vakfı hibe MCB-1715317 tarafından desteklenmiştir.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



