Summary
En mycket parallell metod för att mäta den platsspecifika klyvningen av DNA på singelmolekylnivå beskrivs. Detta protokoll visar tekniken med hjälp av begränsningen endonuclease NdeI. Metoden kan enkelt modifieras för att studera vilken process som helst som resulterar i platsspecifik DNA-klyvning.
Abstract
Site-specifika DNA-klyvning (SSDC) är ett viktigt steg i många cellulära processer, och det är avgörande för genredigering. Detta arbete beskriver en kinetisk analys som kan mäta SSDC i många enstaka DNA-molekyler samtidigt. Bead-bundna substrat DNAs, var och en innehåller en enda kopia av målsekvensen, är beredda i en mikrofluidisk flödeskanal. En yttre magnet applicerar en svag styrka till de paramagnetic pärlorna. Integriteten hos upp till 1000 enskilda DNAs kan övervakas genom att visualisera microbeads under darkfield imaging med hjälp av ett brett fält, låg förstoring mål. Injicering av en begränsning endonuclease, NdeI, initierar klyvningsreaktionen. Videomikroskopi används för att registrera det exakta ögonblicket för varje DNA-klyvning genom att observera den ram där den tillhörande pärlan rör sig upp och ut ur målets fokalplan. Bildruta-för-bildruta pärla räkna kvantifierar reaktionen, och en exponentiell passform bestämmer reaktionshastigheten. Denna metod möjliggör insamling av kvantitativa och statistiskt signifikanta data på enmolekyl SSDC reaktioner i ett enda experiment.
Introduction
Platsspecifik DNA-klyvning (SSDC) är ett viktigt steg i många genomiska transaktioner. Till exempel skyddar systemen bakteriell begränsningsändring (RM)1 och CRISPR2 cellerna från angrepp av fagen och plasmider genom att känna igen och klyver främmande DNA vid specifika sekvenser. I typ II RM känner restriction endonucleases (REs) igen korta 4–8 basparsekvenser (bp) via protein-nukleinsyrainteraktioner3. CRISPR-associerade endonukleaser, såsom Cas9, binder till webbplatser via hybridisering av målplatsen med crRNAs bundna till endonukleaserna4. Skapandet av platsspecifika dubbelsträngade raster (DSB) är också det första steget i många DNA-rekombinationshändelser5. Till exempel kräver den mångfald av antigenbindningsregioner som skapats genom V(D)J-rekombination att specifika målplatser 6 erkänns ochklyvningen. Vissa transposoner är kända för att rikta specifika DNA-sekvenser, samt7. Inte överraskande, många platsspecifika nukleaser som deltar i dessa processer, såsom Cas9, är en viktig komponent i genredigeringsteknik8. Dessutom har även nya platsspecifika endonucleaser (dvs. zinkfingernukleaserna9 och TALENS10) konstruerats för att redigera genom.
Många metoder har använts för att mäta kinetiken hos platsspecifika klyvning av nukleinsyror. Dessa inkluderar gelanalys, fluorescens11,12, och sekvensering baserade metoder13. Ett stort framsteg uppnåddes med tjudra av microbeads, som gör att DSBs i enstaka molekyler av DNA som skall upptäckas genom rörelse av en pärla efter strand separation. I dessa metoder används olika typer av krafter för att säkerställa strandseparation och rörelse av pärlan efter klyvning. I ett fall har optiska fällor använts för att mäta klyvning av DNA genom EcoRV14. I dessa experiment är målsökning målet för undersökningen, med villkor optimerade så att platsspecifika bindande är den takt begränsa steg. En nackdel med optiska fällor är att endast ett enda DNA kan observeras i taget. Dessutom måste en periodisk stor dragkraft appliceras för att testa för strandseparationen.
En annan teknik använder en kombination av flöde och svaga magnetiska krafter för att dra på pärlan på ett kontinuerligt sätt15. På detta sätt mäts diffusion begränsad klyvning av NdeI. Den metod som används möjliggör samtidig mätning av flera hundra DNAs på en gång, vilket möjliggör statistisk signifikans som skall uppnås i ett enda experiment. Experiment baserade på magnetisk pincett har också använts. I en sådan studie studerades en retroviral integrase genom att en DSB togs med i infogningen oligonukleotid16. Framgångsrik integration resulterade i införlivandet av DSB i tjudriga DNA och förlust av den bifogade pärlan. I en liknande studie av atp-beroende typ III begränsning endonuclease EcoPI, tiotals DNAs observerades i ett enda experiment17. Magnetiska pincetter håller fördelen att spänningen, liksom DNA-looping, kan kontrolleras och övervakas under reaktionen.
Presenteras här är en mycket parallell enda molekyl metod för att mäta SSDC kinetik, som drar nytta av de senaste förbättringarna i storskalig tjudra av DNAs. Denna metod är en förbättring och förlängning av tidigare metoder som används för att mäta DNA-replikation18, konturlängd av DNA19, och klyvning av REs15. I denna teknik, linjära DNAs som innehåller en enda kopia av igenkänningssekvensen är beredda med biotin i ena änden och digoxigenin i den andra. Biotinen binder streptavidin, som är kovalent fäst vid en paramagnetisk mikropärla. DNA-bead-komplexen injiceras i en mikrofluidisk kanal som har funktionaliserats med anti-digoxigenin FAB-fragment. DNA-tjuder därefter till ytbehandlatillsatsen pekar via bandet av digoxigeninen till de adsorberade FAB-fragmenten. Svaga magnetiska krafter appliceras med en permanent magnet hålla pärlan från att fastna icke-specifikt till ytan. Prover kan injiceras snabbt (<30 s) i flödeskanalen för att aktivera klyvningsreaktionen. Flödet stängs av under datainsamlingen. Eftersom varje DNA klyvs, kan den exakta tidpunkten för klyvning bestämmas genom att registrera den ram där pärlan rör sig upp och ut ur målets fokalplan och därmed försvinna från videoinspelningen. En bildruta-för-bildruta antal återstående pärlor kan användas för att kvantifiera reaktionsförloppet.
Presenteras nedan är det fullständiga protokollet samt exempel data som samlats in med hjälp av NdeI. Som ett exempel på hur tekniken kan tillämpas mäts klyvningshastigheter för en rad proteinkoncentrationer vid två olika koncentrationer av magnesium, en essentiell metallkofaktor. Även om denna tillämpning av protokollet använder NdeI, kan metoden anpassas för användning med alla platsspecifika nukleas genom att variera DNA-substratets utformning.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Att göra flödescellen
- Tvätta täckskydden
- Placera täcken i färgning burkar och sonikera med etanol (EtOH), sedan med 1 M KOH (för 30 min vardera). För att undvika KOH-nederbörd i EtOH, skölj noga med ddH2O mellan tvättar.
- Upprepa både EtOH och KOH tvättstegen 1x för totalt antal fyra tvättar (två EtOH och två KOH). Förvara rengjorda täcken i ddH2O i färgningsburkar.
- Skär lastnings- och utgångsrör (8 cm långa) med hjälp av en ren rakhyvel och sätt in i hål i en ren glasrutschbana. Använd PE-20 för inlopp och PE-60 för utlopp. Epoxi i 5 min för att säkra slangar och trimma bort eventuella överskottsrör.
OBS: Glasglasen mäter 2'' x 3'' x 1 mm. Hål borras i par 15 mm från varandra. Varje par bildar endera änden av en enda kanal. Slangen med det mindre ID:t används för inloppet, eftersom detta minskar den döda volymen uppströms och därmed den blandningstid som krävs (Bild 1). - Rada upp och applicera förklipp dubbelsidig tejp med kanalmönster utskuret över hål på glasglaset. Släta ut med plast tyns för att uppnå en bra tätning.
OBS: Den dubbelsidiga tejpen som används i detta experiment är 120 μm tjock, och kanalerna är 2 mm breda och 15 mm långa. Kanaler skärs med hjälp av en knivskrivare (Table of Materials). Det är möjligt att passa upp till fyra kanaler på en enda coverslip. Se bild 1 för en bild av flödescellen. - Efter avskalning av underlag, applicera en ren täckkolla (torkad med tryckluft) över tejpen och släta ut igen med plasttång för en bra tätning.
- Epoxy kanten av täckskydd för att täta flödet cellen och låt den bota.
2. Beredning av märkt DNA för tjudra
- I ett PCR-rör, preparera 50 μL PCR-reaktionsmix innehållande 0,02 U/μL high fidelity DNA-polymeras, 200 μM dNTPs, 0,5 μM framåt primer, 0,5 μM omvänd primer och 250 ng av M13mp18 DNA-vektor.
OBS: Här valdes den framåt primer (biotin-CCAACTTAATCGCCTTGC) och omvänd primer (digoxigenin-TGACCATTAKATTTTCGC) för att förstärka en region cirka 1.000 bp lång, som sträcker sig från positioner 6338 till 107 i cirkulär genomet av M13mp18 DNA. Det finns en enda NdeI-plats mitt i den förstärkta regionen. Den framåt primeren är 5ʹ märkt med digoxigenin, som binder anti-digoxigeninen på coverslipen. Den omvända primern är 5ʹ märkt med biotin, som binder streptavidin-belagd pärlor. - Sätt i PCR-röret i thermocycler och följ cykeln enligt tabell 1.
- Rena PCR-produkten med en PCR-saneringssats som följer tillverkarens protokoll.
OBS: Med hjälp av den sats som anges i tabell över material, den typiska DNA-avkastningen är ~ 2 μg.
3. Tjudra av DNA och pärlor
- Förbered 10 mL buffert A (1 M Tris-HCl [pH = 7,5], 50 mM NaCl, 2 mM MgCl2, 1 mg/mL β-Casein, 1 mg/mL Pluronic F-127). Degas i vakuum desiccator i minst 1 h.
- För att funktionalisera flödescellen injicerar du 25 μL anti-digoxigenin FAB-fragment (20 μg/mL) i PBS i flödeskanalen. Använd gel lastning tips för att passa in i PE-60 slangar. Inkubera i rumstemperatur (RT) i 30 min.
- Efter inkubationen spolar du kanalen genom att dra 0,5 mL buffert A genom kanalen med hjälp av en spruta. Var noga med att inte införa luft i kanalen.
- Efter funktionalisering monterar du flödescellen på ett inverterat mikroskop. Haka upp utloppsröret till en sprutpump och sätt inloppsröret i ett mikrocentrifugrör som innehåller buffert A.
- Dra manuellt minst 0,5 mL buffert A för att spola systemet och prime pumpen. Låt pumpen gå på 10 μL/min i minst 5 min för att jämviktsrelibera systemet.
- För att förbereda pärlorna (Tabell of Materials), vortexa lagerflaskan med pärlor och pipett 1,6 μL av 10 mg/mL lager pärlor i 50 μL buffert A, sedan vortex.
- Med hjälp av en magnetisk separator, pipettera ut bufferten och återanvända i 50 μL buffert A, sedan virvel.
- Upprepa steg 3,7 2x för totalt tre tvättar. För den sista tvätten, resuspend i 100 μL av buffert A och vortex för att uppnå en slutlig koncentration på 160 μg/mL.
- För att komplexa DNA och pärlor, först förbereda 480 μL av 0,5 pM märkt DNA-substrat i buffert A. Därefter, pipettering i 20 μL av 160 μg/mL pärla suspension, se till att virvel pärlorna innan pipettering. Placera på en rotator i 3 min.
- Efter 3 min, ladda omedelbart in i kanal vid en flödeshastighet på 10 μL/min i ~15 min eller tills tillräcklig pärla tjudra observeras.
OBS: Pärlor ska inte vara så tätt packade att de interagerar med varandra på ytan (se diskussionsdelen). - För att tvätta kanalen för alla fria pärlor, byt inloppsröret till ett färskt rör av buffert A och flöda in vid 50 μL/min i minst 10 min eller tills inga lösa pärlor observerats.
4. Insamling och analys av uppgifter
- För att förbereda för datainsamling, placera inloppsröret i ett mikrocentrifugrör som innehåller minst 100 μL NdeI (0,25–4,00 U/mL) i buffert A. Sänk permanentmagneten över flödeskanalen, och placera ljuskällan utanför axeln för darkfield-avbildning.
OBS: Två ringformiga sällsynta jordartsmetaller magneter, epoxied tillsammans, hålls 8 mm ovanför den aktiva ytan av flödet kanal med hjälp av en fribärande optisk post under datainsamling. En gås halslampa används för off-axeln ljus kurs. - Använd ett kommersiellt mikroskop, videokamera och datainsamlingsprogram (Table of Materials). I programvaran klickar du på fliken "Exponering" och ställer in "Exponeringstid" till 10 ms. Klicka på "Timelapse" -fliken och ställ in "Image Count" till 600, "Varaktighet" till 20 min, och "Interval" till 2 s. Klicka på "Kör" för att starta datainsamling.
- På en sprutpump, ställ in flödet till 150 μL/min och injektionsvolym till 80 μL. Tryck "Kör" vid 1 min i datainsamling. Efter insprutning, stäng av pumpen och stäng ventilen för att förhindra flöde under datainsamling.
- När data har samlats in, öppna bildanalys programvara( Table of Materials). Under fliken "Arkiv" väljer du "Importera" | "Bildsekvens". Leta upp bildfilerna i popup-menyn och klicka på "Öppna".
- Ställ in tröskeln genom att välja "Justera Theshold" under dennerdragnamenyn " Bild " . Använd skjutreglagets fält för att ställa in tröskelvärdet värde för att identifiera ljusa fläckar som motsvarar pärlor i bilden.
- Räkna de ljusa fläckarna i varje bildruta genom att klicka på "Analysera partiklar" i menyn "Analysera" pull-down. Klicka på" OK", välj sedan "Ja" för att bearbeta alla bilder. Spara resultatfilen.
OBS: Detta kommer att spara en datafil som innehåller antalet pärlor i varje inspelad video ram. - Öppna dataanalysprogramvara (Table of Materials) och importera resultatfilen genom att klicka på " Importera fråntextfil" i "File" pull-down menu. Rita upp pärlräkningsdata kontra tid.
- Passa in antalet pärlor kontra tid genom att klicka på "Kurvpassning" i"Analyze" pull-down menu. Välj ekvationen "Natural Exponent" och klicka på "Try Fit" | "OK".
OBS: Endast de data som registrerats efter insprutning av prov ska ingå i tillpassningsregionen. Den passar parametern i exponenten för passande funktion kommer att vara klyvningshastigheten.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Med hjälp av denna teknik mättes SSDC-satserna för NDEI för en rad proteinkoncentrationer (0,25–4,00 U/mL) vid två olika koncentrationer av magnesium (2 mM och 4 mM). Var och en av dessa villkor replikerades minst två gånger, med några hundra till 1000 bundna DNAs per experiment. Figur 2 beskriver den experimentella designen. I figur 3 visas exempel på uppgifter insamling och analys detaljer. Figur 4 illustrerar hur hastigheten beror på proteinkoncentrationen vid de två koncentrationerna av magnesium. Man kan konstatera att vid tillräckligt låga proteinkoncentrationer är hastigheten proportionell mot protein och oberoende av magnesium. För tillräckligt höga proteinkoncentrationer är hastigheten beroende av magnesium men oberoende av proteinkoncentration.
| Steg | Beskrivning | Temperatur (°C) | Tid (s) |
| 1 | Denaturering | 98 | 30 |
| 2 | Smälta | 98 | 10 |
| 3 | Glödg | 60 | 30 |
| 4 | Utöka | 72 | 30 |
| 5 | Slutlig Extend | 72 | 120 |
Tabell 1: PCR-parametrar. Visas är temperaturer och varaktigheter av termocyklerprogrammet steg som används i steg 2.2 av protokollet. Stegen smälta, glödg och utöka (steg 2, 3 och 4) upprepas 30x.
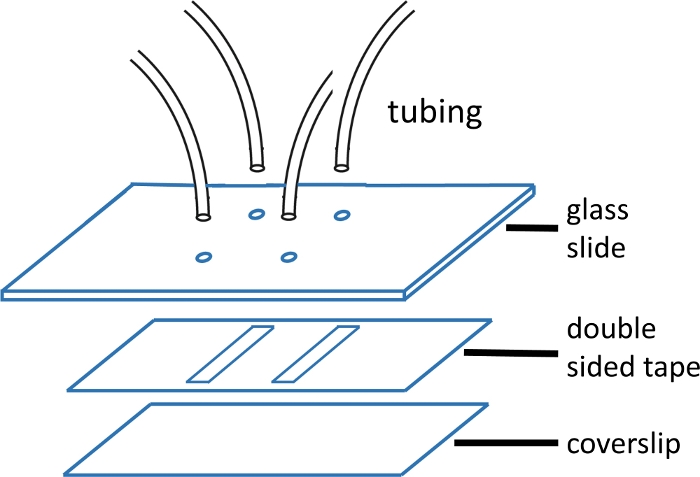
Bild 1: Mikrofluidiskt flöde cellkonstruktion.
Den översta glasglaset (2'' x 3'', 1 mm tjock) är förborrade med hål som matchar kanalmönstret. In- och utloppsrören sätts in i hålen och fixeras med epoxi före fastsättning av tejp och täckglas. Det dubbelsidiga bandet är färdigskuret med kanalmönster. Den nedersta #1 eller #1.5-täckglaset) rengörs tidigare med hjälp av det protokoll som beskrivs i huvudtexten. När den är sammansatt, förseglas täckglasets kant med epoxi. Vänligen klicka här för att visa en större version av denna figur.

Bild 2: Experimentell design.
(A) Metod för DNA-tjudra. Dubbelsträngat DNA (1 kbp) märkt med digoxigenin på 5ʹ änden är fäst på ytan av flödescellen via antidigoxigenin-digoxigenin interaktion. Den 3ʹ änden av DNA, märkt med biotin, är fäst vid en mikropärla via streptavidin-biotin interaktion. NdeI-klyvningsplatsen ligger i mitten av DNA. (B) Experimentell inställning under datainsamling som visar magnet och objektiv position. Permanentmagneten upprätthåller en svag uppåtriktad kraft på pärlan under klyvningsreaktionen. Vänligen klicka här för att visa en större version av denna figur.
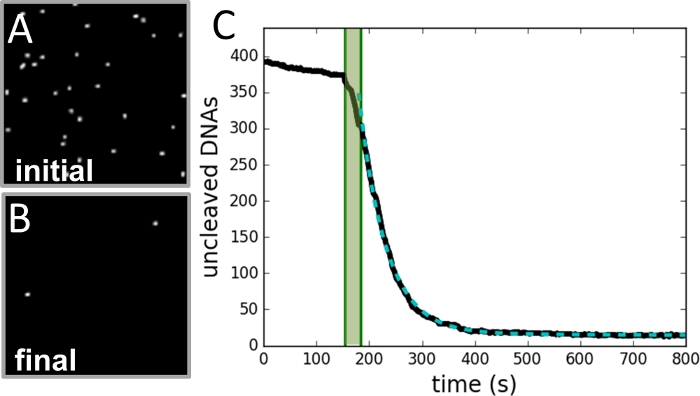
Figur 3: Exempel på insamling och analys av data.
(A) Bild av region av pärlor som tagits innan klyvningsreaktionen inleds. (B) Bild av samma region efter att reaktionen är avslutad. (C) Plot av antal pärlor vs. tid (svart kurva) som bestäms från varje bildruta av videoinspelningen. Det skuggade gröna området markerar perioden av injektion av enzym och ingår inte i passformen. Datan passar en enda exponentiell kurva (streckad grön kurva), med en förfallskonstant som är lika med reaktionshastigheten. Vänligen klicka här för att visa en större version av denna figur.

Figur 4: NDUI-klyvning är beroende av koncentrationer av protein och magnesium.
Tomt av den uppmätta SSDC-hastigheten av NdeI för en rad proteinkoncentrationer vid två olika koncentrationer av magnesium: 2 mM (blåa cirklar) och 4 mM (gröna rutor). Felstaplar representerar SEM. De streckade kurvorna är trendlinjer och representerar inte passar till teori. Vänligen klicka här för att visa en större version av denna figur.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Protokollet kan användas för att mäta kinetiken i alla SSDC-system, förutsatt att strandseparationen observeras under experimentet. Detektion av klyvning påverkas av att observera lossning av den tjudp och markerar därför ögonblick av strandseparation. Alla föregående steg inträffar före detektionen av klyvningen; sålunda registreras endast den totala transittiden.
Flödescellens coverslip är funktionaliserad via icke-specifik adsorption av antikroppsprotein till det rena glaset. Otillräckligt rengjort glas kan påverka antikroppens bindning. I tjudra, bör pärla densitet vara tillräckligt låg så att pärlor inte interagerar. Ytdensiteten hos fästpunkter kan kontrolleras genom koncentrationen av antikropp under funktionalisering. Det totala antalet pärlor beror på storleken på synfältet. I detta fall var några hundra till 1000 bundna pärlor tillräckliga för bra statistik och undvek pärla-pärla interaktioner. Under pärla injektion, ytan tjudra övervakades via live video. Pärl injektion stoppades när uppskattade pärlräkningen var mellan 500 och 1000 pärlor.
De snabbaste klyvningshastigheterna som kan mätas exakt begränsas av blandningstiden för flödescellen. Blandningstiden i laminära flödesceller påverkas av flera faktorer. Diffusion till ytan är ett nyckelsteg; därför beror blandningstiden på reaktantens diffusionskoefficient. Den signifikanta skjuvning som uppstår i entréslangen, som transporterar provet till flödeskanalen från provbehållaren, kan öka den tid som behövs för att säkerställa tillräcklig blandning vid reaktionsytan i kanalen. Det konstaterades att blandningstiden kunde minskas genom att minska den uppströmsdöda volymen och öka flödet. Med en innerdiameter på 380 μm och maxlängd på 8 cm för inloppsslangen (och flödeshastigheten 150 μL/min) konstaterades det att injektionstiden kunde reduceras till ~20 s utan att den uppmätta klyvningshastigheten påverkades. Eftersom blandningstiden beror på reaktantens diffusionskoefficient, bör den bestämmas separat för varje enzym eller klyvningsaktivator som studeras.
Tjudra metoden möjliggör icke-specifika tjuder bristning, förmodligen på grund av antingen dissociation av digoxigenin-antikropp komplex eller frisättning av antikroppen från ytan. Detta resulterar i en reproducerbar bakgrund pärla förlust hastighet närvarande före injektion av enzym på ~ 3 x 10-4 s-1. Denna systematiska effekt kan korrigeras för antingen genom att subtrahera bakgrundshastigheten från den uppmätta klyvningshastigheten, eller genom modellering av bakgrunden i passningsekvationen. Klyvningshastigheter som är lägre än denna nedre gräns kommer dock att mätas på mindre tillförlitligt sätt.
Bristfällig yt passivering kan leda till felaktig tjudra. Detta leder antingen till ökade fraktioner av "fastnat" pärlor, som inte går bort under the experimentet, eller till felaktigt bundna pärlor, som dissociate från ytan mycket långsamt. Detta skapar en högre och eventuellt sluttande baslinje i de bearbetade data. Det konstaterades att med korrekt rengjorda täcka glider och nygjorda β-casein stamlösning, dessa effekter var minimala för de flesta datamängder. För de enstaka datamängder som visar detta, kan ändra passande funktion (för att inkludera en sluttande baslinje) korrigera för denna effekt.
Det aktuella protokollet kan utökas på flera sätt. Ytterligare isolering av mekanistiska steg efter målplatsbindning kan utföras med hjälp av ett förbindande format, där protein injiceras i avsaknad av väsentliga kofaktorer. Denna idé testas genom att injicera NdeI i avsaknad av magnesium. Under dessa förhållanden binder proteinet till sitt konjaksställe men klyver inte DNA:t. Injicera magnesium efter denna bindning steg aktiverar klyvningen resulterar i snabb pärla förlust. Den experimentella setup möjliggör också för kontroll av DNA-konformation och spänning genom att variera magneten konfiguration eller lägga till flöde. Under de låga krafterna i dessa experiment blir DNA:t delvis ihopspolat. Att ändra krafterna något kan ha en dramatisk effekt på konformationen av DNA. Till exempel under buffertförhållanden där målsökning är kursbegränsande, kan varierande konformation av DNA testa för effekten av att hoppa på målsökningen. Under buffertförhållanden där hydrolyssteget är hastighetsbegränsande, kan varierande kraft sondera effekten av DNA-spänningen på fosfodiesterbindningshydrolys. Det bör noteras att under den låga förstoringen som används kan dessa konformationsförändringar inte observeras. De resulterande små rörelserna i pärlläge måste spåras under högre förstoring för att kontrollera att DNA-konformation kan kontrolleras.
Dataanalys kan utökas på en mängd olika sätt. Detta arbete tillämpar en enkel pärlräkningsmetod följt av kurvpassning av en enda exponentiell funktion. Metoder som bygger på uppehållstidsanalys kan användas, liksom20. Fördelningar av uppehållstider av enskilda DNAs kan sedan analyseras via kurvpassning eller genom att använda mer sofistikerade tekniker, såsom den generaliserade metoden för ögonblick21.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Författarna har inga intressekonflikter att avslöja.
Acknowledgments
Detta arbete stöddes av National Science Foundation bidrag MCB-1715317.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



