Summary
שיטה מקבילה מאוד למדידת המחשוף הספציפי לאתר של ה-DNA ברמת מולקולה אחת מתוארת. פרוטוקול זה מדגים את הטכניקה באמצעות ההגבלה אנדונוקלאז Nde. ניתן לשנות את השיטה בקלות כדי ללמוד כל תהליך שתוצאות מחשוף DNA ספציפי לאתר.
Abstract
מחשוף DNA ספציפי לאתר (SSDC) הוא צעד מפתח בתהליכים סלולריים רבים, והוא חיוני לעריכת גנים. עבודה זו מתארת תסה קינטית המסוגלת למדוד SSDC במולקולות DNA יחיד רבות בו זמנית. DNAs של הצוללת עם חרוזים, שכל אחד מהם מכיל עותק יחיד של רצף היעד, מוכנים בערוץ זרימה מיקרו-נוזל. מגנט חיצוני מפעיל כוח חלש על החרוזים הפרמגנטיים. ניתן לפקח על שלמותם של עד 1,000 DNAs בודדים על-ידי הדמיית המיקרו-חרוזים תחת הדמיית Darkfield באמצעות מטרה רחבת-שדה, הגדלה נמוכה. הזרקת אנדונוקליז הגבלה, נדול, יוזמת את תגובת המחשוף. מיקרוסקופ וידאו משמש כדי להקליט את הרגע המדויק של כל מחשוף DNA על ידי התבוננות במסגרת שבה החרוז המשויך נע למעלה והחוצה של מישור המוקד של המטרה. ספירת חרוזים פריים אחר תמונה מכמתת את התגובה, והתאים מעריכי קובע את קצב התגובה. שיטה זו מאפשרת איסוף של נתונים כמותיים ומשמעותיים סטטיסטית על מולקולה אחת תגובות SSDC בניסוי יחיד.
Introduction
מחשוף DNA ספציפי לאתר (SSDC) הוא צעד מפתח בעסקאות גנומיות רבות. לדוגמה, שינוי הגבלה חיידקית (RM)1 ומערכות CRISPR2 להגן על תאים מפני התקפה על ידי phages ופלסמידים על ידי זיהוי ומחשוף של DNA זר ברפים ספציפיים. בסוג II RM, אנדונוקלאס הגבלת (REs) לזהות רצפים קצרים 4-8 בסיס זוג בסיס (bp) באמצעות אינטראקציות חומצה גרעיןחלבון 3. אנדונוקלאסים המשויכים CRISPR, כגון Cas9, לאגד לאתרים באמצעות הכלאה של אתר היעד עם crRNAs מאוגדים endonucleases4. יצירת הפסקות תקועות כפולות ספציפיות לאתר (DSBs) הן גם הצעד הראשון באירועי שילוב DNA רבים5. לדוגמה, המגוון של אזורי איגוד אנטיגן שנוצרו על-ידי V(D)J שילוב דורש זיהוי ומחשוף של אתרי יעד ספציפיים6. כמה transposons ידועים למקד רצפי DNA ספציפיים, כמוגם 7. באופן לא מפתיע, גרעין ספציפי לאתר רבים המעורבים בתהליכים אלה, כגון Cas9, הם מרכיב מרכזי בטכנולוגיות עריכתגנים 8. בנוסף, אנדונוקליאות חדשניות ספציפיות לאתר (כלומר, גרעין אצבעאבץ 9 ו-TALENS10)הונדסו גם הן לעריכת גנומים.
שיטות רבות נוהל כדי למדוד את הקינטיים של מחשוף ספציפי לאתר של חומצות גרעין. אלה כוללים ניתוח ג'ל,פלואורסצנס 11,12, ושיטות מבוססות רצף13. התקדמות משמעותית הושגה עם רצועה של microbeads, המאפשר DSBs במולקולות יחיד של ה-DNA להתגלות על ידי התנועה של חרוז לאחר הפרדת גדיל. בשיטות אלה, סוגים שונים של כוחות מועסקים כדי להבטיח את הפרדת הגדיל ואת התנועה של החרוז לאחר המחשוף. במקרה אחד, מלכודות אופטיות שימשו כדי למדוד מחשוף של ה-DNA על ידי EcoRV14. בניסויים אלה, חיפוש היעד הוא מטרת החקירה, עם תנאים ממוטבים כך שאיגוד ספציפי לאתר הוא השלב המגביל את הקצב. חסרון אחד של מלכודות אופטיות הוא שרק DNA אחד ניתן לצפות בכל פעם. בנוסף, יש להחיל כוח משיכה גדול תקופתי כדי לבדוק את הפרדת הגדיל.
טכניקה נוספת משתמשת בשילוב של זרימה וכוחות מגנטיים חלשים כדי למשוך את החרוז באופן רציף15. בדרך זו, מחשוף מוגבל דיפוזיה על ידי NdeI נמדד. השיטה המועסקת מאפשרת מדידה בו זמנית של כמה מאות DNAs בבת אחת, המאפשר משמעות סטטיסטית להיות מושגת בניסוי אחד. ניסויים המבוססים על פינצטה מגנטית שימשו גם כן. במחקר אחד כזה, integrase רטרוויראלי נחקר על ידי הכללת DSB בהחדרת אוליגונוקלאוטיד16. שילוב מוצלח הביא להתאגדות של DSB ב-DNA קשור ואובדן של החרוז המצורף. במחקר דומה של EcoPI הגבלת אנדונוקליאז סוג III תלוי ATP, עשרות DNAs נצפו בניסוי יחיד17. פינצטה מגנטית להחזיק את היתרון כי מתח, כמו גם לולאות DNA, ניתן לשלוט ולפקח במהלך התגובה.
מוצג כאן היא שיטת מולקולה אחת מקבילה מאוד למדידת קינטיקה SSDC, אשר מנצל את השיפורים האחרונים ברצועה בקנה מידה גדול של DNAs. שיטה זו היא שיפור והרחבה של שיטות קודמות המשמשות למדד שכפול DNA18, אורך קווי מתאר של DNA19, ומחשוף על ידי REs15. בטכניקה זו, DNAs ליניארי המכיל עותק יחיד של רצף הזיהוי מוכנים עם ביוטין בקצה אחד digoxigenin בצד השני. הביוטין קושר סטרפטאבידין, המחובר באופן קוולנטי למיקרו-חרוז פארמגנטי. תסביכות חרוזי ה-DNA מוזרקים לערוץ מיקרו-נוזלים שתפקד עם שברי FAB אנטי-דיגוקסיג'ין. ה-DNA לאחר מכן קשור אל פני השטח נקודות מצורף באמצעות איגוד של digoxigenin לשבבי FAB adsorbed. כוחות מגנטיים חלשים המיושם עם מגנט קבוע לשמור על החרוז מדבק באופן לא ספציפי על פני השטח. ניתן להזריק דגימות במהירות (<30 s) לתוך ערוץ הזרימה כדי להפעיל את תגובת המחשוף. הזרימה מבוטלת במהלך איסוף נתונים. כאשר כל דנ"א מחשוף, ניתן לקבוע את זמן המחשוף המדויק על ידי הקלטת המסגרת שבה החרוז נע למעלה והחוצה מהתוכנית המוקדית של המטרה, ובכך להיעלם מרמת הקלטה. ניתן להשתמש בספירה של מסגרת אחר מסגרת של חרוזים שנותרו כדי לכמת את התקדמות התגובה.
מוצג להלן הפרוטוקול המלא, כמו גם נתונים לדוגמה שנאספו באמצעות NdeI. כדוגמה לאופן שבו ניתן ליישם את הטכניקה, שיעורי מחשוף עבור מגוון ריכוזי חלבון נמדדים בשני ריכוזים שונים של מגנזיום, cofactor מתכת חיוני. למרות יישום זה של הפרוטוקול משתמש NdeI, השיטה יכולה להיות מותאמת לשימוש עם כל גרעין ספציפי לאתר על ידי שינוי עיצוב ה-DNA של הצוללת.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. הפיכת תא הזרימה
- שטיפת כיסויים
- מקום מכסה צנצנות כתמים sonicate עם אתנול (EtOH), אז עם 1 M KOH (עבור 30 דקות כל אחד). כדי למנוע KOH משקעים ETOH, לשטוף ביסודיות עם ddH2O בין שטיפה.
- חזור על EtOH ועל שלבי השטיפה KOH 1x עבור מספר כולל של ארבע שטיפה (שני EtOH ושני KOH). לאחסן כיסויים נקיים בdH2O בצנצנות כתמים.
- חותכים את צינורות הטעינה והיציאה (באורך 8 ס"מ) באמצעות סכין גילוח נקי והכנסו לחורים בשקופית זכוכית נקייה. השתמש PE-20 עבור פנימה ו PE-60 לשקע. אפוקסי למשך 5 דקות כדי לאבטח את הצינורות ולקצץ את כל הצינורות העודפים.
הערה: שקופיות זכוכית מודדות 2'' x 3'' x 1 מ"מ. חורים נקדחו בזוגות 15 מ"מ זה מזה. כל זוג יוצר כל קצה של ערוץ יחיד. הצינורות עם המזהה הקטן יותר משמשים להיכנס, כמו זה מפחית את נפח מת במעלה הזרם ולכן את זמן ערבוב הנדרש(איור 1). - מסדרים בשורה ומחילים סרט דבק דו-צדדי חתוכים מראש עם תבנית ערוץ לגזור מעל חורים על מגלשת הזכוכית. החלק עם מלקות פלסטיק כדי להשיג חותם טוב.
הערה: הקלטת הדו-צדדית המשמשת בניסוי זה היא בעובי של 120 μm, והערוצים ברוחב של 2 מ"מ ובאורך 15 מ"מ. ערוצים נחתכים באמצעות מדפסת סכין(טבלת חומרים). ניתן להתאים עד ארבעה ערוצים על כיסוי אחד. ראה איור 1 לקבלת תמונה של תא הזרימה. - לאחר קילוף הגיבוי, למרוח כיסוי נקי (מיובש באוויר דחוס) על הסרט ולהחליק שוב עם מלקות פלסטיק עבור חותם טוב.
- כדי לאטום את תא הזרימה .ולתת לו לרפא
2. הכנת דנ"א מסומן לרצועה
- בצינור PCR, להכין 50 μL של תמהיל תגובת PCR המכיל 0.02 U /μL פולימראז DNA נאמנות גבוהה, 200 μM dNTPs, 0.5 μM פריימר קדימה, 0.5 μM פריימר הפוך, ו 250 ng של M13mp18 וקטור DNA.
הערה: כאן, פריימר קדימה (ביוטין-CCAACTTATACGCGCCTTGC) פריימר הפוך (digoxigenin-TGACCATTAGATACATTTCGC) נבחרו להגביר אזור כ 1,000 bp ארוך, המשתרע מעמדות 6338 כדי 107 בגנום המעגלי של ה-DNA M13mp18. יש אתר אחד באמצע האזור ההתרופס. פריימר קדמי הוא 5ʹ עם digoxigenin, אשר קושר את אנטי-digoxigenin על כיסוי. פריימר הפוך הוא 5ʹ מתויג עם ביוטין, אשר קושר את החרוזים מצופה סטרפטוודין. - הכנס את צינור ה- PCR לתרמוצ'יקלר ובצע את המחזור כפי שהוצג בטבלה 1.
- נקה את מוצר ה- PCR באמצעות ערכת ניקוי PCR בהתאם לפרוטוקול היצרן.
הערה: באמצעות הערכה שצוינה בטבלת החומרים , תפוקתה-DNA האופיינית היא ~ 2 μg.
3. תחרש דנ"א וחרוזים
- להכין 10 מ"ל של מאגר A (1 M Tris-HCl [pH = 7.5], 50 מ"מ NaCl, 2 מ"מ MgCl2, 1 מ"ג / מ"ל β-Casein, 1 מ"ג / מ"ל F-127 פלורוני). דגה בשואב אבק לפחות שעה אחת.
- כדי לתפקד תא זרימה, להזריק 25 μL של שברי FAB אנטי-digoxigenin (20 μg / מ"ל) ב PBS לתוך ערוץ הזרימה. השתמש בטיפים לטעינת ג'ל כדי להתאים לצינורות PE-60. דגירה בטמפרטורת החדר (RT) במשך 30 דקות.
- לאחר הדגירה, לשטוף את הערוץ על ידי משיכת 0.5 מ"ל של מאגר A דרך הערוץ באמצעות מזרק. תקחותי לא להכניס אוויר לערוץ.
- לאחר פונקציונליזציה, הר את תא הזרימה על מיקרוסקופ הפוך. לחבר את צינור שקע למשאבת מזרק ולשים את צינור מפרצון לתוך צינור microcentrifuge המכיל חוצץ A.
- משוך באופן ידני לפחות 0.5 מ"ל של מאגר A כדי לשטוף את המערכת ולהרים את המשאבה. תנו למשאבה לפעול ב-10 μL/min למשך 5 דקות לפחות כדי לצייד את המערכת.
- כדי להכין את החרוזים(טבלת חומרים),מערבולת בקבוק המניות של חרוזים ופיפטה 1.6 μL של 10 מ"ג / מ"ל חרוזי מניות לתוך 50 μL של מאגר A, אז מערבולת.
- באמצעות מפריד מגנטי, פיפטה החוצה המאגר וssspend ב 50 μL של מאגר A, ולאחר מכן מערבולת.
- חזור על שלב 3.7 2x עבור סך של שלוש שטיפה. לשטיפה האחרונה, resuspend ב 100 μL של מאגר A ומערבולת כדי להשיג ריכוז סופי של 160 μg / מ"ל.
- כדי לתמצת את ה-DNA והחרוזים, תחילה להכין 480 μL של 0.5 pM מסומן כמצע DNA במאגר A. לאחר מכן, פיפטה ב 20 μL של 160 μg / מ"ל השעיית חרוז, הקפד למערבולת החרוזים לפני pipetting. מניחים על מסובב למשך 3 דקות.
- לאחר 3 דקות, מיד לטעון לתוך ערוץ בקצב זרימה של 10 μL / דקה עבור ~ 15 דקות או עד מספיק חרוז קשור נצפתה.
הערה: חרוזים לא צריך להיות כל כך צפוף ארוז שהם אינטראקציה אחד עם השני על פני השטח (ראה סעיף דיון). - כדי לשטוף את הערוץ של כל החרוזים חינם, לעבור את צינור החירום לצינור טרי של חוצץ A ולזרום ב 50 μL / דקה לפחות 10 דקות או עד אין חרוזים רופפים נצפו.
4. איסוף וניתוח נתונים
- כדי להתכונן לאיסוף נתונים, הכן צינור הכנסה לתוך צינור microcentrifuge המכיל לפחות 100 μL של NdeI (0.25-4.00 U/mL) במאגר א' הנמך את המגנט הקבוע מעל ערוץ הזרימה, ומקם את מקור האור מחוץ לציר עבור הדמיה darkfield.
הערה: שני מגנטים נדירים טבעתי כדור הארץ, אפוקסיחד, מוחזקים 8 מ"מ מעל פני השטח הפעיל של ערוץ הזרימה באמצעות הודעה אופטית cantilevered במהלך איסוף נתונים. מנורת צוואר אווז משמשת לקורס האור מחוץ לציר. - השתמש במיקרוסקופ מסחרי, מצלמת וידאו ותוכונת איסוף נתונים(טבלת חומרים). בתוכנה, לחץ על הכרטיסיה "חשיפה" והגדר "זמן חשיפה" ל- 10 ms. לחץ על "Timelapse" tab והגדר "ספירתתמונות " ל- 600, "משך" עד 20 דקות ו "מרווחזמן " ל- 2 שניות. לחץ על "הפעל" כדי להתחיל באיסוף נתונים.
- על משאבת מזרק, להגדיר את קצב הזרימה ל 150 μL / דקה ונפח הזרקה 80 μL. לחץ"לרוץ " ב 1 דקות לתוך איסוף נתונים. לאחר ההזרקה, כבה את המשאבה וסגור את השסתום כדי למנוע זרימה במהלך איסוף הנתונים.
- לאחר איסוף הנתונים, פתח את תוכנת ניתוח התמונה (Table of Materials). תחת הכרטיסיה "קובץ", בחר "ייבוא" | "רצף תמונה". אתר את קבצי התמונה בתפריט המוקפץ ולחץ על "פתח".
- הגדר את הסף על-ידי בחירה באפשרות "התאם את האחיזה"תחת התפריט הנפתח " תמונה ". השתמש בשורת המחוון כדי להגדיר את ערך הסף כדי לזהות נקודות בהירות המתאימות לחרוזים בתמונה.
- ספור את הנקודות הבהירות בכל מסגרת על-ידילחיצה על" נתח חלקיקים"בתפריט הנפתח " נתח". לחץOKעל " אישור ", ולאחר מכןבחר" כן " כדי לעבד את כל התמונות. שמור את קובץ התוצאות.
הערה: אפשרות זו תשמור קובץ נתונים המכיל את מספר החרוזים בכל מסגרת וידאו מוקלטת. - פתח את תוכנת ניתוח נתונים (טבלת חומרים) וייבוא קובץ התוצאות על-ידי לחיצה על "ייבואמקובץטקסט " בתפריט הנפתח " קובץ ". התווה את נתוני ספירת החרוזים לעומת הזמן.
- התאם את מספר החרוזים לעומת הזמן על-ידי לחיצה על "התאמהעקומה"בתפריט הנפתח " נתח ". בחר את המשוואה "מעריך טבעי" ולחץ על "נסה להתאים" | "בסדר" .כן,בסדר.
הערה: יש לכלול רק את הנתונים שנרשמו לאחר הזרקת הדגימה באזור ההתאמה. פרמטר ההתאמה במעריך של פונקציית ההתאמה יהיה קצב המחשוף.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
באמצעות טכניקה זו, שיעורי SSDC של NdeI נמדדו עבור מגוון של ריכוזי חלבון (0.25-4.00 U/mL) בשני ריכוזים שונים של מגנזיום (2 mM ו 4 mM). כל אחד מהתנאים הללו שוכפל לפחות פעמיים, עם כמה מאות עד 1,000 DNAs קשורים לכל ניסוי. איור 2 מתאר את העיצוב הניסיוני. איור 3 מציג דוגמאות של פרטי איסוף וניתוח נתונים. איור 4 ממחיש כיצד הקצב תלוי בריכוז החלבון בשני ריכוזי המגנזיום. ניתן לצפות כי בריכוזי חלבון נמוכים מספיק, הקצב הוא פרופורציונלי לחלבון ולא תלוי במגנזיום. עבור ריכוזי חלבון גבוהים מספיק, הקצב תלוי במגנזיום אך אינו תלוי בריכוז חלבון.
| שלב | תיאור | טמפרטורת (צלסיוס) | זמן (ים) |
| 1 | פירוק (פירוק) | 98 | 30 |
| 2 | להמיס | 98 | 10 |
| 3 | אנאלי (199) | 60 | 30 |
| 4 | להרחיב | 72 | 30 |
| 5 | הארכה סופית | 72 | 120 |
טבלה 1: פרמטרי PCR. מוצגים טמפרטורות ומשכים של שלבי התוכנית thermocycler המשמשים בשלב 2.2 של הפרוטוקול. השלבים המסתים, האנאליים וההרחבה (שלבים 2, 3 ו- 4) חוזרים על עצמם פי 30.
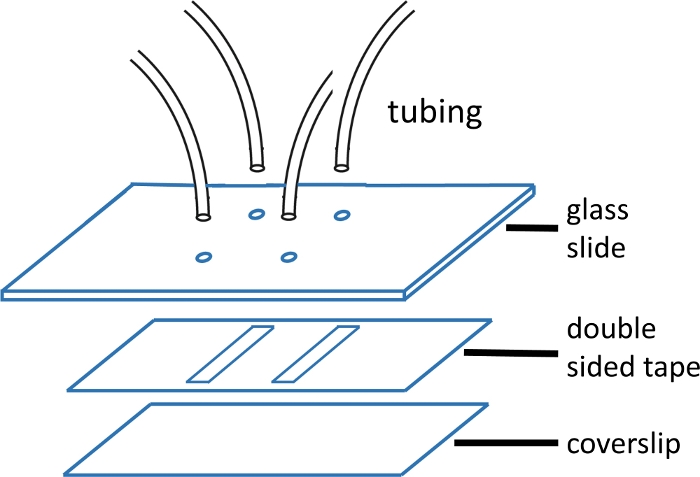
איור 1: בניית תאי זרימה מיקרו-נוזלים.
מגלשת הזכוכית העליונה (2'' x 3', 1 מ"מ עבה) הוא מראש נקדח עם חורים התואמים את תבנית הערוץ. צינורות הכניסה והשקע מוכנסים לתוך החורים וקובעים עם אפוקסי לפני חיבור סרט וזכוכית כיסוי. הקלטת הדו-צדדית נחתכת מראש עם תבנית ערוץ. השקופית התחתונה (#1 זכוכית #1.5) נוקתה בעבר באמצעות הפרוטוקול המתואר בטקסט הראשי. לאחר ההרכבה, קצה זכוכית הכיסוי אטום עם אפוקסי. לחץ כאן כדי להציג גירסה גדולה יותר של נתון זה.

איור 2: עיצוב ניסיוני.
(א)שיטה של תחכום DNA. דנ"א דו-גדילי (1 kbp) המסומן עם digoxigenin בקצה 5ʹ מחובר לפני השטח של תא הזרימה באמצעות אינטראקציה אנטידיגוקסיגן-digoxigenin. הסיום 3ʹ ה-DNA, המסומן בביוטין, מחובר למיקרו-חרוזים באמצעות אינטראקציה סטרפטוודין-ביוטין. אתר המחשוף של NdeI ממוקם במרכז הדנ"א. (ב)התקנה ניסיונית במהלך איסוף נתונים המציגה מיקום מגנט ואובייקטיבי. המגנט הקבוע שומר על כוח חלש כלפי מעלה על החרוז במהלך תגובת המחשוף. לחץ כאן כדי להציג גירסה גדולה יותר של נתון זה.
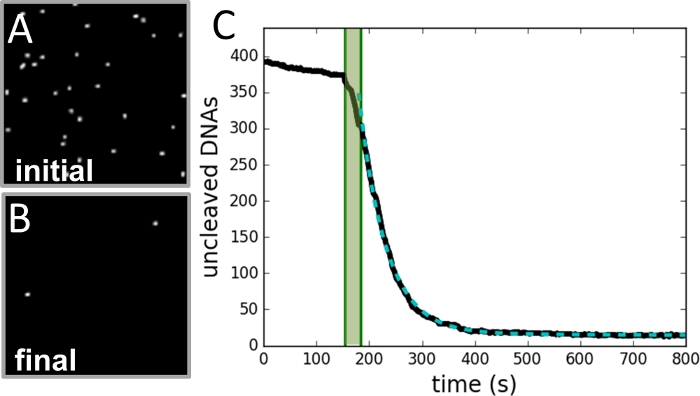
איור 3: דוגמה לאיסוף וניתוח נתונים.
(א)תמונה של אזור חרוזים שצולמו לפני תגובת המחשוף היא יזם. (ב)תמונה של אותו אזור לאחר השלמת התגובה. (ג)מגרש של מספר חרוזים לעומת זמן (עקומה שחורה) כפי שנקבע מכל מסגרת של רשומת וידאו. האזור הירוק המוצל מסמן את תקופת הזרקת האנזים ולא כלול בהתאמה. הנתונים מתאימים לעקומה מעריכית אחת (עקומה ירוקה מקווקות), עם קבוע ריקבון השווה לקצב התגובה. לחץ כאן כדי להציג גירסה גדולה יותר של נתון זה.

איור 4: מחשוף NdeI תלוי בריכוזים של חלבון ומגנזיום.
התוויית קצב SSDC נמדד של NdeI עבור מגוון ריכוזי חלבון בשני ריכוזים שונים של מגנזיום: 2 mM (עיגולים כחולים) ו 4 mM (ריבועים ירוקים). קווי שגיאה מייצגים את SEM. הקימורים המ מקווקווי מגמה אינם מייצגים התאמות בתיאוריה. לחץ כאן כדי להציג גירסה גדולה יותר של נתון זה.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
הפרוטוקול יכול לשמש כדי למדוד את הקינטיים של כל מערכת SSDC, בתנאי שהפרדת הגדיל נצפתה במהלך הניסוי. זיהוי המחשוף מושפע מהתבוננות בניתוק החרוז הרצועה ולכן מסמן את מידת הפרדת הגדילים. כל השלבים הקודמים מתרחשים לפני זיהוי המחשוף; לכן, רק זמן המעבר הכולל נרשם.
כיסוי תא הזרימה הוא פונקציונלי באמצעות ספיגה לא ספציפית של חלבון נוגדן לזכוכית נקייה. זכוכית לא מנוקה מספיק עלולה להשפיע על כריכת הנוגדן. ברצועה, צפיפות חרוזים צריכה להיות נמוכה מספיק, כך חרוזים לא אינטראקציה. צפיפות פני השטח של נקודות מצורף יכול להיות נשלט על ידי ריכוז של נוגדנים במהלך פונקציונליזציה. המספר הכולל של חרוזים תלוי בגודל שדה התצוגה. במקרה זה, כמה מאות עד 1,000 חרוזים קשורים היו מספיקים לסטטיסטיקה טובה ונמנע אינטראקציות חרוזים-חרוז. במהלך הזרקת חרוז, רצועה פני השטח היה במעקב באמצעות וידאו חי. הזרקת חרוזים הופסקה כאשר ספירת חרוזים משוערת הייתה בין 500 ו 1,000 חרוזים.
שיעורי המחשוף המהירים ביותר שניתן למדוד במדויק מוגבלים על ידי זמן הערבוב של תא הזרימה. זמן ערבוב בתאי זרימה למינאר מושפע ממספר גורמים. דיפוזיה אל פני השטח היא שלב מפתח; לכן, זמן הערבוב תלוי במקדם דיפוזיה של המגיב. המיזה המשמעותית המתרחשת בצינורות הכניסה, המעביר את הדגימה לערוץ הזרימה ממאגר המדגם, יכולה להגדיל את הזמן הדרוש כדי להבטיח ערבוב הולם במשטח התגובה בערוץ. נמצא כי זמן ערבוב יכול להיות מופחת על ידי הפחתת נפח מת במעלה הזרם והגדלת קצב הזרימה. עם קוטר פנימי של 380 μm ואורך מרבי של 8 ס"מ עבור צינורות ההזרפה (וקצב זרימה של 150 μL/min), נמצא כי זמן ההזרקה ניתן להפחית ~ 20 s מבלי להשפיע על קצב המחשוף נמדד. מאז זמן ערבוב תלוי מקדם דיפוזיה של המגיב, זה צריך להיקבע בנפרד עבור כל אנזים או מפעיל מחשוף למד.
שיטת הרצועה מאפשרת קרע ברצועה לא ספציפית, ככל הנראה בשל ניתיקות של תסביך הנוגדם digoxigenin או שחרור של הנוגדן מפני השטח. התוצאה היא שיעור אובדן חרוז רקע לשחזור הנוכחי לפני הזרקת אנזים של ~ 3 x 10-4 s-1. ניתן לתקן אפקט שיטתי זה על-ידי הפחתה של קצב הרקע מקצב המחשוף הנמדד, או על-ידי מידול הרקע במשוואה המתאימה. עם זאת, שיעורי מחשוף נמוכים יותר מאשר הגבול התחתון הזה יימדדו פחות אמין.
פסיבציה משטח לא מושלמת יכולה להוביל לרצועה לא תקינה. זה מוביל גם שברים מוגברים של חרוזים "תקועים", אשר אינם נעלם במהלך the הניסוי, או improperly tethered חרוזים קשורים כראוי, אשר לנתק מפני השטח לאט מאוד. פעולה זו יוצרת תוכנית בסיסית גבוהה יותר ואולי משופעת בנתונים המעובדים. נמצא כי עם פתקי כיסוי נקיים כראוי ופתרון מניות β-casein, אפקטים אלה היו מינימליים עבור רוב ערכות הנתונים. עבור ערכות נתונים מזדמנות המוצגות כך, שינוי הפונקציה המתאימה (כך שתכלול תוכנית בסיסית משופעת) יכול לתקן עבור אפקט זה.
ניתן להרחיב את הפרוטוקול הנוכחי במספר דרכים. בידוד נוסף של צעדים מכניים לאחר כריכת אתר היעד יכול להתבצע בפורמט מחייב מראש, שבו חלבון מוזרק בהיעדר קופאקטורים חיוניים. רעיון זה נבדק על ידי הזרקת NdeI בהיעדר מגנזיום. בתנאים אלה, החלבון נקשר לאתר הקוניט שלו אך אינו כובל את ה-DNA. הזרקת מגנזיום לאחר שלב מחייב זה מפעיל את המחשוף וכתוצאה מכך אובדן חרוז מהיר. ההתקנה הניסיונית מאפשרת גם שליטה על קונפורמציה DNA ומתח על ידי שינוי תצורת מגנט או הוספת זרימה. תחת הכוחות הנמוכים בניסויים אלה, ה-DNA הופך לבולע חלקית. שינוי הכוחות מעט יכול להיות השפעה דרמטית על ההתאמה של ה-DNA. לדוגמה, בתנאי מאגר שבהם חיפוש היעד הוא הגבלת קצב, שינוי ההתאמה של ה-DNA יכול לבדוק את ההשפעה של קפיצה על חיפוש היעד. בתנאי חיץ שבהם שלב ההידרוליזה הוא הגבלת קצב, שינוי הכוח יכול לבדוק את ההשפעה של מתח ה-DNA על הידרוליזה אג"ח זרחן. יש לנכון לכך כי תחת הגדלה נמוכה בשימוש, שינויים קונפורמציה אלה לא ניתן לצפות. התנועות הקטנות וכתוצאה מכך במיקום חרוז צריך להיות במעקב תחת הגדלה גבוהה יותר כדי לוודא כי קונפורמציה DNA ניתן לשלוט.
ניתן להרחיב את ניתוח הנתונים במגוון דרכים. עבודה זו מחילה שיטת ספירת חרוזים פשוטה ולאחריה התאמת עקומה של פונקציה מעריכית אחת. ניתן להשתמש בשיטות המבוססות על ניתוח זמן מגורים,כמו גם 20. לאחר מכן ניתן לנתח הפצות של זמני מגורים של DNAs בודדים באמצעות התאמת עקומה או באמצעות טכניקות מתוחכמות יותר, כגון השיטה הכללית שלרגעים 21.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
למחברים אין ניגודי עניינים לחשוף.
Acknowledgments
עבודה זו נתמכה על ידי מענק הקרן הלאומית למדע MCB-1715317.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



