Summary
Um método altamente paralelo para medir o decote específico do DNA no nível da molécula única é descrito. Este protocolo demonstra a técnica utilizando a restrição endonuclease NdeI. O método pode ser facilmente modificado para estudar qualquer processo que resulte em decote de DNA específico do local.
Abstract
O decote de DNA específico do local (SSDC) é um passo fundamental em muitos processos celulares, e é crucial para a edição de genes. Este trabalho descreve um ensaio cinético capaz de medir SSDC em muitas moléculas de DNA únicas simultaneamente. DNAs substratos amarrados por contas, cada um contendo uma única cópia da sequência de destino, são preparados em um canal de fluxo microfluido. Um ímã externo aplica uma força fraca às contas paramagnéticas. A integridade de até 1.000 DNAs individuais pode ser monitorada visualizando as microesferas sob imagens de campo escuro usando um objetivo de ampliação de campo largo e baixa. A injeção de uma restrição endonuclease, NdeI, inicia a reação do decote. A microscopia de vídeo é usada para registrar o momento exato de cada decote de DNA observando o quadro em que a conta associada se move para cima e para fora do plano focal do objetivo. A contagem de contas quadro a quadro quantifica a reação, e um ajuste exponencial determina a taxa de reação. Este método permite a coleta de dados quantitativos e estatisticamente significativos sobre reações SSDC de molécula única em um único experimento.
Introduction
O decote de DNA específico do local (SSDC) é um passo fundamental em muitas transações genômicas. Por exemplo, os sistemas de modificação de restrição bacteriana (RM)1 e CRISPR2 protegem as células contra ataques por phages e plasmídeos, reconhecendo e cortando DNA estranho em sequências específicas. No tipo II RM, as endonucleases de restrição (REs) reconhecem sequências curtas de 4 a 8 pares de bases (bp) através de interações proteicas-nucleicas3. Endonucleases associados ao CRISPR, como o Cas9, ligam-se a locais via hibridização do local alvo com crRNAs vinculados aos endonucleases4. A criação de quebras duplas encalhadas específicas do local (DSBs) também são o primeiro passo em muitos eventos de recombinação de DNA5. Por exemplo, a diversidade de regiões de ligação de antígenos criadas pela recombinação V(D)J requer o reconhecimento e o decote de locais de destino específicos6. Alguns transposons são conhecidos por atingir sequências específicas de DNA, bem como7. Não surpreende que muitos núcleos específicos do site envolvidos nesses processos, como o Cas9, sejam um componente-chave das tecnologias de edição de genes8. Além disso, novos endonucleases específicos do local (ou seja, núcleos de dedo de zinco9 e TALENS10) também foram projetados para editar genomas.
Muitos métodos têm sido empregados para medir a cinética do decote específico do local dos ácidos nucleicos. Estes incluem análise de gel, fluorescência11,,12e métodos baseados em sequenciamento13. Um grande avanço foi alcançado com o amarramento de microesferas, que permite que DSBs em moléculas únicas de DNA sejam detectados pelo movimento de uma conta após a separação da vertente. Nestes métodos, diferentes tipos de forças são empregadas para garantir a separação da vertente e o movimento do pós-decote da conta. Em um caso, armadilhas ópticas têm sido usadas para medir o decote do DNA pelo EcoRV14. Nesses experimentos, a busca de alvos é o objetivo da investigação, com condições otimizadas para que a vinculação específica do local seja a etapa limitante da taxa. Uma desvantagem das armadilhas ópticas é que apenas um único DNA pode ser observado de cada vez. Além disso, uma força de puxar grande periódica deve ser aplicada para testar a separação da vertente.
Outra técnica usa uma combinação de fluxo e forças magnéticas fracas para puxar a conta de forma contínua15. Desta forma, a difusão limitada do decote por NdeI é medida. O método utilizado permite a medição simultânea de várias centenas de DNAs ao mesmo tempo, permitindo que a significância estatística seja alcançada em um único experimento. Experimentos baseados em pinças magnéticas também foram usados. Em um desses estudos, foi estudada uma integração retroviral por meio da inclusão de um DSB na inserção oligonucleotídeo16. A integração bem sucedida resultou na incorporação do DSB no DNA amarrado e na perda da conta anexada. Em estudo semelhante da restrição do tipo III dependente do ATP EcoPI, dezenas de DNAs foram observados em um único experimento17. Pinças magnéticas têm a vantagem de que a tensão, bem como o looping de DNA, podem ser controlados e monitorados durante a reação.
Apresentado aqui é um método de molécula única altamente paralelo para medir cinética SSDC, que se aproveita de melhorias recentes no tethering em larga escala de DNAs. Este método é uma melhoria e extensão dos métodos anteriores usados para medir a replicação de DNA18, comprimento do contorno do DNA19e decote por REs15. Nesta técnica, DNAs lineares contendo uma única cópia da sequência de reconhecimento são preparados com biotina em uma extremidade e digoxigenina na outra. A biotina liga streptavidin, que é covalentemente ligado a uma microesfera paramagnética. Os complexos de contas de DNA são injetados em um canal microfluido que foi funcionalizado com fragmentos da FAB anti-digoxigenina. O DNA então se liga aos pontos de fixação da superfície através da ligação da digoxigenina aos fragmentos da FAB adsorvidas. Forças magnéticas fracas aplicadas com um ímã permanente impedem que a conta grude não especificamente na superfície. As amostras podem ser injetadas rapidamente (<30 s) no canal de fluxo para ativar a reação do decote. O fluxo é desligado durante a coleta de dados. À medida que cada DNA é cortado, o tempo exato do decote pode ser determinado registrando o quadro em que a conta se move para cima e para fora do plano focal do objetivo, desaparecendo assim do registro de vídeo. Uma contagem quadro a quadro de contas remanescentes pode ser usada para quantificar o progresso da reação.
Apresentado abaixo está o protocolo completo, bem como exemplos de dados coletados usando o NdeI. Como exemplo de como a técnica pode ser aplicada, as taxas de decote para uma série de concentrações proteicas são medidas em duas concentrações diferentes de magnésio, um coator metálico essencial. Embora esta aplicação do protocolo use NdeI, o método pode ser adaptado para uso com qualquer nuclease específica do local, variando o design do substrato de DNA.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Fazer a célula de fluxo
- Lavando as tampas
- Coloque manchas em frascos de coloração e sonicate com etanol (EtOH), depois com 1 M KOH (por 30 min cada). Para evitar a precipitação KOH em EtOH, enxágue bem com ddH2O entre as lavagens.
- Repita tanto o EtOH quanto as etapas de lavagem KOH 1x para um número total de quatro lavagens (duas EtOH e duas KOH). Armazene tampas limpas em ddH2O em frascos de coloração.
- Corte os tubos de carga e saída (8 cm de comprimento) usando uma navalha limpa e insira em orifícios em uma lâmina de vidro limpa. Use PE-20 para entrada e PE-60 para tomada. Epóxi por 5 minutos para fixar tubos e cortar qualquer excesso de tubulação.
NOTA: Slides de vidro medem 2'' x 3'' x 1 mm. Os furos são perfurados em pares com 15 mm de distância. Cada par forma cada extremidade de um único canal. O tubo com o ID menor é usado para a entrada, pois isso reduz o volume morto a montante e, portanto, o tempo de mistura necessário(Figura 1). - Alinque-se e aplique fita dupla face pré-cortada com padrão de canal cortado sobre orifícios na lâmina de vidro. Suavize com fórceps plásticos para obter um bom selo.
NOTA: A fita de dupla face usada neste experimento tem 120 μm de espessura, e os canais têm 2 mm de largura e 15 mm de comprimento. Os canais são cortados usando uma impressora de facas(Tabela de Materiais). É possível encaixar até quatro canais em um único coverlip. Consulte a Figura 1 para obter uma imagem da célula de fluxo. - Depois de descascar o apoio, aplique uma mancha de cobertura limpa (seca com ar comprimido) sobre a fita e alise novamente com fórceps plásticos para uma boa vedação.
- Epóxi a borda fora da tampa para selar a célula de fluxo e deixá-la curar.
2. Preparação de DNA rotulado para amarração
- Em um tubo PCR, prepare 50 μL de mix de reação PCR contendo 0,02 U/μL de polimerase de DNA de alta fidelidade, 200 dNTPs μM, 0,5 μM primer para frente, 0,5 μM primer reverso e 250 ng de DNA vetorial M13mp18.
NOTA: Aqui, a cartilha dianteira (biotin-CCAACTTAATCGCCTTGC) e o primer reverso (digoxigenina-TGACCATTAGATACATTTCGC) foram escolhidos para amplificar uma região de aproximadamente 1.000 bp de comprimento, abrangendo das posições 6338 a 107 no genoma circular do DNA M13mp18. Há um único local ndei no meio da região amplificada. A cartilha dianteira é 50 rotulada com digoxigenina, que liga a anti-digoxigenina na tampa. O primer reverso é 50 rotulado com biotina, que liga as contas revestidas de streptavidin. - Insira o tubo PCR no termociclador e siga o ciclo conforme mostrado na Tabela 1.
- Purifique o produto PCR com um kit de limpeza PCR seguindo o protocolo do fabricante.
NOTA: Utilizando o kit especificado na Tabela de Materiais,o rendimento típico do DNA é de ~2 μg.
3. Amarração de DNA e contas
- Prepare 10 mL de tampão A (1 M Tris-HCl [pH = 7,5], 50 mM NaCl, 2 mM MgCl2, 1 mg/mL β-Casein, 1 mg/mL Pluronic F-127). Degas em um desiccator de vácuo por pelo menos 1 h.
- Para funcionalizar a célula de fluxo, injete 25 μL de fragmentos FAB anti-digoxigenina (20 μg/mL) em PBS no canal de fluxo. Use pontas de carregamento de gel para caber na tubulação PE-60. Incubar em temperatura ambiente (RT) por 30 minutos.
- Após a incubação, lave o canal puxando 0,5 mL de buffer A através do canal usando uma seringa. Tome cuidado para não introduzir ar no canal.
- Após a funcionalização, monte a célula de fluxo em um microscópio invertido. Ligue o tubo de saída a uma bomba de seringa e coloque o tubo de entrada em um tubo de microcentrifuuagem contendo tampão A.
- Puxe manualmente pelo menos 0,5 mL de tampão A para lavar o sistema e preparar a bomba. Deixe a bomba funcionar a 10 μL/min por pelo menos 5 minutos para equilibrar o sistema.
- Para preparar as contas(Tabela de Materiais),vórtice a garrafa de caldo de contas e pipeta 1,6 μL das contas de caldo de 10 mg/mL em 50 μL de tampão A, depois vórtice.
- Usando um separador magnético, pipeta para fora do buffer e resuspend em 50 μL de tampão A, em seguida, vórtice.
- Repita o passo 3,7 2x para um total de três lavagens. Para a última lavagem, resuspense em 100 μL de tampão A e vórtice para alcançar uma concentração final de 160 μg/mL.
- Para complexar o DNA e as contas, primeiro prepare 480 μL de 0,5 pM rotulado substrato de DNA no buffer A. Em seguida, pipeta em 20 μL de 160 μg/mL suspensão de contas, certificando-se de vórtice das contas antes da pipetação. Coloque em um rotador por 3 minutos.
- Após 3 min, carregue imediatamente no canal a uma vazão de 10 μL/min por ~15 min ou até que seja observada a amarração de contas suficiente.
NOTA: As contas não devem ser tão densamente embaladas que interagem entre si na superfície (ver seção de discussão). - Para lavar o canal de todas as contas livres, troque o tubo de entrada para um tubo fresco de tampão A e flua a 50 μL/min por pelo menos 10 minutos ou até que não sejam observadas contas soltas.
4. Coleta e análise de dados
- Para se preparar para a coleta de dados, coloque o tubo de entrada em um tubo de microcentrifuuge contendo pelo menos 100 μL de NdeI (0,25-4,00 U/mL) no buffer A. Abaixe o ímã permanente sobre o canal de fluxo e posicione a fonte de luz fora do eixo para imagens de darkfield.
NOTA: Dois ímãs de terras raras anulares, epóxidos juntos, são mantidos 8 mm acima da superfície ativa do canal de fluxo usando um post óptico cantilevered durante a coleta de dados. Uma lâmpada de pescoço de ganso é usada para o curso de luz fora do eixo. - Use um microscópio comercial, câmera de vídeo e software de coleta de dados(Tabela de Materiais). No software, clique na guia "Exposição" e defina "Tempo de Exposição" a 10 ms. Clique em "Timelapse" tab e definir "Contagem de Imagens" para 600, "Duração" a 20 minutos, e "Intervalo" para 2 s. Clique em "Executar" para iniciar a coleta de dados.
- Em uma bomba de seringa, ajuste a taxa de fluxo para 150 μL/min e o volume de injeção para 80 μL.Pressione" Executar " a 1 min na coleta de dados. Após a injeção, desligue a bomba e feche a válvula para evitar o fluxo durante a coleta de dados.
- Uma vez coletados dados, o software de análise de imagens aberta(Tabela de Materiais). Sob a guia "Arquivo", escolha "Importar" | "Sequência de Imagens". Localize os arquivos de imagem no menu pop-up e clique em "Abrir".
- Defina o limiar escolhendo "Ajustar Theshold" sob o menu de retirada "Imagem". Use a barra de controle deslizante para definir o valor do limiar para identificar pontos brilhantes correspondentes às contas na imagem.
- Conte os pontos brilhantes em cada quadro clicando em "Analisar partículas" no menu pull-down "" Analyze". Clique em "OK", e escolha "Sim" para processar todas as imagens. Salve o arquivo de resultados.
NOTA: Isso salvará um arquivo de dados que contém o número de contas em cada quadro de vídeo gravado. - Abra o software de análise de dados(Tabela de Materiais) e importe o arquivo de resultados clicando em "Importar de Arquivo de Texto" no menu de retirada " "arquivo". Plote os dados da contagem de contas versus o tempo.
- Encaixe o número de contas vs. tempo clicando em "Curve Fit" no menu pull-down "Analyze". Escolha a equação "Expoente Natural" e clique em "Try Fit" | "OK".
NOTA: Somente os dados registrados após a injeção da amostra devem ser incluídos na região de montagem. O parâmetro de ajuste no expoente da função de montagem será a taxa de decote.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Utilizando esta técnica, as taxas de SSDC de NdeI foram medidas para uma gama de concentrações proteicas (0,25-4,00 U/mL) em duas concentrações diferentes de magnésio (2 mM e 4 mM). Cada uma dessas condições foi replicada pelo menos duas vezes, com algumas centenas a 1.000 DNAs amarrados por experimento. A Figura 2 descreve o design experimental. A Figura 3 mostra exemplos de detalhes de coleta e análise de dados. A Figura 4 ilustra como a taxa depende da concentração de proteínas nas duas concentrações de magnésio. Observa-se que em concentrações de proteínas suficientemente baixas, a taxa é proporcional à proteína e independente do magnésio. Para concentrações de proteínas suficientemente altas, a taxa depende do magnésio, mas independente da concentração proteica.
| Passo | Descrição | Temperatura (°C) | Tempo (s) |
| 1 | Desnaturação | 98 | 30 |
| 2 | Derreter | 98 | 10 |
| 3 | Anneal | 60 | 30 |
| 4 | Estender | 72 | 30 |
| 5 | Extensão Final | 72 | 120 |
Tabela 1: parâmetros pcr. São mostradas as temperaturas e as durações das etapas do programa termociclador utilizadas na etapa 2.2 do protocolo. Os degraus derretimento, anneal e extensão (etapas 2, 3 e 4) são repetidos 30x.
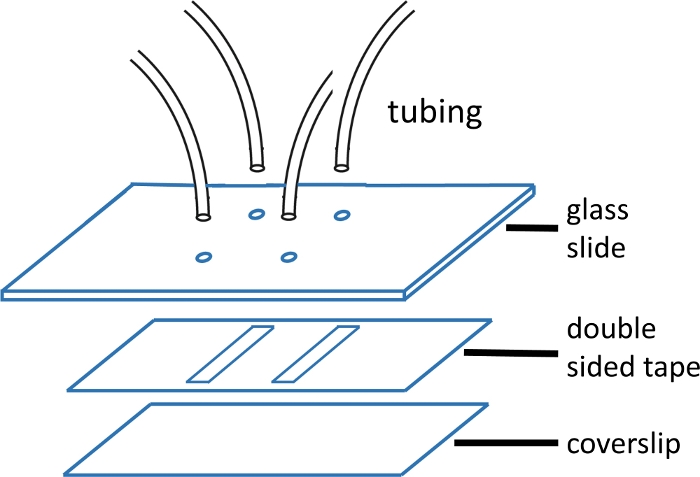
Figura 1: Construção de células de fluxo microfluido.
O slide de vidro superior (2'' x 3'', 1 mm de espessura) é pré-perfurado com furos que correspondem ao padrão do canal. Os tubos de entrada e saída são inseridos nos orifícios e fixados com epóxi antes de fixar fita e cobrir vidro. A fita de dupla face é pré-cortada com padrão de canal. O slide inferior (#1 ou #1,5 de tampa) é previamente limpo usando o protocolo descrito no texto principal. Uma vez montada, a borda do vidro de cobertura é selada com epóxi. Clique aqui para ver uma versão maior desta figura.

Figura 2: Design experimental.
(A) Método de amarração de DNA. DNA de duplaridade (1 kbp) rotulado com digoxigenina na extremidade 50 é anexado à superfície da célula de fluxo através da interação antidigoxigenina-digoxigenina. A extremidade 30 do DNA, rotulada com biotina, é anexada a uma microesfera via interação streptavidin-biotina. O local do decote NdeI está localizado no centro do DNA. (B) Configuração experimental durante a coleta de dados mostrando magnet e posição objetiva. O ímã permanente mantém uma fraca força ascendente na conta durante a reação do decote. Clique aqui para ver uma versão maior desta figura.
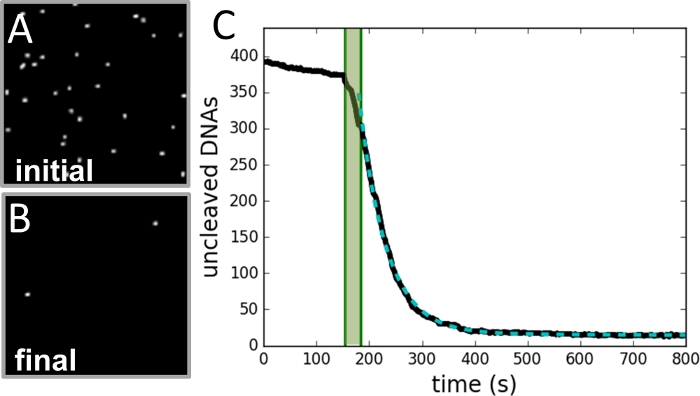
Figura 3: Exemplo de coleta e análise de dados.
(A) Imagem da região das contas tomadas antes da reação do decote ser iniciada. (B) Imagem da mesma região após a reação ser concluída. (C) Gráfico do número de contas vs. tempo (curva preta) conforme determinado a partir de cada quadro do registro de vídeo. A área verde sombreada marca o período de injeção de enzima e não está incluída no ajuste. Os dados se encaixam em uma única curva exponencial (curva verde tracejada), com uma constante de decadência igual à taxa de reação. Clique aqui para ver uma versão maior desta figura.

Figura 4: O decote NdeI depende das concentrações de proteína e magnésio.
Parcela da taxa de SSDC medido de NdeI para uma gama de concentrações proteicas em duas concentrações diferentes de magnésio: 2 mM (círculos azuis) e 4 mM (quadrados verdes). As barras de erro representam a SEM. As curvas tracejadas são linhas de tendência e não representam ajustes na teoria. Clique aqui para ver uma versão maior desta figura.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
O protocolo pode ser usado para medir a cinética de qualquer sistema SSDC, desde que a separação da vertente seja observada durante o experimento. A detecção do decote é afetada pela observação do desprendimento da conta amarrada e, portanto, marca o instante da separação do fio. Todas as etapas anteriores ocorrem antes da detecção do decote; assim, apenas o tempo total de trânsito é registrado.
O deslizamento de cobertura da célula de fluxo é funcionalizado através de adsorção não específica de proteína de anticorpos para o vidro limpo. O vidro insuficientemente limpo pode afetar a ligação do anticorpo. No tethering, a densidade das contas deve ser baixa o suficiente para que as contas não interajam. A densidade superficial dos pontos de fixação pode ser controlada pela concentração de anticorpos durante a funcionalização. O número total de contas depende do tamanho do campo de visão. Neste caso, algumas centenas a 1.000 contas amarradas foram suficientes para boas estatísticas e evitaram interações entre contas de contas. Durante a injeção de contas, a corda superficial foi monitorada via vídeo ao vivo. A injeção de contas foi interrompida quando a contagem estimada de contas estava entre 500 e 1.000 contas.
As taxas de decote mais rápidas que podem ser medidas com precisão são limitadas pelo tempo de mistura da célula de fluxo. O tempo de mistura em células de fluxo laminar é influenciado por vários fatores. A difusão à superfície é um passo fundamental; portanto, o tempo de mistura depende do coeficiente de difusão do reagente. A cisalhamento significativa que ocorre na tubulação de entrada, que transporta a amostra para o canal de fluxo do reservatório de amostra, pode aumentar o tempo necessário para garantir a adequada mistura na superfície de reação no canal. Verificou-se que o tempo de mistura poderia ser reduzido reduzindo o volume morto a montante e aumentando a taxa de fluxo. Com um diâmetro interno de 380 μm e comprimento máximo de 8 cm para a tubulação de entrada (e taxa de fluxo de 150 μL/min), verificou-se que o tempo de injeção poderia ser reduzido para ~20 s sem afetar a taxa de decote medida. Uma vez que o tempo de mistura depende do coeficiente de difusão do reagente, deve ser determinado separadamente para cada enzimático ou ativador de decote estudado.
O método de amarração permite a ruptura não específica da corda, presumivelmente devido à dissociação do complexo digoxigenina-anticorpo ou à liberação do anticorpo da superfície. Isso resulta em uma taxa de perda de contas de fundo reprodutível presente antes da injeção de enzima de ~3 x 10-4 s-1. Este efeito sistemático pode ser corrigido para subtrair a taxa de fundo da taxa de decote medida, ou modelando o fundo na equação de montagem. No entanto, as taxas de decote mais baixas do que este limite inferior serão menos confiáveis.
A passivação imperfeita da superfície pode levar a amarras impróprias. Isso leva tanto ao aumento de frações de contas "presas", que não desaparecem durante o experimento, ou a contas indevidamente amarradas, que se dissociam da superfície muito lentamente. Isso cria uma linha de base maior e possivelmente inclinada nos dados processados. Verificou-se que, com os deslizamentos de cobertura devidamente limpos e a solução de estoque de β-casein recém-feita, esses efeitos foram mínimos para a maioria dos conjuntos de dados. Para os conjuntos de dados ocasionais que mostram isso, modificar a função de montagem (para incluir uma linha de base inclinada) pode corrigir esse efeito.
O protocolo atual pode ser estendido de várias maneiras. Um isolamento adicional das etapas mecanicistas após a vinculação do local de destino pode ser realizado usando um formato pré-vinculação, no qual a proteína é injetada na ausência de cofatores essenciais. Esta ideia é testada injetando NdeI na ausência de magnésio. Nessas condições, a proteína se liga ao seu local cognato, mas não corta o DNA. Injetar magnésio após esta etapa de ligação ativa o decote resultando em perda rápida de contas. A configuração experimental também permite o controle da conformação e tensão do DNA, variando a configuração do ímã ou adicionando fluxo. Sob as baixas forças nesses experimentos, o DNA fica parcialmente enrolado. Mudar um pouco as forças pode ter um efeito dramático na conformação do DNA. Por exemplo, sob condições de buffer em que a pesquisa de destino é limitante de taxas, variando a conformação do DNA pode testar o efeito de saltar na pesquisa de destino. Em condições tampão em que a etapa de hidrólise é limitante, variando a força pode sondar o efeito da tensão do DNA na hidrólise da ligação fosfodiester. Deve-se notar que, sob a baixa ampliação utilizada, essas alterações conformais não podem ser observadas. Os pequenos movimentos resultantes na posição de contas devem ser rastreados sob maior ampliação para verificar se a conformação do DNA pode ser controlada.
A análise dos dados pode ser estendida de várias maneiras. Este trabalho aplica um método simples de contagem de contas seguido pelo encaixe da curva de uma única função exponencial. Podem ser utilizados métodos baseados na análise do tempo de residência, bem como20. As distribuições dos tempos de residência dos DNAs individuais podem então ser analisadas via encaixe de curva ou utilizando técnicas mais sofisticadas, como o método generalizado de momentos21.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Os autores não têm conflitos de interesse para divulgar.
Acknowledgments
Este trabalho foi apoiado pela Fundação Nacional de Ciência do MCB-1715317.
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



