Summary
एकल अणु स्तर पर डीएनए के साइट-विशिष्ट दरार को मापने के लिए एक अत्यधिक समानांतर विधि का वर्णन किया गया है। यह प्रोटोकॉल प्रतिबंध एंडोन्यूक्लिज़ एनडीईआई का उपयोग करके तकनीक को दर्शाता है। साइट-विशिष्ट डीएनए दरार में परिणाम वाली किसी भी प्रक्रिया का अध्ययन करने के लिए विधि को आसानी से संशोधित किया जा सकता है।
Abstract
साइट-विशिष्ट डीएनए क्लीवेज (एसएसडीसी) कई सेलुलर प्रक्रियाओं में एक महत्वपूर्ण कदम है, और यह जीन संपादन के लिए महत्वपूर्ण है। यह काम एक साथ कई एकल डीएनए अणुओं में एसएसडीसी को मापने में सक्षम एक गतिज परख का वर्णन करता है । मनका-सीमित सब्सट्रेट डीएनए, प्रत्येक लक्ष्य अनुक्रम की एक प्रति युक्त, एक माइक्रोफ्लुइडिक प्रवाह चैनल में तैयार किए जाते हैं। एक बाहरी चुंबक पैरामैग्नेटिक मोतियों पर एक कमजोर बल लागू करता है। 1,000 व्यक्तिगत डीएनए तक की अखंडता की निगरानी एक व्यापक क्षेत्र, कम आवर्धन उद्देश्य का उपयोग करके डार्कफील्ड इमेजिंग के तहत माइक्रोमोतियों की कल्पना करके की जा सकती है। एक प्रतिबंध एंडोक्यूलीज, NdeI का इंजेक्शन, दरार प्रतिक्रिया शुरू करता है। वीडियो माइक्रोस्कोपी का उपयोग फ्रेम को देखकर प्रत्येक डीएनए दरार के सटीक क्षण को रिकॉर्ड करने के लिए किया जाता है जिसमें संबद्ध मनका उद्देश्य के फोकल प्लेन से ऊपर और बाहर जाता है। फ्रेम-बाय-फ्रेम मनका गिनती प्रतिक्रिया की मात्रा, और एक घातीय फिट प्रतिक्रिया दर निर्धारित करता है। यह विधि एक ही प्रयोग में एकल अणु एसएसडीसी प्रतिक्रियाओं पर मात्रात्मक और सांख्यिकीय रूप से महत्वपूर्ण डेटा के संग्रह की अनुमति देती है।
Introduction
साइट-विशिष्ट डीएनए क्लीवेज (एसएसडीसी) कई जीनोमिक लेनदेन में एक महत्वपूर्ण कदम है। उदाहरण के लिए, बैक्टीरियल प्रतिबंध-संशोधन (आरएम)1 और CRISPR2 सिस्टम विशिष्ट दृश्यों पर विदेशी डीएनए को पहचानने और क्लीविंग करके कोशिकाओं को फैज और प्लाज्मिड के हमले से बचाते हैं। प्रकार द्वितीय आरएम में, प्रतिबंध एंडो न्यूक्लियस (आरईएस) प्रोटीन-न्यूक्लिक एसिड इंटरैक्शन3के माध्यम से छोटे 4-8 बेस पेयर (बीपी) दृश्यों को पहचानते हैं। CRISPR से जुड़े एंडो न्यूक्लियस, जैसे Cas9, लक्ष्य स्थल के संकरण के माध्यम से साइटों को बांधते हैं, जिसमें एंडोन्यूक्लिस4से बंधे सीआरआरएनए होते हैं। साइट-विशिष्ट डबल फंसे ब्रेक (डीएसबी) का निर्माण भी कई डीएनए पुनर्संयोजन घटनाओं में पहला कदम है5। उदाहरण के लिए, वी (डी) जे पुनर्संयोजन द्वारा बनाए गए एंटीजन बाध्यकारी क्षेत्रों की विविधता के लिए विशिष्ट लक्ष्य स्थलों की मान्यता और दरार की आवश्यकता होती है6। कुछ ट्रांसपोसंस विशिष्ट डीएनए दृश्यों को लक्षित करने के लिए जाना जाता है, साथ ही7। आश्चर्य नहीं कि इन प्रक्रियाओं में शामिल कई साइट-विशिष्ट नाभिक, जैसे Cas9, जीन संपादन प्रौद्योगिकियों का एक प्रमुख घटकहैं 8। इसके अलावा, जीनोम को संपादित करने के लिए उपन्यास साइट-विशिष्ट एंडोन्यूक्लियस(यानी, जिंक फिंगर न्यूक्लियस 9 और टैलैंस10)को भी इंजीनियर किया गया है।
न्यूक्लिक एसिड के साइट-विशिष्ट दरार के काइनेटिक्स को मापने के लिए कई तरीकों को नियोजित किया गया है। इनमें जेल विश्लेषण , फ्लोरेसेंस11,12और अनुक्रमण आधारित विधियां13शामिल हैं । माइक्रोमोतियों के तार के साथ एक बड़ी उन्नति हासिल की गई थी, जो डीएनए के एकल अणुओं में डीएसबी को स्ट्रैंड अलगाव के बाद एक मनका की गति से पता लगाने की अनुमति देता है । इन तरीकों में, विभिन्न प्रकार की ताकतों को स्ट्रैड जुदाई और मनका के बाद दरार की गति सुनिश्चित करने के लिए नियोजित किया जाता है। एक मामले में, EcoRV14द्वारा डीएनए के दरार को मापने के लिए ऑप्टिकल जाल का उपयोग किया गया है । इन प्रयोगों में, लक्ष्य खोज जांच का उद्देश्य है, जिसमें अनुकूलित शर्तें हैं ताकि साइट-विशिष्ट बाध्यकारी दर सीमित कदम हो। ऑप्टिकल जाल की एक खामी यह है कि एक समय में केवल एक ही डीएनए देखा जा सकता है। इसके अलावा स्ट्रैंड सेपरेशन के लिए टेस्ट के लिए आवधिक बड़ी पुलिंग फोर्स लगानी पड़ती है ।
दूसरी तकनीक15को मनका पर खींचने के लिए प्रवाह और कमजोर चुंबकीय बलों के संयोजन का उपयोग करती है । इस तरह, NdeI द्वारा प्रसार सीमित दरार मापा जाता है। नियोजित विधि एक बार में कई सौ DNAs के एक साथ माप के लिए अनुमति देता है, सांख्यिकीय महत्व के लिए एक ही प्रयोग में प्राप्त करने के लिए अनुमति देता है । चुंबकीय चिमटी पर आधारित प्रयोग भी किए गए हैं। ऐसे ही एक अध्ययन में, एक रेट्रोवायरल इंटीग्रल एक्टिंग का अध्ययन प्रविष्टि ओलिगोन्यूक्लियोटाइड16में डीएसबी को शामिल करके किया गया था। सफल एकीकरण के परिणामस्वरूप सीमित डीएनए में डीएसबी का समावेश हुआ और संलग्न मनका की हानि हुई। एटीपी-निर्भर प्रकार III प्रतिबंध एंडोन्यूलेस इकोपी के इसी तरह के अध्ययन में, दसियों डीएनए एक ही प्रयोग17में देखे गए थे। चुंबकीय चिमटी लाभ है कि तनाव, साथ ही डीएनए पाशन, नियंत्रित किया जा सकता है और प्रतिक्रिया के दौरान निगरानी पकड़ो ।
यहां प्रस्तुत एसएसडीसी काइनेटिक्स को मापने के लिए एक अत्यधिक समानांतर एकल अणु विधि है, जो DNAs के बड़े पैमाने पर टेदरिंग में हाल के सुधारों का लाभ उठाती है । यह विधि डीएनए प्रतिकृति 18 , डीएनए19की समोच्च लंबाई और आरईएस15द्वारा दरार को मापने के लिए उपयोग की जाने वाली पिछली पद्धतिों में सुधार और विस्तार है ।15 इस तकनीक में मान्यता अनुक्रम की एक प्रति वाले रैखिक डीएनए को एक छोर पर बायोटिन और दूसरे पर डिगोक्सिजेनिन के साथ तैयार किया जाता है। बायोटिन स्ट्रेप्टाविडिन को बांधता है, जो एक पैरासैग्नेटिक माइक्रोबैस से सहसंबद्ध रूप से जुड़ा होता है। डीएनए-मनका परिसरों को एक माइक्रोफ्लुइडिक चैनल में इंजेक्ट किया जाता है जिसे एंटी-डिगोक्सिजेनिन फैब टुकड़ों के साथ कार्यात्मक किया गया है। डीएनए तो adsorbed फैब टुकड़ों के लिए digoxigenin के बाध्यकारी के माध्यम से सतह लगाव अंक के लिए tethers । एक स्थायी चुंबक के साथ लागू कमजोर चुंबकीय बल मनका को गैर-विशेष रूप से सतह पर चिपकाने से रखते हैं। दरार प्रतिक्रिया को सक्रिय करने के लिए नमूनों को प्रवाह चैनल में तेजी से इंजेक्ट किया जा सकता है (<30 एस) । डेटा संग्रह के दौरान प्रवाह बंद कर दिया जाता है। जैसा कि प्रत्येक डीएनए को क्लीव किया जाता है, दरार का सही समय उस फ्रेम को रिकॉर्ड करके निर्धारित किया जा सकता है जिसमें मनका उद्देश्य की फोकल योजना से ऊपर और बाहर जाता है, इस प्रकार वीडियो रिकॉर्ड से गायब हो जाता है। प्रतिक्रिया प्रगति की मात्रा निर्धारित करने के लिए शेष मोतियों की फ्रेम-बाय-फ्रेम गिनती का उपयोग किया जा सकता है।
नीचे प्रस्तुत पूरा प्रोटोकॉल के साथ-साथ उदाहरण डेटा NDEI का उपयोग करके एकत्र किया गया है। तकनीक को कैसे लागू किया जा सकता है, इसके उदाहरण के रूप में, प्रोटीन सांद्रता की एक श्रृंखला के लिए दरार दरों को मैग्नीशियम, एक आवश्यक धातु कोफैक्टर की दो अलग-अलग सांद्रता पर मापा जाता है। यद्यपि प्रोटोकॉल का यह अनुप्रयोग एनडीईआई का उपयोग करता है, लेकिन डीएनए सब्सट्रेट डिजाइन को अलग करके किसी भी साइट-विशिष्ट नाभिक के साथ उपयोग के लिए विधि को अनुकूलित किया जा सकता है।
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. प्रवाह सेल बनाना
- कवरस्लिप धोना
- धुंधला जार में कवर्लिप्स रखें और इथेनॉल (एटोह) के साथ सोनिकेट करें, फिर 1 एम कोह (30 मिनट प्रत्येक के लिए) के साथ। EtOH में कोह वर्षा से बचने के लिए, वॉश के बीच डीडीएच 2 ओ के साथ अच्छीतरहसे कुल्ला करें।
- चार वॉश (दो एटोह और दो कोह) की कुल संख्या के लिए ETOH और कोह धोने के चरण 1x दोनों दोहराएं। धुंधला जार में डीडीएच2ओ में साफ कवरस्लिप स्टोर करें।
- एक साफ रेजर का उपयोग करके लोडिंग और निकास ट्यूब (8 सेमी लंबे) को काटें और एक साफ ग्लास स्लाइड में छेद में डालें। आउटलेट के लिए इनलेट और पीई-60 के लिए पीई-20 का इस्तेमाल करें। टयूबिंग को सुरक्षित करने और किसी भी अतिरिक्त टयूबिंग को ट्रिम करने के लिए 5 मिनट के लिए एपॉक्सी।
नोट: ग्लास स्लाइड उपाय 2'x 3'' x 1 मिमी। छेद जोड़े 15 मिमी के अलावा में ड्रिल कर रहे हैं। प्रत्येक जोड़ी एक ही चैनल के या तो अंत रूपों । छोटी आईडी के साथ टयूबिंग का उपयोग इनलेट के लिए किया जाता है, क्योंकि यह अपस्ट्रीम डेड वॉल्यूम को कम करता है और इस प्रकार मिश्रण समय आवश्यक(चित्र 1)। - लाइन अप करें और ग्लास स्लाइड पर छेद पर चैनल पैटर्न कट के साथ प्रीकट डबल-तरफा टेप लागू करें। एक अच्छी मुहर प्राप्त करने के लिए प्लास्टिक संदंश के साथ चिकनी।
नोट: इस प्रयोग में उपयोग किया जाने वाला दोतरफा टेप 120 माइक्रोन मोटा है, और चैनल 2 मिमी चौड़े और 15 मिमी लंबे हैं। चैनलों को चाकू प्रिंटर(सामग्री की तालिका) काउपयोग करके काटा जाता है। एक कवरलिप पर चार चैनलों तक फिट होना संभव है। प्रवाह कोशिका की एक छवि के लिए चित्रा 1 देखें। - समर्थन छीलने के बाद, टेप पर एक साफ कवरस्लिप (संकुचित हवा के साथ सूख) लागू करें और एक अच्छी मुहर के लिए प्लास्टिक के संदंश के साथ फिर से चिकनी करें।
- एपॉक्सी प्रवाह सेल को सील करने और इसे ठीक करने के लिए कवरस्लिप से किनारे।
2. टेदरिंग के लिए लेबल डीएनए की तैयारी
- एक पीसीआर ट्यूब में, 0.02 यू/μL उच्च निष्ठा डीएनए बहुलक, 200 μM dNTPs, 0.5 μM फॉरवर्ड प्राइमर, 0.5 μM रिवर्स प्राइमर, और M13mp18 वेक्टर डीएनए के 250 एनजी युक्त पीसीआर प्रतिक्रिया मिश्रण के 50 माइक्रोन तैयार करें।
नोट: यहां, फॉरवर्ड प्राइमर (बायोटिन-सीसीएक्टैटसीजीसीटीसीटीसीटीजीसी) और रिवर्स प्राइमर (डिगोक्सिजेनिन-TGACCATTAGATATATTTCGC) को एम 13एमपी18 डीएनए के परिपत्र जीनोम में 6338 से 107 पदों से फैले लगभग 1,000 बीपी लंबे क्षेत्र को बढ़ाने के लिए चुना गया था। प्रवर्धित क्षेत्र के मध्य में एक ही NdeI साइट है। फॉरवर्ड प्राइमर 5 डिगोक्सिजेनिन के साथ लेबल किया गया है, जो कवरस्लिप पर एंटी-डिगोक्सिजेनिन को बांधता है। रिवर्स प्राइमर 5 बायोटिन के साथ लेबल किया गया है, जो स्ट्रेप्टाविडिन-लेपित मोतियों को बांधता है। - थर्मोसाइकिलर में पीसीआर ट्यूब डालें और तालिका 1में दिखाए गए चक्र का पालन करें।
- निर्माता के प्रोटोकॉल का पालन करते हुए पीसीआर साफ-अप किट के साथ पीसीआर उत्पाद को शुद्ध करें।
नोट: सामग्रीकी तालिका में निर्दिष्ट किट का उपयोग करना, ठेठ डीएनए उपज ~ 2 μg है।
3. डीएनए और मोतियों की टेदरिंग
- बफर ए (1 एम ट्राइस-एचसीएल [पीएच = 7.5], 50 एमएमएल एनएसीएल, 2 एमएमएम एमजीसीएल2,1 मिलीग्राम/एमएल β-केसिन, 1 मिलीग्राम/एमएल प्लूरोनिक एफ-127) की 10 मिलीएल तैयार करें। कम से कम 1 घंटे के लिए वैक्यूम डिसेटर में डेगास।
- प्रवाह कोशिका को क्रियाशील करने के लिए, पीबीएस में एंटी-डिगोक्सिजेनिन फैब टुकड़ों (20 माइक्रोन/एमएल) के 25 माइक्रोन को प्रवाह चैनल में इंजेक्ट करें। पीई-60 ट्यूबिंग में फिट होने के लिए जेल लोडिंग टिप्स का इस्तेमाल करें। 30 मिनट के लिए कमरे के तापमान (आरटी) पर इनक्यूबेट।
- इनक्यूबेशन के बाद, एक सिरिंज का उपयोग कर चैनल के माध्यम से बफर ए के ०.५ एमएल खींच कर चैनल फ्लश । ध्यान रखें कि चैनल में हवा का परिचय न दें।
- कार्यात्मकता के बाद, एक उल्टे माइक्रोस्कोप पर प्रवाह कोशिका माउंट करें। एक सिरिंज पंप के लिए आउटलेट ट्यूब हुक और बफर ए युक्त एक माइक्रोसेंट्रफ्यूज ट्यूब में इनलेट ट्यूब डाल दिया ।
- मैन्युअल रूप से सिस्टम को फ्लश करने और पंप को प्राइम करने के लिए बफर ए के कम से कम 0.5 एमएल खींचें। पंप को सिस्टम को बराबर करने के लिए कम से कम 5 मिनट के लिए 10 माइक्रोन/मिनट पर चलने दें।
- मोतियों(सामग्री की तालिका)तैयार करने के लिए, मोतियों की स्टॉक बोतल और पिपेट 1.6 माइक्रोन की 10 मिलीग्राम/एमएल स्टॉक मोतियों की 50 माइक्रोन में बफर ए, फिर भंवर।
- एक चुंबकीय विभाजक का उपयोग करना, बफर ए के 50 माइक्रोन में बफर और रिसिपेंड को पिपेट करें, फिर भंवर।
- कुल तीन वॉश के लिए 3.7 2x दोहराएं। अंतिम धोने के लिए, 160 माइक्रोग्राम/एमएल की अंतिम एकाग्रता प्राप्त करने के लिए बफर ए और भंवर के 100 माइक्रोन में पुनर्सर्णित करें।
- डीएनए और मोतियों को जटिल बनाने के लिए, पहले बफर ए में डीएनए सब्सट्रेट लेबल 0.5 पीएम के 480 माइक्रोन तैयार करें। फिर, 160 μg/एमएल मनका निलंबन के 20 μL में पिपेट, पिपटिंग से पहले मोतियों भंवर करने के लिए सुनिश्चित कर रही है। 3 मिनट के लिए एक रोटेटर पर रखें।
- 3 मिनट के बाद, तुरंत चैनल में ~ 15 मिनट के लिए 10 μL/min की प्रवाह दर पर लोड करें या जब तक पर्याप्त मनका टेदरिंग नहीं देखी जाती है।
नोट: मोती इतनी घनी पैक नहीं किया जाना चाहिए कि वे सतह पर एक दूसरे के साथ बातचीत (चर्चा अनुभाग देखें) । - सभी मुफ्त मोतियों के चैनल को धोने के लिए, इनलेट ट्यूब को बफर ए की एक ताजा ट्यूब पर स्विच करें और कम से कम 10 मिनट के लिए 50 माइक्रोल/मिनट में प्रवाहित करें या जब तक कि कोई ढीले मोती नहीं देखा जाता है।
4. डेटा संग्रह और विश्लेषण
- डेटा संग्रह के लिए तैयार करने के लिए, इनलेट ट्यूब को एक माइक्रोसेंट्रफ्यूज ट्यूब में रखें जिसमें बफर ए में एनडीईआई (0.25-4.00 यू/एमएल) के कम से कम 100 माइक्रोल होते हैं, और प्रवाह चैनल पर स्थायी चुंबक को कम करें, और डार्कफील्ड इमेजिंग के लिए अक्ष से प्रकाश स्रोत की स्थिति रखें।
नोट: दो वलयाकार दुर्लभ पृथ्वी मैग्नेट, एक साथ epoxied, डेटा संग्रह के दौरान एक ब्रैकट ऑप्टिकल पोस्ट का उपयोग कर प्रवाह चैनल की सक्रिय सतह के ऊपर 8 मिमी आयोजित कर रहे हैं । ऑफ एक्सिस लाइट कोर्स के लिए हंस नेक लैंप का इस्तेमाल किया जाता है। - एक वाणिज्यिक माइक्रोस्कोप, वीडियो कैमरा, और डेटा संग्रह सॉफ्टवेयर(सामग्री की तालिका) का उपयोग करें। सॉफ्टवेयर में ,"एक्सपोजर"टैब पर क्लिक करें, और 10 एमएस करने के लिए"एक्सपोजर टाइम"सेट करें। क्लिक करें'टाइमलैप्स'टैब और सेट'इमेज काउंट','अवधि'से 20 मिनट और'इंटरवल'से 2 एस तक। डेटा संग्रह शुरू करने के लिए'रन'पर क्लिक करें।
- एक सिरिंज पंप पर, 150 μL/मिनट और इंजेक्शन की मात्रा के लिए 80 μL प्रेस"भागो"डेटा संग्रह में 1 मिनट पर प्रवाह दर निर्धारित करें। इंजेक्शन के बाद, पंप बंद करें और डेटा संग्रह के दौरान प्रवाह को रोकने के लिए वाल्व बंद करें।
- एक बार डेटा एकत्र होने के बाद, छवि विश्लेषण सॉफ्टवेयर(सामग्री की तालिका) खोलें। "फ़ाइल"टैब के तहत,"आयात" चुनें। "छविअनुक्रम"। पॉप-अप मेनू में छवि फ़ाइलों का पता लगाएं और "ओपन" पर क्लिक करें।
- "छवि"पुल-डाउन मेनू के तहत"एडजस्ट थेहोल्ड"चुनकर दहलीज निर्धारित करें। छवि में मोतियों के अनुरूप उज्ज्वल स्थानों की पहचान करने के लिए सीमा मूल्य निर्धारित करने के लिए स्लाइडर बार का उपयोग करें।
- "विश्लेषण" पुल-डाउन मेनू में"विश्लेषण कणों"पर क्लिक करके प्रत्येक फ्रेम में उज्ज्वल धब्बे गिनें।Analyze क्लिक करें"ठीक है",तो चुनें"हां"सभी छवियों को संसाधित करने के लिए। परिणाम फ़ाइल को सहेजें।
नोट: यह एक डेटा फ़ाइल को बचाएगा जिसमें प्रत्येक रिकॉर्ड किए गए वीडियो फ़्रेम में मोतियों की संख्या होती है. - डेटा विश्लेषण सॉफ्टवेयर(सामग्री की तालिका) खोलेंऔर "फाइल" पुल-डाउन मेनू में"टेक्स्ट फाइल से आयात"पर क्लिक करके परिणामफ़ाइलआयात करें। समय बनाम मनका गिनती डेटा प्लॉट।
- "विश्लेषण"पुल-डाउन मेनू में"वक्र फिट"पर क्लिक करके मोतियों बनाम समय की संख्या फिट करें। "प्राकृतिकप्रतिपादक"समीकरण चुनें और क्लिक करें"फिट की कोशिश करो"। | "ठीकहै"।
नोट: केवल नमूने के इंजेक्शन के बाद दर्ज डेटा फिटिंग क्षेत्र में शामिल किया जाना चाहिए । फिटिंग फ़ंक्शन के एक्सपोनेंट में फिट पैरामीटर क्लीवेज रेट होगा।
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
इस तकनीक का उपयोग करते हुए, एनडीईआई की एसएसडीसी दरों को मैग्नीशियम (2 mm और 4 mM) के दो अलग-अलग सांद्रता पर प्रोटीन सांद्रता (0.25-4.00 यू/एमएल) की एक श्रृंखला के लिए मापा गया था। इन शर्तों में से प्रत्येक को कम से कम दो बार दोहराया गया था, जिसमें प्रति प्रयोग कुछ सौ से 1,000 सीमित DNAs थे। चित्रा 2 प्रायोगिक डिजाइन का वर्णन करता है। चित्रा 3 डेटा संग्रह और विश्लेषण विवरण के उदाहरण दिखाता है। चित्रा 4 दिखाता है कि कैसे दर मैग्नीशियम की दो सांद्रता पर प्रोटीन एकाग्रता पर निर्भर करता है । यह देखा जा सकता है कि पर्याप्त रूप से कम प्रोटीन सांद्रता पर, दर प्रोटीन के आनुपातिक और मैग्नीशियम से स्वतंत्र है। पर्याप्त रूप से उच्च प्रोटीन सांद्रता के लिए, दर मैग्नीशियम पर निर्भर है लेकिन प्रोटीन एकाग्रता से स्वतंत्र है।
| चरण | विवरण | तापमान (डिग्री सेल्सियस) | समय (एस) |
| 1 | डेनैचेशन | 98 | 30 |
| 2 | पिघल | 98 | 10 |
| 3 | एनील | 60 | 30 |
| 4 | विस्तार | 72 | 30 |
| 5 | अंतिम विस्तार | 72 | 120 |
तालिका 1: पीसीआर पैरामीटर। दिखाया गया है कि प्रोटोकॉल के चरण 2.2 में उपयोग किए जाने वाले थर्मोसाइकिलर प्रोग्राम चरणों का तापमान और अवधियां हैं। पिघल, एनील, और विस्तार चरण (चरण 2, 3, और 4) 30x दोहराया जाता है।
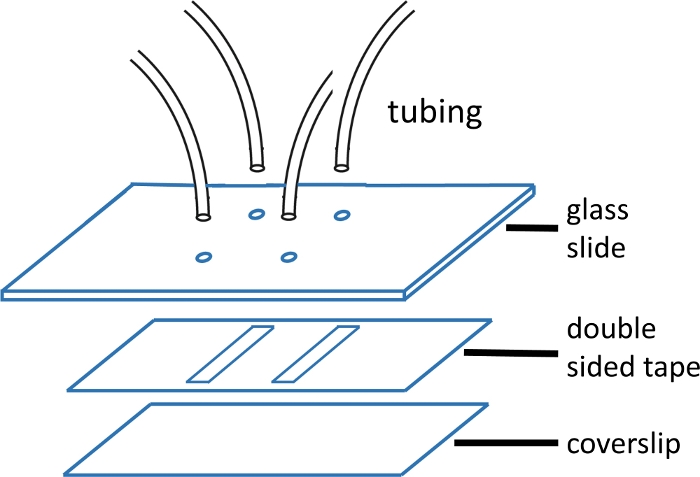
चित्रा 1: माइक्रोफ्लुइडिक प्रवाह सेल निर्माण।
शीर्ष ग्लास स्लाइड (2'x 3', 1 मिमी मोटी) चैनल पैटर्न से मेल खाने वाले छेद के साथ प्री-ड्रिल किया गया है। इनलेट और आउटलेट ट्यूबों छेद में डाला जाता है और टेप और कवर ग्लास संलग्न करने से पहले epoxy के साथ तय की जाती है । दो तरफा टेप चैनल पैटर्न के साथ पूर्व कट है। नीचे स्लाइड (#1 या #1.५ कवर ग्लास) पहले मुख्य पाठ में वर्णित प्रोटोकॉल का उपयोग करके साफ किया जाता है। एक बार इकट्ठा होने के बाद, कवर ग्लास का किनारा एपॉक्सी के साथ बंद हो जाता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

चित्रा 2: प्रायोगिक डिजाइन।
(एक)डीएनए टेदरिंग की विधि। 5 छोर पर डिगोक्सिजेनिन के साथ लेबल किया गया डबल-फंसे डीएनए (1 केबीपी) एंटीडिगोक्सीजेनिन-डिगोक्सिजेनिन इंटरैक्शन के माध्यम से प्रवाह कोशिका की सतह से जुड़ा हुआ है। डीएनए का 3 सिरा, बायोटिन के साथ लेबल, स्ट्रेप्टाविडिन-बायोटिन इंटरैक्शन के माध्यम से एक माइक्रोबैस से जुड़ा हुआ है। NdeI दरार साइट डीएनए के केंद्र में स्थित है। (ख)डाटा संग्रह के दौरान प्रायोगिक सेटअप जिसमें चुंबक और वस्तुनिष्ठ स्थिति दर्शाई गई है । स्थायी चुंबक दरार प्रतिक्रिया के दौरान मनका पर एक कमजोर ऊपर की ओर बल बनाए रखता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
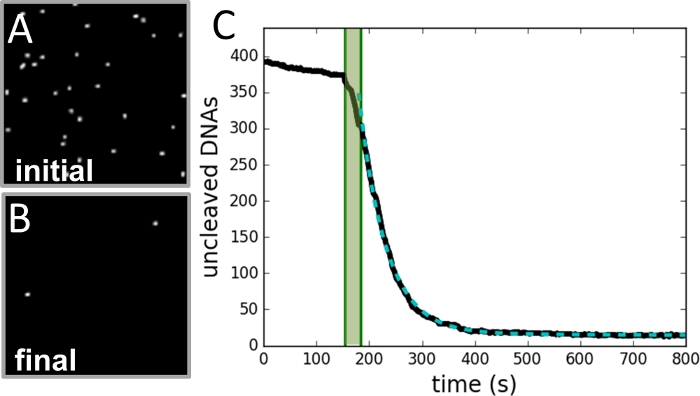
चित्र 3: डेटा संग्रह और विश्लेषण का उदाहरण।
(क)दरार प्रतिक्रिया शुरू होने से पहले ली गई मोतियों के क्षेत्र की छवि । (ख)प्रतिक्रिया पूरी होने के बाद उसी क्षेत्र की छवि । (ग)वीडियो रिकॉर्ड के प्रत्येक फ्रेम से निर्धारित समय (काला वक्र) बनाम मोतियों की संख्या का प्लॉट। छायांकित हरित क्षेत्र एंजाइम के इंजेक्शन की अवधि को चिह्नित करता है और फिट में शामिल नहीं होता है। डेटा एक एकल घातीय वक्र (धराशायी हरे वक्र) फिट बैठता है, जिसमें प्रतिक्रिया दर के बराबर क्षय स्थिर होता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

चित्रा 4: NdeI दरार प्रोटीन और मैग्नीशियम की सांद्रता पर निर्भर है।
मैग्नीशियम की दो अलग-अलग सांद्रता पर प्रोटीन सांद्रता की एक श्रृंखला के लिए एनडीईआई की मापा गया एसएसडीसी दर का प्लॉट: 2 m (ब्लू सर्कल) और 4 mm (ग्रीन वर्ग)। त्रुटि सलाखों एसईएम का प्रतिनिधित्व करते हैं। धराशायी घटता प्रवृत्ति लाइनों रहे है और सिद्धांत के लिए फिट बैठता है प्रतिनिधित्व नहीं करते । कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
Subscription Required. Please recommend JoVE to your librarian.
Discussion
प्रोटोकॉल का उपयोग किसी भी एसएसडीसी प्रणाली के काइनेटिक्स को मापने के लिए किया जा सकता है, बशर्ते कि प्रयोग के दौरान स्ट्रैंड अलगाव मनाया जाता है। दरार का पता लगाने सीमित मनका की टुकड़ी देख कर प्रभावित होता है और इसलिए कतरा जुदाई के तत्काल निशान । दरार का पता लगाने से पहले सभी पूर्ववर्ती चरण होते हैं; इस प्रकार, केवल कुल पारगमन समय दर्ज किया जाता है।
प्रवाह कोशिका कवरस्लिप को साफ ग्लास में एंटीबॉडी प्रोटीन के गैर-विशिष्ट सोखने के माध्यम से कार्यात्मक किया जाता है। अपर्याप्त रूप से साफ ग्लास एंटीबॉडी के बाइंडिंग को प्रभावित कर सकता है। टेदरिंग में, मनका घनत्व काफी कम होना चाहिए ताकि मोती बातचीत न करें। लगाव बिंदुओं की सतह घनत्व कार्यात्मकता के दौरान एंटीबॉडी की एकाग्रता द्वारा नियंत्रित किया जा सकता है। मोतियों की कुल संख्या देखने के क्षेत्र के आकार पर निर्भर करता है। इस मामले में, कुछ सौ से 1,000 सीमित मोती अच्छे आंकड़ों के लिए पर्याप्त थे और मनका-मनका बातचीत से बचा। मनका इंजेक्शन के दौरान, लाइव वीडियो के माध्यम से सतह tethering पर नजर रखी गई थी। मनका इंजेक्शन बंद कर दिया गया था जब अनुमानित मनका गिनती ५०० और १,००० मोती के बीच था ।
सबसे तेजी से दरार दरों है कि सही मापा जा सकता है प्रवाह सेल के मिश्रण समय से सीमित हैं । लेमिनार प्रवाह कोशिकाओं में मिश्रण समय कई कारकों से प्रभावित होता है। सतह पर प्रसार एक महत्वपूर्ण कदम है; इसलिए, मिश्रण समय प्रतिक्रियाकारक के प्रसार गुणांक पर निर्भर करता है। प्रवेश द्वार ट्यूबिंग में होने वाली महत्वपूर्ण कतरनी, जो नमूना जलाशय से प्रवाह चैनल के लिए नमूना परिवहन करती है, चैनल में प्रतिक्रिया सतह पर पर्याप्त मिश्रण सुनिश्चित करने के लिए आवश्यक समय बढ़ा सकती है। यह पाया गया कि अपस्ट्रीम डेड वॉल्यूम को कम करके और फ्लो रेट बढ़ाकर मिक्सिंग टाइम को कम किया जा सकता है । 380 माइक्रोन के एक आंतरिक व्यास और इनलेट ट्यूबिंग के लिए 8 सेमी की अधिकतम लंबाई के साथ (और 150 μL/min की प्रवाह दर), यह पाया गया कि इंजेक्शन समय मापा दरार दर को प्रभावित किए बिना ~ 20 एस करने के लिए कम किया जा सकता है। चूंकि मिश्रण का समय प्रतिक्रियाकार के प्रसार गुणांक पर निर्भर करता है, इसलिए इसे प्रत्येक एंजाइम या दरार सक्रियक के अध्ययन के लिए अलग से निर्धारित किया जाना चाहिए।
तार विधि गैर-विशिष्ट तार टूटना के लिए अनुमति देती है, संभवतः या तो डिगोक्सिजेनिन-एंटीबॉडी कॉम्प्लेक्स के वियोजन या सतह से एंटीबॉडी की रिहाई के कारण। इसके परिणामस्वरूप ~ 3 x 10-4 एस-1के एंजाइम के इंजेक्शन से पहले एक प्रजनन पृष्ठभूमि मनका हानि दर मौजूद है। इस व्यवस्थित प्रभाव को मापा गया दरार दर से पृष्ठभूमि दर को घटाकर या फिटिंग समीकरण में पृष्ठभूमि को मॉडलिंग करके या तो ठीक किया जा सकता है। हालांकि, दरार दरों इस कम बाध्य से कम मज़बूती से मापा जाएगा ।
अपूर्ण सतह पासिवेशन अनुचित टेदरिंग का कारण बन सकता है। यह या तो "अटक" मोतियों के अंशों को जन्म देता है, जो प्रयोग के दौरान दूर नहीं जाते हैं, the या अनुचित रूप से सीमित मोती होते हैं, जो सतह से बहुत धीरे-धीरे अलग होते हैं। यह प्रसंस्कृत डेटा में एक उच्च और संभवतः ढलान वाली आधार रेखा बनाता है। यह पाया गया कि ठीक से साफ कवर स्लिप और हौसले से बनाया β-casein स्टॉक समाधान के साथ, इन प्रभावों को सबसे अधिक डेटा सेट के लिए कम थे । कभी-कभी डेटा सेट के लिए जो इसे दिखाते हैं, फिटिंग फ़ंक्शन को संशोधित करते हैं (एक ढलान वाली बेसलाइन शामिल करने के लिए) इस प्रभाव के लिए सही हो सकते हैं।
वर्तमान प्रोटोकॉल को कई तरीकों से बढ़ाया जा सकता है। लक्ष्य साइट बाध्यकारी के बाद मशीनी कदमों के आगे अलगाव एक पूर्व बाध्यकारी प्रारूप का उपयोग कर किया जा सकता है, जिसमें प्रोटीन आवश्यक cofactors के अभाव में इंजेक्शन है । मैग्नीशियम के अभाव में एनडीईई इंजेक्शन लगाकर इस विचार का परीक्षण किया जाता है। इन परिस्थितियों में प्रोटीन अपनी कॉग्नेट साइट पर बांधता है लेकिन डीएनए को क्लीव नहीं करता । इस बाध्यकारी कदम के बाद मैग्नीशियम इंजेक्शन दरार तेजी से मनका नुकसान में जिसके परिणामस्वरूप सक्रिय करता है । प्रायोगिक सेटअप चुंबक विन्यास या जोड़ने के प्रवाह को अलग करके डीएनए संरचना और तनाव के नियंत्रण के लिए भी अनुमति देता है। इन प्रयोगों में कम ताकतों के नीचे डीएनए आंशिक रूप से कुंडलित हो जाता है। बलों को थोड़ा बदलने से डीएनए की संरचना पर नाटकीय प्रभाव पड़ सकता है । उदाहरण के लिए, बफर परिस्थितियों में, जिसमें लक्ष्य खोज दर सीमित है, डीएनए की संरचना को अलग करने के लिए लक्ष्य खोज पर कूद के प्रभाव के लिए परीक्षण कर सकते हैं । बफर परिस्थितियों में, जिसमें हाइड्रोलिसिस कदम दर सीमित है, अलग बल फॉस्फोडिस्टर बांड हाइड्रोलिसिस पर डीएनए तनाव के प्रभाव की जांच कर सकते हैं । यह ध्यान दिया जाना चाहिए कि कम आवर्धन के तहत, इन अनुरूप परिवर्तनों को नहीं देखा जा सकता है। मनका स्थिति में जिसके परिणामस्वरूप छोटी गति को उच्च आवर्धन के तहत ट्रैक किया जाना चाहिए ताकि यह सत्यापित किया जा सके कि डीएनए संरचना को नियंत्रित किया जा सकता है ।
डेटा विश्लेषण को विभिन्न तरीकों से बढ़ाया जा सकता है। यह काम एक साधारण मनका गिनती विधि लागू होता है जिसके बाद एक ही घातीय कार्य की वक्र फिटिंग होती है। निवास समय विश्लेषण के आधार पर तरीकों का उपयोग किया जा सकता है, साथ ही20। व्यक्तिगत DNAs के निवास समय के वितरण तो वक्र फिटिंग के माध्यम से या अधिक परिष्कृत तकनीकों का उपयोग करके विश्लेषण किया जा सकता है, जैसे21 क्षणोंकी सामान्यीकृत विधि ।
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
लेखकों को खुलासा करने के लिए हितों का कोई टकराव नहीं है ।
Acknowledgments
इस काम को नेशनल साइंस फाउंडेशन ग्रांट एमसीबी-1715317 ने सपोर्ट किया।
Materials
| Name | Company | Catalog Number | Comments |
| 5 minute Epoxy | Devcon | 14250 | |
| anti-digoxigenin FAB fragments | Roche Diagnostics | 11214667001 | |
| camera and software | Jenoptik | GRYPHAX SUBRA | |
| data analysis software | Vernier Inc. | LP | |
| double sided tape | Grace Biolabs | SA-S-1L | |
| Dulbeccos Phosphate Buffered Saline | Corning | 21-031-CV | |
| ethanol 95% | VWR | 89370-082 | |
| forward primer: digoxigenin-CCAACTTAATCGCCTTGC | Integrated DNA Technologies | n/a | |
| image analysis software | National Institutes of Health | ImageJ | |
| inverted microscope | Nikon | TE2000 | |
| knife printer | Silhouette | ||
| M13mp18 DNA | New England Biolabs | N4040S | |
| MyOne streptavidin beads | Thermo Fisher Scientific | 65601 | |
| NdeI enzyme | New England Biolabs | R0111S | |
| PCR cleanup kit | Qiagen | 28104 | |
| pluronic F-127 | Anatrace | P305 | |
| polyethylene tubing PE-20 | BD Intramedic | 427406 | |
| polyethylene tubing PE-60 | BD Intramedic | 427416 | |
| Q5 Mastermix | New England Biolabs | M0492S | |
| rare earth magnet 0.5" OD 0.25" ID | National Imports | NSN0814 | |
| rare earth magnet 0.75" OD 0.5" ID | National Imports | NSN0615 | |
| reverse primer: biotin-TGACCATTAGATACATTTCGC | Integrated DNA Technologies | n/a | |
| syringe pump | Kent Scientific | Genie Plus | |
| β-Casein from bovine Milk | Sigma-Aldrich | C6905 |
References
- Tock, M. R., Dryden, D. T. The biology of restriction and anti-restriction. Current Opinion in Microbiology. 8 (4), 466-472 (2005).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Pingoud, A., Fuxreiter, M., Pingoud, V., Wende, W. Type II restriction endonucleases: structure and mechanism. Cellular and Molecular Life Sciences. 62 (6), 685-707 (2005).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Sadowski, P. D. Site-specific genetic recombination: hops, flips, and flops. The FASEB Journal. 7 (9), 760-767 (1993).
- Schatz, D. G. Antigen receptor genes and the evolution of a recombinase. Seminars in Immunology. 16 (4), 245-256 (2004).
- Craig, N. L. Tn7: a target site-specific transposon. Molecular Microbiology. 5 (11), 2569-2573 (1991).
- Gori, J. L., et al. Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26 (7), 443-451 (2015).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Alves, J., Urbanke, C., Fliess, A., Maass, G., Pingoud, A. Fluorescence stopped-flow kinetics of the cleavage of synthetic oligodeoxynucleotides by the EcoRI restriction endonuclease. Biochemistry. 28 (19), 7879-7888 (1989).
- Deng, J., Jin, Y., Chen, G., Wang, L. Label-free fluorescent assay for real-time monitoring site-specific DNA cleavage by EcoRI endonuclease. Analyst. 137 (7), 1713-1717 (2012).
- Becker, W. R., et al. High-throughput analysis reveals rules for target RNA binding and cleavage by AGO2. Molecular Cell. 75 (4), 741-755 (2019).
- vanden Broek, B., Lomholt, M. A., Kalisch, S. M., Metzler, R., Wuite, G. J. How DNA coiling enhances target localization by proteins. Proceedings of the National Academy of Sciences. 105 (41), 15738-15742 (2008).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Jones, N. D., et al. Retroviral intasomes search for a target DNA by 1D diffusion which rarely results in integration. Nature Communications. 7, 11409 (2016).
- van Aelst, K., et al. Type III restriction enzymes cleave DNA by long-range interaction between sites in both head-to-head and tail-to-tail inverted repeat. Proceedings of the National Academy of Sciences. 107 (20), 9123-9128 (2010).
- Williams, K., et al. A single molecule DNA flow stretching microscope for undergraduates. American Journal of Physics. 79 (11), 1112-1120 (2011).
- Song, D., et al. Tethered particle motion with single DNA molecules. American Journal of Physics. 83 (5), 418-426 (2015).
- Etson, C. M., Todorov, P., Walt, D. R. Elucidating Restriction Endonucleases Reaction Mechanisms via Dwell-Time Distribution Analysis. Biophys Journal. 106 (2), 22 (2014).
- Piatt, S., Price, A. C. Analyzing dwell times with the Generalized Method of Moments. PLoS One. 14 (1), 0197726 (2019).



